2. 中国农业科学院植物保护研究所 植物病虫害生物学国家重点实验室,北京 100193
2. State Key Laboratory for Biology of Plant Diseases and Insect Pests,Institute of Plant Protection,Chinese Academy of Agricultural Sciences,Beijing 100193
栽培花生(Arachis hypogaea L.)作为一种重要的油料作物和经济作物,具有重要的经济价值和营养价值。2015年我国花生的总产量达1 670万t,居世界首位。但是一些生物及非生物胁迫会影响花生的产量和品质,如干旱、盐碱、低温、寒冷、高温及各种病原菌等外界不良环境会延缓植物的生长发育,降低植物的生产效率,甚至在极端条件下,会导致植物死亡。植物的应激反应是动态的,涉及到不同调节水平之间错综复杂的关系,包括新陈代谢的调节、生理上基因表达的调节和形态上的适应等[1]。使用寄主抗性是控制植物病害最有效、经济的方式[2]。
有研究表明,抗病基因(R基因)在识别和抵御病原菌入侵方面具有重要作用。编码具有核苷酸结合位点(NBS)和C-末端富含亮氨酸重复(LRR)的基因,是R基因中最大一类抗性基因[3]。植物NBS-LRR蛋白(nucleotide-binding site-leucine rich re-peat protein)根据氨基酸末端序列可分为两类,卷曲螺旋(CC)- NBS-LRR类蛋白质和TIR-NBS-LRR类蛋白,后一类蛋白在单子叶植物中尚未被鉴别[4, 5],而且TIR-NBS-LRR类蛋白可能比CC-NBS-LRR型蛋白起源更早[6]。抗逆性反应的识别过程是基于NBS-LRR蛋白介导的直接或间接的识别病理分子——无毒蛋白AVR进行的,而AVR蛋白突变会使抗逆性反应过程中识别异常,从而导致抗性异常。也有研究表明,抗性反应的识别过程是通过NBS-LRR蛋白与转录因子的相互作用进行的,Shen 等[7]的研究表明,在大麦中识别白粉病抗性蛋白(Mla10)是通过CC结构域与一个WRKY转录因子结构域的相互作用,进行防御反应。NBS结构域位于NBS-LRR型蛋白的中间区域,存在于真核生物具有结合ATP和GTP的许多蛋白质之中,如ATP合成酶β亚基、核糖体延伸子、腺苷酸激酶以及抗病基因编码蛋白等。NBS结构域可以与核苷酸进行结合,LRR结构域在将感知到的病原体激活方面起着至关重要的作用[8-10]。
花生作为豆科植物家族中的一个重要成员,其NBS-LRR类基因在不同胁迫下的功能分析报道尚不多见。本研究以花生抗逆性基因AhNDrp为研究对象,以本课题组此前获得的花生根转录组测序数据为依据[11],从基因注释结果中筛选到contig281453为花生抗逆性蛋白,获得基因的半长序列,利用电子克隆技术获得AhNDrp全长基因,从而分析该基因在花生不同组织部位(根、茎、叶、幼胚)的表达情况以及干旱和黄曲霉胁迫下的表达特性,旨在为花生抗黄曲霉的机理研究提供依据。
1 材料与方法 1.1 材料 1.1.1 试验材料与处理供试花生栽培品种白沙1016,由山东省花生研究所提供。花生根、茎、叶取自2周龄的组培苗,幼胚子叶为收集果针入土后25-55 d不同大小的花生荚果,剥皮后混合。所有植物材料取样后于液氮中迅速冷冻,在-80℃冰箱保存。
干旱处理:当花生生长至15 d左右时,选择健壮且长势一致的幼苗,用15% PEG6000溶液模拟干旱处理,分别取未处理及处理后2、4、6和8 h的幼苗根,液氮速冻后于-80℃保存,用于总RNA提取和荧光定量PCR分析。试验设3次重复。
黄曲霉处理:花生剥去种皮后种植于温室中,3周后接种黄曲霉孢子。用无菌水收集在PDA培养基上培养2-3 d 的黄曲霉菌孢子,调整孢子浓度为6×108/ mL[12],喷于花生的根部,分别取未处理及处理后 1、3、5和7 d的幼苗根,液氮速冻后,于-80℃保存,用于荧光定量PCR分析。试验设3次重复。
1.1.2 酶及主要试剂Primer Star高保真聚合酶购自TaKaRa公司;2×Taq Mix PCR扩增试剂购自北京博迈得生物技术有限公司;pMD19-T Vector购自TaKaRa公司;DNA Marker DL2000分子量标准购自Promega公司;其他试剂均为国产分析纯产品。PCR 产物测序由北京华大生物技术有限公司完成。反转录和荧光定量PCR试剂盒为北京天根公司FastQuant cDNA第一链合成试剂盒和SYBR Green荧光定量试剂盒。
1.1.3 PCR引物引物的合成由上海生工生物技术有限公司完成,引物序列见表 1。
花生基因组网址(http://peanutbase.org);序列检索采用NCBI blast(http://www.ncbi.nlm.Nih.gcv/blast);蛋白保守结构域分析在(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)进行;三级结构预测在(http://swissmodel.expasy.org/workspace/index.php?func=modelling_simple1)进行;氨基酸同源性比对分析采用DNAMAN软件;系统发育树构建采用MEGA4.0软件。
1.2 方法 1.2.1 花生基因组的提取花生基因组DNA的提取采用改良的CTAB法[13]。将提取基因组放于-20℃储存备用。
1.2.2 NBS-LRR类基因全长获得根据peanutDB上序列,设计引物见序列表 1。以花生栽培品种白沙1016基因组为模板,AhNDrp-f和AhNDrp-r为引物进行PCR扩增。PCR 扩增程序为:94℃ 10 min;94℃ 30 s,50℃ 30 s,72℃ 2 min,30个循环;72℃ 10 min。取PCR产物电泳,选取目的条带,回收、克隆到 pMD19-T Vector,经 PCR 鉴定正确后进行测序拼接。
1.2.3 RNA的提取、cDNA第一链的合成RNA的提取 采用上海生工生物工程有限公司TRIzol试剂,提取方法参见说明书。TaKaRa公司的DNaseⅠ用于消化残留 DNA,cDNA 第一条链的合成用天根的FastQuant cDNA第一链合成试剂盒。
1.2.4 花生AhNDrp的mRNA转录分析分别提取花生2周龄的根、茎、叶和不同生长时期混合幼胚的总RNA及干旱和黄曲霉接种处理下不同时期的花生根总RNA,反转录成cDNA,进行AhNDrp基因表达特性分析。用天根公司的SYBR Green荧光定量试剂盒检测花生AhNDrp基因的表达量。设actin基因为内部参照,AhNDrp-real-time-f和AhNDrp-real-time-r分别为扩增目的基因的上下游引物。PCR 扩增程序为:95℃ 15 min;95℃ 10 s,60℃ 30 s,40个循环。
2 结果 2.1 AhNDrp全长基因的获得与序列分析本实验室经转录组测序,获得一条1 970 bp的EST序列(contig281453),在NCBI上进行比对,注释为NBS-LRR型抗逆性蛋白(NBS-LRR type disease resistance protein)。将该序列与http://peanutbase.org网站上公布的花生基因组序列进行比对,根据比对结果设计引物(AhNDrp-f/r),以花生基因组为模板,扩增得到3 200 bp的片段(图 1),送测序。在NCBI数据库中进行相似性比较,该蛋白与大豆抗逆性蛋白(Glycine soja disease resistance protein RPM1,GenBank No:KHN40407.1)的相似性最高为79%。将该基因命名为AhNDrp基因,编码区为2 832 bp,GenBank登录号为KU128398。AhNDrp基因DNA序列与cDNA序列比对分析发现,AhNDrp基因DNA序列中不包含内含子,编码943个氨基酸,终止密码子为TAG(图 2)。

|
| 图 1 AhNDrp基因的扩增结果 M:DL5000 DNA Marker;CK:阴性对照;1:以花生基因组为模板的PCR扩增产物 |

|
| 图 2 AhNDrp基因的结构示意图 |
使用CDD(conserved domain database)在线数据库进行蛋白功能结构域的预测,发现AhNDrp蛋白有5个典型的抗逆性蛋白保守结构域(图 3)。其中DomainⅠ为RX-CC-like结构域,位于2-127区,与抗病反应的必要因素RanGAP2相互作用;DomainⅡ为AAA-ATP酶结构域,由117个氨基酸组成,包含一个P-Loop(NBS结构域中的保守基序)元件;DomainⅤ位于595-804区,由209个氨基酸组成,是富含亮氨酸重复序列,LRR基序可参加蛋白-蛋白互作,在分子识别过程中发挥重要的作用。这3个保守区域说明AhNDrp属于NBS-LRR型抗逆性蛋白。
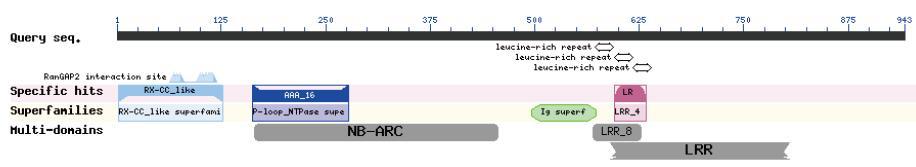
|
| 图 3 花生AhNDrp 蛋白保守结构域预测分析 |
利用同源建模的分析工具SWISS-MODEL对花生NDrp蛋白结构域进行三维结预测。软件自动选择PDB数据库中的2qkwB作为模板,对目标蛋白的605-879氨基酸进行3-D结构预测,如图 4所示,蛋白的胞内结构域是由132个α-螺旋、65个无规则卷曲、52个延伸链和26个β-折叠连接共同构成的复杂结构。

|
| 图 4 花生NDrp 蛋白的三级结构 |
将AhNDrp蛋白序列与豆科植物花生、大豆、野生大豆、鹰嘴豆、菜豆(Phaseolus vulgaris,XP_007158209.1)、苜蓿(Medicago truncatula,AF491998.1)和非豆科植物白梨(P.×bretschneideri,XP_009373508.1)、苹果(Malus domestica,XP_008342592.1)、蔷薇科落叶乔木植物梅(Prunus mume,XP_008238133.1)、可可(Theobroma cacao,XP_007040756.1)、川桑(Morus notabilis,XP_010110397.1)的抗逆性蛋白序列进行多重比对,并构建系统发育树(图 5)。白沙1016号花生AhNDrp蛋白与白梨和苹果的进化关系最近;苜蓿和菜豆等次之;与鹰嘴豆、梅的进化关系最远。
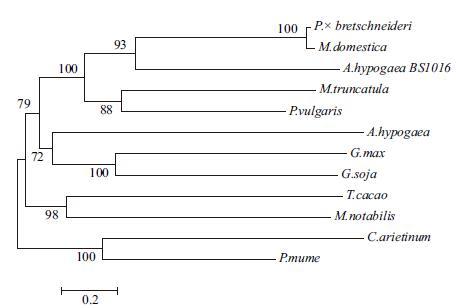
|
| 图 5 12 种植物抗逆性蛋白的系统发育树 |
通过荧光定量PCR方法分析花生品种BS1016中AhNDrp基因的表达情况,结果(图 6-A)显示AhNDrp基因在茎、叶、幼胚中均无表达,仅在根中特异表达。用15% PEG6000溶液模拟干旱胁迫处理,分析0-8 h AhNDrp基因在花生根中的表达量(图 6-B),发现随着干旱处理时间的增加AhNDrp基因表达量也逐渐上升,在处理6 h时表达量最高,处理8 h时表达量降低,但该基因在干旱处理下的表达量始终低于未处理时的植株,说明AhNDrp基因无抗干旱能力。用孢子浓度为6×108/mL的黄曲霉处理花生后,0-3 d,AhNDrp基因的表达量处于上升阶段,在3 d达到最高表达量,为对照表达量的1.62倍;之后开始下降,在第7天时表达量降到最低,明显低于对照组及其他处理组(图 6-C)。

|
| 图 6 AhNDrp基因在花生根、茎、叶、幼胚(A)及干旱(B)和黄曲霉接种处理下(C)花生根的表达分析 |
目前花生基因组测序已经取得重要进展,部分基因组已公布(http://peanutbase.org),为花生新基因克隆及基因功能研究等提供了数据平台。本实验室通过对花生根、叶及未成熟种子的转录组深度测序,构建了含3万多条花生EST序列的数据库。本研究结合转录组数据和基因组测序,成功克隆到了花生AhNDrp全长基因,并利用生物信息学方法对该抗逆性蛋白结构和功能进行了预测,为后续探究花生抗逆性基因的表达调控和作用机制提供参考。
有报道指出,NBS-LRR类基因是在进化过程中通过串联或部分复制祖先基因而得到的。NBS结构域包含几个保守基序:P-loop、kinase 2a、kinase 3a 和GLPL motifs等[14];P-loop被证明在ATP结合能力中起到关键作用[10],在烟草[15]和拟南芥[16]PR5中也得到类似结论;kinase-3a激酶可以结合嘌呤或核糖[14],但是NBS结构域在植物抗病性机制中的作用尚不清楚。LRR结构域在蛋白与蛋白之间的相互作用中发挥重要作用,最新研究结果表明LRR蛋白片段足以启动防御信号[17],是识别病原体衍生的效应分子,随后激活宿主防御反应。LRR蛋白的多种结构域使其可以同时作为病原体探测器、传感器、开关和响应因子[18]。这些NBS-LRR类蛋白质在细胞生长、风化、细胞骨架的形成、囊泡运输和防御反应中起重要作用。目前,已从不同植物中克隆得到一些NBS-LRR类基因,例如,何利等[19]从桑树中克隆到5个NBS类基因分别为CL93、Unigene14278、Unigene26173、Unigene32704、Unigene31320的片段;史静东等[20]利用同源序列法分离小麦NBS-LRR类抗病基因类似片段,获得4个RGAs片段。本研究利用电子克隆的方法,克隆得到的基因全长2 832 bp,命名为AhNDrp基因,含有NBS-LRR类抗病基因典型的保守结构域。
刘建成等[21]从草莓中克隆得到一个NBS-LRR类基因,经半定量 RT-PCR 分析显示,该基因在草莓的所有组织中都表达,但在茎尖分生组织中表达水平最高,且在叶中的表达水平明显受到外源水杨酸和脱落酸处理的影响。本研究中克隆的AhNDrp基因只在花生根部特异性表达,与前期转录组测序结果及半定量RT-PCR的结果一致。赵小波等[22]在花生抗旱品种中克隆到NBS-LRR类PDR1基因,该基因在干旱胁迫处理12 h 后表达量达到峰值。但本研究中,干旱处理后AhNDrp基因的表达量始终低于未处理时的植株,说明该基因的表达产物不参与干旱胁迫的响应,被迫应答干旱胁迫。
刘宇等[23]克隆到花生抗病基因,并测得在黄曲霉侵染后,种皮、籽仁及果皮中基因PnAG3的表达量都上升。而经黄曲霉胁迫后,AhNDrp基因在0-3 d中表达量持续上升,说明该基因可能参与了黄曲霉抗病反应过程。
NBS-LRR类基因是一类抗逆性基因,黄曲霉是一种致病性真菌,而在干旱胁迫下,黄曲霉菌株可能成为土壤中的优势菌株,导致花生黄曲霉毒素污染、品质下降等。所以测定AhNDrp基因在干旱和黄曲霉胁迫下的表达量,为在理想遗传背景下引进高效抗病基因的育种工作提供一定的参考。
4 结论本研究克隆了花生AhNDrp基因的全长序列,含有NBS-LRR保守结构域,该基因在花生根部特异表达,并根据实时荧光定量PCR结果,推测其可能参与黄曲霉胁迫响应。
| [1] | Krasensky J, Jonak C. Drought, salt, and temperature stress-induced metabolic rearrangements and regulatory networks. Journal of Experimental Botany , 2012, 63 (4) : 1593–1608. DOI:10.1093/jxb/err460 |
| [2] | Hulbert SH, Webb CA, Smith SM, et al. Resistance gene complexes:evolution and utilization. Annual Review of Phytopathology , 2001, 39 (4) : 285–312. |
| [3] | Martin GB, Bogdanove AJ, Sessa G. Understanding the functions of plant disease resistance proteins. Annual Review of Plant Biology , 2003, 54 (4) : 23–61. |
| [4] | Bai J, Pennill LA, Ning J, et al. Diversity in nucleotide binding site-leucine-rich repeat genes in cereals. Genome Research , 2003, 12 (12) : 1871–1884. |
| [5] | Meyers BC, Kozik A, Griego A, et al. Genome-wide analysis of NBS-LRR-encoding genes in Arabidopsis. Plant Cell Online , 2003, 15 (4) : 809–834. DOI:10.1105/tpc.009308 |
| [6] | Yue JX, Meyers BC, Chen JQ, et al. Tracing the origin and evolutionary history of plantnucleotide-binding site-leucine-rich repeat(NBS-LRR)genes. New Phytologist , 2011, 193 (4) : 1049–1063. |
| [7] | Shen QH, Saijo Y, Mauch S, et al. Nuclear activity of MLA immune receptors links isolate-specific and basal disease-resistance responses. Science , 2007, 315 (5815) : 1098–1103. DOI:10.1126/science.1136372 |
| [8] | Ooijen GV, Mayr G, Kasiem MMA, et al. Structure function analysis of the NB-ARC domain of plant disease resistance proteins. Journal of Experimental Botany , 2008, 59 (6) : 1383–1397. DOI:10.1093/jxb/ern045 |
| [9] | Takken FL, Albrecht M, Tameling WI. Resistance proteins:molecular switches of plant defence. Current Opinion in Plant Biology , 2006, 9 (4) : 383–390. DOI:10.1016/j.pbi.2006.05.009 |
| [10] | Tameling WIL, Elzinga SDJ, Darmin PS, et al. The tomato R gene products I-2 and MI-1 are functional ATP binding proteins with ATPase activity. Plant Cell , 2002, 14 (11) : 2929–2939. DOI:10.1105/tpc.005793 |
| [11] | Geng LL, Duan XH, Liang C, et al. Mining tissue-specific contigs from peanut(Arachis hypogaea L.)for promoter cloning by deep transcriptome sequencing. Plant & cell physiology , 2014, 55 (10) : 1793–1801. |
| [12] | 梁炫强, 潘瑞炽, 周桂元. 活性氧及膜质过氧化与花生抗黄曲霉侵染的关系. 中国油料作物学报 , 2002, 24 (4) : 19–23. |
| [13] | 宋国立, 崔荣霞. 改良CTAB法快速提取棉花DNA. 棉花学报 , 1998 (5) : 273–275. |
| [14] | TRAUT, Thomas W. The functions and consensus motifs of nine types of peptide segments that form different types of nucleotide-binding sites. European Journal of Biochemistry , 1994, 222 (1) : 9–19. DOI:10.1111/ejb.1994.222.issue-1 |
| [15] | Mestre P, Baulcombe D. Elicitor-mediated oligomerization of the tobacco N disease resistance protein. Plant Cell , 2006, 18 (2) : 491–501. DOI:10.1105/tpc.105.037234 |
| [16] | Ade J, Deyoung BJ, Golstein C, et al. Indirect activation of a plant nucleotide binding site-leucine-rich repeat protein by a bacterial protease. Proceedings of the National Academy of Sciences , 2007, 104 (7) : 2531–2536. DOI:10.1073/pnas.0608779104 |
| [17] | Eitas TK, Dangl JL. NB-LRR proteins:pairs, pieces, perception, partners, and pathways. Current Opinion in Plant Biology , 2010, 13 (4) : 472–477. DOI:10.1016/j.pbi.2010.04.007 |
| [18] | Takken FL, Goverse A. How to build a pathogen detector:structural basis of NB-LRR function. Current Opinion in Plant Biology , 2012, 15 (4) : 375–384. DOI:10.1016/j.pbi.2012.05.001 |
| [19] | 何利. 桑树NBS类抗病基因的克隆与表达分析[D]. 镇江:江苏科技大学, 2014. http://cdmd.cnki.com.cn/Article/CDMD-10289-1014077297.htm |
| [20] | 王海燕, 杨文香, 刘大群. 小麦NBS-LRR类抗病基因同源序列的分离与鉴定. 中国农业科学 , 2006, 39 (8) : 1558–1564. |
| [21] | 刘建成, 段可, 李静, 等. 草莓NBS-LRR家族基因FaNBS1的克隆与表达分析. 果树学报 , 2011 (6) : 1025–1031. |
| [22] | 赵小波, 张廷婷, 闫彩霞, 等. 花生中一个NBS-LRR类基因的克隆和表达分析. 山东农业科学 , 2015 (5) : 1–5. |
| [23] | 刘宇, 闫彩霞, 张延婷, 等. 花生NBS-LRR类抗病基因的克隆及原核表达. 中国农业科技导报 , 2010, 12 (3) : 73–78. |





