2. 中国农业科学院作物科学研究所 国家农作物基因资源与遗传改良重大科学工程 农业部北京大豆生物学重点实验室,北京 100081
2. MOA Key Lab of Soybean BiologyBeijing,National Key Facility of Crop Gene Resource and Genetic Improvement,Institute of Crop Sciences,Chinese Academy of Agricultural Sciences,Beijing 100081
植物开花是植物生长发育的一个重要过程[1]。植物响应并整合环境和内源信号,产生相应的反应信号,然后以网络应答的方式将新信号传递给开花整合因子,再由其促进开花基因LEAFY(LFY)和APETALA1(AP1)表达,最终促使植物开花[2, 3]。AP1对控制植物花分生组织特性与花器官形成具有重要的作用[4, 5],属于MADS-box基因家族[6, 7]。在大豆[8]、苹果[9]、百合[10]、樱桃[11]、建兰[12]和杨树[13]等多种植物中已分别克隆出AP1同源基因。异位表达各种植物的AP1基因,均促进拟南芥或烟草开花,且此性状可遗传到子代[14]。
大豆(Glycine max)起源于中国,是植物性蛋白的主要来源和重要的工业原料[15]。大豆开花影响籽粒产量,对高产育种具有重要作用[16]。传统方法通过人工调控(控制光照、改变温度、使用激素等)花期,而转基因技术较多集中于大豆抗病虫害、抗逆性及大豆油分等方面研究[17],应用转基因控制大豆开花从而提高产量方面研究较少。采用分子遗传学和分子生物学改良品种,将促进开花的AP1基因导入短日照植物中,改变植物开花对短日照的依赖,促进开花,提高产量,可加快育种进程。大豆基因组具有多拷贝特征,AP1同源基因有4个拷贝[18],本研究以其中一个APETAL1(GmAP1)同源基因为目标,利用植物遗传转化技术将该基因转入到拟南芥中,对其功能进行初步分析,探讨其功能与大豆花发育的关系,为进一步研究大豆花发育的分子机理提供理论依据。
1 材料与方法 1.1 材料实验中植物材料为大豆品种天隆1号,种植于中国农业科学院作物科学研究所人工气候室(16 h光照,8 h黑暗,25℃),开花期取花蕾,提取RNA,用于基因克隆,分别取花蕾的花萼、花瓣、雄蕊和雌蕊,提取RNA,用于表达分析。拟南芥(Arabidopsis thaliana)为Columbia(Col-0)生态型背景。拟南芥突变体ap1.3由中国科学院植物研究所孟征研究员惠赠。大肠杆菌DH5α,农杆菌GV3101,载体Fu76-35S、Fu28和Fu39-2[19]均由本实验室保存备用。实验所用的高保真DNA聚合酶,反转录试剂盒、质粒提取试剂盒,琼脂糖凝胶回收试剂盒分别购自TaKaRa公司、博迈德生物公司和天根生物技术公司。实验用引物由华大科技公司合成,序列测定由北京睿博兴科生物技术有限公司完成。
1.2 方法 1.2.1 AP1克隆及表达载体构建根据Phytozome数据库中Glycine max Wm82.a2.v1预测基因序列,设计引物(表 1)扩增GmAP1基因的编码序列,根据北京全式金生物技术有限公司RNA提取试剂盒提取天隆1号RNA,反转录反应总体积10 μL:5× PrimeScript Buffer 2 μL,PrimeScript RT Enzyme Mix I 0.5 μL,Oligo dT Primer 0.5 μL,Random 6 mers 0.5 μL,总RNA 1 μL(500 ng/μL)用RNase Free H2O补足至10 μL。通过37℃ 15 min,85℃ 5 s,完成反转录反应。PCR体系:2×PrimeSTAR GC Buffer 25 μL,dNTP Mixture 4 μL,正反向引物各1 μL(10 μmol/L),模板5 μL,PrimeSTAR HS DNA Polymerase 0.5 μL,用灭菌水补足至50 μL。PCR反应程序为:94℃ 3 min;98℃ 10 s,60℃ 30 s,72℃ 1 min/kb,循环35次;72℃ 7 min。用1.0%琼脂糖凝胶电泳检测扩增产物。回收PCR产物,与Fu28载体连接;连接产物转化大肠杆菌DH5α,挑取单克隆,通过菌落PCR鉴定阳性克隆。阳性克隆委托北京睿博兴科生物技术有限公司测序,序列测序正确的质粒通过LR反应到表达载体Fu39-2,转化大肠杆菌DH5α,挑取单克隆,通过菌落PCR鉴定阳性克隆。阳性克隆委托北京睿博兴科生物技术有限公司测序,最后转化农杆菌GV3101,农杆菌花浸染法转化拟南芥。
在转录本序列设计实时定量PCR特异引物(表 1),扩增长度113 bp。PCR产物转化大肠杆菌DH5α,挑取单克隆,通过菌落PCR鉴定阳性克隆,委托北京睿博兴科生物技术有限公司测序,序列与扩增序列完全匹配。实时定量PCR的反应体系:SYBP Premix Ex Taq 7.5 μL,ROX reference Dye 0.3 μL,正反引物各1.5 μL(2 μmol/L),cDNA 1.5 μL(RNA 325 ng),用超纯水补足15 μL。生物重复3次。实时定量PCR反应程序:95℃ 30 s;95℃ 5 s,60℃ 30 s,40个循环。以ACT11为内参基因,超纯水作模板为阴性对照,采用2-△△Ct法计算相对表达量。
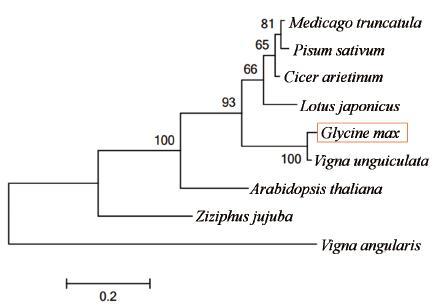
2 结果 2.1 GmAP1编码蛋白聚类分析及其序列特征分析表明GmAP1(基因序列号:Glyma08g36380)编码236个氨基酸(图 1),其中包含MADS-box结构域和K-box结构域(图 2),属于MADS-box基因家族成员。经Blast进行同源序列比对,GmAP1氨基酸序列与豇豆(Vigna unguiculata)、百脉根(Lotus japonicus)、鸡豆(Cicer arietinum)、蒺藜苜蓿(Medicago truncatula)、豌豆(Pisum sativum)、红豆(Vigna angularis)和大枣(Ziziphus jujuba)等物种中的AP1或AP1-like的同源性达到80%以上(图 3)。其中MADS-box结构域和K-box结构域高度保守,C-末端以CF结尾,而不是以法尼基转移酶的识别位点CFAA结尾。

|
| 图 1 GmAP1 编码区的核酸序列及编码的氨基酸序列 |

|
| 图 2 GmAP1 与其他AP1 同源基因氨基酸序列比对 |
|
| 图 3 GmAP1与其他AP1同源基因的聚类分析 |
以拟南芥APETALA1(AP1)基因为参照,根据Phytozome数据库中大豆基因组Glycine max Wm82.a2.v1序列预测的GmAP1基因序列设计引物,以大豆cDNA为模板进行PCR扩增,得到750 bp左右的特异条带(图 4)。测序结果表明该片段长度为711 bp,与Glycine max Wm82.a2.v1中的预测基因序列相同,且和已报道AP1同源基因的同源性较高,表明该片段是大豆的AP1同源基因。

|
| 图 4 GmAP1 CDS全长扩增产物 |
利用实时定量RT-PCR(Real time-PCR)的方法检测GmAP1在大豆不同花器官的表达情况。结果(图 5)表明,在大豆不同花器官中GmAP1的表达有显著差异。GmAP1在萼片中表达量较高,在花瓣中表达量次之,在雄蕊和心皮中也有少量表达量。

|
| 图 5 GmAP1在大豆不同花器官中的表达 |
为进一步分析GmAP1的功能,将由35S启动GmAP1基因的载体转化拟南芥,获得转基因植株,观察p35S∷GmAP1转基因植株和野生型表型。结果(图 6)显示,在长日条件下(16 h光照,8 h黑暗),p35S∷GmAP1转基因植株比野生型植株早花,p35S∷GmAP1转基因植株在播种后13-15 d就开花,此时莲座叶数仅为两片,且株型小,花数量少;而野生型在播种后22-24 d才开花,莲座叶数为9-11片。p35S∷GmAP1转基因植株与野生型的花型不同,拟南芥野生型花有4个花萼,4个花瓣,6个雄蕊,1个雌蕊,而p35S∷GmAP1转基因植株花有4个花萼,8个花瓣,10雄蕊和2个雌蕊(图 7)。

|
| 图 6 GmAP1过表达对植株开花的影响 A:转基因植株(p35S∷GmAP1)与野生型植株开花时间;B:转基因植株(p35S∷GmAP1)与野生型植株莲座叶数 |

|
| 图 7 异位表达GmAP1转基因拟南芥表型分析 A-D:p35S∷GmAP1转基因植株与野生型植株表型;E-H:p35S∷GmAP1转基因植株与野生型植株花器官表型 |
本研究成功克隆得到大豆AP1同源基因GmAP1,其氨基酸序列与许多物种中AP1同源基因编码的氨基酸序列同源性达80%以上,并与豇豆(Vigna unguiculata)、百脉根(Lotus japonicus)、鸡豆(Cicer arietinum)、蒺藜苜蓿(Medicago truncatula)、豌豆(Pisum sativum)等豆科植物在一个分支上。GmAP1的MADS-box和K结构域相对比较保守,这些结果表明,大豆GmAP1属于MADS-box家族基因。但是,GmAP1蛋白C-末端以CF结尾,而不是以法尼基转移酶的识别位点CFAA结尾。
AP1基因属于A类基因[20],调控花萼和花瓣的发育[21-23]。实时定量RT-PCR分析表明,GmAP1在大豆不同花器官中表达量不同,在花萼和花瓣中表达量相对于雄蕊和雌蕊较高。在拟南芥中,AP1同源基因作为花分生组织决定基因,在花原基发育早期阶段发挥作用[6, 24];作为花器官特异基因,决定花器官起始和发育[4, 5]。不同物种的AP1同源基因异位表达均早花,但花器官结构的改变却不相同。在烟草中过表达大豆Jackson品种的AP1-like基因(Glyma16g13070.1),表现出早花,植株矮小,正常花瓣处出现了雄蕊状的花瓣[8]。苹果中AP1-like基因MdMADS5过表达使拟南芥早花,花序较短,花蕾不同,尤其是花萼[9]。在烟草和拟南芥中过表达桦树PsnAP1-1和PsnAP1-2基因,表现出早花,植株矮小,融合终端花,花畸形[25]。本研究在拟南芥中过表达大豆天隆1号品种的GmAP1基因,植株表现出早花,植株矮小,花数量少,花器官(花瓣、雄蕊和雌蕊)数增多。因此,GmAP1基因对大豆开花时间及花器官的形成可能有重要作用。
大豆AP1基因是多拷贝基因[18],Chi等[8]在烟草中过表达大豆Jackson品种的AP1-like基因,表现出早花,植株矮小,正常花瓣处出现了雄蕊状的花瓣。本研究以另一个AP1同源基因为目标,对该基因进行表达分析,以及在拟南芥中过表达该基因进行初步的功能分析,与Chi等早花结果一致,但花器官结构的改变不同。结果说明,大豆中不同GmAP1基因拷贝的功能并不完全相同,它们在进化过程中发生了功能的分化。对这两个GmAP1基因进行比较研究分析,将进一步解析它们在功能上的协作与特化机制。
4 结论本研究克隆获得大豆天隆1号GmAP1基因,并证实该基因参与大豆开花时间以及花器官形态建成的调控。
| [1] | 陈晓, 陈彦惠, 任永哲. 植物开花转换的分子生物学研究. 分子植物育种 , 2005 (4) : 557–565. |
| [2] | Weigel D, Alvarez J, Smyth DR, et al. LEAFY controls floral meristem identity in Arabidopsis. Cell , 1992, 69 (5) : 843–859. DOI:10.1016/0092-8674(92)90295-N |
| [3] | Wagner D, Wellmer F, Dilks K, et al. Floral induction in tissue culture:a system for the analysis of LEAFY-dependent gene regulation. The Plant Journal , 2004, 39 (2) : 273–282. DOI:10.1111/tpj.2004.39.issue-2 |
| [4] | Gustafson-Brown C, Savidge B, Yanofsky MF. Regulation of the Arabidopsis floral homeotic gene APETALA1. Cell , 1994, 76 (1) : 131–143. DOI:10.1016/0092-8674(94)90178-3 |
| [5] | Wagner D, Sablowski RWM, Meyerowitz EM. Transcriptional activa-tion of APETALA1 by LEAFY. Science , 1999, 285 (5427) : 582–584. DOI:10.1126/science.285.5427.582 |
| [6] | Mandel MA, Gustafson-Brown C, Savidge B, et al. Molecular charac-terization of the Arabidopsis floral homeotic gene APETALA1. Nature , 1992, 360 (19) : 273–277. |
| [7] | Weigel D, Meyerowitz EM. Activation of floral homeotic genes in Arabidopsis. Science , 1993, 261 (24) : 1723–1726. |
| [8] | Chi Y, Huang F, Liu H, et al. An APETALA1-like gene of soybean regulates flowering time and specifies floral organs. Journal of Plant Physiology , 2011, 168 (18) : 2251–2259. DOI:10.1016/j.jplph.2011.08.007 |
| [9] | Kotoda N, Wada M, Kusaba S, et al. Overexpression of MdMADS5, an APETALA1-like gene of apple, causes early flowering in transgenic Arabidopsis. Plant Science , 2002, 162 (5) : 679–687. DOI:10.1016/S0168-9452(02)00024-9 |
| [10] | Chen MK, Lin IC, Yang CH. Functional analysis of three lily(Lil-ium longiflorum)APETALA1-like MADS box genes in regulating floral transition and formation. Plant and Cell Physiology , 2008, 49 (5) : 704–717. DOI:10.1093/pcp/pcn046 |
| [11] | Wang J, Zhang X, Yan G, et al. Over-expression of the PaAP1 gene from sweet cherry(Prunus avium L. )causes early flowering in Arabidopsis thaliana. Journal of Plant Physiology , 2013, 170 (3) : 315–320. DOI:10.1016/j.jplph.2012.09.015 |
| [12] | 吴菁华, 吴少华, 杨超, 等. 建兰 AP1 基因的克隆, 表达及其与 MADS-box 转录因子相互作用的分析. 园艺学报 , 2013, 40 (10) : 1935–1942. |
| [13] | 李爽, 郑唐春, 代丽娟, 等. 小黑杨 PsnAP1-1 及 PsnAP1-2 基因的克隆及原核表达分析. 植物研究 , 2014, 34 (4) : 498–504. |
| [14] | Peña L, Martín-Trillo M, Juárez J, et al. Constitutive expression of Arabidopsis LEAFY or APETALA1 genes in citrus reduces their generation time. Nature Biotechnology , 2001, 19 (3) : 263–267. DOI:10.1038/85719 |
| [15] | 邱丽娟, 王昌陵, 周国安, 等. 大豆分子育种研究进展. 中国农业科学 , 2007, 40 (11) : 2418–2436. |
| [16] | 赵双进, 唐晓东, 赵鑫, 等. 大豆开花落花及时空分布的观察研究. 中国农业科学 , 2013, 46 (8) : 1543–1554. |
| [17] | 任海祥, 南海洋, 曹东, 等. 大豆转基因技术研究进展. 东北农业大学学报 , 2012, 43 (7) : 6–12. |
| [18] | 胡瑞波, 范成明, 李宏宇, 等. 大豆MIKC型MADS—box 基因家族分析. 分子植物育种 , 2009, 7 (3) : 429–436. |
| [19] | Wang X, Fan CM, Zhang XM, et al. BioVector, a flexible system for gene specific-expression in plants. BMC Plant Biology , 2013, 13 (198) : 1471–2229. |
| [20] | 胡丽芳, 金志强, 徐碧玉. MADS-box 基因对花的发育及开花早晚的影响. 生命科学研究 , 2004, 8 (4) : 7–12. |
| [21] | Bowman JL, Alvarez J, Weigel D, et al. Control of flower development in Arabidopsis thaliana by APETALA1 and interacting genes. Development , 1993, 119 : 721–743. |
| [22] | Yalovsky S, Rodríguez-Concepción M, Bracha K, et al. Prenylation of the floral transcription factor APETALA1 modulates its function. The Plant Cell , 2000, 12 (8) : 1257–1266. DOI:10.1105/tpc.12.8.1257 |
| [23] | Ng M, Yanofsky MF. Activation of the Arabidopsis B class homeotic genes by APETALA1. The Plant Cell , 2001, 13 (4) : 739–753. DOI:10.1105/tpc.13.4.739 |
| [24] | Kempin SA, Savidge B, Yanofsky MF. Molecular basis of the cauliflower phenotype in Arabidopsis. Science , 1995, 267 (5197) : 522–525. DOI:10.1126/science.7824951 |
| [25] | Zheng TC, Li S, Zang LN, et al. Overexpression of two PsnAP1 genes from Populus simonii× P. nigra causes early flowering in transgenic tobacco and Arabidopsis. PLoS One , 2014, 9 (10) : e111725. DOI:10.1371/journal.pone.0111725 |





