2. 上海出入境检验检疫局,上海 200135;
3. 中国检验检疫科学研究院,北京 100176
2. Shanghai Entry-Exit Inspection and Quarantine Bureau,Shanghai 200135;
3. Chinese Academy of Inspection and Quarantine,Beijing 100176
自1996年国际上首次种植转基因作物至今已有19个年头,转基因作物的发展可谓突飞猛进,截至2014年全球转基因作物种植面积已达1.815亿hm2[1]。由于公众对转基因作物食用安全和环境安全性的担忧,很多国家和地区均实施了标识管理措施,并设定了阈值。如欧盟等国实施强制性的转基因标签制度,欧盟和俄罗斯设定的阈值为0.9%[2, 3],日本和中国台湾为5.0%[4, 5],韩国为3.0%[6]等;美国和加拿大实施自愿性的转基因标签制度;我国目前对转基因产品实施无阈值的强制性标识制度[7]。为了促进标识管理制度的顺利实施,与之相对应的转基因产品精准定量检测方法的研制至关重要。
目前食品中转基因成分的定量检测方法多采用实时荧光PCR方法[8-11],是通过标准物质制备的标准曲线对未知样品进行定量分析的方法[12]。然而,该方法由于影响因素较多,如标准物质和样品间背景不同,标准曲线线性范围有限,低拷贝DNA扩增可重复性差等,常造成定量结果准确性差,偏差大[13-16]。
数字PCR(digital polymerase chain reaction,dP-CR)是近年来在实时荧光PCR基础上发展起来的微量DNA 分子定量检测新技术。dPCR是将PCR体系分配到足够小的反应单元中,实现每个反应单元只有单个模板分子进行PCR扩增,再采用泊松分布原理,根据阳性微滴与阴性微滴数的比例计算目标分子拷贝数,实现绝对定量[17-19]。该方法降低了标准曲线对测量结果产生影响等问题,降低了基体效应,实现了PCR 扩增的样品分离,消除了本底信号的影响,提高了低拷贝DNA的扩增灵敏度[20]。相比实时荧光PCR,数字PCR具有更好的测量独立性,且无需任何校准物,具有更高的特异性、灵敏度、精确性和稳定性。
转基因水稻LL62品系由拜耳公司研发,由于在基因组上重组bar基因从而耐受草铵磷除草剂。目前该水稻品系已在6个国家获得了种植、食用、饲用等不同方面的批准[21]。但我国迄今还未对境外任何转基因水稻品系颁发农业转基因生物安全证书。因此在水稻进境检测中,口岸重点关注我国未批准品系的检测监管,包括LL62品系。为此,本研究基于外源插入片段和水稻基因组DNA的3'端旁侧序列,建立了转基因水稻LL62品系特异性微滴式数字PCR定量检测方法。
1 材料与方法 1.1 材料转基因玉米(Zea mays L.)Bt11、MON98140、NK603和MON810品系,转基因棉花(Gossypium)T304-40、GHB119和281-24-236×3006-210-23品系标准物质购于欧盟联合研究中心标准物质与测量研究所(Institute for Reference Materials and Measurements,IRMM)。转基因棉花MON88913、MON531、MON15985和 MON1445品系,转基因大豆(Glycine max)GTS40-3-2和MON89788品系以及转基因水稻(Oryza sativa)LL62品系(0306-I5)标准物质购于美国油脂化学家协会(American Oil Chemists’ Society,AOCS)。转基因水稻TT51-1、KF6和KMD以及非转基因水稻、棉花、大豆、玉米样品由中国检验检疫科学研究院提供或本实验室储备。
1.2 方法 1.2.1 DNA提取所有转基因与非转基因植物材料均根据植物基因组DNA提取试剂盒(Cat:#DP305-02,TianGen,北京)的操作方法进行基因组DNA提取。利用微量分光光度计(GE公司Nanovue Plus,美国)测定提取DNA溶液的浓度并通过测定A260/A280和A260/A230值来衡量提取的基因组DNA质量。DNA稀释至10 ng/µL保存备用。
1.2.2 混合样品制备为了验证建立的转基因水稻LL62 品系特异性微滴数字PCR定量检测方法的精确度和准确度,本研究分别制备了含有10%、1%和0.1% 3个百分比含量的转基因水稻LL62品系成分混合样品,标记为S1、S2和S3。制备过程为:首先分别吸取20 µL 100%的LL62 DNA标准溶液(10 ng/µL)和180 µL非转基因水稻DNA溶液(10 ng/µL)置于1.5 mL离心管中,使用涡旋震荡仪将其充分混合均匀,制成10.0%混合样品S1;然后分别吸取10 µL混合样品S1和90 µL非转基因水稻DNA溶液(10 ng/µL)置于1.5 mL离心管中,使用涡旋震荡仪将其充分混匀,得到1.0%混合样品S2;最后分别吸取10 µL混合样品S2和90 µL非转基因水稻DNA溶液(10 ng/µL)置于1.5 mL离心管中,充分混匀后得到0.1%混合样品S3。
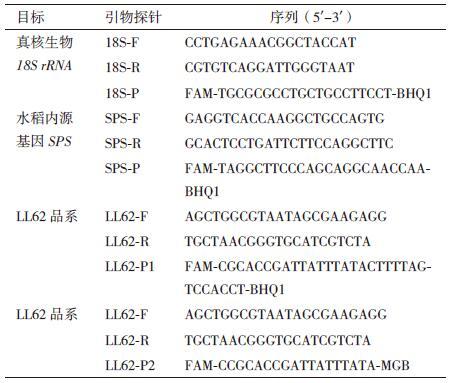
1.2.3 引物和探针设计及筛选在NCBI数据库中查找转基因水稻LL62品系的外源插入基因和基因组DNA邻接区序列信息,得到3'端旁侧序列(GenBank号为NC_008399.2)。由于数字PCR扩增对引物和探针的特殊要求,如扩增效率高、荧光信号强、本底低等,采用PrimerExpress2.0软件设计转基因水稻LL62品系以及水稻内标准基因SPS的特异性引物和探针序列,以混合样品S1(10.0%)DNA为模板,分别对多套转基因水稻LL62品系特异性检测体系进行测试,对比其反应效率的高低、微滴生成的稳定性等因素,筛选适宜的引物和探针,并与欧盟发布的转基因水稻LL62品系特异性检测方法[19]进行比较。同时对水稻内源标准基因SPS的扩增体系也进行了筛选。引物及探针由上海辉睿生物科技有限公司合成并纯化,引物和探针序列见表 1。
微滴式数字PCR反应体系总体积为20 µL,其中1×ddPCRTM Supermix for Probes(no dUTP)(美国Bio-Rad公司,Cat#186-3024)10 µL;上下游引物(10 µmol/L)各1.5 µL;探针(10 µmol/L)0.75 µL;超纯水4.25 µL;DNA模板2 µL。
配制反应混合液,并充分混匀后加入微滴发生板(美国Bio-Rad公司,Cat:#951020362),使用微滴发生器将反应混合液制成约 2×104个微滴。为防止微滴破裂,用排枪缓慢吸取微滴转移入96 孔PCR反应板中进行PCR。反应条件为:95℃ 10 min;95℃ 30 s,60℃ 1 min,40 个循环;98℃ 10 min。将反应后的96孔板放入微滴分析仪(美国Bio-Rad公司)进行荧光测定和数据读取,后采用Quantasoft分析软件(美国Bio-Rad公司)计算微滴生成的个数,并计算出阳性微滴数,换算得到样品浓度(拷贝/µL PCR产物)。
1.2.5 特异性实验采用22种转基因及非转基因植物材料,包括常规水稻、棉花、大豆和玉米样品,转基因水稻LL62、KMD、KF6和TT51-1品系,转基因大豆GTS40-3-2和MON89788品系,转基因玉米Bt11、MON98140、NK603和MON810品系以及转基因棉花T304-40、GHB119、281-24-236×3006-210-23、MON88913、MON531、MON1445和MON15985品系基因组DNA为模板,进行PCR扩增。每个样品重复2次,采用微滴式数字PCR方法进行检测,测试建立方法的特异性。
1.2.6 可重复性实验为测试建立的转基因水稻LL62品系微滴式数字PCR检测方法的可重复性,取含有10%转基因水稻LL62品系的样品DNA溶液(100 ng/µL),用0.1×TE(Tris-HCl,EDTA·Na,pH8.0)缓冲液连续梯度稀释至6个浓度:50、10、2、0.4、0.08和0.001 6 ng/µL,即121 000、24 200、4 840、968、193.6和38.72拷贝/µL水稻基因组DNA(根据1 ng≈2 420拷贝水稻单倍体基因组DNA计算[20]),分别扩增LL62品系特异性序列和内源标准基因SPS,每个浓度每个目标设置3次平行重复,实验共重复3次,共得到9个数值。然后分别计算标准偏差(standard deviation,SD)和相对标准偏差(relative standard deviation,RSD)来判断方法的可重复性。
1.2.7 准确度、精确度测定将含有不同比例转基因水稻LL62成分的混合样品S1(10%)、S2(1%)、S3(0.1%)的DNA,用微滴式数字PCR定量方法分别扩增LL62品系特异性序列和内源标准基因SPS,每个样品重复测定3次,整个实验重复3次。根据内外源基因的拷贝数计算转基因含量,并计算测定值与真实值间的偏差(bias)、各平行重复之间的标准偏差(SD)、相对标准偏差(RSD)判定定量的准确度和精确度。
通过数字PCR仪运行后自动给出的内源标准基因SPS拷贝数(copies/μL)和LL62品系特异性序列的拷贝数(copies/μL),按照下列公式计算样品中转基因水稻LL62的相对含量[25]。

所有提取的植物基因组DNA经微量分光光度计测定,DNA浓度在100-200 ng/µL之间,OD260/OD280值在1.8-2.0之间。结果表明,所提取的基因组DNA符合进一步定量检测的要求。
2.2 引物探针检测体系比较微滴生成是影响微滴式数字PCR(droplet digital PCR,ddPCR)扩增的关键步骤,只有微滴数大于10 000时,DNA分子的分布才能符合泊松分布的统计学原理,系统才可以对阳性和阴性微滴进行有效计数。首先对欧盟发布的方法和本实验设计的方法进行扩增效果比对。两个方法的上下游引物一致,但探针序列不同,分别为LL62-P1和LL62-P2。采用LL62-P1探针的测试结果表明生成的微滴数处于15 264-18 319之间,采用LL62-P2探针测试生成的微滴数处于15 833-18 964之间,均大于10 000,满足ddPCR微滴的分析要求。ddPCR扩增形成的微滴如图 1所示。由图 1-A可见,采用LL62-P1进行的ddPCR反应中,本底荧光值达到7 851.8-8 331.8,区间较宽,而阳性扩增获得的荧光值介于14 197-14 488之间,阳性荧光扩增值与本底间差异约为6 000,差异偏小,阳性微滴和阴性微滴区分界限不明晰,对微滴的计数有一定影响;采用LL62-P2实施的反应中,ddPCR本底荧光值介于3 514.5-3 556.1之间,变化范围较小,且较LL62-P1扩增的本底荧光均值低4 500左右,生成的阳性微滴荧光值处于12 336-12 563之间,二者差异达到9 000左右,阳性微滴和阴性微滴界限分明,系统可更准确判读出阳性微滴和阴性微滴数目(图 1-B)。因此,确定采用本实验设计的LL62-P2扩增体系进一步进行转基因水稻LL62品系ddPCR定量检测方法建立。

|
| 图 1 采用LL62-P1(A)和LL62-P2(B)在微滴数字PCR 测定转基因水稻LL62 品系的微滴生成情况图 |
采用20个常见转基因植物和常规植物基因组DNA为模板,对建立的转基因水稻LL62品系数字PCR定量检测方法的特异性进行测试。当以真核生物18S rRNA引物/探针进行PCR扩增时,所有植物来源材料基因组DNA均有阳性微滴生成,微滴数在12 023-14 792之间,而阴性对照和空白对照无阳性微滴生成,表明提取的 DNA 适于用作微滴数字 PCR 扩增。当以水稻内源基因SPS引物/探针进行PCR扩增时,只有以水稻材料来源的基因组DNA有阳性微滴生成,其他植物来源材料则无阳性微滴生成。当以LL62品系特异性引物/探针进行PCR扩增时,只有LL62品系样品有阳性微滴生成,而其他转基因水稻、转基因大豆、转基因棉花、转基因玉米和非转基因植物样品则没有产生阳性微滴(表 2)。因此,设计的水稻内源SPS和 LL62 品系特异性扩增体系特异性好,可以进一步用于微滴式数字PCR定量检测转基因水稻LL62品系。
定量PCR反应的重复性反映所建立的数字PCR定量体系的稳定性和可靠性。为此,本实验取6个浓度的10%含量转基因水稻LL62样品DNA溶液(50、10、2、0.4、0.08 和 0.016 ng/µL)进行数字PCR反应。根据软件计算的阳性微滴数分析方法的可重复性。结果(表 3)显示,3次重复扩增生成微滴数的SD值介于1.00-264.49之间,RSD值介于0.60%-11.11%之间,DNA模板浓度越高,RSD值越小,RSD值均在可接受范围内(小于25%)[26]。因此,建立的转基因水稻LL62品系ddPCR定量检测方法微滴生成稳定,可重复性好。
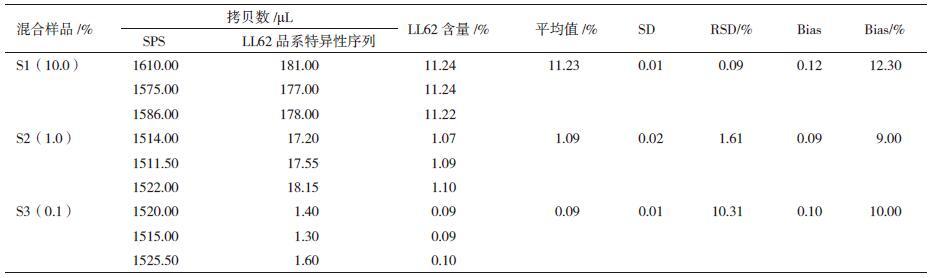
配制3个百分比含量混合样品S1(10.0%)、S2(1.0%)和S3(0.1%),对混合样品中LL62转基因水稻成分的含量进行了定量测试。结果如表 4所示,3个混合样品S1(10.0%)、S2(1.0%)和S3(0.1%)的百分比含量测定值分别为11.23%、1.09%和0.09%;测定值与真实值之间的偏差(bias)分别为0.12、0.09和0.10;每个混合样品的3次重复之间的标准偏差(SD)值介于0.01-0.02,相对标准偏差(RSD)值介于0.09%-10.31%,均在可接受的范围之内,而且偏差较小。结果表明,建立的转基因水稻LL62品系特异性微滴式数字定量PCR检测方法具有较好的特异性、可重复性、准确度和精确度,能够有效的对混合样品中LL62转基因成分进行定性和定量检测分析。
近年来,随着转基因作物商业化的不断发展,转基因产品的标识阈值制度和低水平混杂(low level presence,LLP)政策受到各国的关注,政策的实施很大程度上依靠转基因成分的精准定量。目前对转基因作物及其产品的定量检测方法一直采用基于标准曲线的相对定量,但由于技术的固有问题,很难达到较高的精确度,尤其是转基因成分含量低于1%水平时。近年发展起来的数字PCR定量检测技术,虽然也是基于实时荧光PCR扩增,但由于扩增是在足够小的仅容纳一个反应的微单元中进行,因此极大降低了反应间的相互抑制,提高了反应效率。
目前食品中转基因成分的定量检测方法多采用实时荧光PCR方法。如王渭霞等[9]建立了转基因水稻科丰6号定量检测方法;汪秀秀等[22]建立了转基因棉花GHB119品系特异性定量检测方法;苏长青等[27]建立了Bt63 实时荧光定量方法;吴永彬等[28]建立了转基因大豆 GTS40-3-2、玉米 NK603、油菜 RT73和水稻 TT51-1的定量检测方法等。而这些方法定量下限多在0.5%及以上,有些方法定量下限虽能低至0.1%,但多基于质粒标准分子的定量结果为依据而非植物材料,因此检测下限的局限性也使方法的应用受到了限制。
与实时荧光PCR定量检测方法相比,由于数字PCR是在足够小的反应单元中进行实时荧光PCR扩增,降低了反应间的相互影响和相互抑制,可重复性好,稳定性高;而且数字PCR方法采用直接计数的方法实现绝对定量,使定量结果更加准确,定量下限可低至0.1%或以下,而且由于数字PCR无需制备标准曲线,节省人力物力。数字PCR方法的应用对应对转基因产品阈值制度和低水平混杂措施的实施更加有利。
目前国内外采用数字PCR技术对转基因成分含量进行定量的研究还刚刚起步。Corbisier等[15]比较了数字PCR和实时荧光PCR两种技术应用于转基因玉米MON810品系定量检测上的优劣,认为数字PCR具有计量特性,在定量上有更大的优势。Burns等[29]采用经验证的应用于转基因成分检测的qPCR反应体系,评估了dPCR技术的绝对检测限和定量限,并表明dPCR 可以更精确地测量拷贝数。另外,基于数字PCR技术,国内外专家学者也进行了单细胞基因表达、临床诊断、生物样品种属鉴定和基因拷贝数分析等领域研究[30-33]。
数字PCR作为更精确和更灵敏的DNA定量检测新技术,实现了单分子DNA的绝对定量,为转基因产品的定量分析提供了新的技术手段,也为转基因产品中低含量成分的检测指出了一个新的潜在发展方向。我们期望在不远的将来,这项技术的发展在分子生物学基础研究和应用方面发挥更大的作用。
4 结论本研究基于3'端旁侧序列,建立了转基因水稻LL62品系特异性数字PCR定量检测方法。对方法的适用性鉴定结果表明,建立的数字PCR方法特异于转基因水稻LL62品系检测;方法可重复性好,生成微滴数的RSD值介于0.60%-11.11%之间,小于欧盟定量检测限25%的要求;准确度高,3个混合样品(10%、1%和0.1%)测定值与真实值之间的偏差(bias)分别为0.12、0.09和0.10,相对标准偏差(RSD)值介于0.09%-10.31%。因此,本研究建立的基因水稻LL62 品系特异性微滴数字PCR方法可以用于我国口岸农产品中转基因水稻LL62成分的定性定量分析,加强相关部门对转基因水稻及其制品的监控、安全评价和风险预警,同时也为我国转基因产品标识制度的顺利实施提供技术支持。
| [1] | Clive J. 2014年全球生物技术/转基因作物商业化发展态势. 中国生物工程杂志 , 2015, 35 (1) : 1–14. |
| [2] | European Commission. Regulation(EC)No. 1829/2003 of the European Parliament and of the Council of 22 September 2003 on genetically modified food and feed. Official Journal of the European Union , 2003, L268 : 1–23. |
| [3] | Russian Federation. SanPiN 2. 3. 2. 2227- 07“Additions and Changes #5 to the Sanitary- Epidemiological Rules SanPiN 2. 3. 2. 1078- 01 “Hygiene Requirements to Safety and Nutrition Value of Food Products”. 2007. http://cn.bing.com/academic/profile?id=2020949004&encoded=0&v=paper_preview&mkt=zh-cn |
| [4] | Ministry of Agriculture, Forestry and Fisheries of Japan. Notification 1775. Food and Marketing Nureau[S] . Tokyo, Japan: 2000. |
| [5] | Chiueh LC, Chen YL, Shih DY. Study on the detection method of six varieties of genetically modified maize and processed foods. Journal of Food and Drug Analysis , 2002, 10 (1) : 25–33. |
| [6] | Ministry of Agriculture and Forestry of Korea(MAF). Notification No. 2000- 31, Guidelines for Labeling of Genetically Modified Agricultural Products[S] . Seoul, Korea: 2000. |
| [7] | 中华人民共和国农业部. 农业部第10号令《农业转基因生物标识管理办法》[S] . 2002. |
| [8] | 宋君, 雷绍荣, 刘勇, 等. 转基因玉米NK603品系特异定量PCR检测方法的建立. 生物技术通讯 , 2012, 23 (2) : 238–241. |
| [9] | 王渭霞, 赖凤香, 洪利英, 等. 实时定量PCR检测转基因水稻科丰6号插入拷贝数和转基因含量. 农业生物技术学报 , 2012, 20 (1) : 9–15. |
| [10] | 敖金霞, 高学军, 仇有文, 等. 实时荧光定量PCR技术在转基因检测中的应用. 东北农业大学学报 , 2009, 40 (6) : 141–144. |
| [11] | Li X, Pan LW, Li JY, et al. Establishment and application of event- specific polymerase chain reaction methods for two genetically modified soybean events, A2704-12 and A5547-127. Journal of Agricultural and Food Chemistry , 2011, 59 (24) : 13188–13194. DOI:10.1021/jf202806w |
| [12] | 潘良文, 田风华, 张舒亚. 转基因抗草丁膦油菜籽中Barnase基因的实时荧光定量PCR检测. 中国油料作物学报 , 2006, 28 (2) : 194–198. |
| [13] | Burns M, Burrell A, Foy C. The applicability of digital PCR for the assessment of detection limits in GMO analysis. European Food Research and Technology , 2010, 231 (3) : 353–362. DOI:10.1007/s00217-010-1277-8 |
| [14] | Heyries KA, Carolina T, Michael C, et al. Megapixel digital PCR. Nat Methods , 2011, 8 (8) : 649–651. DOI:10.1038/nmeth.1640 |
| [15] | Corbisier P, Somanath B, Lina P, et al. Absolute quantification of genetically modified MON810 maize(Zea mays L. )by digital polymerase chain reaction. Anal Bioanal Chem , 2010, 396 (6) : 2143–2150. DOI:10.1007/s00216-009-3200-3 |
| [16] | Terry CF, Shanahan DJ, Ballam LD, et al. Real-time detection of genetically modified soya using Lightcycler and ABI 7700 platforms with TaqMan, Scorpion, and SYBR GreenⅠchemistries. J AOAC International , 2002, 85 (4) : 938–944. |
| [17] | Bhat S, Herrmann J, Armishaw P, et al. Single molecule detection in nanofluidic digital array enables accurate measurement of DNA copy number. Anal Bioanal Chem , 2009, 394 (2) : 457–467. DOI:10.1007/s00216-009-2729-5 |
| [18] | 李春勇. 数字PCR技术原理及应用. 生物技术与世界 , 2014 : 9–13. |
| [19] | Sanders R, Huggett JF, Bushell CA, et al. Evaluation of digital PCR fo rabsolute DNA quantification. Anal Chem , 2011, 83 (17) : 6474–6484. DOI:10.1021/ac103230c |
| [20] | 李亮, 隋志伟, 王晶, 等. 基于数字PCR的单分子DNA定量技术研究进展. 生物化学与生物物理进展 , 2012, 39 (10) : 1017–1023. |
| [21] | 赵卫东, 郑文杰, 贺艳. 荧光 PCR 方法定性和定量检测BT63 转基因大米. 食品研究与开发 , 2009, 30 (3) : 133–135. |
| [22] | 汪秀秀, 杨捷琳, 宋青, 等. 转基因棉花GHB119品系特异性定量PCR检测方法的建立. 农业生物技术学报 , 2014, 22 (3) : 380–388. |
| [23] | Joint Research Centre Institute for Health and Consumer Protection Biotechnology & GMOs Unit. Event-specific method for the quantification of rice line LLRICE62 using Real-time PCR(CRLVL05/04VP). 2006. http://cn.bing.com/academic/profile?id=2152702818&encoded=0&v=paper_preview&mkt=zh-cn |
| [24] | National Center for Biotechnology Information. Oryza sativa(rice). [OL] http://www.ncbi.nlm.nih.gov/genome?term=txid4530%5Borgn%5D |
| [25] | Liu J, Guo JC, Zhang HB, et al. Development and in-house validation of the event-specific polymerase chain reaction detection methods for genetically modified soybean MON89788 based on the cloned integration flanking sequence. Journal of Agricultural and Food Chemistry , 2009, 57 (22) : 10524–10530. DOI:10.1021/jf900672d |
| [26] | European Network of GMO Laboratories(ENGL). Definition of minimum performance requirements for analytical methods of GMO testing. 2008. http://www.oalib.com/references/8802981 |
| [27] | 苏长青, 谢家建, 王奕海, 等. 转基因水稻Bt汕优63的整合结构和品系特异性定量PCR方法. 农业生物技术学报 , 2011, 19 (3) : 434–441. |
| [28] | 吴永彬, 肖维威, 张宝, 等. 转基因大豆、玉米、油菜和水稻品系特异性检测质粒标准分子的快速构建及应用. 中国生物化学与分子生物学报 , 2011, 27 (8) : 775–782. |
| [29] | Burns M, Burrell A, Foy C. The applicability of digital PCR for the assessment of detection limits in GMO analysis. European Food Research and Technology , 2010, 231 (3) : 353–362. DOI:10.1007/s00217-010-1277-8 |
| [30] | White AK, Michael V, Oleh IP, et al. High-throughput micro fluidic single-cell RT-qPCR. Proc Natl Acad Sci USA , 2011, 108 (34) : 13999–14004. DOI:10.1073/pnas.1019446108 |
| [31] | Jones MA, Bhide S, Chin E, et al. Targeted polymerase chain reaction-based enrichment and next generation sequencing for diagnostic testing of congenital disorders of glycosylation. Genet Med , 2011, 13 (11) : 921–932. DOI:10.1097/GIM.0b013e318226fbf2 |
| [32] | 胡伟, 陈荣华, 张晨, 等. 微滴式数字PCR技术用于生物样品种属鉴定和绝对定量. 法医学杂志 , 2014, 30 (5) : 342–345. |
| [33] | 姜羽, 胡佳莹, 杨立桃, 等. 利用微滴数字PCR分析转基因生物外源基因拷贝数. 农业生物技术学报 , 2014, 22 (10) : 1298–1305. |