药物的发现及发展需要表达大量正确折叠的重组蛋白。这些药用重组蛋白普遍用于治疗各种疾病,也常作为筛选新药物的靶蛋白。大肠杆菌具有生长速度快、基因操作方便和重组蛋白合成速度快等优点,使它成为表达多种重组蛋白最令人满意的宿主之一[1, 2]。在少数情况下,它能达到非常高的表达水平,其表达量能上升到细胞总蛋白的30%。大肠杆菌是第一个用来生产生物药物的表达载体,它的发展促使了1982年用于糖尿病治疗的人胰岛素得到相关监督部门的批准。1994年牛生长激素(bGH)的批准为生产源自大肠杆菌的药用异源蛋白设立了新标准(表 1)。引人瞩目的一点是胰岛素和牛生长激素都需要氧化蛋白折叠,此外,胰岛素是一种异质二聚体,重组胰岛素和生长激素的成功获得突出了基于大肠杆菌生产的多功能性和有效性。然而,由于大肠杆菌外源基因中存在大量会导致翻译错误的稀有密码子。这些错误若在药用蛋白使用的情况下,即便低水平表达都能导致人体不良的免疫反应,蛋白翻译中存在的细胞错误也可能影响其三级结构,从而间接影响重组蛋白生物活性。为了在大肠杆菌中获得经合适折叠加工且具有生物活性的药用蛋白,这篇综述中,我们将关注关键生化参数“理想调制”的内容,包括大肠杆菌中密码子偏好性、蛋白质翻译、分子伴侣及翻译后修饰。
细胞质中同源氨基酰化tRNA的水平可以显示大肠杆菌中密码子偏好性。较多的密码子在基因组中能普遍存在,也能高水平地进行表达。稀有密码子在基因组中也能经常遇到,其表达水平较低,在大肠杆菌中稀有的密码子在真核生物的基因组中却是丰富存在的。含有稀有密码子的外源基因表达时会出现翻译错误,这是由于在相关位点上存在核糖体衰减,在这些位点上氨基酸和稀有密码子tRNA耦合,这种现象不能避免[3]。此外,由于外源基因上稀有密码子存在而导致的翻译错误可能包括氨基酸替代、翻译的移码突变或提前终止等[4, 5]。Kane等[6]发现在框架内,稀有AGA密码子中两个氨基酸会存在跳跃现象。蛋白质的质量很大程度上被密码子的偏嗜性影响,这是由于赖氨酸并入了AGA密码子处的精氨酸[7]。因此,如果蛋白的质量由于翻译错误存在瑕疵,即使处于非常高的水平,重组蛋白的表达也是无用的。在大肠杆菌中问题最多的稀有密码子是AGA、AGG、CGG、CGA、CGG(精氨酸)、AUA(异亮氨酸)、GGA(甘氨酸)、CUA(亮氨酸)、CCC(脯氨酸)和AAG(赖氨酸)[8]。已经有记录表明,稀有精氨酸密码子AGG和AGA,在大肠杆菌中分别出现的频率为0.14%和0.21%[9]。
现有几种方法可以用来避免大肠杆菌中密码子的偏嗜性。一种方法是基于密码子偏好性去合成全基因组,这是目前首选的能改善大肠杆菌中外源蛋白表达的方法[10],但这一方法全基因合成的花费较高。另一种方法是利用外源基因序列的定点诱变产生密码子,然而,这一方法昂贵且随机性强,因为无论何时总有几个核苷酸需要被修正。第三种策略则通过大肠杆菌菌株与能编码tRNA的质粒进行共转化,最终同源得到稀有密码子[11]。通过增加限制性tRNA的拷贝数,大肠杆菌就能匹配外源基因中密码子的偏好性频率。几种商业化质粒,如pRARE,现在已被用于大肠杆菌中稀有tRNA的共表达。另外,这些质粒中包含p15A复制起始位点,该复制起始位点可以维护存在Cole1复制起始位点的大肠杆菌菌株。几种可用的商业化菌株如BL21(DE3)Codonplus-RIL,BL21(DE3)codonPlus-RP和Rosetta(DE3),这些菌株的质粒含有编码tRNA为稀有密码子的基因序列[12]。这一方法在提高重组人干扰素的生产上已有应用。含有人类干扰素-α2a基因和argU基因的质粒能编码稀有tRNA为精氨酸(AGG/AGA),大肠杆菌BL21(DE3)的共转化会导致IFN-α2a和重组蛋白具有更高水平的表达,这些蛋白可占总蛋白的25%[13]。在另一项研究中发现在替换稀有精氨酸密码子为更常使用的密码子后,人干扰素2 b的产量会增加9.5-11.5倍[14]。含有pRARE质粒的大肠杆菌Rosetta(DE3)菌株也被用来表达不同的人源蛋白,研究表明实验的68种蛋白中的35种其重组蛋白产量都能急剧增加[15, 16]。
2 蛋白质翻译大肠杆菌中有效的翻译起始位点需要一个核糖体结合位点(RBS),核糖体起始位点包括Shine-Dalgarno(SD)序列和翻译起始密码子[5]。Shine-Dalgarno(SD)序列一般位于起始密码子AUG上游的第7-9个核苷酸[17]。当翻译起始位点从含有共识(Shine-Dalgarno,SD)序列AAGGAGG的mRNA开始时会更有效,核糖体结合位点的二级结构对于翻译起始非常关键,而翻译效率通过大量存在的胸腺嘧啶和腺嘌呤会进一步增加[18]。翻译起始位点的效率同样也受到起始密码子后核苷酸的影响,并发现腺嘌呤在高度表达的基因中会普遍存在[19]。故翻译起始会受到各种因素的影响,包括共识的SD序列,起始密码子的核苷酸上游,核糖体结合位点的二级结构在内[20]。阻止核糖体连接的mRNA的二级结构在很大程度上可能会影响蛋白表达[21]。同样也观察到单一碱基的变化会破坏靠近SD区域的二级结构,也会导致RNA噬菌体MS2外壳蛋白表达水平发生500倍的改变。Seo和Park等[22]已开发一种极有效和简单的方法去设计5′-非翻译区(5′ UTR),这能用于大肠杆菌中的可调表达,因为含有SD和AU序列的5′ UTR在蛋白翻译中发挥显著的作用,重装蛋白的表达水平能被5′ UTR区域中简单变化所操纵。研究发现,mRNA中的二级结构能被RNA解旋酶破坏,如大肠杆菌的DEAD蛋白质。
研究表明,从T7启动子起始时,DEAD蛋白质的表达会将β-半乳糖苷酶表达量增加30倍,但在lac启动子起始时,表达水平却没有显著增加[23]。然而当DEAD蛋白质不表达时,从T7启动子起始的β-半乳糖苷酶的表达与lac启动子相比,虽然最终转录率增加10倍,但表达量却减少10倍[24, 25]。所以DEAD-box蛋白质在稳定mRNA时发挥了重要作用,因此DEAD-box蛋白质能被用以改善伴有疑似问题mRNA二级结构的基因表达。
在细菌基因组中,UAA是使用最频繁的终止密码子;其次是UGA和UAG。在翻译过程中,阅读终端密码子中的一个错误会延长蛋白合成,直到mRNA中另一个终止密码子出现。连读会导致含有额外C末端氨基酸的肽的合成,在人IFN-α2b中若用UGA替换UAA,蛋白表达水平会增加2倍[26]。研究表明,转录终止子能通过在mRNA的3′末端产生茎环结构使mRNA稳定。在大肠杆菌中,终止密码子UAA更为普遍地用于翻译终止。通过增加连续终止密码子或使用一个延长的UAAU终止密码子,翻译终止的效率能得到改善[27]。蛋白合成过程中的翻译错误能导致框架迁移突变、过早截断、更低的表达量和氨基酸的错误掺入,会对大肠杆菌中重组蛋白质量产生不利影响,而高水平表达和正确的表观分子质量却不能保证重组蛋白的翻译完整[28, 29]。
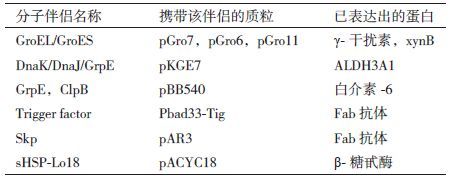
3 优化蛋白折叠的分子伴侣使用大肠杆菌宿主进行生物药物生产时遇到的一个主要问题是:异源蛋白质主要以不溶性聚集物包涵体的形式进行积累。而从这些包涵体中提取重组蛋白是一个冗长、繁琐的过程,需要一系列变性和复性步骤才能获得可溶并正确折叠的重组蛋白。一种改善大肠杆菌中蛋白质溶解度的策略是使用分子伴侣。在蛋白质合成过程中,通过分子伴侣和新生多肽链的相互作用阻止折叠过程中的聚集。一些分子伴侣能阻止蛋白聚集,而一些能促进折叠错误的蛋白重新折叠和溶解[30]。在大肠杆菌中应用最广泛和重要的细胞质伴侣是DnaK、DnaJ、GrpE、GroEL和GroES和触发因素(表 2),这些伴侣能单独及组合使用,用以改善大肠杆菌中蛋白质的溶解度[31, 32]。改善蛋白质再折叠的最有效并已广泛使用的伴侣组合是GroEL-GroES和DnaK-DnaJ-GrpE[33-35]。另外观察到DnaK-DnaJ-GrpE能协助未折叠蛋白的释放,GroEL-GroES能阻止肽的降解,触发因素与GroEL合作能改善GroEL和底物的结合,从而促进蛋白的折叠[36]。其他的伴侣如ClpB(Hsp 100),与DnaK组合后能增溶蛋白并防止其聚集[37]。其他热休克蛋白,如IpbA和IpbB,也能阻止热变性蛋白聚集。为了改善大肠杆菌中重组异源蛋白的生产,应该分析分子伴侣的不同组合,从而确定最有效的组合方式。研究表明,Skp和FkpA伴侣的共表达会增加大肠杆菌中抗体片段的溶解度[38]。相关信息表明GroEL-GroES的共表达会导致65%的抗B型利钠肽单链抗体(scFV)能以可溶形式存在,这一组合的可溶产量会比无伴侣时高2.4倍[39]。
另一种策略为通过降低生长温度来提高重组蛋白的溶解度[40]。在较低温度下生长会降低蛋白合成速度,也会阻止胞浆中折叠中间体的聚集。此外,它也能通过阻止分子内和分子间的疏水性改为相互作用去减少包涵体的形成,从而减少蛋白的聚集[41]。此方法已有效地提高了干扰素α-2、人生长激素及Fab片段等药用蛋白的溶解度[42, 43]。为便于大肠杆菌中可溶重组蛋白的生产,也可优化表达载体、启动子及表达宿主的选择,融合标签的应用、蛋白合成速度、培养基组成、诱导浓度和时间等参 数[44-46]。研究表明,异源蛋白分泌到大肠杆菌的周质中可能会提供一个生产复杂药用蛋白的机会。因为周质中的Dsb蛋白家族会帮助形成二硫键和氧化周质环境,从而促进异源蛋白的正确折叠。除此之外,周质中含有的极少宿主蛋白和低蛋白水解活性物质有利于下游加工,最终能获得高收益的药用蛋白。通过周质分泌,各种药用蛋白已经被研制成功,如商业化Fab片段、Leucentis和Cimza,以及一些全长无糖基化的抗体和scFvs[47]。OmpA、LamB、PhoA、STII、PelB 和木聚糖酶等分泌信号已被用于靶向大肠杆菌周质中的异源蛋白。Yim等[48]利用木聚糖酶信号肽去获得了高水平表达的粒细胞集落刺激因子(G-CSF),其在大肠杆菌周质中的产量能达4.2 g/L。另一个研究报道使用LamB信号肽后胰岛素样生长因子I(IGF)在周质中能高水平表达,其产量能达4.3 g/L[49]。除了信号肽,周质分泌的效率也依赖于大肠杆菌菌株、启动子强度和生长温度。在一些情况下,一些周质伴侣如二硫键氧化酶(DsbA)、异构酶(DsbC)和肽基异构酶的共表达能提高大肠杆菌中异源蛋白的产量。Reilly等[50]报道DsbA和DsbC的过表达能改善周质中全长抗体重轻链的组装效率,也能增加产量从0.1-1.05 g/L。另一个研究报道:当anti-CD20 scFv和周质伴侣Skp共表达时,anti-CD20 scF的产量随着改进的抗原结合亲和力而增加[51]。Lee等[52]报道了大肠杆菌中一种全长IgG有效表达系统的发展,研究表明5′ UTR序列的修饰和DsbC折叠酶的共表达会导致重轻链具有极高的表达水平,并且IgG组件在周质中也被显著改善。在高度优化的条件下,最终制备出全组装和功能上活跃的IgG,其产量高达362 mg/mL。这些研究清楚地表明,未来通过合适的工程加工,大肠杆菌宿主最终能高效及省时生产药用克隆抗体。
4 翻译后修饰生物技术专家多会在药用蛋白大规模生产中选择大肠杆菌。然而,若大肠杆菌中没有翻译后修饰(PTMs)过程,就会限制其生产重组生物药物。糖基化、磷酸化等翻译后修饰对于蛋白的功能活性非常关键,但由于缺乏PTM细胞机器,在大肠杆菌中这些修饰过程不能发生[53, 54]。蛋白质的N-连接糖基化是真核生物中最重要的翻译后修饰方式之一。Wacker等[55]在微生物空肠弯曲菌中发现了一种新的N-连接糖基化途径,也将功能活跃的N-连接糖基化途径成功地转入大肠杆菌中。空肠弯曲菌含有pgl基因簇,这一基因簇在各种糖蛋白的合成中会涉及到。通过向大肠杆菌中成功转入pgl途径,大肠杆菌生产了各种糖基化蛋白。虽然细菌的N-聚糖结构与真核生物中不一样,但把空肠弯曲菌的糖基化途径转入大肠杆菌的分子工程已经为在大肠杆菌中表达糖化蛋白铺平了道路[56, 57]。Valderrama-Rincon 等[56]已经成功在大肠杆菌中设计了真核的糖基化途径,包括酵母尿苷、二磷酸-N-乙酰葡糖胺转移酶Alg13和 Alg14、甘露糖Alg1 和 Alg2,以及从空肠弯曲菌中得到的寡糖PglB在内的4种真核糖基转移酶,在大肠杆菌中共表达用来合成能成功转移到靶向真核蛋白中天冬酰胺残基上的聚糖。这一方法也可用来开发针对多种细菌病原体的糖缀合物疫苗,与目前基于化学物质的疫苗生产方法相比,这种替代方法更高效和方便。目前,使用这一技术开发的痢疾杆菌O1复合糖疫苗已经成功通过I期临床试验。初步的疗效和安全性研究已经证明糖缀合物疫苗虽然安全,但能引起强烈的免疫反应。最终利用空肠弯曲杆菌N-连接糖基化体系生产出同时能针对革兰氏阴性和阳性病原体的糖缀合物疫苗[28, 58-61]。
5 展望目前虽然生产重组蛋白的酵母、哺乳动物细胞系和转基因动植物中存在不同的表达系统,但是改善大肠杆菌表达系统的相关技术仍在不断出现。大肠杆菌系统中遗传操作方便、基因组充分表征、宿主菌株种类多、成本较低、表达水平高等优点促使优化大肠杆菌中异源蛋白的生产。但偏置密码子偏好性、蛋白质溶解度、mRNA稳定性和二级结构等限制因素会妨碍大肠杆菌系统过表达异源基因。因为异源蛋白中稀有密码子存在而出现的翻译错误会导致氨基酸取代或移码突变,最终出现不期待的产品,所以重组蛋白密码子偏好性在确定其表达水平上起着关键作用。在基因组中替换稀有密码子为常见密码子能加强大肠杆菌中治疗蛋白的表达。同样,稀有密码子的tRNA编码基因的共表达能够增加大肠杆菌中治疗蛋白的表达水平。此外,异源蛋白的周质分泌会带来折叠和溶解度适当、纯化方便、蛋白高产量等优点。在大肠杆菌周质中已生产被批准的治疗用抗体片段,表明此方法在商业上可行。最近一系列研究已表明大肠杆菌菌株能针对每种药用蛋白进行特异性改造,从而获得高产量和高质量的产品,最终会为人类的健康作出卓越的贡献。
| [1] | Rodriguez V, Asenjo JA, Andrews BA. Design and implementation of a high yield production system for recombinant expression of peptides. Microbial Cell Fact , 2014, 13 (18) : 1–10. |
| [2] | Sahdev S, Khattar SK, Saini KS. Production of active eukaryotic proteins through bacterial expression systems:A review of the existing biotechnology strategies. Mol Cell Biochem , 2008, 307 (1-2) : 249–264. |
| [3] | McNulty DE, Claffee BA, Huddleston MJ, et al. Mistranslational errors associated with the rare arginine codon CGG in Escherichia coli. Protein Expr Purif , 2003, 27 (2) : 365–374. DOI:10.1016/S1046-5928(02)00610-1 |
| [4] | Kane JF. Effects of rare codon clusters on high-level expression of heterologous proteins in Escherichia coli. Curr Opin Biotechnol , 1995, 6 (5) : 494–500. DOI:10.1016/0958-1669(95)80082-4 |
| [5] | Sorensen HP, Laursen BS, Mortensen KK. Bacterial translation initiation-mechanism and regulation. Dev Biophys Biochem , 2002, 2 : 243–270. |
| [6] | Kane JF, Violand BN, Curran DF, et al. Novel in-frame two codon translational hop during synthesis of bovine placental lactogen in a recombinant strain of Escherichia coli. Nucleic Acids Res , 1992, 20 (24) : 6707–6712. DOI:10.1093/nar/20.24.6707 |
| [7] | Calderone TL, Stevens RD, Oas TG. High-level misincorporation of lysine for arginine at AGA codons in a fusion protein expressed in Escherichia coli. J Mol Biol , 1996, 262 (4) : 407–412. DOI:10.1006/jmbi.1996.0524 |
| [8] | Yarian C, Marszalek M, Sochacka E, et al. Modified nucleoside dependent Watson-Crick and wobble codon binding by tRNALysUUU species. Biochemistry , 2000, 39 (44) : 13390–13395. DOI:10.1021/bi001302g |
| [9] | Kane JF. Effects of rare codon clusters on high-level expression of heterologous proteins in Escherichia coli. Curr Opin Biotechnol , 1995, 6 (5) : 494–500. DOI:10.1016/0958-1669(95)80082-4 |
| [10] | Redwan EM. Optimal gene sequence for optimal protein expression in Escherichia coli:principle requirements. Arab J Biotechnol , 2006, 11 (1) : 493–510. |
| [11] | DieciG, 等. tRNA assisted overproduction of eukaryotic ribosom-al proteins. Protein Expr Purif , 2000, 3 : 346–354. |
| [12] | El-Baky NA, Redwan EM. Therapeutic alpha-interferons protein:structure, production, and biosimilar PREP. Biochem Biotechnol , 2015, 45 (2) : 109–127. |
| [13] | Jeong W, Shin HC. Supply of the argU gene product allows high-level expression of recombinant human interferon-alpha-2a in Escherichia coli. Biotechnol Lett , 1998, 20 (1) : 19–22. DOI:10.1023/A:1005366727366 |
| [14] | Valente CA, Prazeres DMF, Cabral JMS, et al. Translation feature of human alpha 2b interferon production in Escherichia coli. Appl Environ Microbiol , 2004, 70 (8) : 5033–5036. DOI:10.1128/AEM.70.8.5033-5036.2004 |
| [15] | Ferrer-Miralles N, Villaverde A. Bacterial cell factories for recombinant protein production;expanding the catalogue. Microb Cell Fact , 2013, 12 (1) : 113. DOI:10.1186/1475-2859-12-113 |
| [16] | Tegel H, Tourle S, Ottosson J, et al. Increased levels of recombinant human proteins with the Escherichia coli strain Rosetta(DE3). Protein Expr Purif , 2010, 69 (2) : 159–167. DOI:10.1016/j.pep.2009.08.017 |
| [17] | Ringquist S, Shinedling S, Barrick D, et al. Translation initiation in Escherichia coli:sequences within the ribosome-binding site. Mol Microbiol , 1992, 6 (9) : 1219–1229. DOI:10.1111/mmi.1992.6.issue-9 |
| [18] | Laursen BS, Sorensen HP, Mortensen KK, et al. Initiation of protein synthesis in bacteria. Mol Biol Rev , 2005, 1 : 101–123. |
| [19] | Stenstrom C, Jin H, Major L, et al. Codon bias at the 3’-side of the initiation codon is correlated with translation initiation efficiency in Escherichia coli. Gene , 2001, 263 (1-2) : 273–284. DOI:10.1016/S0378-1119(00)00550-3 |
| [20] | Sprengart ML, Porter AG. Functional importance of RNA interactions in selection of translation initiation codons. Mol Microbiol , 1997, 24 (1) : 19–28. DOI:10.1046/j.1365-2958.1997.3161684.x |
| [21] | Etchegaray JP, Inouye M. Translational enhancement by an element example of molecular misreading in Alzheimer disease. Trends Neurosci , 1999, 21 : 331–335. |
| [22] | Seo SW, Yang JS, Cho HS, et al. Predictive combinatorial design of mRNA translation initiation regions for systematic optimization of gene expression levels. Sci Rep , 2014, 4 (3) : 2231–2236. |
| [23] | Iost I, Dreyfus M. mRNA can be stabilized by DEAD-box proteins. Nature , 1994, 372 (6502) : 193–196. DOI:10.1038/372193a0 |
| [24] | Iost I, Bizebard T, Dreyus M. Functions of DEAD-box proteins in bacteria:current knowledge and pending questions. Biochim Biophys Acta , 2013, 1829 (8) : 866–877. DOI:10.1016/j.bbagrm.2013.01.012 |
| [25] | Chen R. Bacterial expression systems for recombinant protein production:E. Biotechnol Adv , 2012, 30 (5) : 1102–1107. DOI:10.1016/j.biotechadv.2011.09.013 |
| [26] | Sanchez JC, Padron G, Santana H, et al. Elimination of an HuIFN alpha 2b readthrough species, produced in Escherichia coli, by replacing its natural translation stop signal. J Biotechnol , 1998, 63 (3) : 179–186. DOI:10.1016/S0168-1656(98)00073-X |
| [27] | PooleES, BrownCM, 等. The identity of the base following the stop codon determines the efficiency of in vivo translational termin-ation in Escherichia coli. EMBO J , 1995, 1 : 151–158. |
| [28] | Fisher AC, Haitjema CH, Guarino C, et al. Production of secretory andextracellular N-Linked glycoproteins in Escherichia coli. Appl Environ Microbiol , 2011, 77 (3) : 871–881. DOI:10.1128/AEM.01901-10 |
| [29] | Overton TW. Recombinant protein production in bacterial hosts. Drug Discovery Today , 2014, 19 (5) : 590–601. DOI:10.1016/j.drudis.2013.11.008 |
| [30] | Carrio MM, Villaverde A. Role of molecular chaperones in inclusion body formation. FEBS Lett , 2003, 537 (1-3) : 215–221. DOI:10.1016/S0014-5793(03)00126-1 |
| [31] | Cui SS, Lin XZ, Shen JH. Effect of co-expression of molecular chaperones on heterologous soluble expression of the cold-active lipase Lip-948. Protein Expres Purif , 2011, 2 : 166–172. |
| [32] | RonezF, ArbaultP, 等. Co-expression of the small heat shock protein, Lo18, with b-glucosidase in Escherichia coli improves solubilization and reveals various associations with overproduced heterologous protein, GroEL/ES. Biotechnol Lett , 2012, 5 : 935–939. |
| [33] | Jhamb K, Sahoo DK. Production of soluble recombinant proteins in Escherichia coli:Effects of process conditions and chaperone co-expression on cell growth and production of xylanase. Bioresource Technol , 2012, 123 (4) : 135–143. |
| [34] | Yan X, Hu S, Guan YX, et al. Co-expression of chaperonin GroEL/GroES markedly enhanced soluble and functional expression of recombinant human interferon-gamma in Escherichia coli. Appl Microbiolb Biotechnol , 2012, 93 (3) : 1065–1074. DOI:10.1007/s00253-011-3599-2 |
| [35] | Voulgaridou GP, Mantso T, Chlichlia K, et al. Efficient E. coli expression strategies for production of soluble human crystalline ALDH3A1. PLoS One , 2013, 8 (2) : 65. |
| [36] | FolwarcznaJ, MoravecT, 等. Efficient expression of human papillomavirus 16 E7 oncoprotein fused to C-terminus of tobacco mosaic virus(TMV)coat protein using molecular chaperones in Escherichia coli. Protein Expres Purif , 2012, 1 : 152–157. |
| [37] | Nausch H, et al. Recombinant production of human interleukin 6 in Escherichia coli. PLoS One , 2013, 1 : 570–579. |
| [38] | Ow DS W, Lim DYX, Nissom PM, et al. Co-expression of Skp and FkpA chaperones improves cell viability and alters the global expression of stress response genes during scFvD1. Microb Cell Fact , 2010, 9 (8) : 1–14. |
| [39] | MaengBH, NamDH, KimYH. Coexpression of molecular chapero-nes to enhance functional expression of anti-BNPscFv in the cytoplasm of Escherichia coli for the detection of B-type natriuretic peptide. Word J Microbiol Biotechnol , 2011, 6 : 1391–1398. |
| [40] | Khattar SK, Kundu PK, Gulati P, et al. Optimization and enhanced soluble production of biologically active recombinant human p38 mitogen-activated-protein kinase(MAPK)in Escherichia coli. Protein Peptide Lett , 2007, 14 (8) : 756–760. DOI:10.2174/092986607781483660 |
| [41] | Fahnert B, Lilie H, Neubauer P. Inclusion bodies:formation and utilization. Adv Biochem Eng Biotechnol , 2004, 89 : 93–142. |
| [42] | Jensen EB, Carlsen S. Production of recombinant human growth hormone in Escherichia coli:Expression of different precursors and physiological effects of glucose, acetate and salts. Biotechnol Bioeng , 1990, 36 (1) : 1–11. DOI:10.1002/(ISSN)1097-0290 |
| [43] | Vasina JA, Baneyx F. Expression of aggregation prone recombinant proteins at low temperatures:a comparative study of the Escherichia coli cspA and tac promoters systems. Protein Expr Purif , 1997, 9 (2) : 211–218. DOI:10.1006/prep.1996.0678 |
| [44] | Lebendiker M, Danieli T. Production of prone-to-aggregate proteins. FEBS Lett , 2014, 588 (2) : 236–46. DOI:10.1016/j.febslet.2013.10.044 |
| [45] | Papaneophytou CP, Kontopidis G. Statistical approaches to maximize recombinant protein expression in Escherichia coli:a general review. Protein Expr Purif , 2014, 94 (2) : 22–32. |
| [46] | Feng Y, Xu Q, Yang T, et al. A novel self-cleavage system for production of soluble recombinant protein in Escherichia coli. Protein Expr Purif , 2014, 99 (4) : 64–69. |
| [47] | Jung ST, Kang TH, Kelton W, et al. Bypassing glycosylation:engineering aglycosylated full-length IgG antibodies for human therapy. Curr Opin Biotechnol , 2011, 22 (6) : 858–867. DOI:10.1016/j.copbio.2011.03.002 |
| [48] | YimS, 等. High-level secretory production of human granulocytes-colony stimulating factor by fed-batch culture of recombinant Esch-erichia coli. Bioprocess Biosyst Eng , 2001, 4 : 249–254. |
| [49] | Joly JC, et al. Overexpression of Escherichia coli oxidoreductases increases recombinant insulin-like growth factor-I accumulation. Proc Natl Acad Sci USA , 1998, 6 : 2773–2777. |
| [50] | Reilly DE, Yansura DG. Production of monoclonal antibodies in E. coli[M]//Shire SJ, Gombotz W, Bechtold-Peters K, et al. Current Trends in Monoclonal Antibody Development and Manufacturing. New York:Springer, 2010:295-308. |
| [51] | Mavrangelos C, Thiel M, Adamson PJ, et al. Increased yield and activity of soluble single-chain antibody fragments by combing high-level expression and the Skp periplasmic chaperonin. Protein Expr Purif , 2001, 23 (2) : 289–295. DOI:10.1006/prep.2001.1506 |
| [52] | Lee YJ, Lee DH, Jeong KJ. Enhanced production of human full-length immunoglobulin G1 in the periplasm of Escherichia coli. Appl Microbiol Biotechnol , 2014, 98 (3) : 1237–1246. DOI:10.1007/s00253-013-5390-z |
| [53] | Jenkins N. Modification of therapeutic proteins:challenges and prospects. Cytotechnol , 2007, 53 (1-3) : 121–125. DOI:10.1007/s10616-007-9075-2 |
| [54] | Walsh G, Jefferis R. Post-translational modifications in the contex of therapeutic proteins. Nat Biotechnol , 2006, 24 (10) : 1241–1252. DOI:10.1038/nbt1252 |
| [55] | Wacker M, Linton D, Hitchen PG, et al. N-linked glycosylation in Campylobacter jejuni and its functional transfer into E. Science , 2002, 298 (5599) : 1790–1793. DOI:10.1126/science.298.5599.1790 |
| [56] | Valderrama-Rincon JD, Fisher AC, Merritt JH, et al. An engineered eukaryotic protein glycosylation pathway in Escherichia coli. Nat Chem Biol , 2012, 8 (5) : 434–436. DOI:10.1038/nchembio.921 |
| [57] | Ollis AA, Zhang S. Engineered oligosaccharyltransferases with greatly relaxed acceptor-site specificity. Nat Chem Biol , 2014, 10 (10) : 816–822. DOI:10.1038/nchembio.1609 |
| [58] | Merritt JH, Ollis AA, Fisher AC, et al. Glycans-by-design:engineering bacteria for the biosynthesis of complex glycans and glycoconjugates. Biotechnol Bioeng , 2013, 6 : 1550–1564. |
| [59] | Guccui J, Wren B. Hijacking bacterial glycosylation for the production of glycoconjugates, from vaccines to humanized glycoproteins. J Pharm Pharmacol , 2015, 67 (3) : 338–350. DOI:10.1111/jphp.2015.67.issue-3 |
| [60] | Ihssen J, Kowarik M, et al. Production of glycoprotein vaccines in Escherichia coli. Microb Cell Fact , 2010, 9 (1) : 98–102. DOI:10.1186/1475-2859-9-98 |
| [61] | Wetter M, Kowarik M, Steffen M, et al. Engineering, conjugation, and immunogenicity assessment of Escherichia coli O121 O antigen for its potential use as a typhoid vaccine component. Glycoc-onj J , 2013, 30 (5) : 511–522. DOI:10.1007/s10719-012-9451-9 |