2. 山东农业大学植病系,泰安 271018
2. Department of Plant Pathology,Shandong Agricultural University,Tai’an 271018
植物激素的发现追溯于19世纪关于向光性和趋向性实验。1880年,Charles Darwin通过实验发现植物具有趋光性现象,推断在植物体内存在一种可传递的物质命名为激素(Auxin),并最终被鉴定为吲哚-3-乙酸(indole-3-acetic acid,IAA)[1]。IAA作为最早被发现的植物生长激素,几乎参与植物生长发育的各个阶段,调节植物细胞的生长、根茎叶的分化及果实发育,在植物生长发育和防御中发挥重要作用[2]。有趣的是许多微生物(既包括植物益生菌,也包括植物致病菌)也具有合成IAA的能力,通过干扰植物体内激素的平衡从而影响寄主植物的发育和抗逆性。例如,许多植物根际促生菌(plant growth promoting rhizobacteria,PGPR)和木霉属(Trichoderma)生防真菌因为产生IAA而具有促进植物生长的潜能;而病原细菌根瘤农杆菌(Agrobacterium tumefaciens)产生的IAA则作为毒力因子诱发根癌病。更重要的是,近期研究发现,IAA也作为信号分子调控细菌基因表达及其与寄主植物的有益或有害互作[3]。本文主要从植物相关细菌IAA的生物合成途径,IAA如何调节细菌的生理活动及参与细菌与植物相互作用,以及影响细菌IAA产生的环境及其遗传因子4个方面进行概述,旨在更好地了解该领域的研究现状及最新研究进展,为IAA 跨界信号机制的系统深入研究提供借鉴。
1 细菌中IAA生物合成途径细菌与植物中IAA的生物合成途径存在高度相似性,分为色氨酸依赖型途径和独立于色氨酸型途径两大类。某些细菌中并存多条IAA生物合成途径,也暗示了IAA对于细菌生理的重要性。以反应中间体命名,包括3种主要的合成途径:吲哚-3-乙酰胺IAM(indole-3-acetamid)途径;吲哚-3-丙酮酸IPyA(indole-3-pyruvic acid)途径和吲哚-3-乙腈IAN(indole-3-acetonitrile)途径,都以色氨酸(tryptophan,Trp)作为前体物质[3]。
1.1 色氨酸依赖型途径(trp-dependent pathway) 1.1.1 吲哚-3-乙酰胺(indole-3-acetamide,IAM)途径IAM主要存在于植物致病菌中,少数植物促生菌也存在该途径。如橄榄节疤病菌Pseudomonas savastanoi pv. savastanoi,冠瘿病菌A. tumefaciens和鹰嘴豆枯萎镰刀菌Fusarium delphinoides等。另外,在植物根际促生菌绿针假单胞菌P. chlororaphis O6中也鉴定存在IAM 途径。IAM途径为色氨酸依赖型途径,主要分为两步:首先Trp在色氨酸-2-单加氧酶(tryptophan-2-monooxygenase,IaaM)的作用下合成吲哚-3-乙酰胺(indole-3-acetamide,IAM),IAM在iaaH基因编码的IAM水解酶(IaaH)作用下合成IAA[4-7]。
1.1.2 吲哚-3-丙酮酸(indole-3-pyruvic acid,IPyA)途径IPyA是植物体内IAA合成的主要途径,研究表明该途径也广泛存在于植物益生菌中,影响细菌适应性定植和植物生长促进。IPyA途径以Trp为前体物质,在氨基转移酶(Aminotransferase)作用下合成吲哚-3-丙酮酸(indole-3-pyruvate,IPyA),IPyA进一步通过吲哚-3-丙酮酸脱羧酶(indole-3-pyruvate decarboxylase,IPDC)脱羧基形成吲哚-3-乙醛(indole-3-acetaldehyde,IAAld),最后IAAld被氧化成IAA。由ipdC基因编码的吲哚-3-丙酮酸脱羧酶IPDC作为IPyA途径中的关键酶,在许多植物细菌中已经被鉴定。例如,植物促生菌分散泛生氏菌(Pantoea dispersa),解淀粉芽孢杆菌(Bacillus amyloliquefaciens)和巴西固氮菌(Azospirillum brasil-ense)等[8-10]。
1.1.3 吲哚-3-乙腈(indole-3-acetonitrile pathway,IAN)途径植物和细菌都存在IAN途径,也属于色氨酸依赖型途径,主要分为3步:首先,Trp在一种类似氧化还原酶的作用下转换为吲哚-3-乙醛肟(indole-3-acetaldoxime,IAOx),IAOx进一步通过吲哚乙醛肟脱水酶(indoleacetaldoxime dehydratase)合成吲哚-3-乙腈(indole-3-acetonitrile,IAN),最后乙腈水解酶将IAN水解形成IAA[1]。如固氮螺菌Azospirillum amazonense基因组中被鉴定存在IAN途径[11]。
1.2 色氨酸独立型途径(trp-independent pathway)通过同位素标记的方法检测发现巴西固氮菌A. brasilense中90%的IAA通过独立于色氨酸的途径合成,只有0.1% 的IAA通过IAM途径合成。尽管目前参与该途径的酶尚未得到鉴定,该研究证明了植物细菌中也存在色氨酸独立的IAA合成途径[12]。
2 IAA参与调节细菌的生理活动IAA不仅作为细菌次生代谢物调节植物生理,近期研究发现IAA还作为信号分子调控细菌基因表达,参与调节细菌多种生物学过程和行为,包括抗逆性、运动性和附生适应性等。
2.1 IAA影响细菌胁迫耐力研究发现,在根瘤固氮菌Sinorhizobium meliloti中,经酸、高渗透压、紫外线和热击等胁迫处理后,添加外源IAA的处理或遗传操纵获得的IAA过表达菌株,其细菌存活率明显高于对照组或野生菌。这些结果表明IAA能够增强细菌的胁迫耐力,使细菌在逆境中获得更好的生存能力和竞争力。进一步分析表明经外源IAA处理的细菌,其胞内海藻糖含量上升。海藻糖作为细菌的碳源,也参与调节细菌渗透压抵抗严寒和干燥。另外,脂多糖、胞外多糖及生物膜形成在IAA过表达菌株中都有所提高,这些都有助于细菌抵御外界不良环境,增强适应性和生存率[13]。值得注意的是,IAA对于细菌胁迫耐力的调节(增强或削弱),随细菌的种类以及具体的胁迫环境不同而有所变化。例如,添加外源IAA能够增强Bacillus subtilis和Escherichia coli对紫外照射的耐受性,但并未增强它们对温度和链霉素的耐受能力;相反,IAA处理后的E. coli对温度和链霉素更加敏感[14]。
2.2 IAA作为信号分子调节细菌基因表达转录组学分析表明,添加外源IAA处理引起固氮菌Bradyrhizobium japonicum大量基因转录物水平的改变。尤其是一些与细菌适应胁迫环境相关的蛋白,包括热休克蛋白、冷休克蛋白、胞外多糖EPS合成和分子伴侣等,其编码基因的转录水平明显上调,揭示了IAA调节细菌胁迫抗性的分子基础。相反,一些参与氨基酸合成、细胞过程、能量代谢和转运等生理活动的基因受到抑制[15]。在A. brasilense sp. 菌株245中,比较分析野生菌和ipdC突变体的基因表达谱发现:ipdC突变体中39个编码核糖体蛋白的基因表达下调,而硝酸盐还原系统相关基因的表达则增加1.4-1.9倍,同时也增加了VI型分泌系统T6SS相关基因的表达。另外,IAA影响细菌的呼吸作用,ipdC突变体中NADH脱氢酶复合体的表达明显下降,添加外源IAA后表达量上升[16]。Spaepen等[17]也报道在Rhizobium etli中IAA可诱导细菌基因表达,包括信号转导、鞭毛和细菌附着相关基因,进而影响细菌运动性和定植能力等。
3 IAA参与细菌与植物的相互作用植物相关细菌分泌的IAA可以与其他细菌毒力因子协同诱发植物病害,或影响植物生长发育。IAA对植物生长的促进或抑制是浓度依赖的,例如 PGPR 菌株P. fluorescens Psd 中IAA产量约为 6.32 μg/OD600,接种植物后表现为促进生长发育。然而,通过将携带iaaM-iaaH基因的质粒导入Psd产生过表达菌株时,IAA产量增至80.32 μg /OD600 则表现为抑制高粱根系的生长[18]。
3.1 IAA与植物-益生菌互作IAA影响固氮菌A. brasilense对植物的促生作用,其IAA合成主要依赖于IPyA途径。研究表明ipdC突变体中IAA产量降低50%,对于植物的促生作用也明显降低[19]。转录组学分析发现细菌感应高水平IAA 刺激后,许多转录因子的表达受到影响,同时VI型分泌系统(T6SS)相关基因的表达上调。大量研究已表明,T6SS在细菌与植物及其与微生物群落的相互作用中扮演重要作用,暗示了细菌的生理活动受植物根际微环境中IAA浓度的影响[16]。
共生根瘤菌Sinorhizobium meliloti RD64既能够固定空气中的氮为宿主植物利用,也合成IAA促进植物生长。IAA水平与固氮能力正相关,并调节TCA循环关键酶的表达。IAA上调聚-β-羟丁酸(poly-β-hydroxybutyrate,PHB)合成酶的活性,导致PHB产量明显增加,同时细菌的存活率也明显提高。当苜蓿接种IAA过表达菌株后茎的干重显著增加[13]。
分离于黄瓜根系的生防菌Bacillus amyloliq-uef-aciens能够抑制镰刀菌枯萎病,刺激植物生长。通过比较分析IAA缺失突变体和野生菌株发现,IAA与其他植物生长调节因子,如挥发性有机化合物(volatile organic compounds,VoCs)和胞外酶等协同作用影响对植物的促生能力[9, 20]。
3.2 IAA与植物-病原细菌互作根瘤农杆菌A. tumefaciens引起多种植物的根癌病,其致病过程首先通过感应植物根际伤口处分泌的大量酚类化合物吸引农杆菌移向这些细胞,激活相关基因的表达,促进Ti质粒整合进入植物细胞。Ti质粒携带的毒力基因表达使植物体IAA和细胞分裂素(Cytokinin)的合成增加,植物局部组织过度增生,导致瘿瘤的形成[5]。
Pantoea agglomerans pv. gypsophilae引起植物瘿瘤病,具有IAM和IPyA两条IAA合成途径,分别参与细菌不同的生理活动。IAM途径主要影响细菌致病力,而IPyA途径与附生能力相关。其中,IAM途径受抑制时可导致瘿瘤减小40%-50%;而抑制IPyA途径对其致病力的影响甚微,但明显影响其附生能力[21]。
Pseudomonas syringage pv. savastanoi产生高浓度的IAA,引起植物橄榄结疤病。该细菌寄生于植物体内,直接将IAA分泌到植物细胞内干扰激素平衡,从而诱导肿瘤的产生[4]。
3.3 IAA与植物防御植物存在两条先天的免疫系统应对细菌感染。第一条基本防御系统识别和应答微生物包括病原菌释放的共有的信号分子,如感应细菌鞭毛。微生物/病原菌相关的分子模式(pathogen/microbial-associa-ted molecular patterns,PAMPs/MAMPs),与病原菌识别受体模式(the plant pathogen recognition receptors,PRRs)互作在激活植物基本防御过程中发挥重要作用。第二条过敏反应(hypersensitive response,HR)应答系统感应病原菌毒力因子,由植物抗病R基因介导,直接或间接影响寄主植物防御[22]。IAA作为植物最重要的激素也参与调节植物防御。Navarro等[23]研究发现植物IAA 信号和抵御病原菌的能力之间存在联系。例如,丁香假单胞菌Pseudomonas syringae以PAMPs模式下调拟南芥IAA 信号,而抑制IAA信号可减缓细菌生长,从而增强了植物对病原细菌的抗病力;相反,添加外源IAA可使植物对该病原细菌的敏感性增强。另外,拟南芥经外源IAA处理后植物病程相关蛋白的表达水平也下调,表明植物体内IAA水平与其抗细菌侵染的能力负相关[24]。已有报道IAA分泌也影响细菌在植物体的附生和适应能力。在P. fluorescens HP72中除了植物促生作用,IAA产量降低的突变体附着植物根系的能力也明显下降,表明植物根际分泌物中的IAA也能够影响细菌的寄生,从而达到防御的目的[25]。
4 细菌IAA生物合成的调控细菌合成的IAA具有多功能性:首先影响寄主植物生理活动,又可以调节细菌自身基因表达、参与细菌的生理和代谢,还作为跨界信号参与植物与微生物的相互作用等,因此细菌合成IAA的水平受到高度精细的调控。影响细菌IAA合成的因素包括pH值和温度、色氨酸浓度、营养等环境因子,更重要的是多层次的遗传因子整合形成复杂的调控网络通过感应和应答环境刺激动态调节IAA产生[3]。
4.1 环境因素细菌IAA的产生受生长环境的影响。首先温度和pH值影响IAA的合成。例如,A. brasilense SM 在30℃时生长最佳,然而温度的波动和增加培养时间都能刺激IAA的产生。另外,pH值也影响IAA合成,但生长最适pH值不是IAA产生的最佳条件。温度和pH值都是通过影响IAA合成相关酶的活性调节IAA的合成[26]。在植物根际促生菌Pseudomonas sp. UW4中IAA主要通过IAM和 IAOx/IAN两条途径合成,其中两种关键性酶腈水解酶(nitrilase)和腈水合酶(nitrile hydratase)的最适温度和最适pH值有所不同。腈水解酶在50℃、pH 6时活性最高;然而腈水合酶在4℃,pH7.5时活性最高,因此在不同温度和pH条件下IAA的产量也有所不同[27]。
除了温度和pH 的影响,营养状况也影响IAA合成。例如,A.brasilense在因饥饿生长受限的条件下,ipdC基因的表达量上升从而导致IAA的产量增加[28]。色氨酸作为细菌IAA合成的主要前体物质,也参与调控IAA的合成,色氨酸可明显提高IAA的产量。例如,在培养基中添加色氨酸可使A. brasilense Sp7 IAA产量提升6-8倍。但不同细菌菌株IAA产量峰值所需的色氨酸浓度不同,同时Trp对IAA合成的影响,也是通过对ipdC基因表达的调控而实现的[29]。由于不同微生物对于碳氮源的喜好不同,也间接影响IAA的合成,研究表明有机碳氮源比无机碳氮源更能提高IAA产量[26]。Dimkpa等[7, 30]研究发现CuO和ZnO纳米粒子也影响P. chlororaphis O6 IAA的产量,但 Fe、A、Cd、Cu和Ni等金属离子能够在不影响生长的情况下降低Streptomyces tendae和S. acidiscabies E13 合成IAA的能力。
另外,植物某些分泌物也能够影响细菌IAA的合成。例如,植物提取物中的黄酮类物质能够刺激植物共生菌Rhizobium sp. NGR23 合成IAA[31]。
4.2 遗传调控 IAA生物合成 4.2.1 自我正调控反馈机制许多细菌IAA的合成可以自我诱导(Autoinduction),形成正反馈机制。例如,在A. brasilense Sp245中控制IAA合成的关键基因为ipdC基因,培养基中添加外源IAA明显刺激ipdC基因的表达,从而提高IAA的产量。由此可见,IAA合成受自身终产物的正调控[32]。研究发现在Azospirillum ipdC基因的上游存在类似于植物激素应答原件AuxRe的区域,能够响应IAA的刺激诱导ipdC基因表达[28]。
4.2.2 遗传调控因子目前已经鉴定了许多遗传调控因子参与调节IAA的生物合成。RpoS作为稳定期σ因子已知调控细菌胁迫耐力和生物膜形成等,从而影响细菌适应性。在Pseudomonas putida GR12-2中ipdC的表达也受RpoS的调控,rpoS突变株IAA产量较野生型发生很大改变[33]。P. chlororaphis O6中也有类似报道,rpoS突变体较野生型IAA产量明显增加,表明RpoS负调控IAA的合成。另外,在该菌株中GacS/GacA双组分系统也负调控IAA的合成[34]。细菌中参与调控氨基酸代谢的TyrR型转录因子也影响IAA产生。例如,Enterobacter cloacae UW5通过IPyA途径合成IAA,培养基中添加芳香族氨基酸(色氨酸、酪氨酸和苯丙氨酸),可促进TyrR上调ipdC基因转录和IAA合成。相反,添加支链氨基酸如缬氨酸和亮氨酸等则使TyrR下调IAA产生[35]。
4.2.3 遗传调控网络研究表明在细菌中IAA可与其他调控因子整合形成相互作用的复杂调控网络精细调节植物与细菌互作。植物瘿瘤病菌P. agglomerans pv. gypsophilae中存在2条IAA合成途径IAM和IPyA。群体感应quorum sensing(QS)系统pagI和pagR与hrp/hrc基因簇调控Ⅲ 型分泌系统(T3SS)相关基因表达影响IAA和细胞分裂素的合成。QS调控子pagI和pagR突变菌株中hrpXY、hrpS和hrpL的转录水平明显降低影响IAA产量。而IAA水平也影响QS及hrp系统相关基因的表达:IAM途径中iaaH突变,使pagI/R和hrpL、hrpS的表达水平也相应降低;而IPyA途径中ipdC突变则使上述基因表达水平升高[21]。以上研究结果表明IAA、QS和T3SS之间存在相互作用;也暗示了经IAM和IPyA途径产生的IAA信号分子在调控QS和Hrp相关基因表达中发挥相反的作用。
植物致病菌Erwinia chrysanthemi 3937中iaaM基因负责IAA合成,转录组学分析iaaM突变菌株发现,与野生菌相比,大量基因的表达水平发生改变。例如,Ⅲ 型分泌系统(T3SS)相关基因和gacA表达水平降低,但翻译抑制子RNA结合蛋白RsmA表达上升。这些结果表明IAA可以整合进入Gac/Rsm信号转导通路,从而获得在转录和翻译等不同层面调控细菌基因表达的能力[36]。
某些IAA产生细菌也能够利用NO作为信号分子参与植物与微生物的相互作用。研究发现IAA和NO在根际存在紧密的联系,相互协调组成复杂的调控网络[37]。例如,在A. brasilense SM中nosR和norBC参与调节NO的合成和代谢,同时也参与调节IAA的合成。在NO合成代谢缺失菌株中IAA的产量明显降低,而在产IAA突变菌株中NO合成代谢相关基因的表达也受到影响,这表明IAA与NO之间存在交流或具有共同的信号机制[38]。
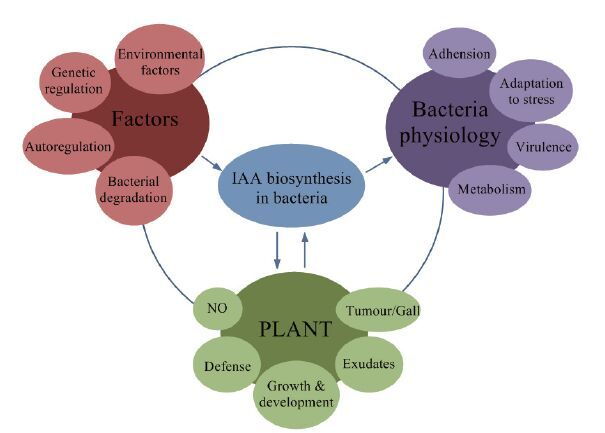
4.3 细菌IAA的降解一些细菌具有双重能力,既能合成IAA,又能分解代谢IAA,维持IAA水平的动态平衡。例如,在Pseudomonas putida 菌株 1290中鉴定细菌中第一个负责降解IAA的基因簇iacABCGEFG,其表达受IAA强烈诱导,同时可以识别IAA和进行趋化性运动。赋予了细菌一定的选择优势,可以利用环境中IAA作为唯一碳氮源,为自身生理活动提供能量,提升了细菌的竞争力和附生植物的能力。细菌通过该机制使IAA在植物、细菌和环境间可以循环利用[39, 40]。Burkholderia phytofirman也能够降解IAA,降解IAA作为细菌附生的一种策略,与IAA降解相关的iacC突变后,对植物的促生能力明显减弱[41]。IAA作为跨界信号介导的细菌与植物及其环境互作的示意图,见图 1。
5 结语综上所述,IAA不仅作为细菌产生的次生代谢物,而且在调节植物生理的同时也作为跨界信号参与调节自身基因表达,生理活动和行为,以及与寄主植物的有益或有害互作。例如,IAA能够影响细菌胁迫耐力,增强细菌对复杂环境的适应能力;还可以作为信号分子调节细菌信号转导、鞭毛形成、附着能力等。随着整合和系统生物学的发展,结合使用新一代高通量测序技术,目前对植物体中IAA的生物合成途径、IAA影响植物生理活动的信号通路和作用机制的认识已经取得了重大进展。相比而言,关于IAA介导的调节植物与微生物互作的跨界信号机制的相关研究才刚刚起步。目前研究主要集中在IAA调控细菌基因表达及生理活动,以及IAA生物合成的调控。IAA、细菌、植物三者构成相互作用、相互协调的有机整体,构成一幅动态的复杂的调控网络。未来更多的目光应该聚集在系统探索IAA作为跨界信号如何整合进入植物与细菌互作的调控网络,精细调节植物与微生物互作。迅速发展的转录组学、蛋白质组学等功能基因组学和代谢组学方法,整合生物信息学和高通量测序技术等将有助于人们通过不断研究探索,将这幅网络描绘得更加清晰和立体。
人口爆炸,土壤干旱、盐渍化和沙漠化,以及大量使用化学农药和化学肥料严重威胁着食品供给和安全性,是目前人类共同面对的全球性挑战。更好地了解该领域的研究动态,阐明植物相关细菌IAA的合成及调控机制,以及IAA参与调节植物与细菌互作的分子基础等,将有助于提高植物抗逆性和产量,为研发环境友好的多功能生物农药/肥料开拓新思路。如通过遗传操纵植物生防菌可以优化其防病促生能力;对于植物病原菌则可以降低其致病力。这对于农业可持续发展、保护环境和生态都具有重要意义。
| [1] | Spaepen S, Vanderleyden J, Remans R. Indole-3-acetic acid in microbial and microorganism-plant signaling. FEMS Microbiol Rev , 2007, 31 (4) : 425–448. DOI:10.1111/j.1574-6976.2007.00072.x |
| [2] | Halliday KJ, Martinez-Garcia JF, Josse EM. Integration of light and auxin signaling. Cold Spring Harbor Perspectives in Biology , 2009, 1 (6) : a001586. |
| [3] | Duca D, Lorv J, Patten CL, et al. Indole-3-acetic acid in plant-microbe interactions. Antonie van Leeuwenhoek , 2014, 106 (1) : 85–125. DOI:10.1007/s10482-013-0095-y |
| [4] | Aragon IM, Perez-Martinez I, Moreno-Perez A, et al. New insights into the role of indole-3-acetic acid in the virulence of Pseudomonas savastanoi pv. FEMS Microbiology Letters , 2014, 356 (2) : 184–192. DOI:10.1111/1574-6968.12413 |
| [5] | Subramoni S, Nathoo N, Klimov E, et al. Agrobacterium tumefaciens responses to plant-derived signaling molecules. Frontiers in Plant Science , 2014, 5 : 322. |
| [6] | Kulkarni GB, Sanjeevkumar S, Kirankumar B, et al. Indole-3-acetic acid biosynthesis in Fusarium delphinoides strain GPK, a causal agent of wilt in chickpea. Applied Biochemistry and Biotechnology , 2013, 169 (4) : 1292–1305. DOI:10.1007/s12010-012-0037-6 |
| [7] | Dimkpa CO, Zeng J, Mclean JE, et al. Production of indole-3-acetic acid via the indole-3-acetamide pathway in the plant-beneficial bacterium Pseudomonas chlororaphis O6 is inhibited by ZnO nanoparticles but enhanced by CuO nanoparticles. Applied and Environmental Microbiology , 2012, 78 (5) : 1404–1410. DOI:10.1128/AEM.07424-11 |
| [8] | Kulkarni GB, Nayak AS, Sajjan SS, et al. Indole-3-acetic acid biosynthetic pathway and aromatic amino acid aminotransferase activities in Pantoea dispersa strain GPK. Letters in Applied Microbiology , 2013, 56 (5) : 340–347. DOI:10.1111/lam.12053 |
| [9] | Shao J, Li S, Zhang N, et al. Analysis and cloning of the synthetic pathway of the phytohormone indole-3-acetic acid in the plant-beneficial Bacillus amyloliquefaciens SQR9. Microbial Cell Factories , 2015, 14 : 130. DOI:10.1186/s12934-015-0323-4 |
| [10] | Jijon-Moreno S, Marcos-Jimenez C, Pedraza RO, et al. The ipdC, hisC1 and hisC2 genes involved in indole-3-acetic production used as alternative phylogenetic markers in Azospirillum brasilense. Antonie van Leeuwenhoek , 2015, 107 (6) : 1501–1517. DOI:10.1007/s10482-015-0444-0 |
| [11] | Cecagno R, Fritsch TE, Schrank IS. The plant growth-promoting bacteria Azospirillum amazonense:genomic versatility and phytohormone pathway. Biomed Research International , 2015, 2015 : 898592. |
| [12] | Prinsen ECA, Michiels K, Vanderleyden J, et al. Azospirillum brasilense indole-3-acetic acid biosynthesis:evidence for a non-tryptophan dependent pathway. Mol Plant Microbe Interact , 1993, 6 : 609–615. DOI:10.1094/MPMI-6-609 |
| [13] | Imperlini E, Bianco C, Lonardo E, et al. Effects of indole-3-acetic acid on Sinorhizobium meliloti survival and on symbiotic nitrogen fixation and stem dry weight production. Applied Microbiology and Biotechnology , 2009, 83 (4) : 727–738. DOI:10.1007/s00253-009-1974-z |
| [14] | Repar J, Sucurovic S, Zahradka K, et al. Stress resistance of Escherichia coli and Bacillus subtilis is modulated by auxins. Canadian Journal of Microbiology , 2013, 59 (11) : 766–770. DOI:10.1139/cjm-2013-0266 |
| [15] | Donati AJ, Lee HI, Leveau JH, et al. Effects of indole-3-acetic acid on the transcriptional activities and stress tolerance of Bradyrhizobium japonicum. PLoS One , 2013, 8 (10) : e76559. DOI:10.1371/journal.pone.0076559 |
| [16] | Van Puyvelde S, Cloots L, Engelen K, et al. Transcriptome analysis of the rhizosphere bacterium Azospirillum brasilense reveals an extensive auxin response. Microbial Ecology , 2011, 61 (4) : 723–728. DOI:10.1007/s00248-011-9819-6 |
| [17] | Spaepen S, Das F, Luyten E, et al. Indole-3-acetic acid-regulated genes in Rhizobium etli CNPAF512. FEMS Microbiology Letters , 2009, 291 (2) : 195–200. DOI:10.1111/fml.2009.291.issue-2 |
| [18] | Kochar M, Upadhyay A, Srivastava S. Indole-3-acetic acid biosynthesis in the biocontrol strain Pseudomonas fluorescens Psd and plant growth regulation by hormone overexpression. Research in Microbiology , 2011, 162 (4) : 426–435. DOI:10.1016/j.resmic.2011.03.006 |
| [19] | Malhotra M, Srivastava S. An ipdC gene knock-out of Azospirillum brasilense strain SM and its implications on indole-3-acetic acid biosynthesis and plant growth promotion. Antonie van Leeuwenhoek , 2008, 93 (4) : 425–433. DOI:10.1007/s10482-007-9207-x |
| [20] | Shao J, Xu Z, Zhang N, et al. Contribution of indole-3-acetic acid in the plant growth promotion by the rhizospheric strain Bacillus amyloliquefaciens SQR9. Biology and Fertility of Soils , 2014, 51 (3) : 321–330. |
| [21] | Chalupowicz L, Barash I, Panijel M, et al. Regulatory interactions between quorum-sensing, auxin, cytokinin, and the Hrp regulon in relation to gall formation and epiphytic fitness of Pantoea agglomerans pv. Molecular Plant Microbe Interactions , 2009, 22 (7) : 849–856. DOI:10.1094/MPMI-22-7-0849 |
| [22] | Abramovitch RB, Anderson JC, Martin GB. Bacterial elicitation and evasion of plant innate immunity. Nature Reviews Molecular Cell Biology , 2006, 7 (8) : 601–611. DOI:10.1038/nrm1984 |
| [23] | Navarro L, Dunoyer P, Jay F, et al. A plant miRNA contributes to antibacterial resistance by repressing auxin signaling. Science , 2006, 312 (5772) : 436–439. DOI:10.1126/science.1126088 |
| [24] | Redman JC, Haas BJ, Tanimoto G, et al. Development and evaluation of an Arabidopsis whole genome Affymetrix probe array. The Plant Journal , 2004, 38 (3) : 545–561. DOI:10.1111/tpj.2004.38.issue-3 |
| [25] | Suzuki S, He Y, Oyaizu H. Indole-3-acetic acid production in Pseudomonas fluorescens HP72 and its association with suppression of creeping bentgrass brown patch. Current Microbiology , 2003, 47 (2) : 138–143. DOI:10.1007/s00284-002-3968-2 |
| [26] | Malhotra M, Srivastava S. Stress-responsive indole-3-acetic acid biosynthesis by Azospirillum brasilense SM and its ability to modulate plant growth. European Journal of Soil Biology , 2009, 45 (1) : 73–80. DOI:10.1016/j.ejsobi.2008.05.006 |
| [27] | Duca D, Rose DR, Glick BR. Characterization of a nitrilase and a nitrile hydratase from Pseudomonas sp. Applied and Environmental Microbiology , 2014, 80 (15) : 4640–4649. DOI:10.1128/AEM.00649-14 |
| [28] | Vande Broek A, Gysegom P, Ona O, et al. Transcriptional analysis of the Azospirillum brasilense indole-3-pyruvate decarboxylase gene and identification of a cis-acting sequence involved in auxin responsive expression. Molecular Plant Microbe Interactions , 2005, 18 (4) : 311–323. DOI:10.1094/MPMI-18-0311 |
| [29] | Zimmer W, Wesche M, Timmermans L. Identification and isolation of the indole-3-pyruvate decarboxylase gene from Azospirillum brasilense Sp7:sequencing and functional analysis of the gene locus. Current Microbiology , 1998, 36 (6) : 327–331. DOI:10.1007/s002849900317 |
| [30] | Dimkpa CO, Svatos A, Dabrowska P, et al. Involvement of siderophores in the reduction of metal-induced inhibition of auxin synthesis in Streptomyces spp. Chemosphere , 2008, 74 (1) : 19–25. |
| [31] | Theunis M, Kobayashi H, Broughton WJ, et al. Flavonoids, NodD1, NodD2, and nod-box NB15 modulate expression of the y4wEFG locus that is required for indole-3-acetic acid synthesis in Rhizobium sp. Molecular Plant Microbe Interactions , 2004, 17 (10) : 1153–1161. DOI:10.1094/MPMI.2004.17.10.1153 |
| [32] | Vande Broek A, Lambrecht M, Eggermont K, et al. Auxins upregulate expression of the indole-3-pyruvate decarboxylase gene in Azospirillum brasilense. Journal of Bacteriology , 1999, 181 (4) : 1338–1342. |
| [33] | Patten CL, Glick BR. Regulation of indoleacetic acid production in Pseudomonas putida GR12-2 by tryptophan and the stationary-phase sigma factor RpoS. Canadian Journal of Microbiology , 2002, 48 (7) : 635–642. DOI:10.1139/w02-053 |
| [34] | Kang BR, Yang KY, Cho BH, et al. Production of indole-3-acetic acid in the plant-beneficial strain Pseudomonas chlororaphis O6 is negatively regulated by the global sensor kinase GacS. Current Microbiology , 2006, 52 (6) : 473–476. DOI:10.1007/s00284-005-0427-x |
| [35] | Parsons CV, Harris DM, Patten CL. Regulation of indole-3-acetic acid biosynthesis by branched-chain amino acids in Enterobacter cloacae UW5. FEMS Microbiology Letters , 2015, 362 (18) : fnv153. DOI:10.1093/femsle/fnv153 |
| [36] | Yang S, Zhang Q, Guo J, et al. Global effect of indole-3-acetic acid biosynthesis on multiple virulence factors of Erwinia chrysanthemi 3937. Applied and Environmental Microbiology , 2007, 73 (4) : 1079–1088. DOI:10.1128/AEM.01770-06 |
| [37] | Koul V, Adholeya A, Kochar M. Sphere of influence of indole acetic acid and nitric oxide in bacteria. Journal of Basic Microbiology , 2015, 55 (5) : 543–553. DOI:10.1002/jobm.v55.5 |
| [38] | Koul V, Tripathi C, Adholeya A, et al. Nitric oxide metabolism and indole acetic acid biosynthesis cross-talk in Azospirillum brasilense SM. Research in Microbiology , 2015, 166 (3) : 174–185. DOI:10.1016/j.resmic.2015.02.003 |
| [39] | Leveau JH, Lindow SE. Utilization of the plant hormone indole-3-acetic acid for growth by Pseudomonas putida strain 1290. Applied and Environmental Microbiology , 2005, 71 (5) : 2365–2371. DOI:10.1128/AEM.71.5.2365-2371.2005 |
| [40] | Scott JC, Greenhut IV, Leveau JH. Functional characterization of the bacterial iac genes for degradation of the plant hormone indole-3-acetic acid. Journal of Chemical Ecology , 2013, 39 (7) : 942–951. DOI:10.1007/s10886-013-0324-x |
| [41] | Zuniga A, Poupin MJ, Donoso R, et al. Quorum sensing and indole-3-acetic acid degradation play a role in colonization and plant growth promotion of Arabidopsis thaliana by Burkholderia phytofirmans PsJN. Molecular Plant Microbe Interactions , 2013, 26 (5) : 546–553. DOI:10.1094/MPMI-10-12-0241-R |
| [42] | Mendes R, Garbeva P, Raaijmakers JM. The rhizosphere microbiome:significance of plant beneficial, plant pathogenic, and human pathogenic microorganisms. FEMS Microbiol Rev , 2013, 37 (5) : 634–663. DOI:10.1111/1574-6976.12028 |