哺乳动物染色体构象在受精过程中发生剧烈变化。高度凝缩的精子染色质在卵母细胞中解凝缩,与卵母细胞染色质融合,形成合子基因组,并激活其转录,最终获得发育的全能性[1, 2]。在植入前胚胎的发育过程中,基因组经整体范围内DNA去甲基化,父本基因组迅速主动的去甲基化,母本基因组则被动去甲基化,随后,在母型合子转变期重新被甲基化[3]。父本基因组的迅速去甲基化过程涉及到DNA的主动去甲基化机制[4]。虽然已报道了很多哺乳动物中的DNA主动去甲基化现象,但是还没有明确是什么物质具有DNA去甲基化的作用[5-7]。并且关于DNA主动去甲基化的机制也并不清楚[7-18]。
AID是一种胞苷脱氨酶,它能介导5mC脱氨基成为T,从而发生碱基对的错配,这样产生的错配可以被碱基切除修复(BER)机制所识别[19]。早期的研究主要集中于其在B淋巴细胞中产生抗体多样性的作用[19-22]。最近,在斑马鱼中发现同时过表达AID和MBD4可以降低其基因组DNA甲基化程度[23]。小鼠中的研究表明AID基因主要表达于多能性的组织[24],其蛋白与成纤维细胞中沉默的甲基化的OCT4和NANOG启动子结合,而不与去甲基化的启动子结合[25]。并且AID基因缺陷小鼠的原始生殖细胞(PGC)的基因组整体范围内的DNA甲基化程度比正常小鼠要高2-3倍[26]。这些研究都证明AID蛋白在高等哺乳动物中具有DNA去甲基化的作用,并成为了新的研究热点。
牛AID基因位于5号染色体,全长4 329 bp(ENSBTAGswjstb-32-7-242018849),有4个外显子,mRNA全长597 bp(ENSBTATswjstb-32-7-242025096),编码198个氨基酸的蛋白。目前关于AID基因的研究都局限于斑马鱼[23]、小鼠[24-26]等模式动物中,而在牛上的研究只是检测了其在GV期和MII期卵母细胞的表达而没有对其功能作进一步的讨论[27]。并且在研究的时间段上,目前研究都局限于原始生殖细胞发生和受精后较短的时间,还未见哺乳动物早期胚胎发育过程中AID蛋白作用的研究报道。本研究首先通过分析AID基因在牛组织中的表达和其启动子区的DNA甲基化状态确定AID基因的T-DMR,然后利用体外受精技术分析在不同发育阶段(卵母细胞和胚胎)AID基因的表达变化及其调节方式,以期揭示胚胎细胞重编程分子机制。
1 材料与方法 1.1 材料 1.1.1 实验材料取成年牛的新鲜心脏、肾脏、肝脏、睾丸组织,切成1 cm3的小块,在灭菌的生理盐水中冲洗三遍,投入液氮中,长期保存在-80℃冰箱中。
1.1.2 主要试剂基因组DNA提取试剂盒、T载体购自Promega;RNAiso Reagent、AMV反转录酶、dNTP、RNase Inhibitor、Random Primer(9 mer)、无酶水(DEPC H2O)和凝胶回收试剂盒购自大连宝生物有限公司;除特别提到的化学试剂,其余均购自Sigma公司。
1.2 方法 1.2.1 成年牛组织中AID基因的表达分析根据Enseml网站中公布的牛AID mRNA序列(ENSBTAT-swjstb-32-7-242025096),设计Real-time PCR引物。正向引物:5′-CGGCTGGAGGAGCAAAAGACCGAAAG-3′;反向引物:5′-GAGCCACGTCCCTGTCGTCGTCTTC-3′,产物长度192 bp。阳性对照GAPDH使用Leutenegger等[28]设计的引物。按照宝生物RNAiso Reagent试剂的说明提取牛心脏、肾脏、肝脏和睾丸组织的总RNA并反转录为cDNA,使用ABI 7300 Real-time PCR仪进行Real-time PCR,反应条件如下:95℃ 30 s,98℃ 5 s,60℃ 31 s,共检测40个循环。Real-time PCR结果通过2-ΔΔCT方法分析得出。AID基因的表达量使用GAPDH的表达量进行归一化处理。
1.2.2 AID基因的DNA序列甲基化分析使用Promega DNA提取试剂盒提取成年牛心脏、肾脏、肝脏和睾丸组织的基因组DNA。分别取5 μg DNA,经Xba I酶切11 h后提纯。加入10 mol/L的NaOH至终浓度为0.3 mol/L,37℃变性15 min。加入终浓度为10 mmol/L氢醌和2.5 mol/L的NaHSO3(pH5.0),55℃避光20 h。加入10 mol/L NaOH至终浓度为0.3 mol/L,37℃脱硫15 min。使用3 mol/L醋酸铵沉淀DNA,20 μL TE(pH8.0)回溶。按以下反应条件进行3次PCR:94℃ 4 min;94℃ 1 min,58℃ 30 s,72℃ 1 min;72℃ 10 min,共30个循环。扩增产物与T载体连接,转化挑选克隆。每组实验至少挑选10个克隆送大连宝生物公司测序。
1.2.3 卵母细胞、体外受精胚胎的获得牛卵巢取自于当地屠宰场,用18 # 注射器抽取卵巢表面的3-8 mm的卵泡。挑选颗粒细胞包裹较完整的卵母细胞,用M199+10% FBS(HyClone)+0.02 IU/mL FSH+1 IU/mL LH+0.01 μg/mL E2和0.06 mg/mL penicillin & 0.05 mg/mL streptomycin在5% CO2,100%湿度,38.5℃培养18-24 h后成熟。37℃水浴中解冻公牛冷冻精液,受精A液(BO液+10 mmol/L Caffeine)离心洗涤(3 000 r/min)两次。离心后,静置2 min,待精子上浮后小心吸取,加入等体积的受精B液(BO液+3 mg/mL BSA+8 µL/mL肝素)制成精子悬浮液。将精子悬浮液做成100 µL受精小滴,每滴放入30-40枚成熟卵母细胞进行体外受精。7 h后,将去除卵丘的受精卵转入发育培养液(CRI)中。分别收集GV期、MⅡ期卵母细胞、2-、4-、8-细胞胚胎、桑椹胚和囊胚保存于液氮中。
PBS(-)+2% BSA清洗收集到的卵母细胞、受精卵和体外受精胚胎。4%多聚甲醛室温固定30 min,1% Triton X-100通透1 h。10%山羊血清+2% BSA的PBS(-)室温封闭1 h。然后用PBS(-)+2% BSA按1∶100稀释AID蛋白一抗(Upstate)4℃孵育过夜。PBS(-)+2% BSA清洗3遍后,用1∶500的FITC标记二抗(Sigma)避光染色2 h。10 μg/mL DAPI染细胞核,PBS(-)+2% BSA清洗3遍后,使用Nikon激光共聚焦显微镜进行检测。所有试验,Nikon激光共聚焦显微镜都使用相同的设置。
1.2.5 统计方法本研究所有数据重复3次以上。所有试验数据都使用SPSS 14.0软件进行统计分析。Real-time PCR试验结果使用单因素分析,DNA甲基化状态使用卡方检验。P<0.05确定为差异的显著性。
2 结果 2.1 AID基因在成年牛组织中的表达变化如 图 1 所示,AID基因在心脏、肾脏、肝脏和睾丸4种组织中均有表达,表达量都较低。其表达水平在不同组织之间呈现不同的状态,其中心脏和肝脏较低,肾脏水平稍高,睾丸组织表达量最高,并明显高于其他3种组织(P<0.05)。

|
| 图 1 AID基因在牛不同组织的表达变化 H:心脏;T:睾丸;L:肝脏;K:肾脏。不同字母代表显著性差异(P< 0.05) |
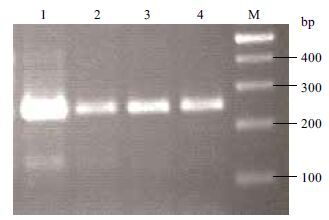
AID基因的转录起始位点以上3 600 bp的区域内共有24个CpG位点,没有CpG岛。根据其序列设计两段BSP引物,分别位于AID基因的-88 bp--431 bp和-1416 bp--1548 bp,共包含11个CpG位点,其中-1416 bp--1548 bp包含有4个CpG位点,-88 bp--431 bp包含有7个CpG位点。分别提取心脏、肝脏、肾脏和睾丸组织的基因组DNA,并经亚硫酸氢盐处理后,使用引物AID BSP 1和AID BSP 2分别进行PCR反应,扩增产物经琼脂糖凝胶电泳后照相观察。结果显示,引物AID BSP 1的PCR产物大小与预期片段大小一致( 图 2 );AID BSP 2的PCR产物大小也与预期片段大小一致( 图 3 )。

|
| 图 2 牛AID BSP 1甲基化PCR结果 M:100 bp DNA Marker;1:0.2 µmol/L Mg2+条件下PCR结果;2:0.4 µmol/L Mg2+条件下PCR结果;3:0.6 µmol/L Mg2+条件下PCR结果;4:0.8 µmol/L Mg2+条件下PCR结果;5:1 µmol/L Mg2+条件下PCR结果 |
|
| 图 3 牛AID BSP 2甲基化PCR电泳结果 1:0.4 µmol/L Mg2+ 条件下PCR结果;2:0.8 µmol/L Mg2+ 条件下PCR结果;3:1.2 µmol/L Mg2+ 条件下PCR结果;4:1.6 µmol/L Mg2+ 条件下PCR结果;M:100 bp DNA Marker |
比较测序结果与原始序列后,发现AID基因5′调控区的11个CpG位点在4种组织中都具有较高的甲基化程度。心脏、睾丸、肝脏、肾脏的DNA甲基化比率分别为81.82%、78.18%、79.09%和78.18%( 图 4-A ),且各组织之间没有明显的差异(P>0.05)。单独分析-88 bp--431 bp这一区段发现其甲基化比率分别为心脏90.0%,睾丸77.1%,肝脏91.4%和肾脏87.1%,各组织之间没有明显的差异( 图 4-C )。在-1416 bp--1548 bp这一区段心脏为65.0%,睾丸为80.0%,肝脏为57.5%,肾脏为65.0%,各组织之间也无明显差异( 图 4-B )。综合分析AID基因在不同组织中的表达量和其在各个区段上的DNA甲基化程度发现,-88 bp--431 bp这一区段的DNA甲基化与牛AID基因的组织特异性表达呈明显的负相关。基于其在AID基因的表达的重要调节作用,可以确定这一区段就是AID基因的T-DMR。

|
| 图 4 牛心脏、睾丸、肝脏、肾脏中AID基因DNA甲基化水平的变化 H:心脏;T:睾丸;L:肝脏;K:肾脏;(A)牛心脏、睾丸、肝脏、肾脏中AID基因启动子区亚硫酸氢盐PCR测序结果。●代表甲基化CpG;○代表未甲基化CpG。(B)AID基因启动子区-1416 bp--1548 bp区段的DNA甲基化水平。(C)AID基因启动子区-88 bp--431 bp区段的DNA甲基化水平 |
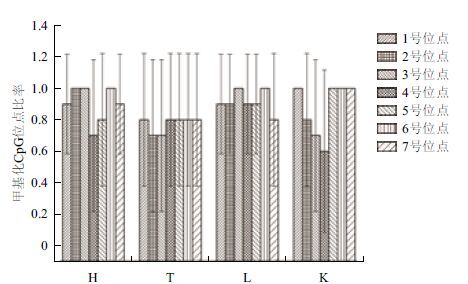
为了更好地分析AID基因T-DMR对AID基因表达的影响,本研究单独分析了AID基因T-DMR的每一个CpG位点的DNA甲基化状态。结果( 图 5 )显示,睾丸组织AID基因T-DMR中7个CpG位点都发生了一定的DNA去甲基化,而其他3种组织却只是在一些个别位点发生了DNA去甲基化。
|
| 图 5 AID基因T-DMR单个CpG位点甲基化情况分析 H:心脏;T:睾丸;L:肝脏;K:肾脏 |
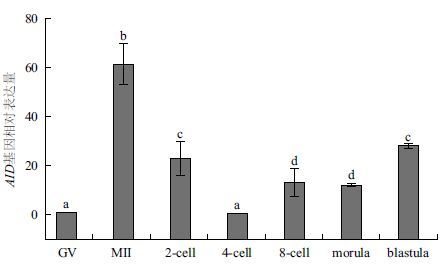
本研究通过Real-time PCR的方法分别检测了GV期卵母细胞、MⅡ期卵母细胞、2-cell、4-cell、8-cell、桑椹胚和囊胚中AID基因的表达。结果( 图 6 )显示,卵母细胞成熟过程中AID基因在逐渐的积累,到MⅡ期卵母细胞达到顶峰。随着精子入卵,受精卵开始细胞分裂,AID基因的表达量急剧下降,直到8-cell时期也即母型合子转变时期AID基因的表达量开始逐渐升高,直到囊胚期。
|
| 图 6 AID基因在牛卵母细胞和不同发育阶段胚胎中的表达 GV:GV期卵母细胞;MⅡ:MⅡ期卵母细胞;2 cell:2-细胞;4-cell:4-细胞;8 cell:8-细胞;morula:桑椹胚;blastula:囊胚。不同字母代表显著性差异(P<0.05) |
为了确定AID在牛早期胚胎发育过程中的作用,本研究使用AID蛋白的特异性抗体,利用免疫荧光细胞化学的方法,检测了牛卵母细胞体外成熟和早期胚胎发育过程中AID蛋白的动态变化。结果( 图 7 )显示,从卵母细胞成熟到早期胚胎发育过程中的2-cell和4-cell阶段,AID蛋白的含量是逐渐降低的。从8-cell开始即母型合子转变时期,AID蛋白的表达量开始逐步上升。从卵母细胞成熟期到桑椹胚期,AID蛋白都是均匀分布于细胞核和细胞质中;而到了囊胚期大量的AID蛋白集中于内细胞团。

|
| 图 7 AID在不同发育阶段牛卵母细胞和胚胎中的动态变化 A,A′:GV期卵母细胞;B,B′:MⅡ期卵母细胞;C,C′:2-细胞;D,D′:4-细胞;E,E′:8-细胞;F,F′:桑椹胚;G,G′:囊胚。DAPI复染DNA,标尺所示50 µm |
随后本研究检测了AID基因T-DMR在卵母细胞成熟和早期胚胎发育过程中DNA甲基化状态。结果( 图 8 )表明,GV期卵母细胞中AID基因T-DMR甲基化比率为82.86%,而在MⅡ期及随后的早期胚胎发育过程中DNA甲基化都未发生变化。

|
| 图 8 牛卵母细胞成熟和早期胚胎发育过程中AID基因DNA甲基化水平的变化 GV:GV期卵母细胞;MⅡ:MⅡ期卵母细胞;2 cell:2-细胞;4 cell:4-细胞;8 cell:8-细胞;morula:桑椹胚;blastula:囊胚。●代表甲基化CpG;○代表未甲基化CpG |
DNA甲基化是主要的调节基因表达的表观遗传修饰方式之一[2]。基因启动子区的DNA被甲基化后可以抑制基因的转录。例如,多能性转录因子c-Myc的识别靶序列CACGTG被甲基化后,FoxA1蛋白就不与其启动子结合转而结合于基因远端调控序列[29],最终导致c-Myc基因转录的失活。哺乳动物基因组存在大量的T-DMR,T-DMR包含有单个细胞或特定组织类型的DNA甲基化信息,决定基因的组织特异性和阶段特异性表达[30, 31]。
在成年牛的不同组织中,T-DMR区的DNA都存在相当高的甲基化水平。但是每一种组织中都有其自己的甲基化模式从而调节其基因的表达,总体上遵循一般的规律,即DNA甲基化程度高的基因表达量就低,相反甲基化程度低的基因表达量高[32]。研究结果表明AID基因在心脏、肾脏、肝脏、睾丸4种组织中均有表达但是其转录水平不同。睾丸组织明显高于其他3种组织。分析4种组织DNA甲基化水平,在-88 bp--431 bp睾丸的DNA甲基化程度较其他3种组织低。因此推断其为AID基因的T-DMR,而T-DMR的DNA甲基化水平与其表达有密切的联系。同时本研究发现AID基因在心脏、肝脏和肾脏中虽然都有少数的个别CpG位点发生了DNA去甲基化,但其组织中AID基因的表达没有明显升高,可以推断AID基因的表达的调控是由于AID基因T-DMR区的所有CpG位点的DNA甲基化状态共同作用的结果,而并非由于某个单独的CpG位点的甲基化所决定。DNA甲基化可能是通过改变了染色质的构象[33, 34],进而影响转录因子与基因启动子区的结合[35]。AID基因这4种组织中的甲基化模式不具有显著性差异,但是其表达水平却存在差异,这就说明在体内还有其它机制共同作用于AID的表达。其相关机制和相互关系有待于进一步研究。
哺乳动物受精后首先发生整体的去甲基化过程,发育相关基因的表达也在特定时间被开关,这种整体的去甲基化状态与母型合子转变中胚胎基因组的激活有关,整体的去甲基化状态发生和持续的时间因动物种类不同而异。体细胞核移植胚也出现同样过程[36]。本研究确定了AID基因的T-DMR(-88 bp--431 bp),这一段DNA的甲基化和其表达水平成紧密相关。在卵母细胞成熟过程中,T-DMR区的第2和第3号CpG位点的DNA甲基化发生了一个明显的去除过程,而其他位点都未发生变化。卵母细胞在成熟过程中AID基因的积累就与这样的DNA甲基化状态的变化相关。而在早期胚胎发育过程中,AID基因的T-DMR区除了第2和第3号CpG位点的DNA甲基化一直都维持去甲基化状态其他位点都是甲基化的状态。相关研究证实,受精后几小时内,鱼精蛋白被组蛋白替换,父源染色质去凝缩,DNA发生去甲基化[37, 38]。在短时间内,父源染色质利用卵母细胞在成熟过程积累的物质发生DNA去甲基化[27]。AID基因表达也具有类似的变化规律,提示AID对于父源染色质DNA的主动去甲基化具有一定的作用。在受精后,AID基因的表达持续下降直到8-cell时期表达量才开始上升,这可能是与母型合子转变中胚胎基因组的激活有关,也说明了AID对于胚胎发育及其干细胞多能性的维持具有重要的作用。在牛早期胚胎发育过程中,虽然AID基因DNA甲基化都未发生变化,但是其基因表达却有明显的改变,这就提示AID基因在这一过程中的阶段特异性表达可能是受到其他表观遗传方面或是转录后的调控。通过免疫荧光检测发现,从卵母细胞成熟期到桑椹胚期,AID蛋白都是均匀分布于细胞核和细胞质中。而囊胚时期大量的AID蛋白集中于内细胞团,这可能对于内细胞团多能性的维持起重要作用。
4 结论卵母细胞成熟中积累的AID作用于受精过程,其启动子区的DNA去甲基化与AID基因的表达有关。胚胎基因组激活后AID基因的表达可能与胚胎发育有关。
| [1] | Tamada H, Thuan NV, Reed P, et al. Chromatin decondensation and nuclear reprogramming by nucleoplasmin[J]. Mol Cell Biol, 2006, 26 : 1259–1271. |
| [2] | Li E. Chromatin modification and epigenetic reprogramming in mammalian development[J]. Nature Reviews Genetics, 2002, 3 : 662–673. |
| [3] | Kass SU, Pruss D, Wolffe AP. How does DNA methylation repress transcription?[J]. Trends in Genetics, 1997, 13 : 444–449. |
| [4] | Reik W, Dean W, Walter Jr. Epigenetic reprogramming in mammalian development[J]. Science, 2001, 293 : 1089–1093. |
| [5] | Jones PA, Liang G. Rethinking how DNA methylation patterns are maintained[J]. Nature Reviews Genetics, 2009, 10 : 805–811. |
| [6] | ZhuJK. Active DNA demethylation mediated by DNA glycosylases[J]. Annu Rev Genet, 2009, 43 : 143–166. |
| [7] | Cortellino S, Xu J, Sannai M, et al. Thymine DNA glycosylase is essential for active DNA demethylation by linked deamination-base excision repair[J]. Cell, 2011, 146 : 67–79. |
| [8] | Ito S, D’Alessio AC, Taranova OV, et al. Role of Tet proteins in 5mC to 5hmC conversion,ES-cell self-renewal and inner cell mass specification[J]. Nature, 2010, 466 : 1129–1133. |
| [9] | Wossidlo M, Arand J, Sebastiano V, et al. Dynamic link of DNA demethylation,DNA strand breaks and repair in mouse zygotes[J]. The EMBO Journal, 2010, 29 : 1877–1888. |
| [10] | Okada Y, Yamagata K, Hong K, et al. A role for the elongator complex in zygotic paternal genome demethylation[J]. Nature, 2010, 463 : 554–558. |
| [11] | Chen T, Li E. Structure and function of eukaryotic DNA methyltransferases[J]. Current Topics in Developmental Biology, 2004, 60 : 55–89. |
| [12] | Schmitz KM, Schmitt N, Hoffmann-Rohrer U, et al. TAF12 recruits Gadd45a and the nucleotide excision repair complex to the promoter of rRNA genes leading to active DNA demethylation[J]. Molecular Cell, 2009, 33 : 344–353. |
| [13] | Wu SC, Zhang Y. Active DNA demethylation:many roads lead to Rome[J]. Nature Reviews Molecular Cell Biology, 2010, 11 : 607–620. |
| [14] | Cannon SV, Cummings A, Teebor GW. 5-Hydroxymethylcytosine DNA glycosylase activity in mammalian tissue[J]. Biochem Biophys Res Commun, 1988, 151 : 1173–1179. |
| [15] | Kantarjian H, Oki Y, Garcia-Manero G, et al. Results of a randomized study of 3 schedules of low-dose decitabine in higher-risk myelodysplastic syndrome and chronic myelomonocytic leukemia[J]. Blood, 2007, 109 : 52–57. |
| [16] | Yang AS, Doshi KD, Choi SW, et al. DNA methylation changes after 5-aza-2’-deoxycytidine therapy in patients with leukemia[J]. Cancer Res, 2006, 66 : 5495–5503. |
| [17] | Mund C, Hackanson B, Stresemann C, et al. Characterization of DNA demethylation effects induced by 5-Aza-2’-deoxycytidine in patients with myelodysplastic syndrome[J]. Cancer Res, 2005, 65 : 7086–7090. |
| [18] | Issa JPJ, Kantarjian HM, Kirkpatrick P. Azacitidine[J]. Nature Reviews Drug Discovery, 2005, 4 : 275–276. |
| [19] | Conticello SG, Langlois MA, Yang Z, et al. DNA deamination in immunity:AID in the context of its APOBEC relatives[J]. Advances in Immunology, 2007, 94 : 37–73. |
| [20] | Maul RW, et al. Chapter six-AID and somatic hypermutation[J]. Advances in Immunology, 2010, 105 : 159–191. |
| [21] | Muramatsu M, Kinoshita K, Fagarasan S, et al. Class switch recombination and hypermutation require activation-induced cytidine deaminase(AID),a potential RNA editing enzyme[J]. Cell, 2000, 102 : 553–563. |
| [22] | Liu M, Schatz DG. Balancing AID and DNA repair during somatic hypermutation[J]. Trends in Immunology, 2009, 30 : 173–181. |
| [23] | Rai K, Huggins IJ, James SR, et al. DNA demethylation in zebrafish involves the coupling of a deaminase,a glycosylase,and Gadd45[J]. Cell, 2008, 135 : 1201–1212. |
| [24] | Morgan H, Dean W, Coker H, et al. Activation-induced cytidine deaminase deaminates 5-methylcytosine in DNA and is expressed in pluripotent tissues[J]. Biological Chemistry, 2004, 279 : 52353–5260. |
| [25] | Bhutani N, Brady JJ, Damian M, et al. Reprogramming towards pluripotency requires AID-dependent DNA demethylation[J]. Nature, 2010, 463 : 1042–1047. |
| [26] | Popp C, Dean W, Feng S, et al. Genome-wide erasure of DNA methylation in mouse primordial germ cells is affected by AID deficiency[J]. Nature, 2010, 463 : 1101–1105. |
| [27] | Pagé-Larivière F, Sirard MA. Spatiotemporal expression of DNA demethylation enzymes and histone demethylases in bovine embryos[J]. Cellular Reprogramming, 2014, 16 : 40–53. |
| [28] | Leutenegger CM, Alluwaimi AM, Smith WL, et al. Quantitation of bovine cytokine mRNA in milk cells of healthy cattle by real-time TaqMan polymerase chain reaction[J]. Vet Immunol Immunopathol, 2000, 77 : 275–287. |
| [29] | Lupien M, Eeckhoute J, Meyer CA, et al. FoxA1 translates epigenetic signatures into enhancer-driven lineage-specific transcription[J]. Cell, 2008, 132 : 958–970. |
| [30] | Shiota K, Kogo Y, Ohgane J, et al. Epigenetic marks by DNA methylation specific to stem,germ and somatic cells in mice[J]. Genes to Cells, 2002, 7 : 961–969. |
| [31] | Wan J, Oliver VF, Zhu H, et al. Integrative analysis of tissue-specific methylation and alternative splicing identifies conserved transcription factor binding motifs[J]. Nucleic Acids Res, 2013, 41 : 8503–8514. |
| [32] | Siegfried Z, Cedar H. DNA methylation:A molecular lock[J]. Current Biology, 1997, 7 : 305–307. |
| [33] | Weinberg MS, Villeneuve LM, Ehsani A, et al. The antisense strand of small interfering RNAs directs histone methylation and transcriptional gene silencing in human cells[J]. RNA, 2006, 12 : 256–262. |
| [34] | Santoro R, Grummt I. Epigenetic mechanism of rRNA gene silencing:temporal order of NoRC-mediated histone modification,chromatin remodeling,and DNA methylation[J]. Mol Cell Biol, 2005, 25 : 2539–2546. |
| [35] | Lehnertz B, Ueda Y, Alwin AHA. Derijck,et al[J]. Curr Biol, 2003, 13 : 1192–1200. |
| [36] | Papp B, Plath K. Epigenetics of reprogramming to induced pluripotency[J]. Cell, 2013, 152 : 1324–1343. |
| [37] | McLay DW, et al. Remodelling the paternal chromatin at fertilizati-on in mammals[J]. Reproduction, 2003, 125 : 625–633. |
| [38] | Rivera RM, Ross JW. Epigenetics in fertilization and preimplantation embryo development[J]. Prog Biophys Mol Biol, 2013, 113 : 423–432. |





