2. 新乡学院生命科学技术学院,新乡 453000;
3. 天津科技大学食品工程与生物技术学院,天津 300457;
4. 华南理工大学轻工与食品学院,广州 510640
2. College of Life Science and Technology,Xinxiang University,Xinxiang 453000;
3. College of Food Engineering and Biotechnology,Tianjin University of Science and Technology,Tianjin 300457;
4. College of Light Industry and Food Sciences,South China University of Technology,Guangzhou 510640
单核细胞增生李斯特菌(Listeria monocytogenes,LM)是一种典型无芽孢细胞内寄生的革兰氏阳性菌,是人畜共患传染病李斯特菌病(Listeriosis)的主要病原菌,常引起脑膜炎、败血症和单核细胞增多等症状。LM能在多种恶劣环境条件下生存和繁殖,导致食品在加工和运输过程中易受其污染,被世界卫生组织列为仅次于大肠杆菌O157∶H7、沙门氏菌、志贺氏菌后的第四大重要的食源性致病菌[1]。
喹诺酮类是由萘啶酸发展起来的全合成抗菌药物,具有独特的作用机制、优秀的药物动力学性能和抗菌活性等特性,在临床和兽医上得到了广泛的应用。一直以来,喹诺酮类药物并没有被用于临床治疗李斯特菌病,但是由于此类药物在临床上的频繁使用,间接的促进了耐喹诺酮类LM的出现[2]。巢国祥等[3]对江苏省8类食品中LM的调查结果显示,菌株对诺氟沙星和环丙沙星的耐药率分别为25.6%和9.3%。Yan等[4]对2005-2007年河北省食源性LM耐药性监测发现17.8%的菌株对环丙沙星表现出耐药性,耐药率仅次于头孢噻肟。
目前细菌对喹诺酮类的耐药机制主要有3种:一是编码DNA促旋酶(gyrA和gyrB编码)和拓扑异构酶ⅠV(parC和parE编码)基因的喹诺酮耐药决定区(Quinolone resistance-determining region,QRDR)发生突变,导致氨基酸置换使药物不能与靶酶结合[5]。QRDR突变是细菌对喹诺酮类药物产生耐药的最主要原因[6]。Lampidis等[7]的研究表明,gyrA基因 QRDR的Ser84→Thr和Asp/Glu88→Phe突变可能导致LM对萘啶酸耐药。二是主动外排系统表达增强[8]。Godreuil等[2]研究发现LM菌株中主要易化子超家族(Major facilitator superfamily,MFS)外排泵lde与LM对喹诺酮耐药密切相关。三是质粒介导的喹诺酮耐药(Plasmid-mediated quino-lone resistance,PMQR)[9]。PMQR基因在肠杆菌科和弧菌科等革兰氏阴性菌中分布广泛,并且能够通过基因水平转移在不同种属细菌间快速传播[10]。
耐喹诺酮类LM菌株不断检出,但是目前关于LM对喹诺酮耐药机制的研究却很少。因此,本研究以分离自食品的15株环丙沙星耐药株为研究对象,通过检测QRDR基因突变、PMQR基因的分布以及外排泵lde在LM喹诺酮耐药中的作用,系统性调查LM对喹诺酮类药物产生耐药的分子机制,旨在为预防和控制LM耐药菌株的出现奠定理论基础。
1 材料与方法 1.1 材料 1.1.1 菌株及质粒食源性单核细胞增生李斯特菌由本实验室保存;大肠杆菌DH5α购自北京博迈德基因技术有限公司;穿梭质粒pKSV7由中科院微生物研究所刘钢教授和浙江大学方维焕教授惠赠。
1.1.2 培养基及主要试剂培养基购自北京陆桥生物技术有限公司;环丙沙星、氨苄西林和利血平购自Sigma公司;Taq DNA聚合酶、dNTPs、pMD18-T载体、限制性内切酶Xba Ⅰ和Pst Ⅰ均购自大连宝生物工程有限公司;细菌RNA提取试剂盒、cDNA第一链合成试剂盒、RealMasterMix(SYBR Green)、细菌基因组DNA提取试剂盒、质粒小提试剂盒购自北京天根生化科技有限公司;T4连接酶购自北京博迈德基因技术有限公司。
1.1.3 仪器与设备DH360型电热恒温培养箱,北京科伟永兴仪器有限公司;THZ-82气浴恒温振荡器,金坛天竟实验仪器厂;TGL-16B型离心机,上海安亭科学仪器厂;ETC811型基因扩增仪,北京东胜创新生物科技有限公司;DYY-8C型电泳仪,北京市六一仪器厂;UNIVERSAL HOOD Ⅱ凝胶成像分析系统、Chromo 4实时荧光定量PCR仪,美国Bio-Rad公司;Eporator电转化仪,德国Eppendorf公司。
1.2 方法 1.2.1 药物敏感性实验采用琼脂二倍稀释法测定LM菌株对环丙沙星的最小抑菌浓度(MIC),结果按照美国临床实验室标准协会(CLSI)的标准[11]进行判读。
1.2.2 PCR扩增QRDR及PMQR基因按照细菌基因组DNA提取试剂盒的步骤提取LM菌株的基因组DNA作为扩增模板。PCR扩增gyrA、gyrB、parC和parE基因所用引物及反应条件参照文献( 表 1 )。扩增产物经测序后,采用DNAman软件进行序列拼接、核正和氨基酸序列的推导,并采用BLAST软件将测序结果与GenBank数据库进行同源性分析,以确定QRDR靶基因突变情况。PMQR相关基因(qnrA、qnrB和qnrS)的引物设计及PCR反应条件,见表 1 。
加入外排泵抑制剂利血平(终浓度为20 μg/mL,该浓度对细菌外排泵的抑制作用明显同时又不影响细菌的正常生长[2, 15, 16])后再检测LM对环丙沙星的药物敏感性,观察MIC值的变化。加入利血平后MIC值下降4倍或4倍以上的菌株视为外排泵阳性菌株,初步判定这些菌株对环丙沙星的耐药与外排泵有关。采用琼脂二倍稀释法检测加入利血平前后LM突变株对环丙沙星的MIC值。
1.2.4 细菌总RNA提取将LM菌株接种于BHI平板上37℃培养18 h,挑取生长良好的单个菌落至2 mL BHI液体培养基中,37℃过夜培养。吸取该培养液0.2 mL至2 mL BHI培养液中,37℃培养至对数期。每个菌株分为3个试验组,第1组为不加任何抗生素(作为对照组),第2组为加入环丙沙星(浓度为0.4×MIC值)后再培养4 h,第3组是在第2组的培养物中加入利血平(20 μg/mL)后再培养4 h。细菌总RNA的提取按照细菌RNA提取试剂盒的使用说明进行,测定RNA的浓度和纯度后用于后续试验。
1.2.5 实时荧光定量PCR利用cDNA第一链合成试剂盒将mRNA合成第一链cDNA,-20℃保存备用。根据GenBank的基因序列设计靶基因lde和参照基因16S rRNA的引物,由上海英骏生物技术有限公司合成( 表 1 )。以反转录反应中得到的第一链cDNA作为PCR反应模板,使用RealMasterMix(SYBR Green)试剂盒进行实时荧光定量PCR的扩增。反应条件为:95℃预变性3 min;95℃变性30 s,50℃退火30 s,72℃延伸20 s,39个循环。采用ΔΔCT法进行目的基因表达量的分析。
1.2.6 lde基因缺失株的构建与分子鉴定 1.2.6.1 引物设计根据GenBank登录的lde基因及上下游同源臂序列,通过DNAMAN软件进行同源性分析,选择保守区域用Primer 5.0软件设计扩增上下游同源臂的特异性引物:上游同源臂的扩增引物为LP1和LP2,全长525 bp;下游同源臂的扩增引物为LP3和LP4,全长712 bp;重叠延伸PCR的引物为LP1和LP4,全长1 237 bp;检测引物LP5和LP6,全长1 852 bp。在LP1和LP4引物的5′端分别加酶切位点Xba Ⅰ和Pst Ⅰ及保护碱基,引物由上海英骏生物技术有限公司合成( 表 1 )。
1.2.6.2 lde基因的缺失以LM基因组DNA为模板,分别用LP1、LP2和LP3、LP4两对引物扩增lde基因的上下游同源臂,PCR反应条件为:94℃预变性5 min;94℃变性40 s,50℃退火40 s,72℃延伸40 s,30个循环;72℃延伸7 min。1%琼脂糖凝胶电泳后PCR产物纯化回收。然后通过SOE-PCR技术融合上下游同源臂,采用25 μL反应体系:纯化的上下游同源臂各3 μL,10×PCR buffer 2.5 μL,10 mmol/L dNTPs 1 μL,水15.3 μL,Taq DNA聚合酶0.2 μL。反应条件:94℃预变性5 min;94℃变性40 s,50℃退火40 s,72℃延伸50 s,8个循环。再加入引物LP1和LP4各1.5 μL及0.2 μL的Taq DNA聚合酶扩增融合片段的全长,反应条件:94℃预变性5 min;94℃变性40 s,50℃退火40 s,72℃延伸90 s,30个循环;72℃延伸7 min。扩增产物经1%琼脂糖凝胶电泳后切胶回收,将回收产物与pMD18-T载体连接,采用10 μL反应体系:pMD18-T载体1 μL,目的片段△lde 4 μL,Solution Ⅰ 5 μL,4℃连接过夜;构建缺失质粒pMD18-T-Δlde,并转化至大肠杆菌DH5α感受态细胞中,经含氨苄西林的LB培养基筛选,挑取单个菌落接种于含氨苄西林的LB液体培养基中37℃过夜培养,用LP5和LP6引物对菌液PCR进行鉴定,将阳性转化株送至北京博迈德基因技术有限公司进行测序。
1.2.6.3 重组穿梭质粒pKSV7-Δlde的构建将测序正确的pMD18-T-Δlde质粒与pKSV7用限制性内切酶Xba Ⅰ和Pst Ⅰ同步双酶切,采用20 μL反应体系:Xba Ⅰ和Pst Ⅰ各1 μL,10×M buffer 2 μL,质粒7 μL,水9 μL。37℃水浴锅中放置4 h。将酶切后的产物分别进行琼脂糖凝胶电泳,切胶回收目的片段,用T4连接酶连接回收片段,采用10 μL反应体系:pKSV7双酶切产物3 μL,酶切后的lde基因缺失片段5 μL,T4连接酶1 μL,10× buffer 1 μL,16℃连接过夜[17];构建重组穿梭质粒pKSV7-Δlde,并转化至大肠杆菌DH5α感受态细胞中,通过菌液PCR和双酶切验证。
1.2.6.4 电转化及L28-Δlde突变株的筛选与鉴定采用青霉素G法制备LM感受态细胞[17]。取10 μL重组穿梭质粒pKSV7-Δlde与100 μL LM感受态细胞混匀,冰浴10 min后2.5 kV电击5 ms,立刻加入900 μL BHI液体培养基于30℃培养2 h。将菌液均匀涂布于含氯霉素(15 μg/mL)的BHI平板上,30℃培养48 h。挑取单个菌落接种至含氯霉素(15 μg/mL)的BHI液体培养基中30℃培养24 h,用LP5和LP6引物对进行菌液PCR筛选鉴定阳性克隆。将鉴定得到的阳性克隆接种至含氯霉素(15 μg/mL)的BHI液体培养基中42℃传代培养15代左右,再将重组菌接种至不含氯霉素的BHI液体培养基中42℃传代培养15代左右,通过菌液PCR检测缺失条带稳定存在,最终得到LM-Δlde突变株。
2 结果 2.1 药物敏感性试验结果LM菌株对环丙沙星的药物敏感性结果,见表 2 。3株敏感菌株对环丙沙星的MIC值为0.5-1 μg/mL;15株耐药菌株对环丙沙星的MIC值为4-16 μg/mL。
加入外排泵抑制剂利血平后,3株敏感菌株对环丙沙星的MIC值不变;15株耐药菌株中,3株对环丙沙星的MIC值没有变化,10株对环丙沙星的MIC值降为原来的1/2,1株降为原来的1/4,1株降为原来的1/8。
2.2 QRDR基因突变检测结果经过DNA序列比对,11株环丙沙星耐药菌株的gyrA、gyrB和parE基因序列与GenBank数据库中的相应序列100%相似,没有发生碱基变化。11株环丙沙星耐药菌株中有2株(L28和L45)的parC基因序列在第351位发生碱基变化(T→A),但是该碱基突变并未引起氨基酸序列变化。
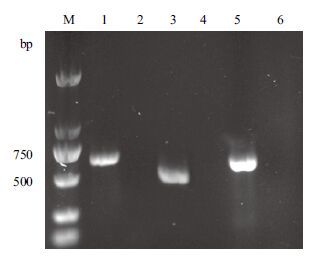
2.3 PMQR基因检测结果18株LM菌株中未检出PMQR基因( 图 1 )。
|
| 图 1 PMQR基因扩增结果 M:DNA分子质量标准;1:qnrA阳性对照;3:qnrB阳性对照;5:qnrS阳性对照;2、4和6:阴性对照 |
通过实时荧光定量PCR技术检测lde基因在LM菌株中的表达量,结果见表 3 。L28和L47代表外排泵阳性菌株;L31和L45代表环丙沙星MIC值没有受到利血平影响的菌株;L69代表环丙沙星敏感菌株。结果显示,lde基因在耐药株和敏感株中均有表达,且表达量相近。在环丙沙星的作用下,lde基因在L28和L47中的相对表达量显著增加(P<0.05),分别为4.17倍和3.93倍,表明该基因的表达可能受到环丙沙星的诱导;lde基因在L31、L45和L69中的相对表达量没有明显变化(P>0.05),分别为1.05倍、1.88倍和1.04倍。加入利血平后,lde基因在L28和L47中的相对表达量明显下降(P<0.01),分别为1.04倍和0.72倍,表明lde基因的表达受到利血平的抑制;lde基因在L31和L45中的相对表达量没有明显变化(P>0.05)。
以L28的基因组DNA为模板,分别用LP1、LP2和LP3、LP4两对引物扩增lde基因的上、下游同源臂,扩增产物经1%琼脂糖凝胶电泳后得到与预期相符的目的条带( 图 2 )。通过SOE-PCR将上游同源臂和下游同源臂连接在一起,经1%琼脂糖凝胶电泳得到长为1 237 bp的片段,与预期结果一致( 图 2 )。目的片段切胶回收后经测序比对发现缺失一段长为1 357 bp的序列,表明lde基因已成功缺失。

|
|
图 2 SOE-PCR扩增结果
A)M:DNA分子质量标准;1和2:上游同源臂;5和6:下游同源臂;3和 4:阴性对照;(B)M:DNA分子质量标准;1-4:重叠延伸PCR扩增产物;5:阴性对照 |
将测序正确的pMD18-T-Δlde质粒与pKSV7经Xba Ⅰ、Pst Ⅰ同步双酶切后,经琼脂糖凝胶电泳得到与预期值相符的目的条带( 图 3 )。回收目的片段,连接转化至大肠杆菌DH5α中,菌液PCR得到预期大小的目的条带。双酶切鉴定得到6 900 bp和1 237 bp两个条带,表明重组穿梭质粒pKSV7-Δlde构建成功。

|
| 图 3 重组穿梭质粒的构建及阳性转化子的筛选 (A)M:DNA分子质量标准;1:pMD18-T-Δlde双酶切产物;3:pKSV7双酶切产物;4:pKSV7质粒。(B)M,DNA分子质量标准;1:pKSV7-Δlde双酶切产物。(C)M:DNA分子质量标准;2:Δlde阳性对照;3和4:lde阳性对照;5:阴性对照;6和7:阳性克隆 |
电转后,用LP5和LP6引物进行菌液PCR检测,筛选得到阳性转化子。阳性转化子在含氯霉素的BHI培养基中42℃传代培养15代左右得到单交换菌株,依然具有氯霉素抗性;在不含氯霉素的BH培养基中42℃传代培养15代左右后得到双交换缺失株,失去氯霉素抗性,菌液PCR扩增只有一条495 bp的片段( 图 3 )。测序结果表明已成功缺失lde基因,获得L28-Δlde突变株。无氯霉素37℃传代培养20代的过程中,每一代菌株均只能扩增出一条495 bp的片段,表明L28-Δlde具有遗传稳定性。
2.6 L28-Δlde对环丙沙星的MIC值LM野生株L28对环丙沙星的MIC值为16 μg/mL。突变株L28-Δlde对环丙沙星的MIC值为1 μg/mL;加入利血平后,L28-Δlde对环丙沙星的MIC值没有发生变化,仍为1 μg/mL。
3 讨论本研究的试验菌株为15株食源性LM环丙沙星耐药菌株,通过研究这些菌株中QRDR基因突变、PMQR基因的分布以及外排泵的作用,旨在阐明LM对喹诺酮产生耐药的分子机制。靶基因DNA促旋酶和拓扑异构酶ⅠV的突变是细菌对喹诺酮类药物最重要的耐药机制。已有报道显示gyrA基因的 QRDR突变可能与LM对萘啶酸耐药有关[7]。本研究中15株LM耐药菌株GyrA、GyrB、parC和ParE亚基的氨基酸序列与敏感株100%相似,表明在这些菌株中不存在靶酶基因突变。此外,PCR检测结果显示在LM耐药菌株中未检出PMQR基因。结合以上两点可初步推断,LM对喹诺酮耐药可能由外排泵介导。
外排泵是细菌的一种自我保护机制,在细菌体内广泛存在,能够除去细菌细胞和胞膜中的有害物质[18]。一些外排泵可以把抗生素、消毒剂、重金属等作为底物排出菌体外使细菌产生耐药性。外排泵的失活能够导致细菌对药物从耐药转为敏感;相反,由于外排泵表达调控蛋白的改变或是基因启动子活性增强能够引起外排泵基因转录水平提高,导致外排泵表达量增加,继而产生耐药[19]。转运蛋白数据库(http://www.membranetransport.org/)的信息显示,LM EGDe中可能存在超过200个外排系统。目前研究者已发现lde外排泵可能与LM对喹诺酮耐药有关[2, 20]。由lde基因编码的多重耐药外排泵lde属于细菌外排系统中常见的MFS型,是一种自足型多药外排转运蛋白,可利用质子偶联交换产生的质子驱动力外排多种化学结构不同的底物,从而降低细胞内相应物质的浓度。lde蛋白由402个氨基酸分子组成,其结构包含12个跨膜区域和一个信号肽,与枯草芽孢杆菌的Bmr,金黄色葡萄球菌的NorA,肺炎链球菌的PmrA和粪肠球菌的EmeA分别具有25%、24%、29%和44%的相似性。
利血平是一种具有降血压活性的小分子生物碱,对MFS类外排泵有较好的抑制作用。外排泵抑制试验表明,加入利血平后大多数LM耐药菌株对环丙沙星的MIC值降低,其中L28和L47对环丙沙星的MIC值分别下降为原来的1/8和1/4,表明在这两株菌中外排泵是介导菌株对环丙沙星耐药的主要原因。在利血平的作用下,其余13株耐药菌株对环丙沙星的MIC值下降不明显甚至没有发生变化,可能是由于在这些菌株中外排泵对环丙沙星耐药贡献不大或者没有贡献,也可能是外排泵的活性没有受到利血平的抑制。实时荧光定量PCR检测lde基因在LM菌株中的相对表达量,结果表明,该基因在耐药株和敏感株中均有表达,且表达量相近;在环丙沙星的作用下,lde基因在耐药株中的相对表达量增加显著,而在敏感株中的表达量基本没有变化;随后加入利血平,可观察到lde基因在耐药株中的表达明显受到抑制。根据这些结果可初步推断外排泵lde可能参与LM对环丙沙星耐药。为了进一步证实我们的推断,明确外排泵lde在LM对喹诺酮耐药中的作用,本研究利用SOE-PCR技术构建lde基因缺失突变株。药敏结果显示,突变株对环丙沙星表现敏感;利血平存在下突变株对环丙沙星的MIC值不受影响。由此可进一步证明外排泵lde介导LM对喹诺酮类药物耐药。
4 结论本研究以分离自食品的15株环丙沙星耐药株为研究对象,系统性调查LM对喹诺酮类药物产生耐药的分子机制。结果表明,LM耐药菌株的GyrA、GyrB、parC和ParE亚基没有发生氨基酸突变;在这些菌株中也没有检出PMQR基因。外排泵抑制试验表明,LM对环丙沙星耐药可能与外排泵有关。实时荧光定量PCR结果表明外排泵lde可能参与LM对环丙沙星耐药。通过构建lde基因缺失突变株,并检测突变株对环丙沙星的MIC值,证明外排泵lde介导LM对喹诺酮类药物耐药。
| [1] | 罗勤, 张晓莉, 李兵, 等. 单核细胞增生李斯特菌PrfA蛋白转录调控毒力基因表达的分子机制[J]. 微生物学通报, 2008, 35(2) : 275–280. |
| [2] | Godreuil S, Galimand M, Gerbaud G, et al. Efflux pump Lde is associated with fluoroquinolone resistance in Listeria monocytogenes[J]. Antimicrobial Agents and Chemotherapy, 2003, 47(2) : 704–708. |
| [3] | 巢国祥, 焦新安, 徐勤, 等. 8类食品单核细胞增生李斯特菌流行特征及耐药性状研究[J]. 中国卫生检验杂志, 2005, 15(5) : 519–521. |
| [4] | Yan H, Neogi SB, Mo Z, et al. Prevalence and characterization of antimicrobial resistance of foodborne Listeria monocytogenes isolates in Hebei province of Northern China,2005-2007[J]. International Journal of Food Microbiology, 2010, 144(2) : 310–316. |
| [5] | Everett MJ, Jin YF, Ricci V, et al. Contributions of individual mechanisms to fluoroquinolone resistance in 36 Escherichia coli strains isolated from humans and animals[J]. Antimicrobial Agents and Chemotherapy, 1996, 40(10) : 2380–2386. |
| [6] | Friedman SM, Lu T, Drlica K. Mutation in the DNA gyrase A gene of Escherichia coli that expands the quinolone resistance-determining region[J]. Antimicrobial Agents and Chemotherapy, 2001, 45(8) : 2378–2380. |
| [7] | Lampidis R, Kostrewa D, Hof H. Molecular characterization of the genes encoding DNA gyrase and topoisomerase IV of Listeria monocytogenes[J]. Journal of Antimicrobial Chemotherapy, 2002, 49(6) : 917–924. |
| [8] | Ruiz J. Mechanisms of resistance to quinolones:target alterations,decreased accumulation and DNA gyrase protection[J]. Journal of Antimicrobial Chemotherapy, 2003, 51(5) : 1109–1117. |
| [9] | Hernández A, Sánchez MB, Martínez JL. Quinolone resistance:much more than predicted[J]. Frontiers in Microbiology, 2011, 2 : 22. |
| [10] | 姜晓冰. 大肠杆菌质粒介导喹诺酮耐药机制研究[D]. 广州:华南理工大学,2013. |
| [11] | CLSI,2012. Performance standards for antimicrobial susceptibility Testing CLSI Document M100-S21. Clinical and Laboratory Standards Institute,Wayne,PA |
| [12] | Wang M, Tran JH, Jacoby GA, et al. Plasmid-mediated quinolone resistance in clinical isolates of Escherichia coli from Shanghai,China[J]. Antimicrobial Agents and Chemotherapy, 2003, 47(7) : 2242–2248. |
| [13] | Jacoby GA, Walsh KE, Mills DM, et al. qnrB,another plasmid-mediated gene for quinolone resistance[J]. Antimicrobial Agents and Chemotherapy, 2006, 50(4) : 1178–1182. |
| [14] | Jiang Y, Zhou Z, Qian Y, et al. Plasmid-mediated quinolone resistance determinants qnr and aac(6’)-Ib-cr in extended-spectrum β-lactamase-producing Escherichia coli and Klebsiella pneumoniae in China[J]. Journal of Antimicrobial Chemotherapy, 2008, 61(5) : 1003–1006. |
| [15] | Markham PN, Westhaus E, Klyachko K, et al. Multiple novel inhibitors of the NorA multidrug transporter of Staphylococcus aureus[J]. Antimicrobial Agents and Chemotherapy, 1999, 43(10) : 2404–2408. |
| [16] | 邝小佳, 蔡杏珊, 张宇青, 等. 利血平对耐药结核分枝杆菌临床株的作用[J]. 热带医学杂志, 2008, 8(2) : 128–129. |
| [17] | 彭叶龙, 乔军, 孟庆玲, 等. 单核细胞增生李斯特菌ncRNA rli60基因缺失株的构建研究[J]. 石河子大学学报, 2014, 32(2) : 148–152. |
| [18] | Piddock LJV. Multidrug-resistance efflux pumps? not just for resistance[J]. Nature Reviews Microbiology, 2006, 4 : 629–636. |
| [19] | Poole K. Efflux-mediated resistance to fluoroquinolones in gram-negative bacteria[J]. Antimicrobial Agents and Chemotherapy, 2000, 44(9) : 2233–2241. |
| [20] | Jiang X, Zhou L, Gao D, et al. Expression of efflux pump gene lde in ciprofloxacin-resistant foodborne isolates of Listeria monocytogenes[J]. Microbiology and Immunology, 2012, 56(12) : 843–846. |