2. 吉林农业大学 生物反应器与药物开发教育部工程研究中心,长春 130118;
3. 通化市第一中学,通化 134000
2. Ministry of Education Bioreactor and Drug Development Research Center,Jilin Agricultural University,Changchun 130118;
3. Tonghua No.1 Senior High School,Tonghua 134000
植物非特异脂质转运蛋白(Non-specific lipid transfer protein,nsLTP)是一类胞外分泌性蛋白,分子量约为9 kD的碱性小分子蛋白[1]。脂质转运蛋白在不同物种之间的同源性较低,但均存在脂质转运蛋白的明显特征。脂质转运蛋白是一个多基因家族蛋白,如在拟南芥中已分离到100多个脂质转运蛋白基因,在辣椒中已经分离出6个脂质转运蛋白基因,而在水稻全基因组测序后发现了53个脂质转运蛋白基因,同时同一物种中脂质转运蛋白基因的表达具有较强的发育和组织特异性[2, 3]。
LTP 蛋白主要存在于植物地上器官的表皮组织中,许多研究表明,不同的nsLTP基因表达对生物胁迫及非生物胁迫产生不同响应[4]。国内外对水稻、菠菜、玉米、花椰菜、绿豆和拟南芥等多种植物的脂质转运蛋白已有较深入的研究,具备多种复杂的生物学功能,主要包括脂类物质转运、角质层蜡质合成[5, 6],低温[7-10]、高温[9, 11]或重金属[12]和病原菌侵害[13],另外,在花药和花粉发育[14, 15]、干旱和盐[16-19]的胁迫过程中也发挥重要作用。例如,小麦TaLTP1的表达受PEG、NaCl、H2O2、伤害胁迫诱导[20],芝麻SiLTP2与SiLTP4受NaCl、甘露醇诱导[16],腊梅nsLTP基因响应低温胁迫其表达增高[21],这些结果表明,nsLTP在植物对非生物胁迫和生物胁迫的反应过程中可能起着重要作用,但关于这些基因的具体生物学功能还有待进一步研究。
红花(Carthamus tinctorius L.)为菊科、红花属植物,对高温干旱逆境具有较强的耐性,种子耐储藏,是一种药食两用的经济作物。本课题前期已开展红花种子、花、叶的转录组测序分析(SRA0472-79.2)及红花功能基因的研究[22],在此基础上本研究克隆1个红花脂质转运蛋白基因cDNA序列并进行生物信息学分析和不同器官中的表达特性检测,旨在为深入研究CtLTP基因的功能,揭示脂质转运蛋白在红花抵抗逆境胁迫中的作用提供依据。
1 材料与方法 1.1 材料样品采自吉林农业大学种植基地,经吉林农业大学胡全德教授鉴定为菊科植物红花(Carthamus ti-nctorius L.)。取红花种子、花、茎、叶、根、胚轴和子叶迅速放于液氮,锡箔纸包好于-80℃保存备用。
1.2 方法 1.2.1 RNA提取和cDNA合成取红花各组织置于液氮中研碎,总RNA提取方法按照Trizol试剂说明书进行,提取后进行纯度及浓度测定;于-80℃保存备用。cDNA合成根据Biotek公司反转录试剂盒操作进行,反转录产物分离纯化后于-20℃保存备用。
1.2.2 红花LTP基因ORF片段的克隆根据Solexa测序获得的红花转录组序列中功能注释为植物脂质转运蛋白的Unigene23752,利用NCBI 网站(http://www.ncbi.nlm.nih.gov/)提供的ORF finder 软件对开放阅读框进行分析,确定起始密码子及终止密码子。按照引物设计原则,采用引物设计软件 Primer5.0,对红花转录组序列中的1个脂质转运蛋白基因的ORF序列设计引物(LPT-F和LPT-R)(表 1)。以反转录后的cDNA为模板进行PCR扩增,反应条件为:94℃预变性5 min;94℃ 30 s,58℃ 30 s,72℃ 30 s,30个循环;72℃ 8 min,4℃终止反应。对PCR产物进行回收,连接pEASY-T1载体,并转化DH5α感受态细胞,通过蓝白斑筛选后,挑取白色菌落过夜培养,提取质粒,PCR、双酶切(EcoRⅤ和Hind Ⅲ)鉴定重组子,然后测序(由生工生物工程(上海)股份有限公司完成)。
1.2.3 生物信息学分析ORF Finder在线软件分析红花LTP基因相应的氨基酸序列。将获得的红花LTP基因ORF序列通过BLAST搜索NCBI的蛋白质和核苷酸数据库,并通过DNAMAN软件翻译成氨基酸序列,利用DNAMAN软件对来源于各种植物的LTP的相关氨基酸序列进行同源比对。相对分子质量与理论等电点(pI)预测采用ExPASy在线服务器的Compute Pi/Mw 工具,二级结构预测使用 Dublin 大学的Porter 服务器,三级结构预测使用Swiss-Model服务器,结构功能域分析采用ExPASy在线务器Prosite Scanprosite。利用 SignalP4.1Server(http://www. cbs.dtu.dk/services/SignalP/)对红花LTP进行信号肽预测。利用TMHMM Server v. 2.0在线软件进行跨膜区预测。利用MEGA5.0软件中的Neighbor-Joining(邻位相连法,NJ)构建系统进化树等。
1.2.4 红花LTP基因的组织表达差异分析取红花种子、花、茎、叶、根提取RNA,并反转录为cDNA用于Real-time PCR分析。定量PCR反应体系为:SYBR Premix Ex Taq(Tli RNaseH Plus)(2×)10 μL;上下游引物(表 1)各0.4 μL;ROX Reference Dye Ⅱ(50×);DNA模板2 μL;ddH2O 6.8 μL。PCR反应条件为:95℃预变性30 s;95℃变性5 s,60℃退火20 s,40个循环。
从吉红1号种子中提取总RNA,凝胶电泳检测结果(图 1)显示,28S rRNA和18S rRNA 条带清晰,说明所提取的RNA完整性较好;经核酸蛋白检测仪测得A260/A280=1.93,A260/A230=1.98,表明RNA纯度较高,可用于RT-PCR 扩增。

|
| 图 1 红花种子总RNA电泳图 1,2:RNA 样品 |
根据CtLTP 基因编码区序列特异引物(LTP-F,LTP-R),以红花种子cDNA为模板扩增出一条294 bp的条带(图 2-A)。经回收纯化后,PCR产物与pEASY-T1载体连接,通过菌液PCR验证(图 2-B)和质粒的 EcoRⅤ和Hind Ⅲ双酶切验证(图 2-C),测序结果正确,命名该基因为CtLTP(GenBank 登录号为KT692604)。
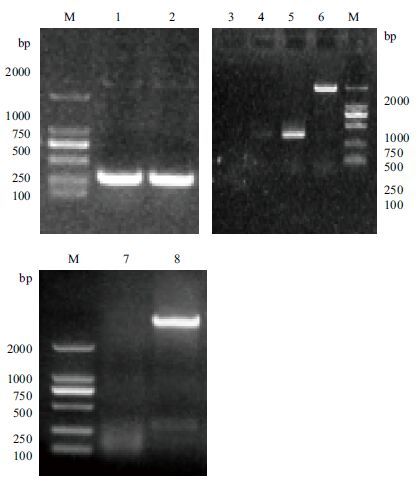
|
| 图 2 红花LTP基因的克隆 M:DNA Marker;1,2:LTP基因RT-PCR电泳;3-6:LTP基因菌液PCR电泳;7,8:LTP基因酶切验证 |
将推导出的红花脂质转运蛋白LTP氨基酸序列利用SignalP4.1软件进行信号肽分析,发现CtLTP1蛋白氨基端的前29个氨基酸为信号肽序列,属于分泌蛋白。用推测的氨基酸序列进行BLASTp比较,发现CtLTP属于LTP蛋白家族(lipid-transfer protein family)。TMHMM Server v. 2.0软件分析结果表明CtLTP1蛋白是一个跨膜蛋白,位于7-29处存在一个跨膜区,N端位于膜内,C端位于膜外。
利用ProtParam 软件对去除信号肽的红花LTP成熟蛋白进行基本特性分析,结果显示CtLTP1含有原子总数为1 029,分子式为C316H513N97O94S9,其分子量为7 463.6 Da,理论等电点为8.91。
用DictyOGlyc在线工具对蛋白的糖基化位点进行预测。根据P值大于0.5即为存在糖基化位点为评分标准,LTP蛋白仅存在1个糖基化位点,是位于第46位的丝氨酸(Ser)。这些修饰可能与LTP蛋白的生物学功能密切相关。
采用 SWISS-MODELSOPMA 进行LTP的编码蛋白二级结构分析,结果表明LTP蛋白二级结构主要由α-螺旋(Alpha helix,39.18)、延伸链(Extended strand,10.31)、无规则卷曲(Random coil,40.21)、β-转角(Beta turn,10.31)组成。
在SWISS-MODEL依据保守结构域作图工具中,对CtLTP编码蛋白进行三维结构建模,结果(图 3)显示,CtLTP编码蛋白是由3个α-螺旋和一个β-转角简单的缠绕在无规则卷曲上的简单结构。

|
| 图 3 LTP蛋白三级结构预测图 |
红花LTP蛋白氨基酸序列与其他植物LTP蛋白进行比对,结果显示在植物LTP中一些保守的氨基酸在红花中同样存在,但只有7个保守的半胱氨酸残基。利用DNAMAN软件对红花LTP进行同源性分析显示,与其他物种比较CtLTP蛋白与甘蓝型油菜一致性最高,为47.42%。结果表明,CtLTP是一个新的红花脂质转运蛋白。
为了进一步了解CtLTP蛋白与其他物种LTP蛋白间的进化关系,采用MEGA5.0软件的NJ法构建系统进化树(图 4)。通过与14种植物的LTP氨基酸聚类分析发现,脂质转运蛋白在烟草、野生大豆、甘蓝型油菜、拟南芥等植物中均以多种形式存在,且序列差异较大,在系统进化树分散在遗传距离较远的分支上。红花LTP蛋白与拟南芥、野生大豆、油橄榄、千里光以及苜蓿同属于一个分支。

|
| 图 4 红花LTP与其他物种LTP的聚类分析 |
利用实时荧光定量PCR技术检测CtLTP基因在不同组织中的相对表达量。组织差异表达可为研究基因的功能和调控机制提供依据,本实验选取红花的种子、花、茎、叶、根、胚轴和子叶7个组织对红花脂质转运蛋白基因进行了组织差异表达分析。图 5显示,CtLTP在不同组织的表达水平具有显著差异,在种子和花中呈现高表达,而在其他组织中低表达。

|
| 图 5 红花LTP基因的组织表达分析 *表示差异显著(P<0.05);**表示差异极显著(P<0.01) |
红花在食品、药品领域起着重要作用,是我国重要的油料和经济作物。红花病害的发生和流行一直是影响红花黄色素及红花油产量的重要因素,特别是根腐病、锈病等病害严重影响了红花的产量及品质。因此,通过生物技术手段提高红花品种的抗性,确保红花食品、药品的安全是十分必要的。
脂质转运蛋白在植物中普遍存在,目前已经从单子叶植物和双子叶植物的很多器官和组织中发现了脂质转运蛋白,其中以在拟南芥、水稻、玉米、小麦、棉花、辣椒、以及番茄等作物中研究较为深入。本研究根据红花转录组测序结果,克隆到1个脂质转运蛋白基因CtLTP,其编码的蛋白属于Non-specific lipid-transfer proteinⅡ(nsLTP2)亚家族,其具有 LTP蛋白的理化性质,包括8个保守的半胱氨酸残基、碱性等电点、低分子量以及N端典型信号肽结构等[24]。通过对13种植物与克隆得到的红花LTP氨基酸序列进行多重比对发现,他们的同源性差异较大,说明LTP蛋白的一级结构在不同物种间存在差异,具有多态性。LTP能被病原菌诱导而高水平表达,并能抑制病原菌的生长和诱导植物系统抗性的产生[25]。拟南芥LTP3作为一个新的负调控因子,通过调控ABA-SA的平衡参与植物免疫[26]。过量表达LTPs基因也能提高转基因作物对病原菌的抗性,能够增强作物的抗病和适应不良环境的能力;小麦TdLTP4基因在拟南芥中过表达能够提高其抗旱和抗盐能力,同时转基因拟南芥离体叶片表现出增强对交链孢菌和灰葡萄孢真菌性[27];小麦TdLTP3能够增强拟南芥抗旱性及氧化应激能力[28];超表达脂质转运蛋白AZI能够增强植物对盐的耐受性,体内外实验证明其通过与有丝分裂原活化的蛋白激酶MPK3相互结合发挥作用[29];转LTP基因甘蓝型油菜对菌核病的抗病性增强[30]。因此,通过基因工程的手段过量表达LTPs基因培育作物抗逆新品种,具有广泛的应用前景。
本研究克隆到1个脂质转运蛋白基因CtLTP基因,并主要在红花种子及花瓣中表达,具有显著的组织表达特异性,为进一步利用红花的脂质转运蛋白LTP提高红花的抗性研究奠定基础,以推动红花转基因育种的进程。下一步实验会将CtLTP基因构建植物表达载体并转化到植物中进行表达及功能验证,期望获得抗病抗逆能力增强的转基因植物;同时,亦可构建原核表达载体,获得纯化的LTP蛋白,在体外验证该蛋白的功能。
4 结论本研究成功克隆1个红花脂质转运蛋白LTP基因cDNA序列并进行生物信息学分析及组织特异性表达分析,结果表明,红花LTP基因在不同组织的表达水平具有显著性差异,在种子和花瓣中较高,而在其他组织中表达量较低。
| [1] | 田爱梅, 曹家树. 植物脂质转移蛋白[J]. 细胞生物学杂志, 2008, 30(4) : 483–488. |
| [2] | 李长生, 赵传志, 李爱芹, 等. 植物脂质转运蛋白的研究[J]. 山东农业科学, 2009(8) : 13–17. |
| [3] | Guo L, Yang HB, Zhang XY, et al. Lipid transfer protein 3 as a target of MYB96 mediates freezing and drought stress in Arabidopsis[J]. J Exp Bot, 2013, 64(6) : 1755–1767. |
| [4] | Jang CS, Kim DS, Bu SY, et al. Isolation and characterization of lipid transfer protein(LTP)genes from a wheat-rye trans-location line[J]. Plant Cell Rep, 2002, 20(10) : 961–966. |
| [5] | Debono A, Yeats TH, Rose J K, et al. Arabidopsis LTPG is aglycosyl phosphatidylinositol-anchored lipid transfer protein required for export of lipids to the plant surface[J]. Plant Cell, 2009, 21 : 1230–1238. |
| [6] | Cameron KD, Teece MA, Smart LB. Increased accumulation of cuticular wax and expression of lipid transfer protein in response to periodic drying events in leaves of tree tobacco[J]. Plant Physiol, 2006, 140 : 176–183. |
| [7] | Gaudet DA, Laroche A, Frick M, et al. Cold induced expression of plant defensin and lipid transfer protein transcripts in winter wheat[J]. Physiol Plantarum, 2003, 117(2) : 195–205. |
| [8] | Yubero SEM, Moyano E, Medina EN, et al. Identification of a straw- berry gene encoding a non-specific lipid transfer protein that responds to ABA,wounding and cold stress[J]. J Exp Bot, 2003, 54(389) : 1865–1877. |
| [9] | Wu GH, Robertson AJ, Liu XJ, et al. A lipid transfer protein gene BG-14 is differentially regulated by abiotic stress,ABA,anisomycin,and sphingosine in bromegrass(Bromus inermis)[J]. J Plant Physiol, 2004, 161(4) : 449–458. |
| [10] | Hwang EW, Kim KA, Park SC, et al. Expression profiles of hot pepper(Capsicum annuum)genes under cold stress conditions[J]. J Biosci, 2005, 30(5) : 657–667. |
| [11] | Oshino T, Abiko M, Saito R, et al. Premature progression of anther early developmental programs accompanied by comprehensive alterations in transcription during high- temperature injury in barley plants[J]. Mol Genet Genomics, 2007, 278(1) : 31–42. |
| [12] | Gorjanovi? S, Su?njevi? D, Beljanski M, et al. Barley lipid-transfer protein as heavy metal scavenger[J]. Environ Chem Lett, 2004, 2(3) : 113–116. |
| [13] | Maldonado AM, Doerner P, Dixon RA, et al. A putative lipid transfer protein involved in systemic resistance signalling in Arabidopsis[J]. Nature, 2002, 419 : 399–403. |
| [14] | Zhang D, Liang W, Yin C, et al. OsC6,encoding a lipid transfer protein,is required for postmeiotic anther development in ricer[J]. Plant Physiol, 2010, 154 : 149–162. |
| [15] | Liu X, Shangguan Y, Zhu J, et al. The rice OsLTP6 gene promoter directs anther-specific expression by a combination of positive and negative regulatory elements[J]. Planta, 2013, 238 : 845–857. |
| [16] | Choi AM, Lee SB, Cho SH, et al. Isolation and characterization of multiple abundant lipid transfer protein isoforms in developing sesame(Sesamum indicum L[J]. Plant Physiol Biochem, 2008, 46(2) : 127–139. |
| [17] | Liu KH, Lin TY. Cloning and characterization of two novel lipid transfer protein I genes in Vigna radiate[J]. DNA Seq, 2003, 14(6) : 420–426. |
| [18] | Jung HW, Kim KD, Hwang BK. Identification of pathogen-responsive regions in the promoter of a pepper lipid transfer protein gene(CALTPI)and the enhanced resistance of the CALTPI transgenic Arabidopsis against pathogen and environmental stresses[J]. Planta, 2005, 221(1) : 361–373. |
| [19] | Gonorazky AG, Regente MC, de la Canal L. Stress induction and antimicrobial properties of a lipid transfer protein in germinating sunflower seeds[J]. J Plant Physiol, 2005, 162(6) : 618–624. |
| [20] | Jang CS, Lee HJ, Chang SJ, et al. Expression and promoter analysis of the TaLTP1 gene induced by drought and salt stress in wheat(Triticum aestivum L[J]. Plant Sci, 2004, 167 : 995–1001. |
| [21] | 郑明. 植物非特异性脂转移蛋白基因的多抗性功能初步研究[D]. 长春:吉林大学,2009. |
| [22] | Li HY, Dong YY, Yang J, et al. De novo transcriptome of safflower and and the identification of putative genes for oleosin and the biosynthesis of flavonoids[J]. PLoS One, 2012, 7 : 1–10. |
| [23] | Van Loon LC, Van Strien EA. The families of pathogens is related proteins,their activities and comparative analysis of PR/L1 type proteins[J]. Physiol Mol Plant Pathol, 1999, 55 : 85–97. |
| [24] | Chae K, Gonong BJ, Kim SC, et al. A multifaceted study of stigma/style cysteine-rich adhesin(SCA)-like Arabidopsis lipid transfer protein(LTPs)suggests diversified roles for these LTPs in plant growth and reproduction[J]. J Exp Bot, 2010, 61 : 4277–4290. |
| [25] | Kirubakaran SI, Begum SM, Ulaganathan K, et al. Characterization of a new antifungul lipid transfer protein from wheat[J]. Plant Physiol Biochem, 2008, 46 : 918–927. |
| [26] | Gao S, Guo W, Feng W, et al. LTP3 contributes to disease susceptibility in Arabidopsis by enhancing abscisic acid(ABA)biosynthesis[J]. Mol Plant Pathol, 2015, 29. |
| [27] | Safi H, Saibi W, Alaoui MM, et al. A wheat lipid transfer protein (TdLTP4)promotes tolerance to abiotic and biotic stress in Arabidopsis thalianas[J]. Plant Physiol Biochem, 2015, 89 : 64–75. |
| [28] | Wang F, Zang XS, Kabir MR, et al. A wheat lipid transfer protein 3 could enhance the basal thermotolerance and oxidative stress resistance of Arabidopsis[J]. Gene, 2014, 550(1) : 18–26. |
| [29] | Pitzschke A, Datta S, Persak H. Salt stress in Arabidopsis: lipid transfer protein AZI1 and its control by mitogen-activated protein kinase MPK3[J]. Mol Plant, 2014, 7(4) : 722–738. |
| [30] | Fan Y, Du K, Gao Y, et al. Transformation of LTP gene into Bras-sica napus to enhance its resistance to Sclerotinia sclerotiorum[J]. Genetika, 2013, 49(4) : 439–447. |





