芸薹属植物自交不亲和反应(self-incompatibil-ity,SI)是沿着SCR(S cysteine-rich,S-富含半胱氨酸蛋白[1])→SRK(S receptor kinase,S-受体激酶[2])→ARC1(armadillo-repeat containing protein 1)→Exo70A1→MOD[3]的途径进行的。SRK作为柱头S基因,由胞外S结构域、跨膜结构域和激酶结构域的编码区构成,其同SCR识别的胞外S结构域编码区变异性较大[4-6],而SRK蛋白完整的胞外域包括B-Lection结构域,SLG结构域,PAN-APPLE结构域。在SI反应途径中,ARC1与Exo70A1的相互作用起着承上启下的关键作用。研究表明,ARC1的C端含有一个由6-7个臂重复区组成的ARM功能域,N端含有一个UND功能域,中间有一个U-box功能域[7]。ARC1的C端结构域是与SRK激酶域发生相互作用的区域[7],而U-box保守结构域是70多个氨基酸残基构成的UFD2(泛素融合蛋白质),其主要作为泛素连接酶-E3起作用,在泛素化系统中特异性识别底物蛋白质[8]。Exo70A1是SI信号传导途径中ARC1的下游底物,是Samuel等[9]运用酵母双杂交文库筛选技术从甘蓝型油菜的柱头细胞中分离出的一种新型SI信号传导元件。Exo70A1蛋白的C端含有一个典型的Exo70结构域[10]。油菜的Exo70A1基因含有12个外显子及11个内含子,研究表明Exo70Al可能是促进花粉水合、萌发及花粉管生长的亲和因子,在SI反应途径中被ARC1参与的泛素/26S蛋白酶体途径降解,从而导致柱头对自花花粉的拒绝[11]。
Stone等的研究表明[7],在ARC1和Exo70A1发生互作时,ARC1蛋白质N端的U-box是不可缺少的结构域;而杨佳等[11]的研究进一步表明羽衣甘蓝ARC1与Exo70A1相互作用区段是ARC1的N端(UND区段)和Exo70A1的N端(此蛋白质的1-270个氨基酸)。而目前根据S位点基因序列构建基因树做进化分析的文章也日益增多[12-14]。在这些研究的基础上,本研究以高度自交不亲和的结球甘蓝为研究对象,并利用其编码区序列分别对ARC1-Exo70A1互作区和非互作区编码DNA进行进化分析,以期为选育的甘蓝自交不亲和材料在整个植物中初步建立起进化位置和进化关系。
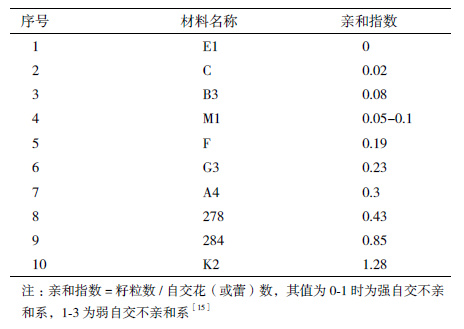
1 材料与方法 1.1 材料近年来选育的高度自交不亲和结球甘蓝(表 1),由西南大学园艺园林学院蔬菜实验室提供;大肠杆菌感受态DH5α、EasyPfu Taq DNA polymerase、胶回收试剂盒和TranScript First-strand cDNA Synthesis SuperMix、Mark Ⅲ(目录号:MD103)购自北京全式金生物技术有限公司;T4 DNA 连接酶、pMDTM19-T Vector、Sac I与Sma I购自TaKaRa大连公司;RNA提取试剂盒RNAprep Pure Plant Kit(目录号:DP432)购自天根生化科技(北京)有限公司;其他常规试剂均为国产分析纯;引物合成与序列测定由北京华大基因公司完成。
运用Primer Premier 6.0软件,依照Genbank中ARC1和Exo70A1已有的序列分别设计结球甘蓝ARC1引物和Exo-70A1引物。所选择的ARC1上游引物是(F-primer)5'-CCGGAATTCCCGATGGCCACTGATTCAGCAATGTTCGCAT-3',下游引物是(L-primer)5'-ACGCGTCGACGTTATCTCTGTGTTTTCTGGTCGCAC-3';Exo70A1上游引物是(F-primer)5'-GGGAACATATGTACATGGCCGTCGATAGCGGAAT-3',下游引物是(L-primer)5'-ACGCGTCGACCTTTACCGTCGTGGTTCATTCATAGACT-3'。
收集开花前1 d结球甘蓝花蕾的雌蕊,提取总RNA。以TranScript First-strand cDNA Synthesis Supe-rMix反转录合成进cDNA第一链;再选用引物对,以所得cDNA为模板,对应的扩增ARC1和Exo70A1基因。扩增体系均为50 μL:模板4 μL,2 mmol/L dNTPs 5 μL,上下游引物各2 μL,10×Easypruf Bu-ffer 5 μL,ddH2O 31 μL,TransTaq酶1 μL。PCR扩增程序参数为:94℃预变性2 min;94℃预变性30 s,退火30 s,72℃延伸90 s,35个循环;于72℃反应5 min后再4℃ 5 min,反应终止(T:ARC1为60℃,Exo70A1为62℃),反应结束后,置于4℃冰箱。PCR产物经1%(W/V)琼脂糖凝胶电泳,凝胶成像仪拍照,切胶,试剂盒回收纯化产物。胶回收产物(ARC1、Exo70A1)分别进行TA克隆,连接到pMDTM19-T载体;重组载体转化感受态细胞DH5α,LB固体培养基培养过夜,蓝白斑筛选阳性克隆子,挑取阳性克隆并经PCR鉴定后,分别抽提质粒pMDTM19-T-ARC1、pMDTM19-T-Exo70A1,抽提质粒送北京华大科技公司测序。将测序的结果进行拼接获取结球甘蓝材料的序列。
1.2.2 ARC1-Exo70A1互作区与非互作区编码DNA的确定不包括U-box区域的N端编码序列(UND结构域)是ARC1-Exo70A1的互作编码区段[8, 10, 11]。结合NCBI在线网站查询序列的结构域,利用MEGA5.05软件对ARC1序列和Exo70A1序列进行剪切,分别保留所需的互作编码区和非互作编码区于本地文件夹中。
1.2.3 结球甘蓝ARC1和Exo70A1序列生物信息学分析利用CLUATAL-W,Vector NTI Advance 10 进行多重序列比对分析;dnasp5软件进行序列多态性分析。采用MEGA5.05构建N-J模型进化树,仅在构建进化树时将the bootstrap probabilities此项参数修改为1 000,其他步骤均采用默认设置。
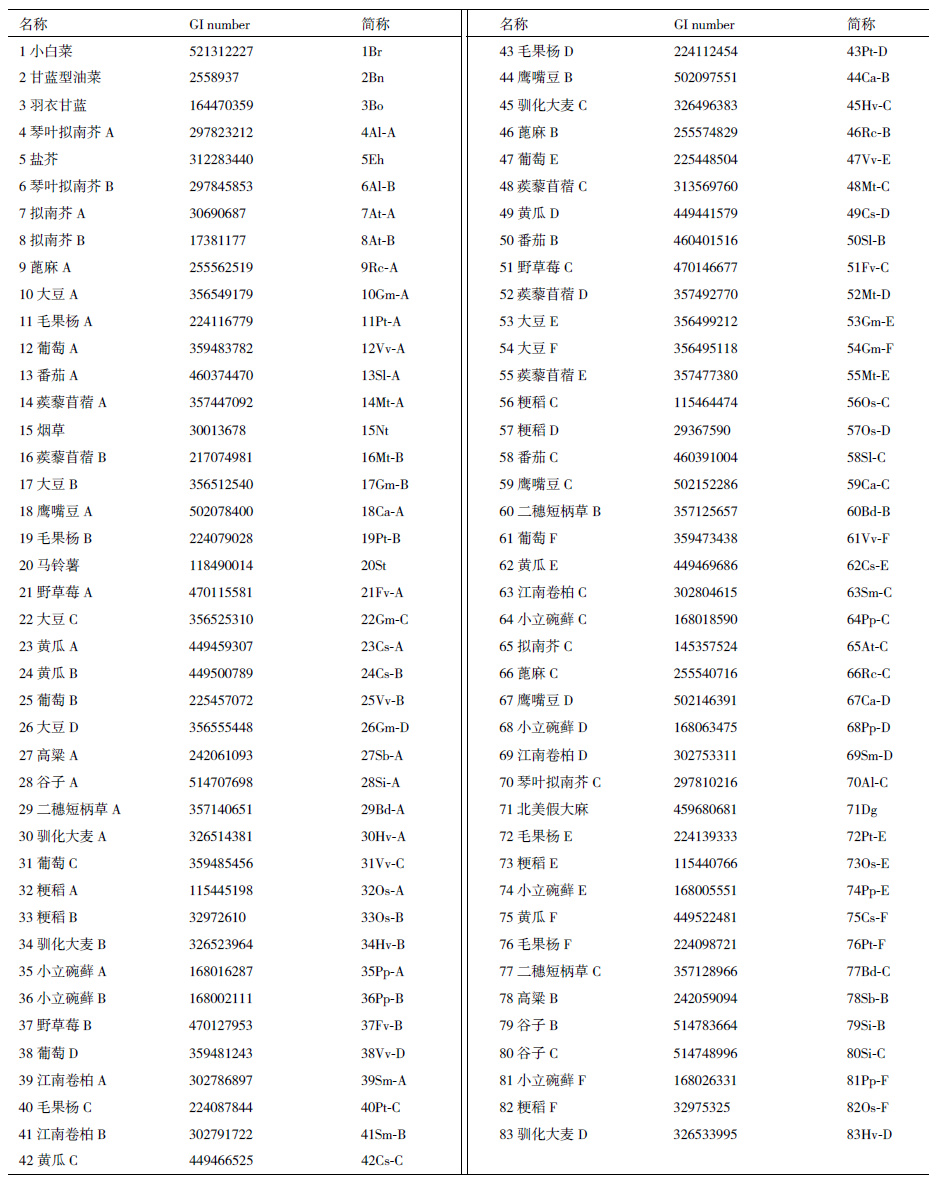
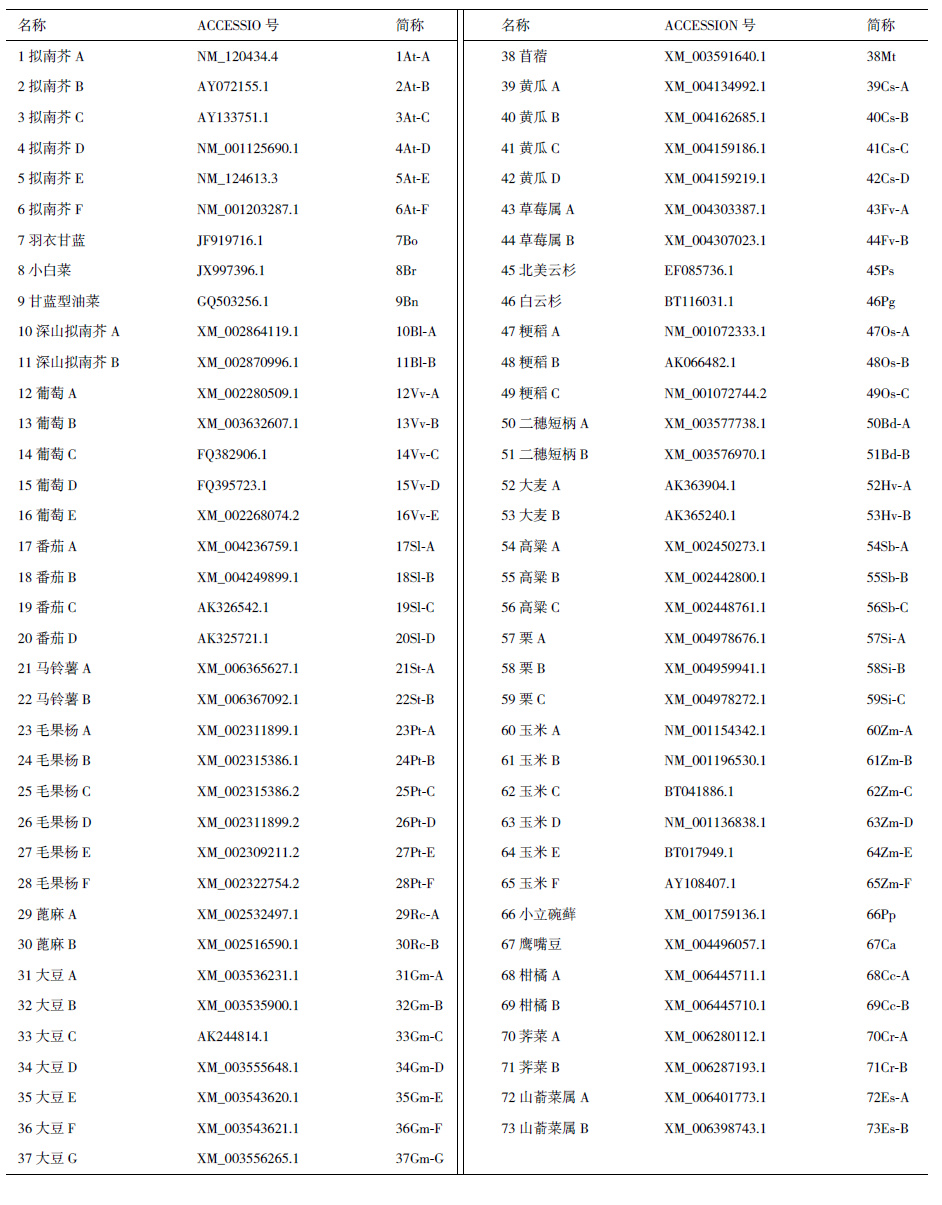
1.2.4 ARC1和Exo70A1相似序列的获取NCBI及Brassica Database(http://brassicadb.org/)等网站分别获取与结球甘蓝ARC1和Exo70A1核酸或氨基酸序列相似性(Similarity)大于70%的ARC1(表 2),Exo70A1核酸序列(表 3)。结球甘蓝所得材料序列命名为序列名称加基因名,如E1-ARC1,E1-Exo70A1。对于同一种属的不同序列在名称后加上大写字母A,B,C等加以区分,如序列1命名1Arabidopsis thaliana A,简称为1At-A。根据聚类情况来反映树的可信度。
提取RNA后,经反转录后获得10种材料的cDNA。以cDNA为模板,分别扩增获得相应10种材料的ARC1和Exo70A1的CDS(图 1);分别连接到pMDTM19-T载体进行测序,拼接后获得所需序列数据,经过Vector NTI分析可知,ARC1均为1 992 bp和 Exo70A1均为1 918 bp,与图 1中电泳结果条带约在2 000 bp处相符。

|
| 图 1 ARC1和Exo70A1基因PCR电泳图 M1,M2:Marker Ⅲ;1-10:ARC1基因;11-20:Exo70A1基因 |
10种材料的ARC1核酸序列相似度均高达97%以上,存在84个差异位点,氨基酸差异位点37个,且多为同义突变;10种材料的Exo70A1核酸序列相似度则均高于98%,有62个差异位点,氨基酸差异位点为23,也多为同义突变。图 2为ARC1与Exo70A1编码区核酸序列与氨基酸序列局部比对图(此部分为该序列差异位点最多的100 bp碱基区段),可以看出ARC1的核酸序列变异位点更多。综上说明在自交不亲和的结球甘蓝中ARC1和Exo70A1序列高度相似,且Exo70A1比ARC1更为保守。

|
| 图 2 局部序列比对图 |
在线网站共收集获得现有的83条ARC1和73条Exo70A1本身及类似序列(表 2,表 3)。ARC1序列来自21个不同种属,如芸薹属,拟南芥等;而Exo70A1则来自24个不同种属。两种基因的序列均可分为3大类:单子叶(水稻、小麦和玉米等),双子叶植物(甘蓝、白菜、拟南芥和大豆等)和低等植物(小立碗藓和江南卷柏),但是未收集到江南卷柏的Exo701序列。序列比对结果表明:83条ARC1核酸序列相似度在41%-100%之间,存在677个差异位点;73条Exo70A1核酸序列相似度在43%-100%之间且大部分高于ARC1的相似度,有529个差异位点。
2.2 ARC1-Exo70A1互作、非互作编码区DNA的确定在所获得的73条Exo70A1的CDS序列中,其C-端均含有一个保守的Exo70结构域。由文献[11]可知:用ARC1 N端的UND功能域与Exo70A1的1-85氨基酸或1-270氨基酸互作均可激活报告基因,但与1-270氨基酸互作时其相互作用强度更大。据此认为所获得的Exo70A1序列CDS的前840 bp为Exo70A1蛋白与ARC1蛋白的主要互作编码区。此外根据杨红等[5]研究结果可知:RING-finger type结构与U-box蛋白质相似,所以认为其RING-finger结构域功能类似于U-box结构域;在NCBI网站预测结球甘蓝ARC1结构域,可知其存在U-box结构域,且位于844-1 041 bp。
综上可知ARC1-Exo70A1的互作区段(图 3),对序列进行剪切。ARC1以U-box保守域的第一个上游核苷酸为剪切点,对于无U-box的序列以RING-finger结构域(表 2中编号为43、45、47、48、52、53、58和61)的第一个上游核苷酸为剪切点,获得93条互作区和非互作区ARC1序列;Exo70A1以序列840 bp处为剪切点,得到83条互作区和81条非互作区Exo70A1序列(其中编号为42和65的序列,因其非互作区不完整未使用)。
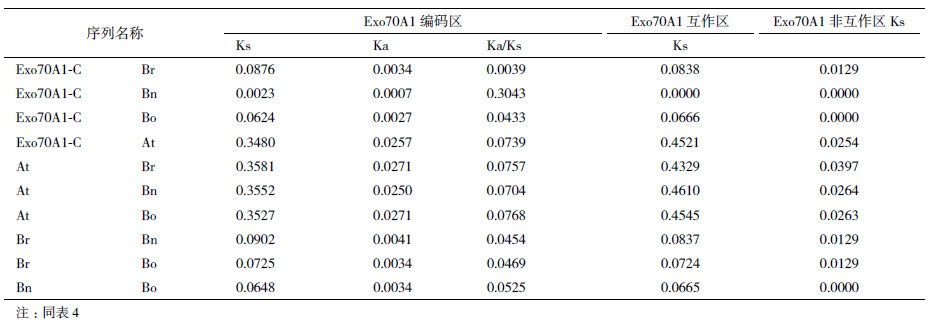
2.3 芸薹属与拟南芥属序列的相对进化速率分析10种材料结球甘蓝序列相似性极高,利用其共有序列(命名为ARC1-C、Exo70A-C)进行相关分析。芸薹属及拟南芥属序列进行多态性分析的结果,如表 4和5所示。
非同义突变率(Ka)与同义突变率(Ks)的比值是选择效应和选择方向的代表[16]。Ka/Ks大于1则说明基因受到正向选择影响,为快速进化基因;Ka/Ks等于1则说明同义突变与非同义突变频率相同,未受到选择的影响;Ka/Ks小于1则说明基因受负向选择影响,为相对保守基因。芸薹属与拟南芥属的ARC1和Exo70A1序列的Ka与Ks及其比值均小于1(表 4和表 5),说明在芸薹属与拟南芥属中,ARC1与Exo70A1这两个基因都受负向选择影响,是相对保守的基因。
比较不同物种同源或同类大分子的一级结构可以得出其相对进化速率[17],其公式为:
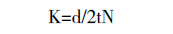
其中,K代表相对进化速率,2t代表进化时间,d代表替换的大分子数,N代表总的大分子数。
运用同义替换率估算分歧时间。序列间的同义突变(Ks)率如表 4和5所示,且已知芸薹属与拟南芥的分歧发生在200万年前(million years ago,MYA)[18],根据上述公式可以得出K值:ARC1中Bo与At的同义突变率为1.478 7,得出KARC1为0.037 0[12],同理可知KExo70A1为0.008 8,说明ARC1的相对进化速率比Exo70A1快,其保守性要低。而根据编码区内的互作区与非互作区不同区段序列的同义突变率可知,KARC1互作为0.043 1、KARC1非互作为0.031 1、KExo70A1互作为0.011 4、KExo70A1非互作为0.000 7;说明在ARC1-Exo70A1互作中,核酸序列互作区的进化速率比非互作区要快。
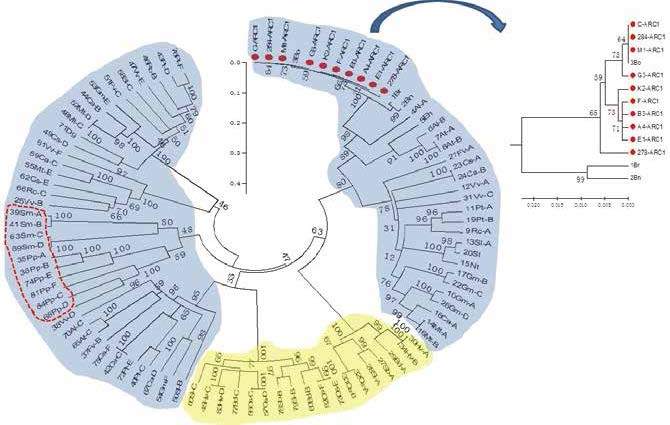
2.4 10种材料的ARC1与Exo70A1的平行进化关系进化树图 4-图 7中大部分同一种属的序列位置均较近,说明此进化树具有一定的可靠性。但个别同一种属的序列分布在不同位置,,这可能是因为基因分化先于了物种分化。
互作编码区ARC1进化树(图 4-A)中单子叶植物与双子叶植物均分为不同分支,而小立碗藓和江南卷柏这两个低等植物序列则在双子叶部分独立于一个分支下,说明ARC1的N端在进化过程中在双子叶植物与单子叶植物中已经发生了分化。结球甘蓝序列与羽衣甘蓝序列3聚类于同一树枝下,说明其亲缘关系很近。
Exo70A1互作区进化树(图 5-A)的分支较短、大部分聚类到一处,说明Exo70A1保守性较高。该进化树可大体分为3大部分:上部的双子叶分支,中间的单子叶分支,下部的双子叶分支。结球甘蓝序列与甘蓝型油菜序列9聚类到芸薹属的这一树枝下,与实际情况植物分类相符合。从进化树的分支情况可以看到芸薹属分支与拟南芥属分支位置临近,说明芸薹属与拟南芥属的Exo70A1的N端在进化中是很保守的,具有很近的亲缘关系。序列66即小立碗藓这一低等植物聚类于双子叶富集区。总体上来说Exo70A1在芸薹属植物的种间及属间高度保守。

|
| 图 5 互作区Exo70A1的进化树 注:图5-A为Exo70A1互作进化树整体;图5-B为Exo70A1互作区进化树芸薹属部分 |
根据ARC1互作区芸薹属部分放大图(图 4-B)可知:芸薹属序列均聚类到同一树枝下,说明芸薹属中ARC1的N-端保守性较好;拟南芥靠近,说明与拟南芥属的亲缘关系较为接近;芸薹属序列分为3部分,其中结球甘蓝测序序列也分为3部分,但未发现其分支情况与亲和指数之间的相关性。而观察Exo70A1互作区的芸薹属部分(图 5-B)可发现:序列9甘蓝型油菜与其他结球甘蓝序列聚类在一起,这与ARC1互作区的分支情况存在差异;材料278,E1与序列7羽衣甘蓝的亲缘关系更近,并与其他结球甘蓝材料聚类于不同分支下。
比较ARC1与Exo70A1互作编码区DNA的进化树,可发现3个主要的共同点:第一,单子叶植物与双子叶植物的序列均分别聚类于不同分支之下,说明ARC1与Exo70A1的互作编码区DNA均在单子叶植物与双子叶植物之间已经发生了分化;第二,芸薹属与拟南芥属的序列位置都十分靠近,说明ARC1与Exo70A1的互作编码区DNA在进化过程中至今仍然有着很近的亲缘关系;第三,芸薹属序列分支情况类似;第四,低等植物序列均聚类于双子叶富集区。综上说明ARC1与Exo70A1的互作区存在协同进化。
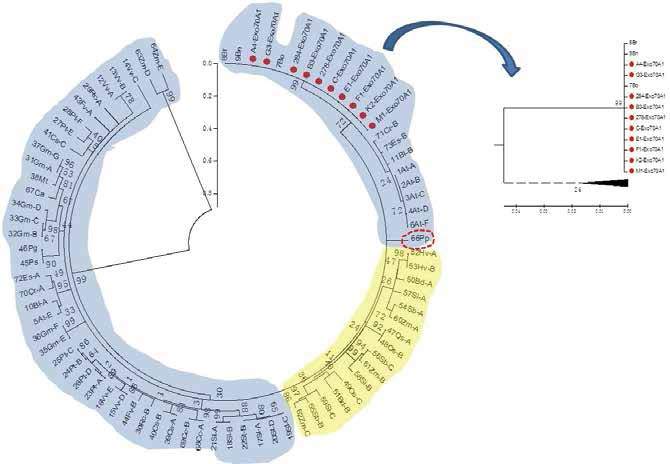
2.4.2 ARC1-Exo70A1非互作编码区DNA的进化分析在ARC1与Exo70A1非互作编码区进化树(图 6、图 7)中,不论ARC1还是Exo70A1,芸薹属的非互作编码区序列都在同一分支下;非互作区进化树中双子叶植物与单子叶植物也分别聚类于不同分支下,这与互作区进化树情况相同。

|
| 图 6 ARC1非互作区进化树 |
|
| 图 7 Exo70A1非互作区进化树 |
ARC1非互作编码区构建的进化树中(图 6):与芸薹属亲缘关系最近的是拟南芥(Arabidopsis),而大部分同一亚种的序列均聚类在同一分支下,表现出相同的亚种间同源性更高的情况;其芸薹属序列的分支情况与互作区部分相一致,而低等植物的序列则独立于一个分支下,说明ARC1非互作区序列差异性更大;分支长度短于互作区,即进化速率慢。Exo70A1非互作编码区构建的进化树中(图 7):整体分支情况类似于互作区;分支长度也短于互作区;芸薹属序列未出现亚枝,说明其保守性非常好。
非互作区进化树表明:芸薹属序列分支情况存在一定的差异性;其进化速率均低于互作区,与2.3中结果一致;低等植物的分支存在差异性。以上分析说明ARC1-Exo70A1互作中非互作区的DNA编码序列比互作区比较而言更为保守。
3 讨论本研究以自交不亲和结球甘蓝为研究材料,结合生物信息分析发现,结球甘蓝ARC1与其他芸薹属ARC1序列相似性极高;结球甘蓝Exo70A1与芸薹属Exo70A1序列也存在很高相似性及类似的结构域,由此可知结球甘蓝存在ARC1和Exo70A1的同源序列。
所选用的10种结球甘蓝的亲和指数在0-1.28之间,其ARC1和Exo70A1的核酸序列无明显差异存在,亲缘关系很近。说明在高度自交不亲和系的材料中ARC1和Exo70A1序列的保守性较好,具有保守的功能来维持自交不亲和信号通路。因此,在实际SI系选育过程中,对于亲和指数略高于1的品系不应立即舍弃,可多观察几代自交不亲和情况后再决定是否保留该品系。与芸薹属亲缘关系较近的拟南芥属中,拟南芥A. thaliana生态型均为自交亲和型,而琴叶拟南芥A. lyrata却有天然的自交不亲和型[20];但是在本文进化分析中发现ARC1和Exo70A1在芸薹属和拟南芥属中却具有很近的亲缘关系和较高的相似度,但与禾本科等单子叶植物则进化差异较大,考虑到自交不亲和机制出现在20 MYA之后,故可以推则ARC1在不同生态型拟南芥中对SI的贡献不同,ARC1在拟南芥A. thaliana中较完整,在拟南芥A. lyrata中则变异加大。在禾本科等单子叶植物中则变异更大,可能承担着其他功能。因此,所选育甘蓝品系的亲和指数高低可能反映着ARC1参与SI反应的强弱程度,而Exo70A1似乎没有这些差异。
本研究所用材料是经过多代人工选育所得,10个品种的不结球甘蓝因受人为因素控制,导致其变异趋同,如体现在10种材料的ARC1和Exo70A1的基因编码序列具有极高的相似度,说明在同一选择压下基因的变异及进化会向着类似的方向进行。在Vekemans等[14]文献中曾提到在环境条件的限制下,人工选育SI系,可能导致其自交亲和性的加强。如干旱等不利条件或有利于其向SC系过渡,而顺境中SI杂种优势可大大抵消其向SC系的转变。在逆境条件下,人工选育的SI系其S位点多态性偏少,这可能是材料更为集中,基因差异较低的原因之一。因此人工选育SI系过程中应该尽量保持良好环境,在获得SI系纯种的基础上尽量减少自交次数。本研究所用材料虽是不同来源的亲本,但其选择范围、选育条件等都可能在所测定的序列中起到了选择作用,从而被固定为某种变异序列,导致10种自交不亲和结球甘蓝其ARC1和Exo70A1序列相似度高,存在平行进化关系,均聚类于芸薹属分支下。这说明短时期的逆境选育并未造成ARC1和Exo70A1编码区DNA序列的改变,因此这种转向是生理性的。但是选育自交不亲和系最好在顺境条件下进行,因为只有在顺境条件下,SI所表现的异交优势,才不会被逆境掩盖,自交不亲和性也才能得以延续下去,也才具有选育SI系的遗传和生理基础。
ARC1和Exo70A1互作编码区存在协同进化与其功能相适应。本研究认为ARC1与Exo70A1蛋白质的相互作用可能促成了ARC1-Exo70A1互作编码区DNA的协同进化,即二者在进化过程中相互影响,从而具有相类似的进化方向。说明由于蛋白质之间的相互作用需要不变的构象,这可能在一定程度上约束了序列的变异程度,也就是说,当一方发生改变时,另一方也相应会发生改变,这样才能保持蛋白质之间仍旧存在互作所需的构象,最终形成协同进化这一趋势。这与Goh等[21, 22]提出的互作蛋白质通常有相似的进化关系且表现为互作区域有相似的进化树这一观点相切合。此外,根据相对进化速率分析得知ARC1比Exo701的进化速度快,这与进化树分析结果相一致,说明ARC1基因的变化性更大,其在植物中可能具有多样性的功能。
ARC1-Exo70A1互作编码区的变异速率比非互作编码区更快。首先,在上游的SCR-SRK互作中,SRK的胞外域(S domain)起了主要作用[4-6],然而S domain却具有多态性,其氨基酸序列表现出一定的差异[12]。根据Kitashiba的文章[23],可知不同胞外结构域的SRK属于不同S单倍型,且S单倍型的分化先于物种分化,那么SRK胞外结构域与SCR的进化都应受到单倍型约束,其分化也应该先于物种分化;对于上游的蛋白质而言,其可变性虽然较大,但还存在其他机制来保证反应的进行,如SLG(S-glycoprotein,S-糖蛋白[24])与同一单倍型SRK的S domain存在90%的氨基酸相似性[23],它与SRK之间可能存在一个机制以保证SI反应的发生。其次,在SRK-ARC1互作中ARC1的臂重复(ARM)区域(与Exo70A1的非互作区)是互作区,由本研究结果可知这个区段进化较慢,说明在SI途径中SRK-ARC1互作占了比较重要的位置,需要较为保守的结构域来维持反应的进行。最后,在下游互作蛋白ARC1-Exo70A1中互作区段序列也具有很大的可变性,说明其可能只是SI下游反应的一个分支,或存在其他机制来保障SI反应的顺利进行,而在杨佳等[10]的文章中提到破坏ARC1-Exo70A1途径并不能完全打破SI,认为SI信号网络可能存在其他的分支。此外,Exo70A1的Exo结构域(与ARCI的非互作区)其进化慢,可能是与其他蛋白质互作的区段,猜测其应具有极其重要的作用;而目前已经明确在酵母中Exo70结构域能够通过与Rho3 GTPase发生相互作用,来介导细胞的极性运输[25]。Vekemans等[14]认为SI向SC的过渡是由许多独立事件造成的,那么这些变异可能是分布于SI途经中SCR、SRK、ARC1等相关蛋白的。短期内,这些变异应该是体现为随机的序列变异,结合本研究,可知这些变异可能更加显著地积累于重要蛋白的互作区核酸序列。
本研究分析了SI信号传导途径中蛋白质ARC1和Exo-70A1,如果加入其他相关的互作蛋白进行分析,或许能发现SI途径相关蛋白质编码DNA的进化规律,以期为其功能研究及SI信号传导途径研究提供参考。而目前已经有许多软件可以通过分子测年的方法来构建系统发生树的时序图,那么下一步通过构建时序图便可获得节点分歧时间,进而得到序列在各种物种中的相对进化速率,从而定量的分析不同大分子(核酸,蛋白质等)的进化速率[19],来说明SI途径相关大分子在不同选择压力下的进化快慢,为SI的相关进化研究提供新的内容。
4 结论10种结球甘蓝自交不亲和材料之间存在平行进化关系;ARC1和Exo70A1在芸薹属和拟南芥属中是相对较保守基因;在整个植物中ARC1和Exo70A1互作编码区存在协同进化,但变异速率却比非互作编码区快。
| [1] | Schopfer CR, Nasrallah ME, Nasrallah JB. The male determinant of self-incompatibility in Brassica[J]. Science, 1999, 286(5445): 1697–1700. |
| [2] | Stein JC, Howler B, Boyes DC, et al. Molecular cloning of a putative receptor protein kinase gene encoded at the self-incompatibility locus of Brassica oleracea[J]. Proc Natl Acad Sci USA, 1991, 88(19): 8816–8820. |
| [3] | 吴志刚. 甘蓝自交不亲和相关基因MOD的克隆及其与ARC1的FISH定位研究[D]. 重庆:西南大学, 2011. |
| [4] | Shimosato H, Yokota NH, Iwano M, et al. Characterization of the SP11/SCR high-affinity binding site involved in self/nonself recognition in Brassica self-incompatibility[J]. Plant Cell, 2007, 19(1): 107–117. |
| [5] | 杨红, 朱利泉, 张贺翠, 等. 利用酵母双杂交系统鉴定甘蓝SCR与SRK胞外域片段间的相互作用[J]. 中国农业科学, 2011, 44(9): 1953–1962. |
| [6] | Tantikanjana T, Nasrallah ME, Nasrallah JB. Complex networks of self-incompatibility signaling in the Brassicaceae[J]. Current Opinion in Plant Biology, 2010, 13(5): 520–526. |
| [7] | Stone SL, Erson EM, Mullen RT, et al. ARC1 is an E3 ubiquitin ligase and promotes the ubiquitination of proteins during the rejection of self-incompatible Brassica pollen[J]. Plant Cell, 2003, 15(4): 885–898. |
| [8] | 杨昆, 张贺翠, RichardC, 等. 甘蓝自交不亲和信号转导元件ARC1与Exo70A1的相互作用[J]. 作物学报, 2011, 37(12): 2136–2144. |
| [9] | Samuel MA, Chong YT, Haasen KE, et al. Cellular pathways regulating responses to compatible and self-incompatible pollen in Brassica and Arabidopsis stigmas intersect at Exo70A1, a putative component of the exocyst complex[J]. Plant Cell, 2009, 21(7): 2655–2671. |
| [10] | 杨佳, 李玉花, 蓝兴国. 芸苔属植物自交不亲和性S-受体激酶的内吞作用及信号传递网络[J]. 植物生理学报, 2012, 48(3): 211–216. |
| [11] | 杨佳. 羽衣甘蓝ARC1相互作用蛋白及授粉过程中差异表达蛋白的研究[D]. 哈尔滨:东北林业大学, 2012. |
| [12] | Ryuichi I, Takeshi N. Phylogenetic analysis of Brassiceae based on the nucleotide sequences of the S-locus related gene, SLR1[J]. Theoretical and Applied Genetics, 2002, 105(8): 1159–1165. |
| [13] | Edh K, Widén B, Ceplitis A. The evolution and diversification of S-locus haplotypes in the Brassicaceae family[J]. Genetics, 2009, 181(7): 977–984. |
| [14] | Vekemans X, Poux C, Goubet PM, et al. The evolution of selfing from outcrossing ancestors in Brassicaceae:what have we learned from variation at the S-locus?[J]. Evol Biol, 2014, 27(7): 1372–1385. |
| [15] | 张桂玲. 甘蓝自交不亲和性快速鉴定方法的研究[D]. 哈尔滨:东北农业大学, 2003. |
| [16] | Nielsen R. Molecular signatures of natural selection[J]. Annual Review of Genetics, 2005, 39: 197–218. |
| [17] | 张昀. 生物进化[M]. 北京: 北京大学出版社, 1998.207. |
| [18] | Koch M, Haubold B, Mitchell-Olds T. Molecular systematic of the Brassicaceae:evidence from coding plastidic MatK and nuclear Chs sequences[J]. Am J Bot, 2001, 88(3): 534–544. |
| [19] | 黄原. 分子系统发生学(第1版)[M]. 北京: 科学出版社, 2012.1-21. |
| [20] | 郝艾馨, 蓝兴国, 王宇, 等. SRK-SCR转基因拟南芥自交不亲和性的研究进展[J]. 植物生理学报, 2013, 11: 1113–1120. |
| [21] | Goh CS, Bogan AA, Joachimiak M, et al. Co-evolution of proteins with their interaction partners[J]. Mol Biol, 2000, 299(2): 283–293. |
| [22] | Goh CS, Cohen FE. Co-evolutionary analysis reveals insights into protein-protein interactions[J]. Mol Biol, 2002, 1: 177–192. |
| [23] | Kitashiba H, Nasrallah JB. Self-incompatibility in Brassicaceae crops:lessons for interspecific incompatibility[J]. Breed Sci, 2014, 64(1): 23–37. |
| [24] | Watanabe M, Takasaki T, Toriyama K, et al. A high degree of homology exists between the protein encoded by SLG and the S receptor domain encoded by SRK in self-incompatible Brassica campestris L[J]. Plant Cell Physiol, 1994, 8: 1221–1229. |
| [25] | Robinson NG, Guo L, Imai J, et al. Rho3 of Saccharomyces cerevisiae, which regulates the actin cytoskeleton and exocytosis, is a GTPase which interacts with Myo2 and Exo70[J]. Mol Cell Biol, 1999, 19(5): 3580–3587. |