2. 天津北洋百川生物技术有限公司,天津 300457;
3. 天津现代职业技术学院生物工程学院,天津 300350;
4. 天津市轻工业化学研究所有限公司,天津 300350
2. Tianjin Peiyang Biotrans Biotech Co.,Ltd,Tianjin 300457;
3. School of Biotechnology,Tianjin Modern Vocational Technology College,Tianjin 300350;
4. Tianjin Research Institute of Light Industrial Chemistry,Tianjin 300350
聚苹果酸(poly malic acid,PMLA)属于聚酯类聚合物,是一种以苹果酸为唯一单体的均聚高分子聚合物。PMLA具有高度的水溶性、生物可吸收性、生物相容性、生物可降解性、可化学衍生性及无免疫原性等优良的性能的水溶性脂肪族聚酯,是一种具有广泛应用前景的功能高分子材料[1, 2]。出芽短梗霉相较于其他菌株合成的PMLA产率及分子量较高,故被广泛选为PMLA生产菌株。目前普遍认为微生物发酵积累 PMLA 是以 L-苹果酸为前体[3]。苹果酸的生物合成途径主要有4种,分别为:(1)丙酮酸途径;(2)6-磷酸葡萄糖途径;(3)TCA循环;(4)乙醛酸循环。然而对于哪一条途径是合成PMLA的前体物苹果酸的主要途径还没有一个明确的定论,程若东等[4]通过添加代谢抑制剂推断出芽短梗霉积累PMLA与TCA循环和乙醛酸途径有关。乔长晟等[5]通过对出芽短梗霉代谢通量及关键酶活性分析推断丙酮酸羧化途径和乙醛酸途径为 PMLA 的主要合成途径。
代谢物组学(metabolomics/metabonomics)是一种通过定性定量测定生物体代谢产物及种类的变化,分析系列关联代谢物的综合差异,从而发现生物系统对基因以及环境变化响应的研究方法。它是伴随基因组学和蛋白质组学的兴起而逐渐发展起来的又一门主要组学,完善了组学研究系统。通过在转录组、代谢组和蛋白组水平上整体分析环境干扰或基因,可以更全面地了解并分析某一生物或细胞全面而复杂系统的生理生化结构和机理[6, 7]。目前,代谢组学分析技术主要采用3种分析方法[8, 9]:基于核磁共振的分析方法[10];基于质谱的分析方法,通常情况下与色谱联用,如GC-MS、LC-MS、CE-MS等[11];基于光谱的方法,如傅立叶变换红外光谱、拉曼光谱等[12, 13]。其中,GC-MS技术因其有完整的标准图库,代谢物谱图可检索性较强,定性鉴定方便等优点,从而备受国内外代谢组学研究人员的青睐并被广泛应用与代谢组学的研究中。
微生物代谢物组学需要高效可靠的样品前处理,这样才能够确保胞内代谢物分析的准确性[14, 15]。由于实验菌株间具有差异性,若未进行条件优化直接使用文献报道方法进行处理,难免会对实验结果产生影响。为确保所用实验样品真实反映细胞的特定代谢特征,研究合理有效的细胞内代谢物的提取分离方法就显得尤为重要。目前常用的方法有,使用冷甲醇和冷乙醇等有机溶剂,瞬间停止细胞的代谢,再进一步用预冷的有机溶剂提取代谢物[16, 17]。本研究着重对比分析了细胞淬灭剂、提取溶剂和溶解剂对出芽短梗霉胞内代谢物的影响。从细胞淬灭、代谢物提取和代谢物溶解3个方面,对出芽短梗霉代谢物组学样品前处理方法进行优化,为准确分析出芽短梗霉代谢物组学奠定基础。
1 材料与方法 1.1 材料 1.1.1 菌种出芽短梗霉Aureobasidium pullulans CGMCC3337和Aureobasidium pullulans TKPM10017,天津科技大学生化工程研究室保藏。
1.1.2 主要试剂甲氧基铵盐酸盐(色谱纯)与吡啶(色谱纯);N-甲基 -N-(三甲基硅烷)三氟乙酰胺(色谱纯),购自SIGMA化学有限公司。色谱纯乙腈、甲醇、乙醇、四氢呋喃、二甲基甲酰胺、氯仿。实验用水为 Milli-Q超纯水。
1.1.3 仪器与设备GC/MS 气相色谱-质谱联用仪,配有7683 自动进样器与 6980 气相色谱仪(GC,Agilent Technologies,Palo Alto,CA,USA)。Milli-Q Advantage A10超纯水制备系统(美国Millipore公司)。
1.2 方法 1.2.1 不同淬灭方法比较取对数生长期后期的发酵液,离心(7000 r/min,5 min,4℃),弃上清液。然后用4℃的0.9%生理盐水洗菌体两次。分别取 5 mL 不同淬灭液[(1)100%甲醇(-40℃);(2)60%甲醇(-40℃);(3)60%乙醇(-40℃);(4)60%甲醇/0.9%氯化钠(-40℃)],快速加入装有沉淀的离心管中,用涡旋震荡器快速混匀,离心(10 000 r/min,5 min,-4℃),将上清液置于-80℃ 保存直到分析,标记为胞外样本。将淬灭后的细胞液氮研磨 20 min,至细粉状,称取 50 mg 于 1.5 mL 离心管中用于小分子代谢物的提取,每个取样点取 3个平行,液氮研磨后的样品加入1 mL 预冷的提取溶剂(70%的冷甲醇)反复冻融法提取,将上清液置于-80℃ 保存直到分析,标记为胞内样本。萃取液中加入10 μL 1 mg/mL的d4-丁二酸作为内标。萃取液冷冻干燥,储存在-20℃直到GC-MS分析。每个灭活方法做3个重复。
1.2.2 细胞完整性的测定淬灭处理后的细胞沉淀重悬浮于4 mL 1% PBS 缓冲液(8 g/L NaCl,0.2 g/L KCl,1.44 g/L Na2HPO4和 0.24 g/L KH2PO4),置于冰上。通过检测 OD600减少值评估细胞的裂解情况。OD回收率计算公式如下:
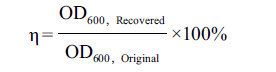
其中 OD600,Recovered为淬灭后菌体重悬浮的吸光度;OD600,Original为未经淬灭菌体重悬浮的吸光度。
1.2.3 灭活过程中代谢物渗出实验取10 mL菌体处于指数生长期的发酵液(OD620约为9.0),10 000 r/min,5 min,4℃离心得到菌体再使用0.9%氯化钠润洗细胞一次。将菌体重悬在2 mL不同灭活剂中,6 000 r/min,10 min,-4℃离心得到上清。将水洗后的细胞重悬在0.9%和2.3% NaCl中离心得到的上清作为阴性对照样品,将水洗后的细胞重悬在热0.9% NaCl(70℃,30 min)中离心得到的上清作为阳性对照样品。测定上述方法得到的上清在260 nm和280 nm的吸光度,分别代表胞内核酸和蛋白质的渗出量。同时测定了灭活后水洗液中的代谢物。
1.2.4 细胞破碎和胞内小分子提取取对数生长期后期的发酵液,离心(7 000 r/min,5 min,4℃),弃上清液。然后用 4℃ 的 0.9%生理盐水洗菌体两次。向清洗后的细胞中加入60%甲醇/0.9%氯化钠(-40℃),用涡旋震荡器快速混匀,离心(10 000 r/min,5 min,-4℃),-80℃保存备用。将淬灭后的细胞液氮研磨 20 min,至细粉状,称取 50 mg 于 1.5 mL 离心管中用于小分子代谢物的提取,每个取样点取 3 个平行,液氮研磨后的样品加入1 mL 预冷的不同提取溶剂[(1)100%甲醇;(2)60%乙醇;(3)60%甲醇;(4)甲醇/氯仿(1∶1,V/V)]反复冻融法提取,将上清液置于-80℃保存直到分析,标记为胞内样本。萃取液中加入10 μL,1 mg/mL的d4-丁二酸作为内标。萃取液冷冻干燥,储存在-20℃直到GC-MS分析。每个提取方法做3个重复。
1.2.5 不同溶解剂的比较将冷冻干燥后的样品,加50 μL甲氧基铵盐酸盐 / 不同溶解剂溶液(吡啶、乙腈、四氢呋喃、二甲基甲酰胺)(20 mg/mL),充分溶解样品,置于40℃水浴中,反应90 min;(2)加80 μL N-甲基 -N-(三甲基硅烷)三氟乙酰胺,混合均匀,置于40℃水浴中,反应90 min;(3)将衍生后的样品 10 000 r/min 离心 5 min;取上清液 100 μL加入进样瓶中,并编号,于室温放置 2 h,准备进行 GC-MS 检测。
1.2.6 GC/MS检测GC条件[18]:Agilent HP5 30 m×0.25 mm×0.25 μm毛 细 管 柱;进 样 口 温 度:280℃,升温程序:初始温度 70℃保持 2 min,以5℃/min 程序升温至 290℃保持 5 min;载气:高纯氦气,恒压模式 91 kPa,流速 1 mL/min;进样量1 μL;柱后分流比 10∶1。
MS 条件[18]:接口温度:280℃;电离方式:EI;电离方式:电子轰击电离(EI+);离子源温度:250℃;电子轰击能量:70 eV;电子电流:40 μA;扫描质量范围:50-800 m/z。
1.2.7 数据处理将 GC/MS 数据导入 MSD 增强型化学工作站,提取离子峰并用 NIST08 数据库进行比对进行定性,通过将所有的离子峰与内标物质的峰面积相比使数据归一化,再对数据进行中心化后导入 SIMCA 软件进行 PCA 分析[19, 20]。
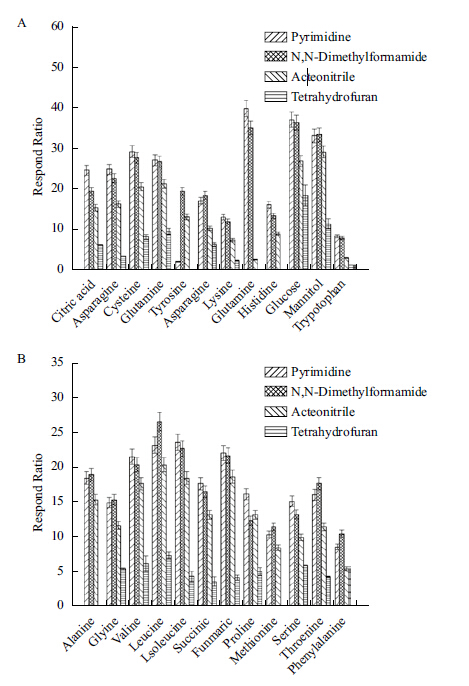
2 结果 2.1 不同溶解剂对代谢物溶解效率的影响出芽短梗霉胞内物质种类繁多,其中主要有氨基酸类,有机酸类,糖类等。因此,分析比较了4种不同的溶解剂吡啶、乙腈、四氢呋喃、二甲基甲酰胺对标准品溶液的溶解效果。运用GC-MS分析衍生后不同溶解剂溶解后的标品,比较不同溶解剂溶解后样品的出峰个数和峰面积。结果如图 1所示,不同溶解剂溶解后样品的出峰个数和峰面积有不同程度的差异,其中四氢呋喃和乙腈效果相对较差,其他两组效果较好,但二甲基二酰胺溶剂延迟较吡啶长,同时据文献报道吡啶能溶解大部分非极性和极性的化合物[21],因此在代谢组学文献中吡啶被广泛用于提取剂,综合以上原因选择吡啶为本实验的提取剂。
|
|
图 1 不同溶解剂对各种代谢物峰面积的影响
|
测定不同灭活溶液上清液中核酸和蛋白质的渗出情况,核酸和蛋白质分别在260 nm和280 nm处有特征吸收峰。此外,还测定了灭活后在水洗过程中代谢物的渗出情况。
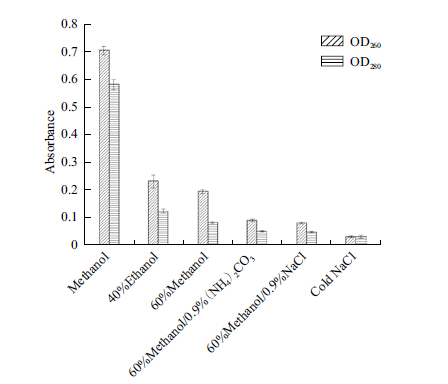
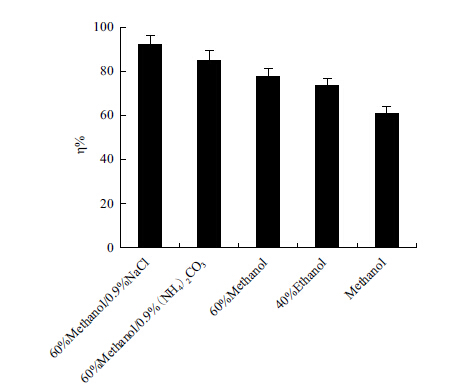
图 2为不同淬灭剂条件下核酸和蛋白质的渗漏情况,以间接反映代谢物的渗出情况。从图中可以看出,100%甲醇作为淬灭剂时蛋白质的渗出情况相对比较严重,证明其不适合作为淬灭剂使用;而采用其他3组淬灭剂使蛋白质的渗出得到大幅改善。其中甲醇和氯化钠组灭活后吸光度最低,表明代谢物渗出最少。同时利用监控淬灭处理后细胞 OD 值的减少来反映整体细胞水平的细胞损伤,OD 回收率越大表明细胞受到的损伤越小。由图 3可知,60%甲醇/0.9%氯化钠(-40℃)OD 回收率最大,结果表明低浓度的甲醇相对于高浓度的甲醇可以减轻细胞的损伤,同时加入盐溶液可以明显减轻细胞的损伤。
|
|
图 2 不同淬灭剂条件下胞内核酸和蛋白质的渗出分析
|
|
|
图 3 不同淬灭方法处理后的OD 回收率
|
综合上述两个实验结果表明使用有机溶剂对细胞灭活过程中有代谢物渗出,其原因可能是冷有机溶剂对细胞壁的冷冲击作用对细胞壁有一定的破坏性,同时有机溶剂可以在一定程度上改变细胞膜的流动性,使得胞内代谢物渗出胞外。60%甲醇/0.9% NaCl组相对于其他组分淬灭处理后的OD回收率最大,细胞完整性最好,并且代谢物渗出最少。综上所述,选择60%甲醇/0.9% NaCl为淬灭剂。
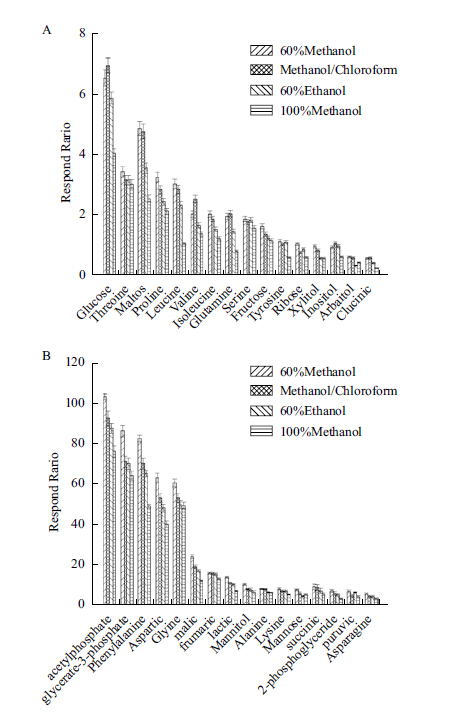
2.3 不同提取剂对代谢物提取效率的影响在已鉴定代谢物中随机选出部分氨基酸、有机酸、糖类等物质进行比较分析,结果(图 4)显示,70%甲醇和甲醇/氯仿(1∶1)两组提取效果较好,但由于氯仿具有一定的毒性,因此选择70%甲醇为提取剂。
|
|
图 4 不同提取方法处理得到的胞内代谢物水平
|
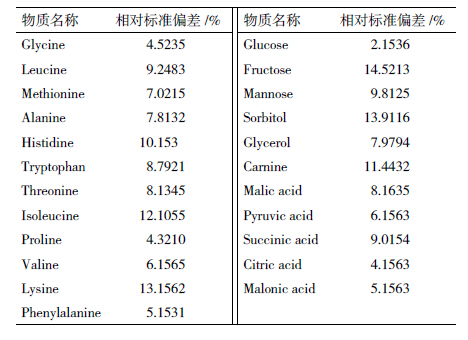
平行制备出样本4份,分别进行GC-MS检测。结果(表 1)显示,对4个平行样品进行相对定量计算后发现不同物质的相对含量的相对标准偏差(RSD)值均小于15%,因此该方法适用于代谢组学样品的检测和进一步分析。
出芽短梗霉作为聚苹果酸和普鲁兰多糖的生产菌株,传统的发酵优化手段已经无法满足现代工业的要求,对菌株的定向改造进一步提高产物产量降低副产物的产出的要求日益迫切。代谢组学作为一种系统的研究手段,在目的产物相关代谢物轮廓分析,代谢物合成途径的发现中具有无与伦比的优势。近年来,对于微生物代谢组学的研究飞速发展,但尚无出芽短梗霉代谢组学研究方法的文献报道,而现存的代谢物组学研究方法中没有一种可以满足所有微生物的研究。所以本研究对出芽短梗霉代谢物样品制备过程进行了系统优化。
预冷的有机溶剂在接触细胞的瞬间就可以使细胞失去活性,很好的满足了代谢组样品制备快速灭活的要求,但是有机溶剂对于细胞膜的破坏和对细胞渗透压的冲击使得胞内小分子代谢物容易渗出胞外,从而影响实验结果的准确性。本研究比较了4种不同的灭活剂,结果发现在有机溶剂中加入适合浓度的盐溶液可以很好的减少胞内代谢物的渗出,60%甲醇/0.9% NaCl组代谢物渗出最少并且灭活后细胞完整性最好,最终选择60%甲醇/0.9% NaCl为淬灭剂。代谢物组数据分析过程中,需要定性和定量尽可能多的物质来全面的描述代谢途径、代谢调节及描述机制。由于代谢物在不同的提取溶剂中的提取效率不同,本研究比较了 4 种提取溶剂的提取效率,结果发现用70%甲醇提取的胞内的代谢物质最全面,而且提取含量最高。
4 结论本研究以出芽短梗霉的代谢组为研究对象,比较了不同淬灭剂,提取剂和溶解剂条件下代谢物组的分析结果,最终确定了出芽短梗霉代谢物样品制备的最适方法:采用60%甲醇/0.9% NaCl溶液淬灭、乙腈∶甲醇∶水(2∶2∶1)提取胞内代谢物、含有甲氧基铵盐酸盐的吡啶溶液溶解冻干样品、最后使用N-甲基-N-(三甲基硅烷)三氟乙酰胺对样品进行衍生化。采用本方法从出芽短梗霉中共检测到300多种代谢物,经鉴定包括大量的氨基酸有机酸和糖类物质。
| [1] | Ding H, Portilla-Arias J, Patil R, et al. The optimization of polymalic acid peptide copolymers for endosomolytic drug delivery[J]. Biomaterials, 2011, 32(22): 5269–5278. |
| [2] | Ding H, Klibanov AM. Inhibition of brain tumor growth by intravenous poly(β-L-malic acid)nanobioconjugate with pH-dependent drug release[J]. Proceedings of the National Academy of Sciences, 2010, 107(42): 18143–18148. |
| [3] | Schmidt A, Windisch C, Holler E. Nuclear accumulation and homeostasis of the unusual polymer β-poly(L-malate)in plas modia of Physarum polycephalu[J]. European Journal of Cell Biology, 1996, 70(4): 373–380. |
| [4] | 程若东, 王浩, 周华, 等. 出芽短梗霉积累聚苹果酸途径及调控研究[J]. 化工学报, 2012, 63(11): 3639–3644. |
| [5] | 乔长晟, 郑志达, 孟迪, 等. 出芽短梗霉发酵生产聚苹果酸的代谢通量及关键酶活性分析[J]. 现代食品科技, 2014, 7(7): 74–80. |
| [6] | King RD, Whelan KE, Jones FM, et al. Functional genomic hypothesis generation and experimentation by a robot scientist[J]. Nature, 2004, 427(6971): 247–252. |
| [7] | Kitano H. Systems biology:A brief overview[J]. Science, 2002, 295(5560): 1662–1664. |
| [8] | Dunn WB, Broadhurst DI, Atherton HJ, et al. Systems level studies of mammalian metabolomes:the roles of mass spectrometry and nuclear magnetic resonance spectroscopy[J]. Chem Soc Rev, 2011, 40(1): 387–426. |
| [9] | 彭超, 黄和, 肖爱华, 等. 代谢组学分析技术平台及方法研究进展[J]. 食品科技, 2008, (9): 220–223. |
| [10] | Girjesh G. Metabonomics:A new frontier of nuclear magnetic resonance(NMR)[J]. National Academy Science Letters, 2004, 27: 289–299. |
| [11] | Detterm K, Aronov PA, Hammock BD. Mass spectrometry-based metabolomics[J]. Mass Spectrometry Review, 2007, 26(1): 51–78. |
| [12] | Lasch P, Boese M, Pacifico A, et al. FT-IR spectroscopic investigations of single cells on the sub cellular level[J]. Vibrational Spectroscopy, 2002, 28(1): 147–157. |
| [13] | Jürgen S, Michael B, Angelika B, et al. Identification of scrapie infection from blood serum by Fourier transforms infrared spectroscopy[J]. Analytical Chemistry, 2002, 74(15): 3865–3868. |
| [14] | Somerville GA, Proctor RA. At the crossroads of bacterial metabolism and virulence factor synthesis in staphylococci[J]. Microbiology and Molecular Biology Reviews, 2009, 73(2): 233–248. |
| [15] | Bolten CJ, Kiefer P, Letisse F, et al. Sampling for metabolome analy amino acid analysis in five phylogenetically different yeasts[J]. Analytical Chemistry, 2007, 79(10): 3843–3849. |
| [16] | Villas-Bôas SG, Højer-Pedersen J, Åkesson M, et al. Global metabolite analysis of yeast:evaluation of sample preparation methods[J]. Yeast, 2005, 22(14): 1155–1169. |
| [17] | Maharjan RP, Ferenci T. Global metabolite analysis:the influence of extraction methodology on metabolome profiles of Escherichia coli[J]. Analytical Biochemistry, 2003, 313(1): 145–154. |
| [18] | Bo T, Liu M, Zhong C, et al. Metabolomic analysis of antimicrobial metabolomics of epsilon-Poly-L-lysine on Saccharomyces cerevisiae[J]. Journal of Agricultural and Food Chemistry, 2014, 62(19): 4454–4465. |
| [19] | Fiehn O. Metabolomics-the link between genotypes and phenotypes[J]. Plant Molecular Biology, 2002, 48(1-2): 155–171. |
| [20] | 阿基业. 代谢组学数据处理方法——主成分分析[J]. 中国临床药理学与治疗学, 2010, (5): 481–489. |
| [21] | 李晓雪, 田锡炜, 王永红, 等. 拟干酪乳杆菌胞内代谢产物的GC—MS分析方法的建立[J]. 食品工业科技, 2013, (18): 169–173. |