肝细胞癌(hepatocellular carcinoma)是人类最常见的恶性肿瘤之一[1, 2]。MiRNA是一种内源性非编码RNA,长度在22个核苷酸左右,它主要是通过特异性结合靶基因3'非翻译区(3'UTR)抑制靶基因的翻译或者直接诱导靶基因的降解从而调控下游靶基因参与的生物进程[3]。研究表明miRNAs参与了由DNA损伤应答相关的多种细胞生物过程,包括细胞增殖、细胞周期调控和细胞凋亡等[4, 5]。Andrea等[6]发现,原癌基因miR-27a在人肺腺癌细胞A549 DNA损伤反应中起到重要的作用,其主要通过直接调控ATM基因进而抑制肺癌细胞的增殖。小RNA测序技术(small RNA sequencing)作为一种高通量的miRNA检测技术,目前广泛应用于miRNA表达谱的检测。本研究利用抗癌药物阿霉素处理肝癌细胞株HepG2,使细胞产生特异的DNA损伤,并运用小RNA测序技术检测阿霉素处理前后肝癌细胞株HepG2的miRNAs表达谱,筛选出DNA损伤前后差异性表达miRNAs,旨在为进一步研究肝细胞癌的DNA损伤应答机制提供理论依据。
1 材料与方法 1.1 材料人肝癌细胞HepG2由本实验室保管;阿霉素购自sigma公司;DMEM培养基购自Gibco公司;100 U/mL青霉素和100 U/mL链霉素以及胎牛血清购自Hyclone公司;Trizol Regent购自Invitrogen公司;测序由华大基因公司完成。
1.2 方法 1.2.1 细胞培养人肝癌细胞HepG2用含有10%胎牛血清(Hyclone),100 U/mL青霉素和100 U/mL链霉素(Hyclone)的DMEM培养基(Gibco)培养,并置于372饱和湿度的细胞培养箱中进行培养。取对数期5.0×106个细胞接种于60 mm培养皿中,待长至80%左右,加入0.2 µg/mL的阿霉素(北京华丰)处理HepG2细胞,24 h后收集细胞。
1.2.2 RNA提取及质量检测收集经阿霉素处理前后HepG2细胞,按TRIzol(Invitrogen)实验说明书抽提细胞总RNA。1%琼脂糖凝胶电泳和酶标仪(百泰克)鉴定RNA的纯度及完整性。
1.2.3 SmallRNA文库构建及测序 使用15%PAGE胶分离不同片段大小的RNA,回收18-30 nt的片段在T4 RNA连接酶作用下加5'和3'测序接头将产物反转录为双链并扩增。使用PAGE胶对PCR扩增产物切胶回收及纯化,回收产物溶于EB溶液中,完成文库建立。使用Agilent 2 100 Bioanalyzer和StepOnePlus Real-Time PCR System对构建好的small RNAs文库进行质量及产量评估。参照Illumima HiSeq 2 500测序仪说明书完成small RNAs文库测序。
1.2.4 SmallRNA测序数据分析 Illumina HiSeq 2 500测序得到长度为49 nt的序列,通过数据处理去除3'端缺失、5'端污染、插入片段缺失、含polyA等序列后获得高质量clean reads。对clean reads的长度、质量和分布等进行统计、分类和注释。通过bowtie将sRNA定位到基因组,分析reads在基因组上的分布及表达情况。使用bowtie将reads与miRBase数据库进行比对,注释已知miRNAs。通过bowtie将sRNA和GeneBank、Rfam数据库比对,注释除miRNA以外的sRNA,没有比对到注释信息的sRNA用unann表示。
1.2.5 差异表达miRNAs分析筛选实验组与对照组文库差异表达miRNA,将两个文库中的miRNA归一化处理后进行倍数变化值(fold change)和P值统计计算。筛选显著性差异表达miRNA的具体标准如下:(1)reads数大于10;(2)|log2(fold change)|>1;(3)P<0.05。
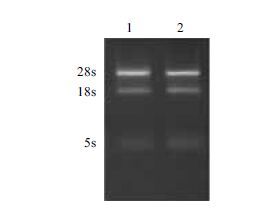
1.2.6 miRNAs靶基因预测、功能分析 2 结果 2.1 总RNA提取和检测使用0.2 µg/mL阿霉素处理HepG2细胞0 h和24 h后,TRIzol试剂抽提样本中总RNA,用1%琼脂糖凝胶电泳和酶标仪检测总RNA含量和质量。各组样本总RNA的OD260/OD280均在1.8-2.0之间,样品浓度在500-1 000 ng/µL之间。1%琼脂糖凝胶电泳结果(图 1)显示2个样品的28S和18S电泳条带清晰,灰度比大于1.9,质量合格,可用于后续测序实验。
|
| 图 1 总RNA 琼脂糖凝胶电泳图 1 :未处理HepG2 细胞株样本的总RNA 电泳条带;2 :阿霉素处理HepG2细胞株样本的总RNA 电泳条带 |
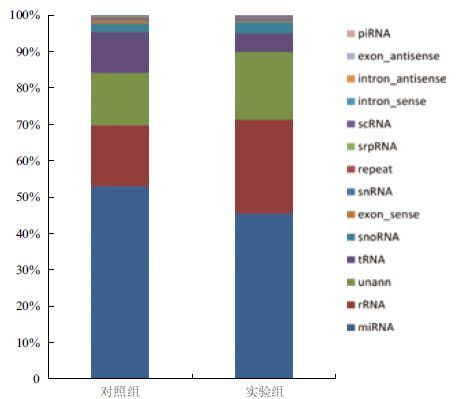
测序得到对照组和实验组总reads分别为11 519 591条和12 110 255条,高质量数据占总数的98.95%和97.96%。通过bowtie将sRNA与miRbase、Rfam和GenBank等数据库比对进行sRNA种类注释。正如预期,sRNA中miRNA占有最高的比例,对照组和实验组中miRNA占比分别为53.01%和45.51%(图 2)。对照组样本中检测到888种成熟miRNAs,实验组样本中检测到790种成熟的miRNA,两个样本中均表达的有679种(图 3)。
|
| 图 2 对照组和实验组样本中small RNA 占比柱状图 |

|
| 图 3 对照组与实验组样本miRNA 种类的比较 |
将阿霉素处理组和对照组miRNA的表达量丰度进行比较后共得到127个差异表达miRNA(P<0.05)(图 4)。通过进一步筛选得到显著差异表达miRNA 共68条(13条上调miRNA,55条下调miRNA)(表 1)。上调最为显著的是hsa-miR-449c-5p(上调50.14倍)。miR-17-92基因簇中的miR-19b、miR-20a、miR-18a、miR-92a-1在本实验中均呈现下调趋势。

|
| 图 4 差异表达miRNAs 散点图 |
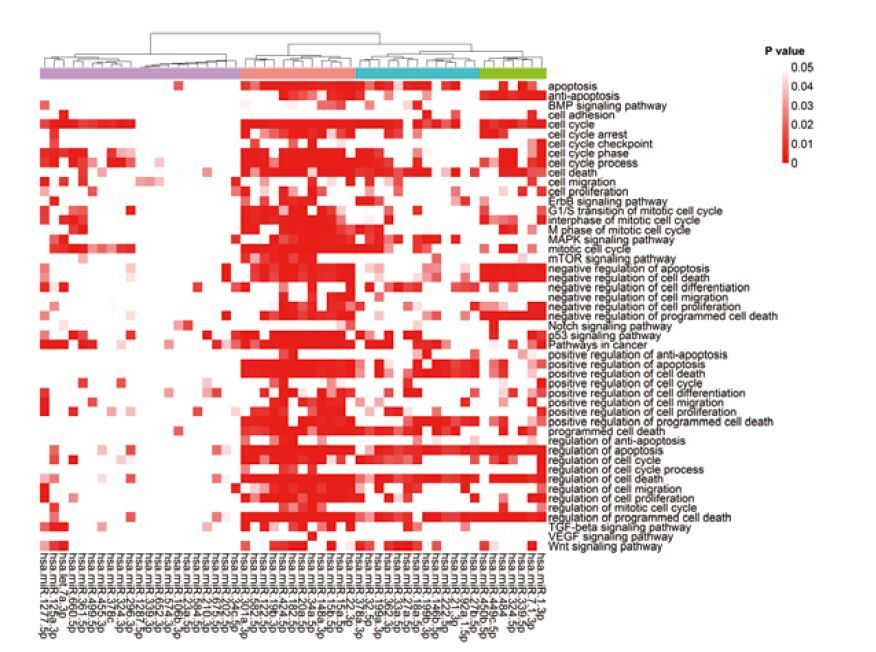
运用Targetscan、miRanda、miRDB和TarBase四种软件组合预测了差异表达miRNAs的靶基因(参照材料与方法部分)。通过GO富集分析和KEGG信号通路分析对每个表达差异的miRNA靶基因进行功能注释(P<0.05)。我们发现68条表达差异miRNAs中有53条miRNAs的靶基因显著参与细胞增殖、细胞凋亡、细胞周期调控、细胞迁移等与DNA损伤应答相关的生物进程和信号通路,如p53信号通路(最小P值=6.90E-09)、MAPK信号通路(最小P值=2.60E-05)、癌症中通路(pathways in cancer)(最小P值=5.10E-10)等(图 5)。其中hsa-miR-148a-3p(最小P值= 1.20E-08)、hsa-miR-122-5p(最小P值= 1.80E-06)、hsa-miR-182-5p(最小P值= 2.50E-11)、hsa-miR-19b-3p(最小P值= 3.70E-06)、hsa-miR-20a-5p(最小p值= 1.20E-11)高度富集于多个与DNA损伤相关的生物学进程和信号通路中(图 5)。
|
| 图 5 miRNA-靶基因参与癌症相关的生物过程和信号通路热图 |
本研究选用抗癌药物阿霉素诱导肝癌细胞株HepG2 DNA损伤,通过对处理前后HepG2细胞的sRNA进行高通量测序,得到与DNA损伤应答相关的差异表达miRNAs,并分析这些miRNA靶基因参与肝细胞癌DNA损伤应答相关的生物学进程与信号通路的情况。研究发现,miRNA介入多种与DNA损伤以及由DNA损伤诱发的细胞生物过程中,包括细胞增殖、细胞周期、细胞凋亡等,其在DNA损伤引起的肿瘤生物学进程中发挥重要功能[13, 14]。
本研究筛选出具有显著性差异表达miRNAs 68个,其中表达上调的13个,表达下调的55个。上调miRNAs中hsa-miR-122-5p、hsa-miR-1-3p等参与细胞凋亡、细胞增殖和侵袭等多个与DNA损伤相关的生物进程和信号通路。最新研究表明,在肝癌细胞系HepG2中过表达miR-122可以有效促进肝癌细胞凋亡[15];Xu等[16]也报道miR-122作为肿瘤抑制因子,通过负调控M2型丙酮酸激酶(PKM2)抑制肝癌细胞生长,这与本实验阿霉素诱导DNA损伤后激活p53相关信号通路并上调miR-122进而促进肝癌细胞凋亡和抑制肝癌细胞增殖的结果相吻合。另外,miR-1已被证实为肿瘤抑制因子,它通过下调恶性肿瘤发展相关的内皮素1(EF1)从而抑制肝癌细胞的增殖[17]。本实验中,我们检测到miR-27a-5p下调,Andrea等[6]发现,原癌基因miR-27a在人肺腺癌细胞A549的DNA损伤反应中起到重要的作用,研究结果表明DNA损伤会引起miR-27a的下调并间接促进miR-27a靶基因——抑癌基因ATM的表达进而抑制肺癌细胞的增殖和生存,本研究与之前的研究一致。我们的结果显示,miR-17-92基因簇的miR-18a-5p、miR-92a-1-5p、miR-20a-5p和miR-19b-3p 表达均下调。研究表明miR-17-92基因簇是极为重要的致癌基因,在肝癌中高表达[18]。Yan等[19]发现低氧环境导致的DNA损伤会诱导p53过表达进而下调原癌基因miR-17-92表达,抑制细胞的增殖并促进细胞凋亡过程。miR-17-92基因簇具有原癌基因的功能,参与调控细胞周期、促进细胞增殖、抑制细胞凋亡等生理进程,Zhu等[20]通过RT-PCR和原位杂交实验证实了miR-17-92基因簇在肝癌组织中高表达,阿霉素处理后会激活p53调控的凋亡通路,因此miR-17-92基因簇的下调会有效抑制肝癌细胞的增殖。综上所述,这些受肝细胞癌DNA损伤影响的差异表达miRNAs在阿霉素诱导的DNA损伤应答进程中起着重要的作用。
4 结论本研究分析了阿霉素诱导下肝细胞癌细胞系HepG2中miRNA差异表达情况,共检测出显著差异表达的miRNA 68条,其中上调13个,下调55个。MiRNA靶基因功能结果显示,53条miRNAs显著富集于与DNA损伤应答关联的生物进程和信号通路中,其中hsa-miR-148a-3p(最小P值= 1.20E-08)、hsa-miR-122-5p(最小P值= 1.80E-06)、hsa-miR-182-5p(最小P值= 2.50E-11)、hsa-miR-19b-3p(最小P值= 3.70E-06)、hsa-miR-20a-5p(最小P值= 1.20E-11)高度富集于多个DNA损伤应答相关的通路中,预示这些miRNA在肝细胞癌HepG2的DNA损伤应答过程中发挥着至关重要的作用。
| [1] | Waller LP, Deshpande V, Pyrsopoulos N. Hepatocellular carcinoma: A comprehensive review[J]. World J Hepatol, 2015, 7(26): 2648–2663. |
| [2] | Torre LA, Bray F, Siegel RL, et al. Global cancer statistics, 2012[J]. CA Cancer J Clin, 2015, 65(2): 87–108. |
| [3] | Bartel DP. MicroRNAs :target recognition and regulatory functions[J]. Cell, 2009, 136(2): 215–233. |
| [4] | Wouters MD, van Gent DC, et al. MicroRNAs, the DNA damage response and cancer[J]. Mutat Res, 2011, 717(1-2): 54–66. |
| [5] | Hu H, Gatti RA. MicroRNAs :new players in the DNA damage response[J]. J Mol Cell Biol, 2011, 3(3): 151–158. |
| [6] | Di Francesco A, De Pitta C, Moret F, et al. The DNA-damage response to gamma-radiation is affected by miR-27a in A549 Cells[J]. Int J Mol Sci, 2013, 14(9): 17881–17896. |
| [7] | Agarwal V, Bell GW, Nam JW, et al. Predicting effective microRNA target sites in mammalian mRNAs[J]. Elife, 2015, 4. |
| [8] | Betel D, Koppal A, Agius P, et al. Comprehensive modeling of microRNA targets predicts functional non-conserved and noncanonical sites[J]. Genome Biol, 2010, 11(8): R90.. |
| [9] | Wong N, Wang X. MiRDB :an online resource for microRNA target prediction and functional annotations[J]. Nucleic Acids Res, 2015, 43: D146–D152.. |
| [10] | Vlachos IS, Paraskevopoulou MD, Karagkouni D, et al. DIANATarBase v7[J]. Nucleic Acids Res, 2015, 43: D153–D159. |
| [11] | Huang DW, Sherman BT, Lempicki RA. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J]. Nat Protoc, 2009, 4(1): 44–57. |
| [12] | Huang DW, Sherman BT, Lempicki RA. Bioinformatics enrichment tools :paths toward the comprehensive functional analysis of large gene lists[J]. Nucleic Acids Res, 2009, 37(1): 1–13. |
| [13] | Wang Y, Taniguchi T. MicroRNAs and DNA damage response : implications for cancer therapy[J]. Cell Cycle, 2013, 12(1): 32–42. |
| [14] | Wan G, Mathur R, Hu X, et al. miRNA response to DNA damage[J]. Trends Biochem Sci, 2011, 36(9): 478–484. |
| [15] | Huang H, Zhu Y, Li S. MicroRNA-122 mimic transfection contributes to apoptosis in HepG2 cells[J]. Mol Med Rep, 2015, 12(5): 6918–6824. |
| [16] | Xu Q, Zhang M, et al. MicroRNA-122 affects cell aggressiveness and apoptosis by targeting PKM2 in human hepatocellular carcinoma[J]. Oncol Rep, 2015, 34(4): 2054–2064. |
| [17] | Li D, Yang P, Li H, et al. MicroRNA-1 inhibits proliferation of hepatocarcinoma cells by targeting endothelin-1[J]. Life Sci, 2012, 91(11-12): 440–447. |
| [18] | Yang H, Lan P, Hou Z, et al. Histone deacetylase inhibitor SAHA epigenetically regulates miR-17-92 cluster and MCM7 to upregulate MICA expression in hepatoma[J]. Br J Cancer, 2015, 112(1): 112–121. |
| [19] | Yan HL, Xue G, Mei Q, et al. Repression of the miR-17-92 cluster by p53 has an important function in hypoxia-induced apoptosis[J]. Embo J, 2009, 28(8): 2719–2732. |
| [20] | Zhu H, Han C, Wu T. MiR -17-92 cluster promotes hepatocarcinogenesis[J]. Carcinogenesis, 2015, 36(10): 1213–1222. |





