几丁质酶可以催化几丁质的水解,广泛存在于高等动植物、微生物以及某些病毒体内[1]。自Skujins等[2]首次发现来源于链霉菌(Streptomyces)的几丁质酶可以分解黑曲霉(Aspergillus niger)和腐皮镰刀菌(Fusarium solani)的细胞壁以来,利用几丁质酶来防治植物真菌病害已经成为最主要的应用研究领域。几丁质酶和产生几丁质酶的菌株除了可以单独用于植物病虫害防治以外,也可以通过和其他抗生素与杀虫剂共同使用来减少其他化学试剂的用量,进而减少对环境和人类健康的危害。已有研究表明将内切几丁质酶和苏云金芽孢杆菌的杀虫晶体蛋白同时使用可以将δ-毒素的杀虫效力提高近10倍[3, 4]。此外,由于几丁质降解所得到的几丁寡糖具有抗菌、调节免疫力和抗癌等功能,其在功能食品和保健药品等方面具有广阔的应用前景。例如,几丁六糖和几丁七糖已被证明具有良好的抗肿瘤活性[5, 6]。与其他方法相比,几丁质酶解产生几丁寡糖的方法具有反应专一性强,不会引起结构破坏,副反应少等优点。因此利用几丁质酶来生产几丁寡糖也成为几丁质酶的应用研究领域之一。
本研究以获得产几丁质酶的芽孢杆菌出发,从土壤中分离出7株菌株。经过比较分析后筛选出一株侧孢短芽孢杆菌,将其几丁质酶基因克隆表达于大肠杆菌中,并通过对该几丁质酶的酶学性质研究,旨在为几丁质酶制剂的开发和废弃几丁质资源的有效利用奠定基础。
1 材料与方法 1.1 材料 1.1.1 植物病原真菌尖孢镰刀菌(F. oxysporum ACCC209139),禾谷丝核菌(Rhizoctonia solani CICC40529)和腐皮镰刀菌(F. solani CICC2603),分别购于中国农业微生物菌种保藏管理中心(ACCC)和中国工业微生物菌种保藏中心(CICC)。
1.1.2 培养基分离纯化培养基(g/L):胶体几丁质 7.0,酵母提取物 2.0,KH2PO4 3.0,K2HPO4 2.0,NH4Cl 1.0,MgSO4·7H2O 0.2,琼脂 15。酶活测定培养基:分离纯化培养基不添加琼脂。细菌计数培养基:牛肉膏蛋白胨培养基。大肠杆菌培养基:LB培养基。病原真菌培养与拮抗能力检测培养基:PDA培养基。上述培养基[7]均于1.01×105 Pa灭菌30 min后使用。
1.2 方法 1.2.1 胶体几丁质的制备将30 g几丁质粉末(Sigma-Aldrich,USA)缓慢加入至300 mL预冷的浓盐酸中,磁力搅拌2 h后放入4℃静置24 h。然后将该混合液缓慢加入至2 L预冷的蒸馏水中搅拌均匀。5 000 ×g离心10 min收集白色的胶体几丁质沉淀,反复冲洗至pH约为6.5。最后用500 mL蒸馏水重悬,即获得浓度为6%的胶体几丁质母液。
1.2.2 菌株的筛选土壤样品为棉田和菜园土壤,采自于湖北武汉市周边。采集土壤样品后,取10.0 g土样加入90 mL无菌水中制备土壤悬液。经过80℃处理20 min后梯度稀释。将稀释液涂布在分离培养基上,28℃恒温培养。待菌落周围出现透明圈后反复划线分离,得到纯培养。
1.2.3 菌种的16SrRNA基因序列测定 将菌株接种至液体牛肉膏蛋白胨培养基中,培养至稳定生长期后使用TIANamp细菌基因组抽提试剂盒(Tiangen Biotech,China)抽提获得基因组DNA。采用通用引物27F和1492R进行16S rRNA基因的PCR扩增。PCR产物经过纯化后TA克隆至载体pMD18-T中测序。将测序所得序列提交至GenBank中。
1.2.4 几丁质酶活力的测定使用DNS法[8](3D-D-N-乙酰氨基葡萄糖的还原糖所需的酶量定义为1个酶活力单位。
1.2.5 拮抗病原真菌能力的测定采用平板对峙培养法[9]:分别将尖孢镰刀菌,禾谷丝核菌和腐皮镰刀菌培养2-3 d,然后从菌落边缘取下直径5 mm大小的菌饼置于新的PDA平板中央。使用打孔器取下滤纸片,然后将滤纸片贴至距中央3 cm处。定量滴加菌悬液(或酶溶液)后,置于30℃培养并观察抑菌情况。培养7 d后测量菌落半径和抑菌圈半径(x±s),分别用D和H来表示。根据抑菌圈半径和菌苔半径的比值大小判断菌株(或几丁质酶)的抑菌能力。
1.2.6 几丁质酶基因的克隆与异源表达根据侧孢短芽孢杆菌DSM25和GI9全基因组序列信息设计兼并引物,以菌株CDY64总基因组DNA为模板,PCR扩增得到除信号肽之外的ORF片段,引物序列见表 1。将得到的不含信号肽的ORF片段通过Sac Ⅰ和Xho Ⅰ酶切后,连接进入表达载体pET28a中。将酶连产物转化E. coli DH5α,转化子经过PCR以及酶切验证后,所得到的正确的重组质粒命名为pETChi72A。
将重组质粒pETChi72A转化进入E. coli BL21(DE3)中。筛选转化子后使用液体LB过夜培养获得种子液,然后以1%接种量接种至新的LB液体培养基中(含50 mg/mL硫酸卡那霉素),37600达到0.4时,加入终浓度为0.1 mmol/L的IPTG。然后28℃培养8 h诱导几丁质酶的表达。由于重组蛋白N端含有六聚组氨酸标签,因此使用Ni-琼脂糖蛋白纯化试剂盒(Tiandz,China)对异源表达的几丁质酶进行纯化。菌体的高压破碎、蛋白上样与洗脱均参照试剂盒手册进行。使用葡聚糖凝胶除盐后进行SDS-PAGE电泳,检测是否得到纯化的几丁质酶。所获得的几丁质酶溶液经过Bradford法[10]定量后用于酶学特性研究。
1.2.8 酶学性质的测定最适温度和pH值的测定:分别于20-80℃以及pH为3.0-10.0条件下测定Chi72A的活性,并以相对酶活的百分比为纵坐标,温度为横坐标进行作图。
热稳定性的测定:将Chi72A置于20-80℃下水浴3 h后检测酶活,并以相对酶活的百分比为纵坐标,以温度为横坐标进行作图。
酶促动力学参数的测定:在几丁质酶的最适反应条件下,使用纯酶液与不同浓度的胶体几丁质(1-25 mg/mL)反应,测定其酶促反应的初始速度。通过双倒数法作图确定Km和Vmax值。几丁质酶的催化常数由方程kcat = Vmax/[E]计算得出,[E]代表几丁质酶的浓度。
金属离子对几丁质酶酶活的影响:向酶促反应体系中分别加入Mg2+、K+、Ca2+、Fe2+、Fe3+和Cu2+共6种金属离子至终浓度为10 mmol/L。定量测定不同金属离子对Chi72A酶活的影响。
2 结果 2.1 产几丁质酶芽孢杆菌的分离使用平板计数法对不同土壤样品中可培养细菌总数进行测定后,发现不同土壤样品的可培养细菌总数介于0.8×108-3.3×108 cfu/g之间。与此同时,将不同土壤悬液80℃处理20 min后,涂布至以胶体几丁质为唯一碳源的筛选平板上。28℃培养3-7 d后,有多种细菌菌落产生水解圈。能分泌几丁质酶产生水解圈的菌落数量平均占可培养细菌总数的1.4%。本实验共筛选纯化得到7个不同菌株。
2.2 菌株产酶能力与拮抗病原真菌能力的测定将上述菌株接种至100 mL酶活测定培养基中,使用摇瓶发酵的方法分别在12、24、36和48 h取样测定不同菌株的产几丁质酶能力。来源于菜园土壤的菌株(Paenibacillus sp. W1,Paenibacillus sp. A14,Br. laterosporus CDY64和Brevibacillus sp. CDY118)平均酶活较高,其中CDY118酶活最高达到63.7 U/L;而来源于棉田的菌株(Paenibacillus sp. H316,B. aryabhattai H2-16和Brevibacillus sp. L2)平均酶活较低,菌株H316整个发酵周期内所测得的最高酶活仅为24.3 U/L。不同菌株的产几丁质酶能力如表 2所示(x±s)。
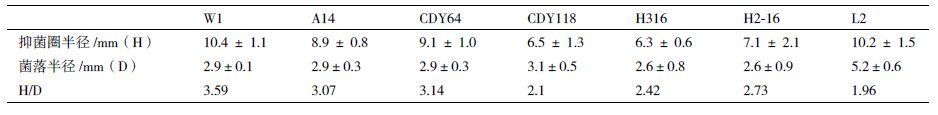
不同菌株拮抗尖孢镰刀菌的能力如表 3所示。所有被试菌株均表现出了对植物病原真菌的拮抗性能。
使用菌株CDY64的基因组DNA为模板,扩增获得全长的几丁质酶基因(chi72A,KT205398)。经过TA克隆后测序发现,chi72A全长为1 950 bp,所编码蛋白质分子量为72.23 kD。对该基因所编码蛋白质进行结构域分析后发现,该几丁质酶存在两个结构域,分别为N端催化结构域和C端的几丁质结合结构域。由于Chi72A的活性中心(F169EGIDIDYE177)与PROSITE数据库中第18糖苷酶家族活性中心的氨基酸序列([LIVMFY]-[DN]-G-[LIVMF]-[DN]-[LIVMF]-[DN]-x-E)一致,因此可以判断该几丁质酶属于第18糖苷酶家族。多序列比对结果(图 1)表明,Chi72A催化区域的氨基酸序列与侧孢短芽孢杆菌DSM25所编码的几丁质酶表现出了97.4%的相似性;与蜡状芽孢杆菌ATCC4342 的几丁质酶ChiC,环状芽孢杆菌WL-12的几丁质酶ChiA1以及粘质沙雷氏菌2170的几丁质酶ChiA的序列相似性分别为49%、51%和40%。

|
| 图 1 几丁质酶Chi72A催化区域的多序列比对图谱 所选取几丁质酶的氨基酸序列为来自于Br. laterosporus DSM25(DSM25,WP_003336146.1),B. cereus(ChiC,EEK85987.1),B. circulans(ChiA1,AAA81528.1)和S. marcescens(ChiA,BAA31567.1)的几丁质酶。保守氨基酸残基用深色背景表示。图中数字表示每行第一个氨基酸在肽链上的位置 |
此外,多序列比对还发现,Chi72A的几丁质结合结构域除了与其他几种细菌几丁质酶表现出序列相似性以外,还与链霉菌所编码的纤维素结合结构域表现出了较高的序列相似性。
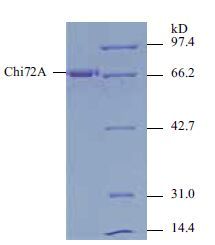
2.4 异源表达几丁质酶的纯化为了进一步研究Chi72A的酶学特性与影响因素,将除信号肽以外的ORF序列酶切后连接进入表达载体pET28a中。重组菌株使用0.1 mmol/L IPTG,28℃诱导8 h后,使用高压破碎仪进行破碎。然后12 000 ×g离心10 min取上清。最后使用Ni-琼脂糖和葡聚糖凝胶进行分离纯化。所获得纯品几丁质酶的检测结果如图 2所示。
|
| 图 2 Chi72A纯化后的SDS-PAGE电泳检测图谱 1:纯化后的Chi72A;M:蛋白质Marker |
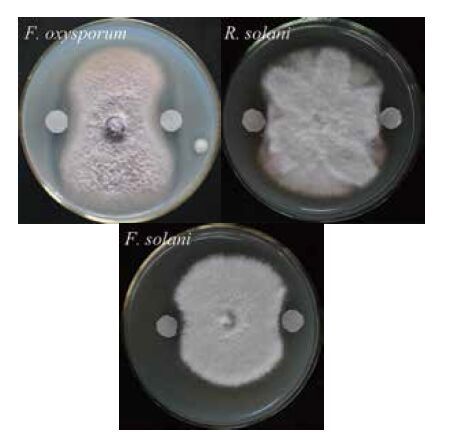
将纯化后的几丁质酶溶液50 μL缓慢滴加至滤纸片上,30℃培养3 d后观察抑菌情况。结果(图 3)显示,该几丁质酶除对尖孢镰刀菌表现出了很强的拮抗能力以外,还显著抑制了禾谷丝核菌以及腐皮镰刀菌的生长。这也进一步证实,该几丁质酶为菌株CDY64产生真菌拮抗能力的重要原因之一。
|
| 图 3 Chi72A对植物病原真菌拮抗作用的示意图 |
温度与pH值对纯化后Chi72A的影响如图 4所示。Chi72A在pH为6.0-8.0的范围内比较稳定;当反应缓冲液pH分别为3.0与10.0时,Chi72A酶活力分别下降至最高值的15%和27%(图 4-A)。图 4-B表明,Chi72A的最适反应温度为60℃。当温度为20℃时,Chi72A的酶活为最高值的23%;而当反应温度为80℃时,Chi72A酶活仅为最高值的18%。为了进一步研究Chi72A对高温的耐受能力,将酶溶液分别置于20-80℃预处理3 h,然后在60℃以及pH=8.0的条件下测定酶溶液的残余酶活力。图 4-C表明,Chi72A在50℃条件下处理3 h后,其酶活力没有受到显著影响。而当预处理温度提高至60℃时,其酶活力仅下降9%,具有较强的热稳定性。

|
| 图 4 Chi72A的酶学特性研究 A:pH值变化对Chi72A酶活力的影响;B:反应温度对Chi72A酶活力的影响;C:Chi72A对温度的耐受性研究 |
金属离子往往有助于维持酶的三维结构,甚至直接参与酶的催化过程。因此,本研究分别检测了Mg2+、K+、Ca2+、Fe2+、Fe3+和Cu2+,6种金属离子对纯化后Chi72A酶活力的影响。结果(表 4)显示,当向反应液中添加CaCl2至终浓度为10 mmol/L时,Chi72A催化活性升高了10.5%;Fe3+和Cu2+对Chi72A表现出了抑制能力;尤其是添加Cu2+至终浓度为10 mmol/L时,Chi72A的活性下降了49%;而Mg2+、K+和Fe2+对Chi72A的酶活力没有显著影响。
分别用不同浓度的胶体几丁质(1-25 mg/mL),在最适反应条件下对纯化后Chi72A的起始反应速度进行测定。以底物浓度的倒数为横坐标,酶促反应速度的倒数为纵坐标作图,获得酶促动力学参数。结果(图 5)表明,Chi72A的Km值为5.85 μmol/L,而其催化常数,kcat为29.27 S-1(即每秒钟可以产生相当于29.27个N-乙酰氨基葡萄糖的还原性末端)。

|
| 图 5 Chi72A酶促反应的Lineweaver-Burk图谱 |
具有几丁质降解能力的细菌在自然界碳/氮循环过程中具有非常重要的作用。此类菌株以及相关降解基因的分离也有助于几丁质酶在工农业上的应用。本实验所筛选菌株以胶体几丁质作为底物时,培养液中的几丁质酶的酶活力介于24.3 U/L和63.7 U/L之间,这与Trivedi等[11]所筛选的菌株酶活相似。由于侧孢短芽孢杆菌在微生物肥料安全通用技术准则(NY1009-2006)和农业部发布的《饲料添加剂目录(2013)》中均属于生物安全菌种,同时侧孢短芽孢杆菌 CDY64又具有较高的产几丁质酶能力以及拮抗病原真菌的能力,因此本研究以CDY64为目的菌株,对其所编码的几丁质酶展开进一步研究。
侧孢短芽孢杆菌往往存在于多种水体、土壤或某些昆虫的体表,能够产生多种与杀虫和拮抗病原真菌有关的蛋白质和抗生素[12]。然而,侧孢短芽孢杆菌中几丁质酶的酶学性质和酶促动力学参数尚未有报道。大多数细菌所产生的几丁质酶属于第18糖苷酶家族,而第18糖苷酶家族的几丁质酶又可以分为A、B和C三个亚类[13]。根据氨基酸序列相似性,Chi72A可归类于A亚类。酶学特性研究表明Chi72A的最适反应pH范围为6.0-8.0,与地衣芽孢杆菌(B. licheniformis)SK-1[14]所编码的几丁质酶类似。迄今为止,大多数几丁质酶的最适反应pH值介于4.0-10.0之间,只有少部分几丁质酶的最佳pH超出这一范围。例如,Pradeep等[15]发现一株链霉菌所产几丁质酶的最适pH值为12.5。由于高温酶促催化过程有助于降低污染风险以及提高酶促反应的速率,因此很多工业上的酶促反应是在高温下进行的。本研究中所涉及的几丁质酶Chi72A具有良好的热稳定性,这将有助于该酶在几丁质废弃物生物转化方面的推广与应用。
4 结论本研究在不同土壤样品中共分离得到7株不同的产几丁质酶菌株,16S rRNA序列分析发现,这些菌株可归类于类芽孢杆菌、短芽孢杆菌和芽孢杆菌3个属。酶学研究表明,侧孢芽孢杆菌CDY64所编码的几丁质酶(Chi72A)具有良好的热稳定性,其最佳反应温度为60℃;而且在50℃条件下预处理3 h没有造成显著的酶活丧失。此外,该几丁质酶在体外对多种植物病原真菌具有拮抗作用。
| [1] | Morimoto K, Karita S, Kimure T, et al. Cloning sequencing and expression of the gene encoding Clostrium paraputrificum chitinase ChiB and analysis of the functions of novel cadherin-like domains and a chitin-binding domain[J]. Journal of Bcteriology, 1997, 179: 7306–7314. |
| [2] | Manjeet K, Purushotham P, Neeraja C, et al. Bacterial chitin binding proteins show differential substrate binding and synergy with chitinases[J]. Micorbiological Reaearch, 2013, 168: 461–468. |
| [3] | Ding X, Luo Z, Gao B, et al. Improving the insecticidal activity by expression a recombinant cry1A gene with chitinase-encoding gene in a crystalliferous Bacillus thuringiensis[J]. Current Microbiology, 2008, 56: 442–446. |
| [4] | Regev A, Keller M, Strizhov N, et al. Synergistic activity of a Bacillus thuringensis delta-endotoxin and a bacterial endochitinase against Spodoptera littoralis larvae[J]. Applied Environmental Microbiology, 1996, 62: 3581–3586. |
| [5] | Nanjo F, Sakai K, Ishikawa M, et al. Properties and transglycosylation reaction of a chitinase from Nocardia orientalis[J]. Agricultural and Biological Chemistry, 1989, 53: 2189–2196. |
| [6] | Usui T, Matsui H, Isobe K. Enzymic synthesis of chitooligosaccharides utilizing transglycosylation by chitinolytic enzymes in a buffer containing ammonium sulfate[J]. Carbohydrate Research, 1990, 203: 65–77. |
| [7] | 赵斌, 何绍江. 微生物学实验[M]. 北京: 科学出版社, 2002.85-88. |
| [8] | Miller GL. Use of dinitrisalicylic acid reagent for determination of reducing sugar[J]. Analytical Chemistry, 1959, 31: 426–428. |
| [9] | Roberts WK, Selitrennikoff CP. Isolation and partial characterization of two antifungal proteins from barley[J]. Biochimica et Biophysica acta, 1986, 880: 161–170. |
| [10] | Bradford MM. A rapid and sensitive method for the quantization of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72: 248–254. |
| [11] | Trivedi P, Spann T, Wang N. Isolation and characterization of beneficial bacteria associated with Citrus roots in Florida[J]. Microbial Ecology, 2011, 62: 324–336. |
| [12] | Ruiu L. Brevibacillus laterosporus,a pathogen of invertebrates and a broad-spectrum antimicrobial species[J]. Insects, 2013, 4: 476–492. |
| [13] | Terahara T, Ikeda S, Noritake C, et al. Molecular diversity of bacterial chitinase in arable soils and the effects of environmental factors on the chitinolytic bacterial community[J]. Soil Biology and Biochemistry, 2009, 41: 473–480. |
| [14] | Kudan S, Pichyanqkura R. Purification and characterization of thermostable chitinase from Bacillus licheniformis SK-1[J]. Applied Biochemistry and Biotechnology, 2009, 157: 23–35. |
| [15] | Pradeep GC, Choi YH, Choi YS, et al. An extremely alkaline novel chitinase from Streptomyces sp[J]. Process Biochemistry, 2014, 49: 223–229. |