2. 武汉中科光谷绿色生物技术有限公司,武汉 430075
2. Wuhan Zhongke Optics Valley Green Biotechnology Co., Ltd., Wuhan 430075
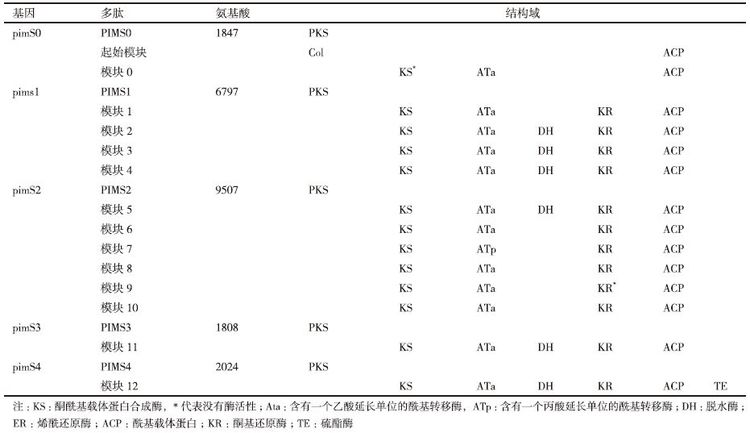
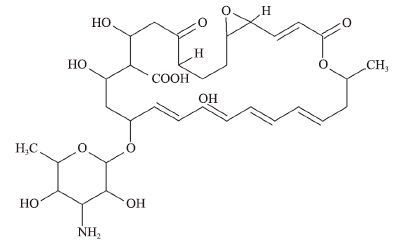
纳他霉素是一种多烯类大环内酯抗生素(图 1),是一种抗菌范围广、效率高的真菌抑制剂,主要由恰塔努加链霉菌(Streptomyces chmanovgensis)、褐黄孢链霉菌(Streptomyces gilvosporeus)和纳塔尔链霉菌(Streptoyces natalensis)等发酵合成[1]。它不仅能够与膜上麦角固醇发生特异性作用来抑制真菌的生长[2],还能够有效地抑制黄曲霉毒素的形成。由于对动物细胞毒性较低[3],广泛应用于食品防腐剂和真菌角膜炎的治疗等领域[4]。
|
| 图 1 纳他霉素的分子结构 |
纳他霉素主要在纳塔尔链霉菌生长的稳定期中合成。在此期间,若某种营养素缺乏或受到其他环境信号的刺激,特异性调节子和总体调节子会被激活,从而通过级联反应或总体交互作用来激活纳他霉素合成基因簇的表达[5]。
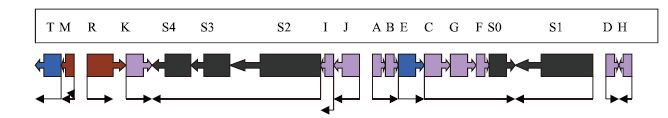
1 纳他霉素在纳塔尔链霉菌中的合成 1.1 参与纳他霉素合成的基因簇2000年,Aparicio等[6]已经基本完成了对参与纳他霉素合成的基因测序,目前已知该序列共有19个基因,横跨约100 kb。在纳他霉素的合成过程中,首先由pimS0-pimS4编码的Ⅰ类聚酮合成酶(polyketidesynthase,PKS)合成纳他霉素骨架环;再由pimC、pim G、pimD、pimJ、pimK和pimⅠ编码的附加蛋白逐步对纳他霉素骨架环进行加工修饰;最后由pimA、pimB和pimH编码的蛋白将完整的纳他霉素转运出细胞;而pimE、pimM和pimR则编码转录调节子,负责调控纳他霉素合成基因的表达(图 2)。
纳他霉素大环内酯环的合成是由Ⅰ类PKS催化的,PKS包括13个同源的模块,这些模块分布在5个由pimS0-pimS4编码的大的多功能酶PIMS0-PIMS4中。如表 1所示,PIMS0由起始模块和模块0组成;PIMS1包括4个模块,模块1-4;PIMS2包括模块5-10;PIMS3和PIMS4分别由模块11和12构成。骨架环的合成由PIMS0催化起始,它的结构为CoA连接酶-ACP-KS-AT-ACP。合成开始时,CoA连接酶结构域催化乙酸盐形成酰基腺苷酸,再由ACP结构域将其转移到相邻的KS结构域,KS会将生长的聚酮化合物链转移到C端ACP上,为PimS1模块1的KS提供乙酰单位,作为延长的第一步[7]。
合成起始后,PIMS1的4个模块分别完成骨架链延长的前4个循环,并构成大部分的多烯发色团。当4个延伸步骤完成后,骨架链将转移到PIMS2上继续合成,PIMS2、PIMS3、PIMS4的8个模块依次催化8个羧酸基本成分的结合,并对其进行修饰,产生纳他霉素骨架环。其中PIMS2的模块9的KR是非活性的,不能将C9的酮基还原成羰基。因此,C9酮基通过和C13羧基之间的相互作用,构成了成熟纳他霉素所需的半缩酮环。最后,PIMS4 C末端的硫酯酶结构域(TE)在丝氨酸残基活性位点酰化,产生酰基-酶中间体,催化骨架延伸及环化的结束[8]。
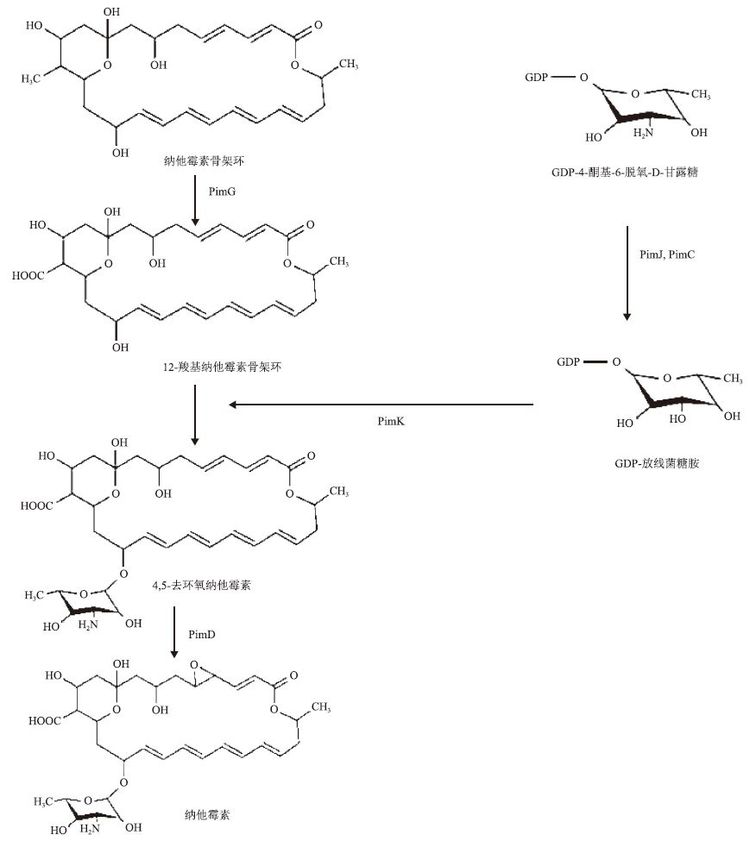
1.3 纳他霉素骨架环的修饰如表 2和图 3所示,纳他霉素骨架环合成后,PimG催化纳他霉素骨架环的C4-C5双键发生环氧化反应形成环外羧基,而GDP-甘露糖被PimJ、PimC催化构成GDP放线菌糖胺,最后由PimK催化放线菌糖胺连接在4,5-去环氧纳他霉素骨架环上,构成4,5-去环氧纳他霉素。
|
| 图 3 纳他霉素骨架环的加工修饰过程 |
pimD失活的纳塔尔链霉菌突变株只能合成4,5-去环氧纳他霉素,这说明PimD是作为一种细胞色素P450单氧酶催化C4-C5双键形成一个环氧,将4,5-去环氧纳他霉素转化成纳他霉素[9, 10]。这也是纳他霉素合成的最后一步[11]。
pimⅠ还编码一种硫酯酶,但它与pim4编码的硫酯酶同源性很差。因此,这两种酶来源不同。后来发现,这两种酶的作用也不同,PimⅠ代表了Ⅱ型硫酯酶,参与从聚酮化合物中除去非生产性脱羧基延长酰基,促进产物的正确积累[12]。
1.4 纳他霉素的转运完整的纳他霉素形成后,由PimA、PimB所编码的ABC跨膜转运子负责将其运出胞外[13]。pimH基因位于纳他霉素合成基因簇的最右端,由于其编码蛋白与耻垢分枝杆菌乙胺丁醇抗性区域的膜蛋白Orf4有36%的同源性,Aparicio等[6]据此推测,它也可能编码一种流出泵参与纳他霉素的转运。近年来,人们发现nysH、nysG(pimA、pimB的同源基因)失活的诺尔斯链霉菌突变体也可将制霉菌素运出胞外,证明链霉菌中存在除ABC转运子外的其他转运系统,从而大大提高了PimH参与转运纳他霉素的可信度(表 2)。
2 纳他霉素合成的调控机制纳他霉素合成的调控机制较为复杂,主要包括途径特异性调控和总体调控等类型。途径特异性调控基因位于调控网络的最底端,通过激活相关基因簇的表达调节纳他霉素的合成。而总体多效调控基因一般不与纳他霉素合成基因簇联系在一起,它们可以对多种营养与环境压力作出应答,调控多个代谢途径[14, 15]。
2.1 转录调节子的调节PimR和PimM是由纳他霉素基因簇编码的两种转录调节子,其编码基因pimR和pimM位于纳他霉素合成基因簇的最左端[16]。PimR属于链霉菌抗生素调节蛋白(streptomyces antibiotic regulatory proteins,SARPs)型调节子,是一种途径特异性调节蛋白,它的N端有SARP样结构域,C端有鸟苷酸环化酶的半同源结构域和一个LuxR家族的大的ATP结合调节子[17]。PimM是一个高度保守的转录调节子,其N端含PAS结构域,C端含LuxR HTH模体[18],其中LuxR是一个DNA结合结构域,而PAS结构域是一个感应结构域,会限制DNA结合结构域对其靶启动子的亲和力[19, 20]。
pimM失活的纳塔尔链霉菌突变体不能产生纳他霉素,而在纳塔尔链霉菌野生型中扩增pimM会大大提高纳他霉素产量。因此,PimM是纳他霉素合成的激活子。Santos-Aberturas等[21]用RT-PCR和电泳迁移率分析实验发现,PimR通过调节PimM来调节纳他霉素的合成。PimR作为一个转录调节子直接结合于pimM与pimR的整合区域,激活pimM表达PimM蛋白,PimM结合于纳他霉素合成基因pimK、pimS2S3S4、pimI、pimJ、pimAB、pimE、pimS1和pimD的启动子上,通过与RNA聚合酶的相互作用调控这些基因的表达。同时,PimM也激活其他分级调节子,从而调节pimC基因的表达。在这些蛋白的共同作用下,纳他霉素开始合成[22]。
2.2 途径特异性调节途径特异性调节是通过一种丁内酯受体蛋白(butyrolactone receptor protein,Brp)应答其同源丁内酯分子,从而激活一系列途径特异性调节基因编码蛋白,这些蛋白被统称为链霉菌抗生素调控蛋白家族[25, 26]。
2.2.1 PI因子的调节作用Recio等[27]发现,在纳塔尔链霉菌中存在一种新型的群体感应自诱导子(PI因子),它是一种纳他霉素诱导子(图 4),在微摩尔浓度下即可诱导不产纳他霉素的突变体合成纳他霉素。据推测,PI因子的这种诱导作用可能是由γ-丁内酯型受体介导的[28]。
|
| 图 4 PI(2,3-二氨基-2,3-双(羟甲基)-1,4-丁二醇)的分子结构 |
后来,Vicente等[29]研究发现,PI因子可以诱导pimT基因的表达,而pimT失活的纳塔尔链霉菌突变体中PI因子的表达水平降低了50%,纳他霉素产量降低了65%。由此可见,PimT是PI因子的转运子,细胞在营养限制等条件下产生PI因子,随后被PimT转运出胞外,作为识别其他细胞的一种信号分子,其他菌株整合这种外来群体信号后,纳他霉素基因开始表达。
2.2.2 甘油、乙二醇、1,2或1,3-丙二醇的调节作用除PI因子外,甘油、乙二醇、1,2或1,3-丙二醇也可以作为纳他霉素合成的诱导因子或前体,诱导并提高纳他霉素的合成[30]。后续实验结果发现,它们与PI诱导子的诱导机制不同,也不是PI因子的直接前体。据Li等[31]分析,这些物质能提高纳他霉素的产量的原因有两种,一是丙醇作为前体,为纳他霉素大环内酯的合成提供了三碳单元;二是丙醇通过调节代谢的关键位点,使乙酰辅酶A的积累,并抑制了TCA循环、糖酵解途径,以此促进纳他霉素的合成。
2.2.3 胆固醇氧化酶的调节作用pimE基因位于纳他霉素基因簇的中间,它是一种胞外型胆固醇氧化酶,对菌株的生长、分化没有影响[23]。Mendes等[24]发现,pimE失活的菌株不能产生纳他霉素,而基因互补却恢复了纳他霉素的合成,证明PimE是纳他霉素合成的一种信号蛋白。据此推测,PimE与其他胆固醇氧化酶可能作为一种真菌传感器作用于麦角固醇(真菌细胞膜中主要的固醇类物质)并引起细胞应答,再通过一系列信号转导级联反应诱导纳他霉素的合成。
2.3 总体调控总体调控主要是通过双组分系统来应答一系列外部压力信号,从而调控次级代谢过程。培养基中磷酸的浓度对纳塔尔链霉菌的初级代谢和次级代谢均产生极大的影响,磷酸浓度会干扰一系列基因的表达,关系到链霉菌由初级代谢向次级代谢的转变[32, 33]。当培养基中无机磷酸盐浓度达到1 mmol/L时,纳他霉素产量急剧下降;10 mmol/L时,所有纳他霉素合成基因的表达都处于抑制状态。Mendes等[35]克隆并测序了phoU-phoR-phoP 基因簇编码的PhoR-PhoP双组分系统,构建了phoP 缺失突变体及phoR-phoP 缺失突变体,二者都降低了对磷酸的敏感性,并且纳他霉素产量都增加了80%。对上述两种突变体进行PT-PCR及功能分析,结果表明,磷酸对纳他霉素合成的调控是通过phoR-phoP修饰其他合成基因介导的。Rodríguez等[34]也提出,链霉菌中的双组分系统是调节抗生素复杂代谢途径中的关键。
PhoR蛋白是一个标准的跨膜传感激酶,它仅含有一个完整的跨膜结构域和一个额外的短的疏水尾部,可以感应外部信号。PhoP是OmpR家族的一类DNA结合应答调节子,其C端含有DNA结合域[36]。phoU-phoR-phoP 基因位于纳他霉素合成基因簇之外,PhoP检测到外部磷酸信号时会结合于phoU-phoR整合区域启动子-35区域附近的PHO盒(PhoP结合序列)上,并通过招募RNA聚合酶及稳定RNA聚合酶-DNA启动子复合物进行自调节,诱导phoU-phoR基因大量转录,随后PhoP通过一些途径特异性调节子的级联反应,发生磷酸化和去磷酸化,抑制纳他霉素合成基因pimS1、pimS4、pimC和pimG的表达,导致纳他霉素产物的减少[35]。
2.4 交叉调节在链霉菌代谢中,某些营养限制信号,如磷酸[37]、氮源[38]、葡萄糖或其他容易利用的碳源[39],通过转换成总体调节子,激活途径特异性调节子来调控纳他霉素合成的初级和次级代谢过程。Borodina等[40]研究发现,磷酸果糖激酶基因在调控天蓝色链霉菌的次级代谢中发挥重要作用,而这种酶又是糖酵解途径的关键酶。因此,糖酵解过程中的磷酸化中间产物可能作为碳调控和磷酸调控的整合分子将两种信号途径串联起来。
在磷酸饥饿或氮饥饿引起细胞初级代谢不平衡的情况下,细胞会产生一系列的应答机制去清除过剩的营养,并产生次级代谢产物。它们之间的交互作用较为复杂,不仅影响参与氮源转运和同化的基因的表达,还直接影响了氮源调节子的表达[41]。有研究报道,双组分系统中的PhoP蛋白会结合到某些参与氮同化作用基因的启动子上,调控氮的代谢[42]。然而,在纳塔尔链霉菌中碳源、氮源、磷酸之间的交叉调节机制尚不甚清楚,有待进一步研究。
2.5 胞内ROS的调节众所周知,纳他霉素的产量在很大程度上依赖于溶解氧的水平,低浓度O2会限制菌株的生长及纳他霉素的合成,而高浓度O2会诱导胞内活性氧(reactive oxygen species,ROS)的升高和细胞的氧化损伤。因此,适当增加溶氧量可以提高纳他霉素的产量[43]。2011年,Beites等[44]阐明了胞内ROS调节纳他霉素合成的机制,氧气消耗会诱导细胞产生更多的ROS,尤其是胞内H2O2将PimM蛋白作为直接或间接靶标,通过氧化还原机制调控并提高纳他霉素的合成。然而,当胞内H2O2超过某个阈值时,会对细胞产生毒性作用,降低纳他霉素的产量。2013年,Tiago等[45]发现胞内NADPH/NADH比例和通过BACC(支链氨基酸)代谢的生物合成的前体细胞的有效性是氧化应激条件下纳他霉素合成的重要影响因素。
3 展望提高纳他霉素产量一直是微生物发酵产纳他霉素研究的核心内容。目前,国内主要采用诱变技术进行菌株选育,这在一定程度上可以提高纳他霉素的产量,但增幅程度受到该技术自身局限性的限制。研究纳他霉素的合成及其分子调控机制,为传统诱变技术向基因工程技术过渡奠定了理论基础。基因工程技术能够有目的地对相关基因进行定向改造,不仅从源头上提高纳他霉素基因的表达水平及其产量,还可降低诱变技术的盲目性和随机性。因此,基因工程菌种技术很可能代表纳他霉素未来的研究和开发方向。
| [1] | Wang S, Liu F, Hou Z, et al. Enhancement of natamycin production on Streptomyces gilvosporeus by chromosomal integration of the Vitreoscilla hemoglobingene(vgb)[J]. World J Microbiol Biotechnol, 2014, 30(4): 1369–1376. |
| [2] | te Welscher YM, ten Napel HH, Balagué MM, et al. Natamycin blocks fungal growth by binding specifically to ergosterol without permeabilizing the membrane[J]. J Biol Chem, 2008, 283(10): 6393–6401. |
| [3] | Prajna NV, Mascarenhas J, Krishnan T, et al. Comparison of natam-ycin and voriconazole for the treatment of fungal keratitis[J]. Arch Ophthalmol, 2010, 6: 672–678. |
| [4] | 王春艳, 刘树立. 纳他霉素的研究概况及其在食品工业中的应用[J]. 中国食品添加剂, 2007, (2): 169–173. |
| [5] | Martín JF, Liras P. Reprogramming microbial metabolic pathways[M]. Netherlands: Springer Netherlands, 2012.115-138. |
| [6] | Aparicio JF, Fouces R, Mendes MV, et al. A complex multienzyme system encoded by five polyketide synthase genes is involved in the biosynthesis of the 26-membered polyene macrolide pimaricin in Streptomyces natalensis[J]. Chem Biol, 2000, 7(11): 895–905. |
| [7] | Aparicio JF, Colina AJ, Ceballos E, et al. The biosynthetic gene cluster for the 26-membered ring polyene macrolide pimaricin a new polyketide synthase organization encoded by two subclusters separated by functionalzation genes[J]. J Biol Chem, 1999, 274(15): 10133–10139. |
| [8] | Sharma KK, Boddy CN. The thioesterase domain from the pimaricin and erythromycin biosynthetic pathways can catalyze hydrolysis of simple thioester substrates[J]. Bioorg Med Chem Lett, 2007, 17(11): 3034–3037. |
| [9] | Kells PM, Ouellet H, Santos-Aberturas J, et al. Structure of cytochrome P450 pimd suggests epoxidation of the polyene macrolide pimaricin occurs via a hydroperoxoferric intermediate[J]. Chem Biol, 2010, 17(8): 841–851. |
| [10] | Santos-Aberturas J, Engel J, Dickerhoff J, et al. Exploration of the substrate promiscuity of biosynthetic tailoring enzymes as a new source of structural diversity for polyene macrolide antifungals[J]. ChemCatChem, 2015, 7(3): 490–500. |
| [11] | Mendes MV, Recio E, Fouces R, et al. Engineered biosynthesis of novel polyenes:A pimaricin derivative produced by targeted gene disruption in Streptomyces natalensis[J]. Chem Biol, 2001, 8(7): 635–644. |
| [12] | Kim BS, Cropp TA, Beck BJ, et al. Biochemical evidence for an editing role of thioesterase Ⅱ in the biosynthesis of the polyketide pikromycin[J]. J Biol Chem, 2002, 277(50): 48028–48034. |
| [13] | Davidson AL, Dassa E, Orelle C, et al. Structure, function, and evolution of bacterial ATP-binding cassette systems[J]. Microbiol Mol Biol R, 2008, 72(2): 317–364. |
| [14] | Liu G, Chater KF, Chandra G, et al. Molecular regulation of antibiotic biosynthesis in streptomyces[J]. Microbiol Mol Biol R, 2013, 77(1): 112–143. |
| [15] | Yoon V, Nodwell JR. Activating secondary metabolism with stress and chemicals[J]. J Ind Microbiol Biot, 2014, 41(2): 415–424. |
| [16] | Jang BY, Hwang YI, Choi SU. Effects of pimM and pimR on the increase of natamycin production in Streptomyces natalensis[J]. J Korean Soc Appl Bi, 2011, 54(1): 141–144. |
| [17] | Antón N, Martin JF, Mendes MV, et al. Identification of PimR as a positive regulator of pimaricin biosynthesis in Streptomyces natalensis[J]. J Bacteriol, 2004, 186(9): 2567–2575. |
| [18] | Antón N, Santos-Aberturas J, Mendes MV, et al. PimM, a PAS domain positive regulator of pimaricin biosynthesis in Streptomyces natalensis[J]. Microbiology, 2007, 153(9): 3174–3183. |
| [19] | Santos-Aberturas J, Payero TD, Vicente CV, et al. Functional conservation of PAS-LuxR transcriptional regulators in polyene macrolide biosynthesis[J]. Metab Eng, 2011, 13(6): 756–767. |
| [20] | Vicente CM, Santos-Aberturas J, Payero TD, et al. PAS-LuxR transcriptional control of filipin biosynthesis in S. avermitilis[J]. Appl Microbiol Biot, 2014, 98(22): 9311–9324. |
| [21] | Santos-Aberturas J, Vicente CV, Payero TD, et al. Hierarchical control on polyene macrolide biosynthesis:PimR modulates pimaricin production via the PAS-LuxR transcriptional activator PimM[J]. PLoS ONE, 2012, 7(6): e38536. |
| [22] | Santos-Aberturas J, Vicente CM, Guerra SM, et al. Molecular control of polyene macrolide biosynthesis direct binding of the regulator pimm to eight promoters of pimaricin genes and identification of binding boxes[J]. J Biol Chem, 2011, 286(11): 9150–9161. |
| [23] | Aparicio JF, Martn JF. Microbial cholesterol oxidase:Bioconversion enzymes or signal proteins?[J]. Mol Biosyst, 2008, 4(8): 804–809. |
| [24] | Mendes MV, Recio E, Antón N, et al. Cholesterol oxidases act as signaling proteins for the biosynthesis of the polyene macrolide pimaricin[J]. Chem Biol, 2007, 14(3): 279–290. |
| [25] | Tezuka T, Ohnishi Y. Microbial production[M]. Japan: Springer Japan, 2014.179-190. |
| [26] | Martín JF, Liras P. Engineering of regulatory cascades and networks controlling antibiotic biosynthesis in Streptomyces[J]. Curr Opin Microbiol, 2010, 3(3): 263–273. |
| [27] | Recio E, Colina A, Rumbero A, et al. PI factor, a novel type quorum sensing inducer elicits pimaricin production in Streptomyces natalensis[J]. J Biol Chem, 2004, 279(40): 41586–41593. |
| [28] | Nodwell JR. Are you talking to me? A possible role for γ-butyrola-ctones in interspecies signaling?[J]. Mol Microbiol, 2014, 94(3): 483–485. |
| [29] | Vicente CM, Santos-Aberturas J, Guerra SM, et al. PimT, an amino acid exporter controls polyene production via secretion of the quorum sensing pimaricin-inducer PI-factor in Streptomyces natalensis[J]. Microb Cell Fact, 2009, 8(1): 426–433. |
| [30] | Recio E, Aparicio JF, Rumbero A, et al. Glycerol, ethylene glycol and propanediol elicit pimaricin biosynthesis in the PI-factor defective strain Streptomyces natalensis npi287 and increase polyene production in several wild type actinomycetes[J]. Microbiology, 2006, 152: 3147–3156. |
| [31] | Li M, Chen S, Li J, et al. Propanol addition improves natamycin biosynthesis of Streptomyces natalensis[J]. Appl Biochem Biotechnol, 2014, 172(7): 3424–3432. |
| [32] | Sanche S, Demain AL. Metabolic regulation of fermentation processes[J]. Enzyme Microb Tech, 2002, 31(7): 895–906. |
| [33] | Nieselt K, Battke F, Herbig A, et al. The dynamic architecture of the metabolic switch in Streptomyces coelicolor[J]. BMC Genomics, 2010, 11(1): 150–155. |
| [34] | Rodríguez H, Rico S, Díaz M, et al. Two-component systems in Streptomyces:key regulators of antibiotic complex pathways[J]. Microb Cell Fact, 2013, 12(4): 491–494. |
| [35] | Mendes MV, Tunca S, Antón N, et al. The two-component phoR-phoP system of Streptomyces natalensis:inactivation or deletion of phoP reduces the negative phosphate regulation of pimaricin biosynthesis[J]. Meta Eng, 2007, 9(2): 217–227. |
| [36] | Martín JF. Phosphate control of the biosynthesis of antibiotics and other secondary metabolites is mediated by the PhoR-PhoP system:an unfinished story[J]. J Bacteriol, 2004, 186(16): 5197–5201. |
| [37] | Rodríguez-García A, Barreiro C, Santos-Beneit F, et al. Genome-wide transcriptomic and proteomic analysis of the primary response to phosphate limitation in Streptomyces coelicolor M145 and in a DphoP mutant[J]. Proteomics, 2007, 7(14): 2410–2429. |
| [38] | Tiffert Y, Supra P, Wurm R, et al. The Streptomyces coelicolor GlnR regulon:identification of new GlnR targets and evidence for a central role of GlnR in nitrogen metabolism in actinomycetes[J]. Mol Microbiol, 2008, 67(4): 861–880. |
| [39] | Rigali S, Titgemeyer F, Barends S, et al. Feast or famine:the global regulator DasR links nutrient stress to antibiotic production by Streptomyces[J]. EMBO Rep, 2008, 9(7): 670–675. |
| [40] | Borodina I, Siebring J, Zhang J, et al. Antibiotic overproduction in Streptomyces coelicolor A3 2 mediated by phosphofructokinase deletion[J]. J Biol Chem, 2008, 283(37): 25186–25199. |
| [41] | Martín JF, Sola-Landa A, Santos-Beneit F, et al. Cross-talk of global nutritional regulators in the control of primary and secondary metabolism in Streptomyces[J]. Microb Biotechnol, 2011, 4(2): 165–174. |
| [42] | Rodríguez-García A, Sola-Landa A, Apel K, et al. Phosphate control over nitrogen metabolism in Streptomyces coelicolor:direct and indirect negative control of glnR, glnA, glnⅡ and amtB expression by the response regulator PhoP[J]. Nucleic Acids Res, 2009, 37(10): 3230–3242. |
| [43] | el-Enshasy HA, Farid MA, el-Sayed el SA. Influence of inoculum type and cultivation conditions on natamycin production by Streptomyces natalensis[J]. J Basic Microb, 2000, 40(5-6): 333–342. |
| [44] | Beites T, Pires SDS, Santos CL, et al. Crosstalk between ROS homeostasis and secondary metabolism in S. natalensis ATCC 27448:modulation of pimaricin production by intracellular ROS[J]. PLoS One, 2011, 6(11): e27472. |
| [45] | Beites T, Rodríguez-García A, Moradas-Ferreira P, et al. Genome-wide analysis of the regulation of pimaricinproduction in Streptomyces natalensis by reactive oxygen species[J]. Appl Microbiol Biot, 2014, 98(5): 2231–2241. |