2. 农业部水产品贮藏保鲜质量安全风险评估实验室,上海 201306;
3. 上海水产品加工及贮藏工程技术研究中心,上海 201306
2. Laboratory of Quality & Safety Risk Assessment for Aquatic Production on Storage and Preservation, Ministry of Agriculture, Shanghai 201306;
3. Shanghai Engineering Research Center of Aquatic Product Processing & Preservation, Shanghai 201306
近年来随着分子生物学技术的迅速发展,出现了一系列的食源性致病菌流行病学调查方法,如脉冲电场凝胶电泳(PFGE)[1]、扩增片段长度多态性(AFLP)[2]、随机扩增多态性(RAPD)[3]、重复元素多态性(REP-PCR)[4],以及多位点序列分型(MLST)[5]等技术。其中,ERIC-PCR是基于“肠道细菌基因间重复序列”(Enterobacterial repetitive intergenic conse-nsus,ERIC)的分型方法,具有分辨率较高、准确性好、简便快捷、无需特殊仪器等优点[6]。此外,变性梯度凝胶电泳(Denaturing gradient gel electrophoresis,DGGE)是一种广泛应用于不同领域的微生物群落多样性分析和种群动态监测的分子生物学技术。近几年,DGGE技术也被应用于食源性致病微生物的快速检测与分型[7]。
基于16S rDNA中V3区的PCR-DGGE常用于微生物群落多样性分析和食源性致病菌的快速检测与分型的研究[8, 9]。但此方法同时存在一定的缺陷,如单一菌可能在DGGE指纹图谱中含有多谱带[10]。然而,RNA聚合酶β亚基的编码基因rpoB具有高度的保守性,单拷贝以及更高分辨率的特点,与PCR-DGGE技术相结合使得单一菌在DGGE指纹图谱中只存在单谱带,从而使得PCR-DGGE方法用于研究微生物群落多样性,微生物检测与分型的结果更为准确[11]。为提高DGGE技术对DNA片段序列差异分辨率,通常在一侧引物5´端连接一段30-50 bp富含G、C碱基的DNA片段(GC夹)。Sheffield等[12]发现40 bp的GC夹足够满足DGGE对微生物多样性的研究。然而,目前关于GC夹的变化对微生物群落多样性和检测分型结果影响的研究较少。
本研究以5株副溶血性弧菌(Vibrio parahaemo-lyticus),5株单增李斯特菌(Listeria monocytogenes)和3株沙门氏菌(Salmonella spp.)为研究对象,讨论不同GC夹和GC夹的不同引物连接位置(正反引物的5´端)对3种(13株)食源性致病菌的rpoB-PCR-DGGE检测与分型结果的影响,并与V3-PCR-DGGE和ERIC-PCR两种方法的结果进行比较。
1 材料与方法 1.1 材料 1.1.1 菌株5株副溶血性弧菌:3株来源于美国典型菌种保藏中心(ATCC):ATCC 33846、ATCC 33847、ATCC17802;1株来源于实验室分离:F18;1株来源于临床分离:O3:K6。5株单增李斯特菌均来自于ATCC:ATCC 19112、ATCC 19114、ATCC 19115、ATCC 19116、ATCC 19118。3株沙门氏菌:2株来源于中国医学微生物菌种保藏管理中心(CMCC):肠炎沙门氏菌 CMCC 50041、乙型副伤寒沙门氏菌 CMCC 50094;1株来源于中国工业微生物菌种保藏中心(CICC):鼠伤寒沙门氏菌 CICC 21484。
1.1.2 主要仪器与试剂PCR扩增仪,德国Eppen-dorf公司;DGGE电泳仪The DcodeTM Universal Muta-tion Detection System,美国Bio-Rad公司;TIANamp Bacteria DNA Kit,北京Tiangen公司;Premix rTaq,宝生物工程(大连)有限公司。胰蛋白胨大豆肉汤(TSB)、溶菌肉汤(LB)、脑心浸液(BHI)、弧菌选择性琼脂(TCBS)、李斯特菌选择性琼脂(PALC-AM)、沙门氏菌选择性琼脂(BS),北京路桥技术责任有限公司。
1.2 方法 1.2.1 细菌DNA提取将13株菌种(5株副溶血性弧菌,5株单增李斯特菌,3株沙门氏菌)分别划线接种于选择性培养基TCBS、PALCAM、BS上,于37℃恒温培养箱下培养18-24 h。依次挑取单菌落接种于TSB(含3% NaCl)、BHI、LB中,于37℃恒温摇床培养16 h,连续活化2次。收集菌体沉淀,使用TIANamp Bacteria DNA Kit进行细菌DNA提取,详细步骤见试剂盒说明书。其中,蛋白酶K处理时间延长至1 h,溶菌酶处理时间延长至0.5 h[13]。
1.2.2 PCR扩增本研究所用的引物由上海生物工程技术有限公司合成,详细序列见表 1和表 2。
PCR扩增体系溶液总体积为25 µL,其中包含Premix TaqTM DNA聚合酶12.5 µL(其中,Taq酶0.625 U;2× dNTP各0.4 mmol/L;2× Taq Buffer 4 mmol/L Mg2+),正反引物各1 µL(各20 µmol/L),DNA模板1 µL,ddH2O 9.5 µL。rpoB降落PCR反应程序:94℃预变性5 min;94℃ 1 min,53-48℃ 30 s,72℃ 1 min(其中每个循环后复性温度下降0.5℃);10个循环后进入第二个循环程序:94℃ 1 min,50℃ 30 s,72℃ 1 min,25个循环后;72℃持续7 min。V3-PCR反应程序:95℃预变性 3 min;95℃ 1 min,55℃ 1 min,72℃ 30 s,共30个循环;最后72℃持续5 min。ERIC-PCR反应程序:95℃预变性7 min;94℃ 1 min,52℃ 1 min,65℃ 8 min,共30个循环;最后65℃维持16 min。检测方法:取5 µL PCR反应产物用2%(rpoB-PCR,V3-PCR)和1%(ERIC-PCR)琼脂糖凝胶,于5 V/cm电压下电泳30 min后于凝胶成像仪下观察结果。
1.2.3 变性梯度凝胶电泳(DGGE)DGGE指纹图谱构建采用DcodeTM系统(Bio-Rad)。V3区扩增产物在8%聚丙烯酰胺凝胶中分离,变性梯度为40%-60%。其运行条件:1×TAE电泳缓冲液,温度60℃,电压60 V,时间16 h,每个样品点样量为15 µL PCR产物,电泳结束后用15 mL 1×SYBR Green染色,在紫外灯下用凝胶成像仪拍照成像。rpoB基因扩增产物在8%聚丙烯酰胺凝胶中分离,变性梯度为35%-55%。其运行条件:1×TAE电泳缓冲液,60℃,100 V,10 h,每个样品点样量为15 µL PCR产物,电泳结束后用15 mL 1×SYBR Green染色,在紫外灯下用凝胶成像仪拍照成像。
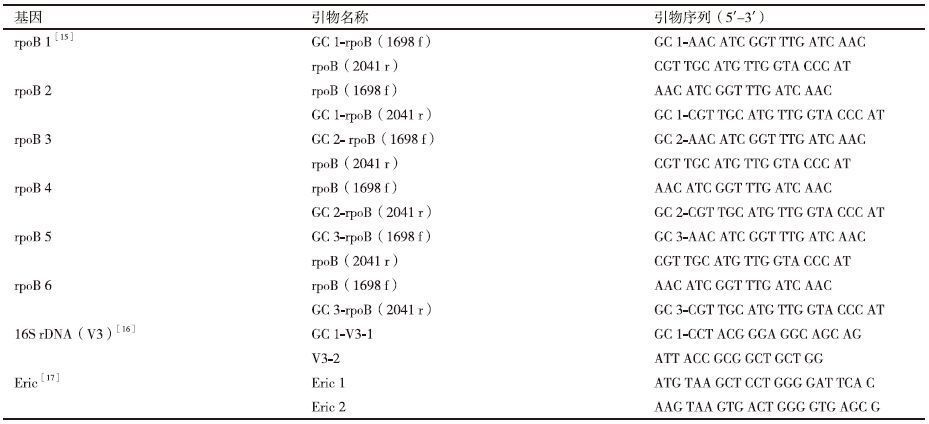
1.2.4 DGGE 图谱和 ERIC-PCR 图谱分析应用Quantity One(Bio-Rad)凝胶图谱分析软件对成像所得DGGE图谱和ERIC-PCR图谱进行分析,通过泳道和条带的检测与匹配,获得条带相对电泳距离值(Rf values),从而构建图谱泳道之间的相似性矩阵。然后应用PAST ver.1.79分析软件选择Paired group算法对图谱进行聚类分析[18]。使用Hunter和Gaston的方法计算出分型方法的分辨力指数[19]。公式如下:
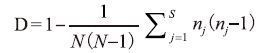 (公式1)
(公式1)
其中,N表示实验中总菌株数,S表示聚类分析的总类型数,表示第j个类型中含有的菌株总数。
2 结果 2.1 三种GC夹、两种连接位置的 rpoB-PCR-DGGE 指纹图谱的构建及分析总体观察指纹图谱(图 1),rpoB-PCR-DGGE能将3种食源性致病菌完全区分开。然而,5株副溶血性弧菌中,只有ATCC 33846被区分开。对于5株单增李斯菌,则情况不一。最多分为4类:如图 1-C和图 1-F所示。最少分为3类:如图 1-A,1-B,1-D,1-E所示。且图 1-A和1-B中,ATCC 19112没有谱带出现。对于3株沙门氏菌,图 1-A,1-C,1-E中没有区分开,而图 1-B,1-D,1-F中 CICC 21484被区分开。
|
| 图 1 三种食源性致病菌的 rpoB-PCR-DGGE 指纹图谱 A-F:GC-1 连接正引物, GC-1 连接反引物, GC-2 连接正引物, GC-2 连接反引物, GC-3 连接正引物, GC-3 连接反引物。 1-5 :副溶血性弧菌:ATCC 33846, ATCC 33847, ATCC 17802, F18, O3:K6 ; 6-10 :单增李斯特菌:ATCC 19112, ATCC 19114, ATCC 19115, ATCC 19116, ATCC 19118; 11-13 :沙门氏菌:CMCC 50041, CMCC 50094, CICC 21484 |
纵向比较图 1-A、1-C、1-E以及图 1-B、1-D、1-F发现,基于不同GC夹连接于引物的相同位置的rpoB-PCR-DGGE,对5株副溶血性弧菌和3株沙门氏菌的分辨效果无显著差别。但是,对于5株单增李斯特菌,其分型效果差别较大。菌株ATCC 19115和ATCC 19116始终处于同一水平位置上,菌株ATCC 19118谱带位置处于前两株菌株的上方。另外两株菌ATCC 19112和ATCC 19114谱带位置存在变化。基于GC-2连接于正引物(图 1-C)和GC-3连接于反引物(图 1-F)的rpoB-PCR-DGGE将5株单增李斯特菌区分成了四类。
横向比较图 1-A和1-B、图 1-C和1-D,图 1-E和1-F观察到,基于相同GC夹连接于引物不同位置的rpoB-PCR-DGGE,对5株副溶血性弧菌分型效果无任何影响。而对5株单增李斯特菌,影响较大。其中,菌株ATCC 19115、ATCC19116和 ATCC 19118的谱带在DGGE图谱中的位置无变化,菌株ATCC 19112和ATCC 19114的谱带位置变化明显。对于3株沙门氏菌,基于GC连接于正引物的rpoB-PCR-DGGE(图 1-A、1-C、1-E)未将其区分开,且谱带均呈弥散状态。而基于GC连接于反引物的DGGE图谱中(图 1-B、1-D、1-F),菌株CICC 21484被区分开,谱带无弥散。
经以上比较与分析,可知应用rpoB-PCR-DGGE对13株食源性致病菌检测与分型效果最好的图谱为图 1-F,即GC-3连接于rpoB 的反引物上。
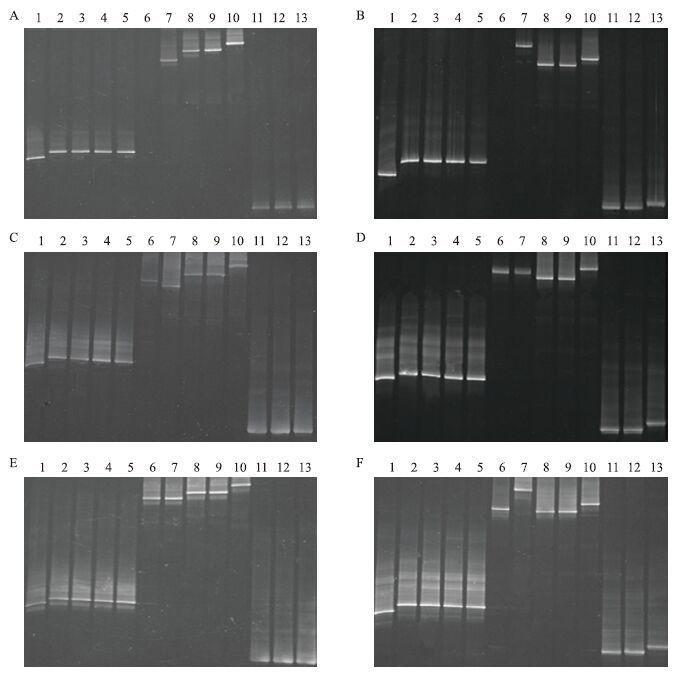
2.2 V3-PCR-DGGE的指纹图谱的构建及分析13株食源性致病菌基于V3-PCR-DGGE所构建的图谱(图 2)显示,副溶血性弧菌和沙门氏菌中出现单一菌株存在多条谱带的现象,然而,单增李斯特菌单一菌株都只对应1条谱带。5株副溶血性弧菌株中,在DGGE图谱上只出现1条谱带的是O3:K6,出现2条谱带的是ATCC 33846和ATCC 17802,出现3条谱带的是ATCC 33847,出现5条谱带的是F18。对于3株沙门氏菌,菌株CMCC 50041只出现1条谱带,菌株CMCC 50094出现约8条谱带,菌株CICC 21484出现5条谱带。DGGE图谱中,5株副溶血性弧菌的谱带位置与3株沙门氏菌谱带位置约处于同一水平线上。而5株单增李斯特菌的谱带位置一致,且位于其它两种菌的谱带上方。
|
| 图 2 三种食源性致病菌的 V3-PCR-DGGE 指纹图谱 1-5:副溶血性弧菌;6-10:单增李斯特菌;11-13:沙门氏菌 |
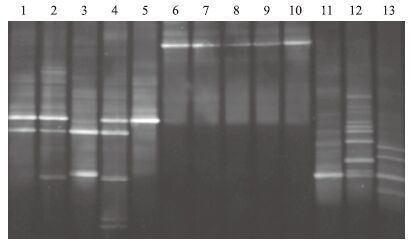
13株食源性致病菌经ERIC-PCR扩增,结果(图 3)表明,得到了谱带丰富,分辨率较好的DNA指纹图谱。5株副溶血性弧菌的谱带最为丰富,为9-12条。5株单增李斯特菌谱带数为4-7条。3株沙门氏菌谱带数为7-10条。种间比较,3种食源性致病菌的指纹图谱之间差距大。种内比较,5株副溶血性弧菌的指纹图谱之间差别不明显,而5株单增李斯特菌和3株沙门氏菌的指纹图谱间差别较显著。

|
| 图 3 三种食源性致病菌的 ERIC-PCR 指纹图谱 1-5:副溶血性弧菌;6-10:单增李斯特菌;11-13:沙门氏菌 |
运用PAST ver. 1.79软件对上述3种方法的指纹图谱(图 1-F、图 2、图 3)进行聚类分析(图 4),凡相似度大于80%者为同一基因型,小于80%者为不同的基因型[18]。如图 4-A所示,13株食源性致病菌可被rpoB-PCR-DGGE至少分为8个基因类型,菌株2、3、4、5为同一型,菌株8、9为同一型,菌株11、12为同一型,其他菌株各为不同型。使用Hunter和Gaston的方法计算出此分型方法的分辨力指数为0.90。图 4-B显示,13株食源性致病菌可被V3-PCR-DGGE至少分为6个型,菌株1、2、4、5为同一型,菌株6、7、8、9、10为同一型,其它菌株各为不同型。此分型方法的分辨力指数为0.80。图 4-C显示,13株食源性致病菌可被ERIC-PCR至少分为8个型,菌株1、2、3、4、5为同一型,菌株8、10为同一型,菌株11、13为同一型,其他菌株各为不同型。此分型方法的分辨力指数为0.90。
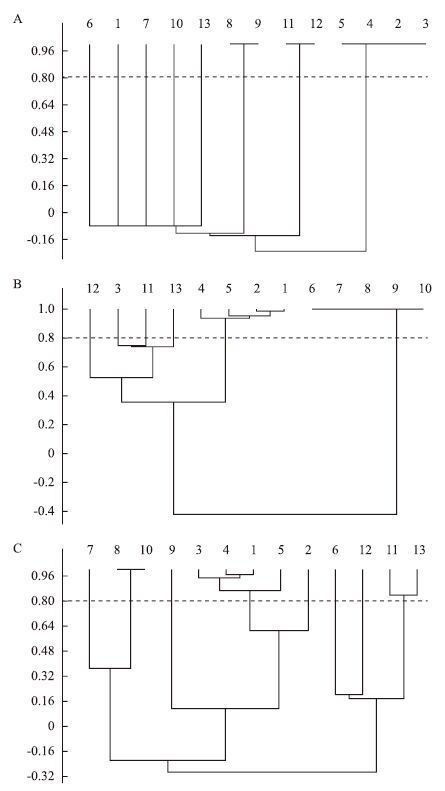
|
| 图 4 三种食源性致病菌的三种分型方法指纹图谱聚类分析 A:rpoB-PCR-DGGE 方法;B:V3-PCR-DGGE 方法;C:ERIC-PCR 方法 |
本研究采用3种不同的GC夹(GC-1、GC-2、GC-3),并且将同种GC夹分别连接在正反引物上,共合成6对rpoB引物。针对3种(13株)食源性致病菌进行rpoB-PCR-DGGE分析,获得6张DGGE指纹图谱。Rettedal等[20]研究表明GC夹中序列的变化,会引起GC%和Tm的变化,从而引起PCR结果的改变。本研究中,GC-1与GC-2的序列存在16个碱基的差异,GC-1和GC-3存在15个碱基的差异。最终获得的DGGE图谱间都存在一定的差异。Sheffield等[12]报道称碱基序列中存在多个毗连的鸟嘌呤(G)会影响DNA序列合成或是PCR反应的结果。Murphy等[21]研究表明多个相邻的鸟嘌呤(G)易导致形成畸形的空间结构,而且该结构能抑制DNA聚合酶η的作用。GC-1中存在2次两个连续,2次三个连续和1次四个连续的碱基G;GC-2中只存在1次两个连续的碱基G;而GC-3中则不存在连续的碱基G。因此,图 1-F显示DGGE的分型效果最好与上述具有一定的相关性。另外,相同的GC夹子分别连接在正反引物上,同样对rpoB-PCR-DGGE图谱产生了一定的影响,如连接于正引物上沙门氏菌谱带弥散,而连接于反引物上沙门氏菌谱带正常且使菌株CICC 21484与其它两株沙门氏菌区分开。近来,并未有相关研究对此现象进行解释。但可能仍与序列中存在多个连续的鸟嘌呤有关。因为正引物rpoB(1698 f)中出现1次两个连续的G,而反引物rpoB(2041 r)则未出现,分别与GC夹连接后进行PCR而产生不同的反应结果。本研究中,基于GC-3连接在ropB基因反引物上的DGGE图谱的分辨效果最佳(图 1-F),因此在选择或设计GC夹时,应当避免序列中出现连续的鸟嘌呤(G)以及连接于反引物的5´端。
3.2 rpoB-PCR-DGGE与V3-PCR-DGGE结果比较分析PCR-DGGE技术已被广泛应用于微生物群落多样性的研究,该技术常基于16S rDNA中保守区域片段,如V1-V3区[22]、V3区[23]、V6-V8区[24]。近年来,PCR-DGGE也被应用于食源性致病菌的检测与分型[25]。但根据文献[26]报道,单一菌的16S rDNA存在多拷贝或异源双链,导致DGGE图谱中单一菌出现多条谱带,从而使得微生物群落多样性的结果评价过高或者检测结果不准确。本实验应用了V3-PCR-DGGE对13株食源性致病菌进行分型研究。结果(图 2)表明,4株副溶血性弧菌和2株沙门氏菌在DGGE图谱中均出现了多谱带现象。而5株单增李斯特菌均只有单谱带出现。对于多谱带的单一菌,基于16S rDNA V3区PCR-DGGE具有重复性好的特点,不同菌株谱带数目与位置具有特异性。因此,该方法可针对性作为一种潜在的分型方法。但对于副溶血性弧菌与沙门氏菌的混合样品,可能无法将二者同时准确检测出。
编码RNA聚合酶β亚基基因(rpoB)是一种单拷贝的管家基因,与DGGE技术相结合,逐渐被应用于微生物群落多样性的研究中[27]。因此,基于rpoB的PCR-DGGE图谱中,一种菌只有单谱带,比基于16S rDNA的PCR-DGGE更为准确地反映微生物群落多样性。然而,未见有文献报道关于rpoB-PCR-DGGE技术对食源性致病菌进行快速分型的研究。本研究应用rpoB-PCR-DGGE技术对13株食源性致病菌进行检测与分型,同时与V3-PCR-DGGE的结果进行比较。从本研究的图谱(图 1-F,图 2)与图谱聚类分析结果(图 4-A,4-B)明显看出,rpoB-PCR-DGGE检测和分型的效果优于V3-PCR-DGGE。基于rpoB的DGGE图谱中,13种菌各均只有单谱带。与此同时,副溶血性弧菌、单增李斯特菌及沙门氏菌的谱带都能较好地被区分开,同种菌的不同亚种也被不同程度的区分。根据Hunter和Gaston的分辨力指数计算方法可知,rpoB-PCR-DGGE分辨力(0.90)高于V3-PCR-DGGE分辨力(0.80)。
3.3 rpoB-PCR-DGGE与ERIC-PCR分子分型结果比较与分析研究发现,肠道细菌中ERIC序列是一段长126 bp的反向重复序列,位于基因组内可转录的非编码区域或与转录有关的区域[28]。ERIC序列分布于整个基因组中,具有极强的保守性,用ERIC-PCR得到的DNA谱带特征能反映出细菌整个基因组结构的差异能区别包含有ERIC序列的不同细菌的种和株,因此具有很强的鉴别种乃至菌株的能力[29]。与其它分子分型方法相比,ERIC-PCR具有操作简单,结果重复性好,分辨力强的优势。本研究中,同样应用了ERIC-PCR对13株食源性致病菌进行了分型的研究,并与rpoB-PCR-DGGE的结果进行比较。对图谱结果(图 1-F,图 3)和图谱聚类分析结果(图 4-A,4-C)分析可知,ERIC-PCR图谱中每株菌的谱带呈现出不同的多态性,副溶血性弧菌谱带数最多,沙门氏菌次之,单增李斯特菌最少。ERIC-PCR的分辨力指数为0.9,与rpoB-PCR-DGGE分辨力相同。以上结果说明与ERIC-PCR相比,rpoB-PCR-DGGE同样具有操作简单,结果重复性好,分辨力强的特点。并且,对于实际样品,后者能直接进行快速检测与分型,而前者需要先分离出单一菌株。
4 结论不同序列的GC夹以及GC夹不同的引物连接位置均会对13株食源性致病菌的rpoB-PCR-DGGE检测与分型结果产生影响,其分辨力与ERIC-PCR的相等并优于V3-PCR-DGGE。因此,本研究得到了基于特定GC夹以及GC夹连接引物特定位置的rpoB-PCR-DGGE能有效提高食源性致病菌进行快速检测与分型效果的结论,从而为提高食品安全质量起到积极的促进作用。
| [1] | Karami N, Helldal L, Welinder-Olsson C, et al. Sub-typing of extended-spectrum-β-Lactamase-producing isolates from a nosocomial outbreak:application of a 10-Loci generic Escherichia coli multi-locus variable number tandem repeat analysis[J]. PLoS One, 2013, 8(12): e83030. |
| [2] | Parisi A, Latorre L, Fraccalvieri R, et al. Occurrence of Listeria spp. in dairy plants in Southern Italy and molecular subtyping of isolates using AFLP[J]. Food Control, 2013, 29(1):91-97. |
| [3] | Al-Harthi OM, Abdelkader HS, Halawani EM. Molecular typing of Salmonella enterica serovars typhimurium and enteritidis isolated from Taif area of Saudi Arabia by random amplified polymorphic DNA-polymerase chain reaction(RAPD-PCR)[J]. African Journal of Microbiology Research, 2015, 9(9): 651–661. |
| [4] | Taylor J, Doyle D, Blackall P, et al. Use of REP-PCR and 16S rRNA gene sequencing for comparison of Mannheimia haemolytica isolates obtained from fatal cases of bovine respiratory disease in the USA and Australia[J]. Aust Vet J, 2014, 92(1-2): 15–23. |
| [5] | Boonsilp S, Thaipadungpanit J, Amornchai P, et al. A single multilocus sequence typing(MLST)scheme for seven pathogenic Leptospira species[J]. PLoS Negl Trop Dis, 2013, 7(1): e1954. |
| [6] | Fendri I, Hassena AB, Grosset N, et al. Genetic diversity of food-isolated Salmonella strains through Pulsed Field Gel Electrophoresis(PFGE)and Enterobacterial Repetitive Intergenic Consensus(ERIC-PCR)[J]. PLoS One, 2013, 8(12): e81315. |
| [7] | Vashin I, Stoyanchev T, Iliev M, et al. Application of polymerase chain reaction and denaturing gradient gel electrophoresis assay of the flagellin gene for direct detection and subtyping of Campylobacter jejuni and Campylobacter coli in avian faecal samples[J]. Bulgarian Journal of Veterinary Medicine, 2012, 15(1): 22–29. |
| [8] | Lan WQ, Xie J. Characterization of the dynamic changes of microorganisms in cutlassfish(Trichiurus haumela)under the cold storage with composite natural preservatives based on culture-dependent and 16S rRNA-DGGE technology[J]. Advanced Materials Research, 2012, 554: 1498–1506. |
| [9] | Wen CQ, Lai XT, Xue M, et al. Molecular typing and identification of Bdellovibrio-and-like organisms isolated from seawater shrimp ponds and adjacent coastal waters[J]. Journal of Applied Microbiology, 2009, 106(4): 1154–1162. |
| [10] | Kang Y, Cheng J, Mei L, et al. Multiple copies of 16S rRNA gene affect the restriction patterns and DGGE profile revealed by analysis of genome database[J]. Microbiology, 2010, 79(5): 655–662. |
| [11] | Shu QL, Jiao NZ. Developing a novel approach of rpoB gene as a powerful biomarker for the environmental microbial diversity[J]. Geomicrobiology Journal, 2013, 30(2): 108–119. |
| [12] | Sheffield VC, Cox DR, Lerman LS, et al. Attachment of a 40-base-pair G+ C-rich sequence(GC-clamp)to genomic DNA fragments by the polymerase chain reaction results in improved detection of single-base changes[J]. Proceedings of the National Academy of Sciences, 1989, 86(1): 232–236. |
| [13] | Liao C, Peng ZY, Li JB, et al. Simultaneous construction of PCR-DGGE-based predictive models of Listeria monocytogenes and Vibrio parahaemolyticus on cooked shrimps[J]. Letters in Applied Microbiology, 2015, 60(3): 210–216. |
| [14] | Muyzer G, De Waal EC, Uitterlinden AG. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695–700. |
| [15] | Dahllöf I, Baillie H, Kjelleberg S. rpoB-based microbial community analysis avoids limitations inherent in 16S rRNA gene intraspecies heterogeneity[J]. Applied and Environmental Microbiology, 2000, 66(8): 3376–3380. |
| [16] | Cocolin L, Comi G. Use of a culture-independent molecular method to study the ecology of Yersinia spp. in food[J]. International Journal of Food Microbiology, 2005, 105(1): 71–82. |
| [17] | Jeršek B, Gilot P, Gubina M, et al. Typing of Listeria monocytogenes strains by repetitive element sequence-based PCR[J]. Journal of Clinical Microbiology, 1999, 37(1): 103–109. |
| [18] | 刘海泉, 朱颖, 姜文洁, 等. ERIC-PCR技术对单增李斯特菌的溯源分析[J]. 食品工业科技, 2013, 34(8): 49–51. |
| [19] | Hunter PR, Gaston MA. Numerical index of the discriminatory ability of typing systems:an application of Simpson’s index of diversity[J]. J Clin Microbiol, 1988, 26(11): 2465–2466. |
| [20] | Rettedal EA, Clay S, Brözel VS. GC-clamp primer batches yield 16S rRNA gene amplicon pools with variable GC clamps, affecting denaturing gradient gel electrophoresis profiles[J]. FEMS Microbiology Letters, 2010, 312(1): 55–62. |
| [21] | Murphy CT, Gupta A, Armitage BA, et al. Hybridization of G-quadruplex-forming peptide nucleic acids to guanine-rich DNA templates inhibits DNA polymerase η extension[J]. Biochemistry, 2014, 53(32): 5315–5322. |
| [22] | Probst M, Walde J, Pümpel T, et al. Lactic acid fermentation within a cascading approach for biowaste treatment[J]. Applied microbiology and Biotechnology, 2015, 99: 2029–2040. |
| [23] | 张振华, 鲍王波, 余冉, 等. 鹌鹑肠道微生物PCR-DGGE方法的优化[J]. 生物技术通报, 2015, 31(1): 73–78. |
| [24] | 兰阿峰, 杨曼, 郭素芬, 等. 免培养法对大鲵肠道微生物多样性的研究[J]. 微生物学通报, 2014, 41(7): 1342–1349. |
| [25] | Xiao J, Liu Y, Wang M, et al. Detection of Mycoplasma pneumoniae P1 subtype variations by denaturing gradient gel electrophoresis[J]. Diagnostic Microbiology and Infectious Disease, 2014, 78(1): 24–28. |
| [26] | Wu B, Zhang Q, Liu Z, et al. Bacterial communities in alfalfa and corn silages produced in large-scale stack and bunker silos in China[J]. Grassland Science, 2014, 60(4): 247–251. |
| [27] | Perumbakkam S, Craig AM. Design and in vitro evaluation of new rpoB-DGGE primers for ruminants[J]. FEMS Microbiology Ecology, 2011, 76(1): 156–169. |
| [28] | Versalovic J, Koeuth T, Lupski R. Distribution of repetitive DNA sequences in eubacteria and application to finerpriting of bacterial enomes[J]. Nucleic Acids Res, 1991, 19(24): 6823–6831. |
| [29] | Chen W, Xie Y, Xu J, et al. Molecular typing of Vibrio parahaemolyticus isolates from the middle-east coastline of China[J]. Int J Food Microbiol, 2012, 153(3): 402–412. |