2. 中国科学院生态环境研究中心城市与区域生态国家重点实验室,北京 100085
2. State Key Laboratory of Urban and Region Ecology, Research Center for Eco-Environmental Sciences, Chinese Academy of Sciences, Beijing 100085
甲烷是一种重要的温室气体,其全球增温潜势是二氧化碳的25倍,我国将甲烷列为重点控制的温室气体之一[1]。研究表明,自前工业化时期以来,大气中甲烷的排放对全球变暖的贡献率约为20%[2]。据估算,全球每年通过自然源和人为源排放至大气的甲烷为500-600 Tg[3],而垃圾填埋场是最为重要的人为甲烷排放源之一,其年甲烷释放量为35-69 Tg,垃圾填埋场的甲烷减排技术受到越来越多的关注[1]。填埋场中大量的可生物降解垃圾是其甲烷释放的主要来源。填埋场的甲烷产量会达到峰值后缓慢下降,但释放量会在其封场后仍然持续几十年[4]。我国的生活垃圾卫生填埋场虽然有导气和填埋气收集装置,但也仅能收集60%-80%的填埋气,仍有大量的甲烷排放到环境中。土壤中的甲烷氧化微生物在维持大气中的甲烷浓度平衡中具有非常重要的作用,有研究表明土壤中的甲烷氧化菌贡献了甲烷氧化总量的5%-15%,是重要的全球甲烷汇之一[5]。填埋场的存量垃圾和表层覆土中都有甲烷氧化菌的存在,因此强化其甲烷氧化功能、削减垃圾填埋场中无组织排放的甲烷,已成为垃圾处理、温室气体减排、生态及环境工程微生物等领域的研究热点[6]。
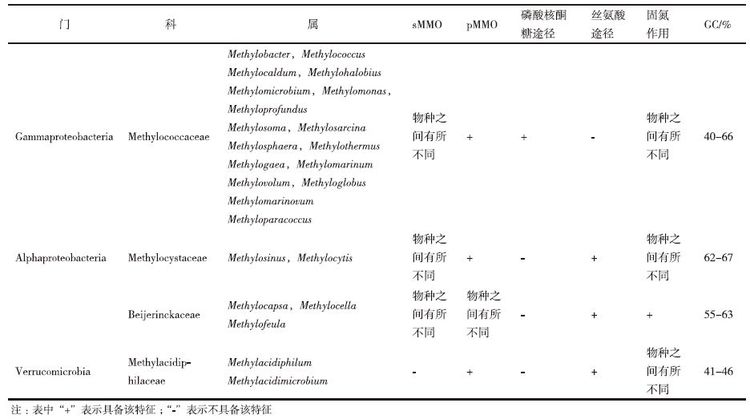
1 甲烷氧化菌类型及其特征甲烷氧化菌是一类以甲烷为唯一碳源和能源的甲基营养型微生物[7],可将甲烷经一系列酶促反应氧化成二氧化碳,于1906 年首次被分离出来。根据代谢途径、膜结构、主要磷脂脂肪酸成分等系列特征,甲烷氧化菌分为TypeⅠ、TypeⅡ和Type X三种类型。TypeⅠ型甲烷氧化菌归属于γ-变形菌门(gammaproteobacteria),通过磷酸核酮糖途径同化甲醛;TypeⅠ型甲烷氧化菌中的Methylococcus和Methylocaldum也被称为Type X型甲烷氧化菌,是一类耐热的甲烷氧化菌[8]。与Ⅰ型一样,X型利用磷酸核酮糖途径同化甲醛。X 型和Ⅰ型的不同之处在于 X型含有低水平的丝氨酸途径酶,生长温度要比Ⅰ型和Ⅱ型高,且DNA中的GC%比大多数Ⅰ型高。而TypeⅡ型甲烷氧化菌归属于α-变形菌门(Alphaproteobacteria),通过丝氨酸途径同化甲醛[9]。随着研究的深入,人们又发现了属于疣微菌门(Verrucomicrobia)的极端嗜酸嗜热甲烷氧化菌[10-14],其同化甲醛的途径也为丝氨酸途径。目前已知的甲烷氧化菌有24个属[15-19],主要类型及其生物学特征,见表 1。虽然甲烷氧化菌被认为是以甲烷为唯一碳源和能源的甲基营养型微生物,但随着研究的深入,科学家发现了Methylocella silvestris[20]等兼性甲烷氧化菌,这些菌除能利用甲烷外,还能利用多碳化合物、乙醇和烷烃作为碳源[21, 22]。
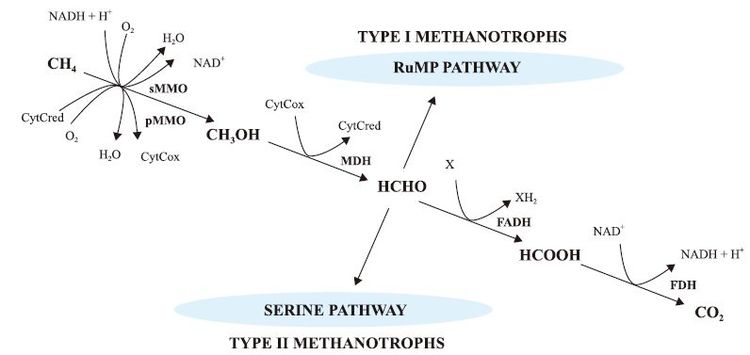
虽然甲烷氧化菌的种类和生存环境多种多样,但通过其生物化学作用氧化甲烷的原理基本一致。Hanson等[7]解析了甲烷的生物氧化机理,如图 1所示。甲烷首先被氧化为甲醇,催化该过程的关键酶是甲烷单加氧酶(methane monooxygenase,MMO),该酶有两种不同类型,一种是颗粒状或膜结合甲烷单加氧酶(particulate methane monooxygenase,pMMO),几乎存在于所有已发现的甲烷氧化菌中;另一种是位于细胞质中的可溶性甲烷单加氧酶(soluble methane monooxygenase,sMMO),仅存在于部分甲烷氧化菌中。虽然它们在细胞内具有相似的功能,但这两种酶的基因和结构明显不同[23]。由于MMO 是甲烷氧化菌的功能酶系,且几乎所有好氧甲烷氧化菌都含有pMMO,因此已广泛利用MMO作为生物标记物进行甲烷氧化菌生态学研究。接着甲烷氧化产生的内源甲醇和由几丁质及木质素降解形成的外源甲醇在甲醇脱氢酶(methanoldehydrogenase,MDH)的作用下氧化成甲醛。然后甲醛在甲醛脱氢酶的作用下氧化成甲酸。同时,Ⅰ型甲烷氧化菌可以利用磷酸核酮糖途径同化甲醛,作为其生长繁殖需要的碳源和能源;Ⅱ型甲烷氧化菌则通过丝氨酸途径完成这个同化作用[24]。最后甲酸在甲酸脱氢酶作用下氧化为二氧化碳。
3 甲烷氧化菌多样性的研究方法甲烷氧化菌多样性研究可以很好地反应甲烷氧化菌在环境样品中的物种丰富度、各类遗传信息及其所执行的功能和过程,为我们全面了解甲烷氧化菌及其氧化降解甲烷的功能奠定了基础。研究甲烷氧化菌多样性的方法主要有培养法、分子生物学法和稳定同位素示踪法[25]。因传统的培养法得到的甲烷氧化菌分离培养物并不能代表填埋土里真正的甲烷氧化菌群落,会造成大量信息的丢失,特别是不可培养的甲烷氧化菌,所以目前研究者多采用分子生物学法和稳定同位素示踪法。
研究甲烷氧化菌常用的分子标记物主要是16S rRNA和功能基因。16S rRNA作为研究最多和最透彻的基因序列,因其高度保守性和特异性,通过高通量测序Hiseq、Miseq等技术,已被广泛地应用于甲烷氧化菌的检测及相关的分子生态学领域。功能基因因其在复杂的环境样品中具有高度敏感性和可识别不可培养的功能微生物的优点也被越来越被广泛使用[26]。pmoA(编码颗粒性甲烷单加氧酶的α-亚基)已在大多数甲烷氧化菌中被测序,是目前研究最为深入的功能基因。另外,mmoX(编码可溶性甲烷单加氧酶)、mxaF(编码甲醇脱氢酶的较大亚基)[27]和nifH(编码固氮酶的关键部分双氮还原酶)[28, 29]等功能基因也较为常用。
末端限制性片段长度多态性分析(terminal restriction fragment length polymorphism,T-RFLP)、变性梯度凝电泳(denaturing gradient gel electrophoresis,DGGE)是常用的分析环境样品中甲烷氧化菌多样性的方法,与实时定量PCR技术结合使用,可以定量地分析环境样品中甲烷氧化菌群落特征。传统的定量方法——最大释然法(most-probable-number,MPN)已被用来探测沼泽[30]、湿地[31]、稻田[32]等环境样品中的甲烷氧化菌的数量,但因其取决于特定培养基中甲烷氧化菌的生长情况,具有很大的偏好性。
随着分子生物技术的发展,许多新兴的技术也相继出现。荧光原位杂交技术(fluorescence in situ hybridization,FISH)是利用带有荧光标记的探针与甲烷氧化菌的特征基因序列进行杂交,来探测同源的核酸序列,最终来鉴定环境样品中甲烷氧化菌的存在及丰度。它具有无需培养和无需单独分离基因的优点,但同时也存在其目标物的16S rRNA需为已知的缺点[26]。
稳定同位素示踪技术(stable isotope probing,SIP)和微生物标记物结合使用,可以有效的分析甲烷氧化菌的群落结构和功能多样性。这项技术主要有DNA-SIP[33]、RNA-SIP[34]、磷脂脂肪酸(PLFA)-SIP[35]和最新的mRNA-SIP[36]。PLFA-SIP由于灵敏度高于DNA-SIP,经常被用于甲烷氧化菌的检测。PLFA是活体微生物细胞膜的重要组分,它在自然生理条件下相对稳定,而通常细胞外的磷脂含量较少,当微生物死亡时,磷脂会被迅速代谢[25]。因土壤中不同微生物的特征性PLFA组成和含量有所不同,所以与稳定同位素示踪技术结合可以指示特定微生物群落的结构特征与生物量。土壤中Ⅰ型甲烷氧化菌的特征性PLFA以14C-PLFA和16C-PLFA为主,而Ⅱ型甲烷氧化菌则以18C-PLFA 为主[37, 38]。但PLFA-SIP方法本身具有一定的局限性,其对微生物种类的判断主要依赖于样品PLFA图谱和已知的纯培养甲烷氧化菌PLFA图谱的比对,难以对环境样品中甲烷氧化菌种类进行精确的鉴定[39]。
另外,以pmoA为目标基因的微阵列(microarray)、基因芯片技术,也已应用于检测和定量垃圾填埋覆土中的甲烷氧化菌[40, 41],它可以清晰地反映垃圾填埋场在不同深度的空间趋势。另外,纳米二次离子质谱(nanoSIMS)[42]、微流体数字PCR[43]等技术也被用来探究甲烷氧化菌群的功能特征。
4 填埋土层中甲烷氧化菌活性影响因素影响垃圾填埋场覆土层甲烷氧化活性的主要因素有土壤成分、含水率、温度、甲烷及氧气浓度、NH4+、土壤pH值等。
4.1 土壤成分增加土壤有机质的含量,可以明显提高甲烷的氧化活性[44]。因有机质可以为甲烷氧化菌提供充足的营养物质,使其数量和活性显著增加[45]。Kightley等[46]通过在填埋覆土中添加富含有机质的活性污泥发现,与对照组相比,甲烷氧化能力提高了26%,而添加无机盐K2HPO4的覆土,甲烷氧化能力变化不大。同时,有研究表明,有机质含量高的生物覆盖层与普通的泥沙覆土相比,前者不仅具有较高的甲烷氧化活性,可达441 µg CH4 h-1· g· dw-1,而且甲烷氧化菌的数量也相对较高[47]。
4.2 含水率当填埋场覆土层含水率达到最适时,气体易于扩散,微生物活性较高,甲烷氧化活性最高;含水率过高时,甲烷和氧气在土壤中的扩散受到限制,使底物供应受阻,从而降低了甲烷氧化能力[48];含水率过低,则会使甲烷氧化菌的活性降低,其甲烷氧化性能显著降低[49]。Scheutz[50]认为填埋场覆土层中甲烷氧化的最佳含水率在25%左右。而Zhang等[51]研究添加活性污泥的填埋覆土发现,含水率与甲烷氧化活性呈倒U型关系,在24%-36%时,达到最大值。
4.3 温度一般认为甲烷氧化的最适温度为25-35℃[49]。研究发现,在2℃时,甲烷氧化作用仍然存在[50, 52],表明即使在冬季甲烷氧化菌仍然可以有效地氧化甲烷,降低垃圾填埋场的甲烷排放量,但冬季垃圾填埋场甲烷的氧化效率要明显低于夏季。Borjesson[53]发现,在低温环境中发现的甲烷氧化菌都属于Ⅰ型,表明温度具有选择效应,可以确定哪种类型的甲烷氧化菌在给定的环境系统中起主导作用。同时,研究表明填埋场受所处气候带的影响,不同气候带中甲烷氧化菌的最适温度也存在差异[54]。
4.4 甲烷及氧气浓度填埋场覆土层内甲烷和氧气的分布是由下层甲烷的产生和上层氧气扩散共同影响的。Li等[55]研究了不同浓度的甲烷对覆土层中甲烷氧化菌的影响发现,当甲烷浓度为5.0 ×104 ppmv和1.0 ×105 ppmv时,甲烷氧化菌的丰富度最高。且随着甲烷和氧气浓度的增高,甲烷氧化活性也不断提高,最大可达98.7 ×1016 mol·cell-1·h-1。Pawlowska[56]与He等[57]研究指出,覆土层内好氧甲烷氧化速率最大的深度在10-20 cm之间,且随着深度不断增加,氧化效率逐渐减小,这应与氧气的浓度减小有关。
4.5 NH4+无机氮对填埋场覆土层中甲烷氧化效果的影响较为复杂,正面或负面的影响都可能存在。现普遍认为NH4+对甲烷氧化有抑制作用[58, 59],其抑制机理可能在于NH4+和CH4具有相似的分子结构,竞争甲烷氧化菌酶系统相同的位点或降低甲烷氧化酶活性,从而起到抑制甲烷氧化的作用[50]。Vissche等[60]研究发现,当甲烷浓度不同时,NH4+起到的作用也不同,低甲烷浓度时,NH4+可起到40%的抑制作用,且NH4Cl和(NH4)2SO4的抑制效果也大体一致,高甲烷浓度时,NH4+则起到促进作用。
4.6 土壤pH值填埋场覆土层甲烷氧化的最适pH值一般取决于土壤的材料。Zhang[61]通过实验发现,在添加了矿化垃圾的覆土中,甲烷氧化最佳pH为6.5-8.0。而甲烷氧化菌在沙质脱钙土壤中要求的pH一般在7以下,最低可到4.5[50]。现一般认为,在自然环境条件下,甲烷氧化菌氧化甲烷的最佳pH为中性,pH值范围在6.5-7.5[51],而纯培养时,甲烷氧化菌的最适宜pH值为6.6-6.8[7]。
4.7 植被填埋场覆土层的植被可以有效提高甲烷氧化效率。原因可能是覆土层植被的根系可疏松土壤,形成气体传输通道,增加气体扩散,从而形成适于甲烷氧化菌生长的环境来提高其甲烷氧化能力[62]。Bohn等[63]在覆土中种植豆科植物发现,与对照组相比,甲烷氧化能力显著提高,这应该与豆科植物的固氮作用有关,同时,他还认为植物的蒸腾作用可以减少垃圾渗滤液,从而调节了覆土的含水率。
5 垃圾填埋场的甲烷生物减排针对垃圾填埋场大部分填埋气不能有效利用、甲烷排放浓度高等环境问题,目前除了对甲烷气体进行收集燃烧或作为燃料开发利用外,研究主要通过生物手段控制甲烷排放,主要包括检测甲烷氧化优势菌,改良垃圾填埋覆土层以扩大甲烷氧化菌培养,抑制产甲烷菌和准好氧填埋等生物减排技术。
5.1 填埋土层中的甲烷氧化优势菌的检测分析Gebert等[64]利用末端限制性片段长度多态性分析技术和微列阵技术研究了德国5个垃圾填埋场的覆土的甲烷氧化菌的群落结构发现,甲烷氧化菌的组成在这5个垃圾填埋场的不同填埋深度都基本相似。Ⅱ型甲烷氧化菌中的Methylocystis和Ⅰ型甲烷氧化菌中的Methylobacter和Methylococcus为优势菌属,这与Stralis-Pavese等[41, 65, 66]的研究结果较为一致。而Chang等[67]通过借助pmoA的基因芯片技术研究种植不同植物的垃圾填埋土发现,Ⅹ型甲烷氧化菌Methylocaldum占主导作用,而Methylocystis在种植光棍树(milk bush)的深层土壤中为优势菌。另外,Singh等[68]研究发现Ⅰ型甲烷氧化菌在种植牧草的垃圾覆土中占主导作用,Ⅱ型甲烷氧化菌在种植灌木林的垃圾覆土中发挥重要作用。这可能与Ⅰ型甲烷氧化菌对氧浓度更加敏感,更喜欢高氧浓度,而Ⅱ型甲烷氧化菌对甲烷浓度更敏感,更适应于高甲烷浓度下生长有关。Lee等[69]研究了实验室模拟垃圾填埋生物覆盖层的甲烷氧化菌,在不同深度土层的样品中,利用荧光定量PCR技术发现甲烷氧化菌的数量无显著差异。在上层(0-10 cm)样品中,Ⅰ型和Ⅱ型甲烷氧化菌都发挥重要作用,而在中层(10-40 cm)和底层(40-50 cm)样品中,Ⅰ型比Ⅱ型甲烷氧化菌更占主导地位。
5.2 甲烷氧化菌的强化根据已知的影响填埋土层中甲烷氧化菌的活性因素,在填埋覆土层添加矿化垃圾、活性污泥等可以有效地强化甲烷氧化菌的生物活性,加快甲烷的氧化速度[70, 71]。矿化垃圾是在填埋场中填埋多年,基本达到稳定化,已可进行开采利用的垃圾,在长期的生物降解过程中,其表面附着了数量庞大、种类繁多、代谢能力极强的微生物群落。因其本身富含甲烷氧化菌(1.25×108-1.25×109 CFU/g),且成本低、氧化效果明显,并可增加再生填埋库容,使填埋场空间循环利用,所以是甲烷氧化覆盖层的一种很好材料[72]。Lou等[73]通过实验发现,添加矿化垃圾和活性污泥,甲烷去除率分别可达78.7%和66.9%,且发现当用14年以上的矿化垃圾处理时,效果最好。Zhang等[51]研究发现,以6:4的比例添加矿化垃圾和活性污泥到覆土中,甲烷氧化效率可达到最佳。此外,研究者还发现粉煤灰、陶粒等材料含水率低、孔穴丰富、比表面积大的优点[74],可添加到矿化垃圾和活性污泥中,配合使用来增强甲烷氧化效果。另外,种植植物也是改良填埋覆土的一种有效方法,目前填埋场覆土种植的植物主要集中在芒草、苜蓿和杨树等[63, 65, 75]。有研究发现,利用矿化垃圾中的硫酸盐还原菌进行甲烷的共氧化是甲烷自然减排的另一种途径[76]。
甲烷氧化菌除了能够氧化甲烷作为生长所必需的碳源和能量外,由于甲烷单加氧酶的底物非特异性,使得还可以氧化一些碳氢化合物及部分卤代烃。因垃圾填埋气中除了甲烷和二氧化碳外,还有约占填埋气总体积1%的各种非甲烷有机气体,如氯乙烯、苯及甲苯等,且大多有毒。所以甲烷氧化菌的这一性质对于垃圾填埋气中非甲烷有机气体的去除也有着重要意义。另外,对甲烷氧化菌的开发利用也取得了一定进展,有报道利用甲烷氧化混合菌合成聚β-羟基丁酸酯(PHB)[77](一种生物可降解的仿生塑料)、利用混合菌为催化剂实现丙烯氧化生产环氧丙烷[78]、去除水体中的污染物三氯乙烯[79]以及废水脱氮[80]等。
5.3 产甲烷菌的抑制与甲烷氧化菌不同,产甲烷菌是一类能够将有机或无机化合物经厌氧消化作用转化成甲烷和二氧化碳的古细菌,为严格厌氧菌,生存条件较为苛刻,也是垃圾填埋场甲烷气体的主要生产菌。目前,研究主要通过添加甲烷抑制剂多为多卤素化合物,如氯代甲烷、三氯乙炔、溴氯甲烷等抑制产甲烷菌的代谢活性,从而减少甲烷释放量。Zhao等[81]通过餐厨垃圾厌氧发酵过程中甲烷抑制的实验发现,同样浓度的二氯甲烷、氯仿和四氯化碳,氯仿的抑制作用要远远强于二氯甲烷和四氯化碳。当浓度都为20 mg/kg时,氯仿对甲烷的抑制率可达到98.1%,而四氯化碳和二氯甲烷的抑制率分别为42.7%和43.2%。虽然,卤素化合物对产甲烷菌有较好的抑制作用,但因其本身具有毒性,实际应用时,还具有一定局限性。对填埋场进行好氧通风也是抑制微生物产甲烷的有效途径之一,准好氧填埋是代表性的技术之一[82]。准好氧填埋是通过渗滤液收集管的不满流设计,在填埋场内外温差的作用下使空气自然通过渗滤液收集管末端进入填埋场,从而使得垃圾内部部分区域处于好氧状态,使垃圾降解的速度得到提高,并且减少了甲烷的排放[72]。
6 结语甲烷氧化菌在甲烷减排、全球温室气体控制及碳、氮、硫等元素的地球生物化学循环中都起到了非常重要的作用。垃圾填埋领域的甲烷氧化研究正在受到越来越广泛的关注。目前国内外对于甲烷氧化菌的研究主要集中在好氧甲烷氧化菌、氧化机理、填埋场覆土应用等方面,以下几方面还有待进一步的探索。
6.1 垃圾填埋甲烷氧化复合微生物菌剂研究微生物菌剂在促进有机物发酵、除臭、降解污染物等方面具有广泛的应用,但目前还没有甲烷氧化菌的复合微生物菌剂的应用。在传统的分离培养方法中,甲烷氧化菌生长非常缓慢、细胞密度低、发酵周期较长,很难满足工业上的大规模生产。这就需要对甲烷氧化菌的培养工艺进行更深入的研究,强化氧气、甲烷传递速率,提高甲烷氧化菌的生长速度和酶活稳定性,为开发处理甲烷的微生物菌剂奠定基础。另外,除臭菌剂主要是靠微生物降解作用去除NH3,即以NH4+的形态被微生物吸收,作为营养物质通过新陈代谢作用为微生物所分解、利用,最终将其氧化分解为NO2-、NO3-等。而NH4+和CH4因具有相似的分子结构,可以竞争甲烷氧化菌酶系统相同的位点或降低甲烷氧化酶活性,起到抑制甲烷氧化作用,所以可利用除氨气的微生物和甲烷氧化菌相结合,开发同时处理甲烷与氨气的复合微生物菌剂,以解决垃圾填埋场温室气体排放和恶臭污染的问题。
6.2 甲烷氧化菌的互生关系研究及其应用目前已有研究表明,甲烷氧化菌同氨氧化菌、填埋土表面植物之间存在某种互生关系,例如氨氧化菌可以削减NH4+以减少其对甲烷氧化功能的抑制,填埋土表面植物可以为甲烷氧化菌提供必要的好氧环境。目前常规的实验技术手段和方法,较难分离得到纯种甲烷氧化菌,这可能预示着在土壤生态系中,甲烷氧化菌与其他生物或环境之间存在着密切的依存关系,它们对甲烷的协同作用,降解机理还不是十分清楚。在未来的研究中,分子技术、同位素示踪等新的研究手段,有望进一步解析甲烷氧化菌同其他微生物或植物间的互生、共生、拮抗关系,对于掌握其耦合作用机制,提升甲烷协同氧化能力,具有非常重要的理论和应用价值。
6.3 厌氧型甲烷氧化菌的机理与应用研究生活垃圾填埋场堆体内部主要是厌氧环境,而厌氧是甲烷产生和释放的必要条件。目前除了通过管道通风换气,抑制厌氧发酵甲烷的产生,同时促进好氧甲烷氧化菌对甲烷的分解外,则主要通过好氧型甲烷氧化菌及填埋层覆土材料的选择与工程优化等方面进行综合甲烷减排。厌氧甲烷氧化菌的作用机理研究刚刚起步,也仅有较少的菌株被筛选出来。有研究表明,厌氧甲烷氧化古菌可参与到有机废水的“反硝化”过程,为反硝化细菌还原 NO2-和NO3-提供电子和所需物质等,帮助其最终完成反硝化过程,亦可达到甲烷减排的目的。因此,厌氧甲烷氧化菌在填埋场的工程应用研究同样具有一定的开发前景。
| [1] | IP CC. Climate Change 2007:the Physical Science Basis. Contribution of Working Group I to the Fourth Assessment Report of the Intergovernmental Panel on Climate Change[M]. Cambridge and New York: Cambridge University Press, 2007. |
| [2] | Kirschke S, Bousquet P, Ciais P, et al. Three decades of global methane sources and sinks[J]. Nature Geoscience, 2013, 6(10): 813–823. |
| [3] | Conrad R. The global methane cycle:recent advances in understanding the microbial processes involved[J]. Environmental Microbiology Reports, 2009, 1(5): 285–292. |
| [4] | Albanna M, Warith M, Fernandes L. Kinetics of biological methane oxidation in the presence of non-methane organic compounds in landfill bio-covers[J]. Waste Manage, 2010, 30(2): 219–227. |
| [5] | Aronson E, Allison S, Helliker BR. Environmental impacts on the diversity of methane-cycling microbes and their resultant function[J]. Frontiers in Microbiology, 2013, 4: 225. |
| [6] | 梅娟, 赵由才. 填埋场甲烷生物氧化过程及甲烷氧化菌的研究进展[J]. 生态学杂志, 2014, 33(9): 2567–2573. |
| [7] | Hanson RS, Hanson TE. Methanotrophic bacteria[J]. Microbiological Reviews, 1996, 60(2): 439–471. |
| [8] | 贠娟莉, 王艳芬, 张洪勋. 好氧甲烷氧化菌生态学研究进展[J]. 生态学报, 2013, 33(21): 6774–6785. |
| [9] | Semrau JD, Dispirito AA, Yoon S. Methanotrophs and copper[J]. FEMS Microbiology Reviews, 2010, 34(4): 496–531. |
| [10] | Dunfield PF, Yuryev A, Senin P, et al. Methane oxidation by an ext-remely acidophilic bacterium of the phylum Verrucomicrobia[J]. Nature, 2007, 450(7171): 879–882. |
| [11] | Pol A, Heijmans K, Harhangi HR, et al. Methanotrophy below pH 1 by a new Verrucomicrobia species[J]. Nature, 2007, 450(7171): 874–878. |
| [12] | Islam T, Jensen S, Reigstad LJ, et al. Methane oxidation at 55℃ and pH 2 by a thermoacidophilic bacterium belonging to the Verrucomicrobia phylum[J]. Proce Natl Acad Sci USA, 2008, 105(1): 300–304. |
| [13] | Op den Camp HJ, Islam T, Stott MB, et al. Environmental genomic and taxonomic perspectives on methanotrophic Verrucomicrobia[J]. Environmental Microbiology Reports, 2009, 1(5): 293–306. |
| [14] | van Teeseling MC, Pol A, Harhangi HR, et al. Expanding the verrucomicrobial methanotrophic world:description of three novel species of Methylacidimicrobium gen. nov[J]. Applied and Environmental Microbiology, 2014, 80(21): 6782–6791. |
| [15] | Kalyuzhnaya MG, Puri AW, Lidstrom ME. Metabolic engineering in methanotrophic bacteria[J]. Metab Eng, 2015, 29: 142–152. |
| [16] | Deutzmann JS, Hoppert M, Schink B. Characterization and phylogeny of a novel methanotroph, Methyloglobulus morosus gen. nov., spec. nov.[J]. Syst Appl Microbiol, 2014, 37(3): 165–169. |
| [17] | Tavormina PL, Hatzenpichler R, McGlynn S, et al. Methyloprofu-ndus sedimenti gen. nov., sp. nov., an obligate methanotroph from ocean sediment belonging to the'deep sea-1'clade of marine methanotrophs[J]. Int J Syst Evol Microbiol, 2015, 65(1): 251–259. |
| [18] | Hirayama H, Abe M, Miyazaki M, et al. Methylomarinovum caldi-curalii gen. nov., sp. nov., a moderately thermophilic methanotroph isolated from a shallow submarine hydrothermal system, and proposal of the family Methylothermaceae fam. Nov[J]. Int J Syst Evol Microbiol, 2014, 64(3): 989–999. |
| [19] | Hoefman S, van der Ha D, Iguchi H, et al. Methyloparacoccus mur-rellii gen. nov., sp. nov., a methanotroph isolated from pond water[J]. Int J Syst Evol Microbiol, 2014, 64(6): 2100–2107. |
| [20] | Theisen AR, Ali MH, Radajewski S, et al. Regulation of methane oxidation in the facultative methanotroph Methylocella silvestris BL2[J]. Molecular Microbiology, 2005, 58(3): 682–692. |
| [21] | Semrau JD, DiSpirito AA, Vuilleumier S. Facultative methanotrophy:falseleads, true results, and suggestions for future research. FEMS Microbiology Letters, 2011, 323(1):1-12. |
| [22] | Dunfield PF, Dedysh SN. Methylocella:a gourmand among methanotrophs[J]. Trends Microbiol, 2014, 22(7): 368–369. |
| [23] | Murrell JC, Gilbert B, McDonald IR. Molecular biology and regulation of methane monooxygenase[J]. Archives of Microbiology, 2000, 173(5-6): 325–332. |
| [24] | Chang YS, HaIsey JL. Detection of methanotrophs in groundwater by PCR[J]. Applied and Environmental Microbiology, 1999, 65(2): 648–651. |
| [25] | 黄梦青, 张金凤, 杨玉盛, 等. 土壤甲烷氧化菌多样性研究方法进展[J]. 亚热带资源与环境学报, 2013, 8(2): 42–48. |
| [26] | McDonald IR, Bodrossy L, Chen Y, et al. Molecular ecology techniques for the study of aerobic methanotrophs[J]. Applied and Environmental Microbiology, 2008, 74(5): 1305–1315. |
| [27] | McDonald IR, Murrell JC. The methanol dehydrogenase structural gene mxaF and its use as a functional gene probe for methanotrophs and methylotrophs[J]. Applied and Environmental Microbiology, 1997, 63(8): 3218–3224. |
| [28] | Auman AJ, Speake CC, Lidstrom M E. nifH sequences and nitrogen fixation in type Ⅰ and typeⅡ methanotrophs[J]. Applied and Environmental Microbiology, 2001, 67(9): 4009–4016. |
| [29] | Dedysh SN, Ricke P, Liesack W. NifH and NifD phylogenies:an evolutionary basis for understanding nitrogen fixation capabilities of methanotrophic bacteria[J]. Microbiology, 2004, 150(5): 1301–1313. |
| [30] | Daniel NM, Joseph BY, Eugene LM, et al. Methanotrophic activity, abundance, and diversity in forested swamp pools:dynamics and influences on methane fluxes[J]. Geomicrobiology Journal, 2004, 21(4): 257–271. |
| [31] | Horz HP, Raghubanshi AS, Heyer J, et al. Activity and community structure of methane-oxidizing bacteria in a wet meadow soil[J]. FEMS Microbiology Ecology, 2002, 41(3): 247–257. |
| [32] | Bosse U, Frenzel P. Activity and distribution of methane oxidizing bacteria in flooded rice soil microcosms and in rice plants(Oryzasativa)[J]. Applied and Environmental Microbiology, 1997, 63(4): 1199–1207. |
| [33] | Radajewski S, Ineson P, Parekh NR, et al. Stable-isotope probing as a tool in microbial ecology[J]. Nature, 2000, 403(6770): 646–649. |
| [34] | Manefield M, Whiteley AS, Griffiths RI, et al. RNA stable isotope probing, a novel means of linking microbial community function to phylogeny[J]. Applied and Environmental Microbiology, 2002, 68(11): 5367–5373. |
| [35] | Bull ID, Parekh NR, Hall GH, et al. Detection and classification of atmospheric methane oxidizing bacteria in soil[J]. Nature, 2000, 405(6783): 175–178. |
| [36] | Dumont MG, Pommerenke B, Casper P, et al. DNA-, rRNA-and mRNA-based stable isotope probing of aerobic methanotrophs in lake sediment[J]. Environmental Microbiology, 2011, 13(5): 1153–1167. |
| [37] | Bowman JP, Sly LI, Nichols PD, et al. Revised taxonomy of the methanotrophs:Description of Methylobacter gen. nov. emendation of Methylococcus, validation of Methylosinus and Methylocystis species, and a proposal that the family Methylococcaceae includes only the group Ⅰ methanotrophs[J]. Int J Syst Bacteriol, 1993, 43(4): 735–753. |
| [38] | Dedysh SN, Khmelenina VN, Suzina NE, et al. Methylocapsa acidiphila gen. nov., sp. nov., a novel methane-oxidizing and dinitrogen-fixing acidophilic bacterium from Sphagnum bog[J]. Int J Syst Bacteriol, 2002, 52(1): 251–261. |
| [39] | 蔡元锋, 贾仲君. 土壤大气甲烷氧化菌研究进展[J]. 微生物学报, 2014, 54(8): 841–853. |
| [40] | Cebron A, Bodrossy L, Stralis-Pavese N, et al. Nutrient amendments in soil DNA stable isotope probing experiments reduce the observed methanotroph diversity[J].` Applied and Environmental Microbiology, 2007, 73(3): 798–807. |
| [41] | Stralis-Pavese N, Sessitsch A, Weilharter A, et al. Optimization of diagnostic microarray for application in analyzing landfill methanotroph communities under different plant covers[J]. Environmental Microbiology, 2004, 6(4): 347–363. |
| [42] | Li TL, Wu TD, Mazéas L, et al. Simultaneous analysis of microbial identity and function using NanoSIMS[J]. Environmental Microbiology, 2008, 10(3): 580–588. |
| [43] | Hashsham SA, Gulari E, Tiedje JM. Microfluidic systems being adapted for microbial, molecular biological analyses[J]. Microbe-American Society for Microbiology, 2007, 2(11): 531–536. |
| [44] | Sadasivam BY, Reddy KR. Landfill methane oxidation in soil and bio-based cover systems:a review[J]. Reviews in Environmental Science and Bio/Technology, 2014, 13(1): 79–107. |
| [45] | Scheutz C, Mosbaek H, Kjeldsen P. Attenuation of methane and volatile organic compounds in landfill soil covers[J]. Journal of Environmental Quality, 2004, 33(1): 61–71. |
| [46] | Kightley D, Nedwell DB, Cooper M. Capacity for methane oxidation in landfill cover soils measured in laboratory scale soil microcosms[J]. Appl Environ Microbiol, 1995, 61(2): 592–601. |
| [47] | Ait-Benichou S, Jugnia LB, Greer CW, et al. Methanotrophs and methanotrophic activity in engineered landfill biocovers[J]. Waste Management, 2009, 29(9): 2509–2517. |
| [48] | Cabral AR, Tremblay P, Lefebvre G. Determination of the diffusion coefficient of oxygen for a cover system including a pulp and paper by-product[J]. Geotechnical Testing Journal, 2004, 27(2): 184–197. |
| [49] | Scheutz C, Kjeldsen P, Bogner JE, et al. Microbial methane oxidation processes and technologies for mitigation of landfill gas emissions[J]. Waste Management Research, 2009, 27(5): 409–455. |
| [50] | Scheutz C, Kjeldsen P. Environmental factors influencing attenuation of methane and hydrochlorofluorocarbons in landfill cover soils[J]. J Environ Qual, 2004, 33(1): 72–79. |
| [51] | Zhang HH, Yan XF, Cai BC, et al. The effects of aged refuse and sewage sludge on landfill CH4 oxidation and N2O emissions:Roles of moisture content and temperature[J]. Ecological Engineering, 2015, 74: 345–350. |
| [52] | Christophersen M, Linderod L, Jensen PE, et al. Methane oxidation at low temperatures in soil exposed to landfill gas[J]. Journal of Environmental Quality, 2000, 29(6): 1989–1997. |
| [53] | Borjesson G, Sundh I, Svensson B. Microbial oxidation of CH4 at different temperatures in landfill cover soils[J]. FEMS Microbiology Ecology, 2004, 48(3): 305–312. |
| [54] | Christopherson M, Kjeldsen P, Holst H, et al. Lateral gas transport in soil adjacent to an old landfill:Factors governing emissions and methane oxidation[J]. Waste Management and Research, 2001, 19(6): 595–612. |
| [55] | Li H, Chi ZF, Lu WJ, et al. Sensitivity of methanotrophic community structure, abundance, and gene expression to CH4 and O2 in simulated landfill biocover soil[J]. Environmental Pollution, 2014, 184: 347–353. |
| [56] | Pawlowska M, Stepniewski W. Biochemical reduction of methane emissions from landfills[J]. Environmental Engineering Science, 2006, 23(4): 666–672. |
| [57] | He PJ, Yang N, Fang WJ, et al. Interaction and independence on methane oxidation of landfill cover soil among three impact factors:water, oxygen and ammonium[J]. Frontiers of Environmental Science and Engineering in China, 2011, 5(2): 175–185. |
| [58] | Bender M, Conrad R. Microbial oxidation of methane, ammonium and carbon monoxide, and turnover of nitrous oxide and nitric oxide in soils[J]. Biogeochemistry, 1994, 27(2): 97–112. |
| [59] | King GM, Schnell S. Ammonium and nitrite inhibition of methane oxidation by Methylobacter albus BG8 and Methylosinus trichosporium OB3b at low methane concentrations[J]. Applied and Environmental Microbiology, 1994, 60(10): 3508–3513. |
| [60] | Visscher AD, Cleemput OV. Induction of enhanced CH4 oxidation in soils:NH4+ inhibition patterns[J]. Soil Biology and Biochemistry, 2003, 35(7): 907–913. |
| [61] | Zhang HH, Li WX, Cai ZC, et al. Landfill CH4 oxidation and N2O emissions by aged refuse:effects of wastewater NH4+-N incubation, heavy metals and pH[J]. Ecological Engineering, 2013, 53: 243–249. |
| [62] | Keppler F, Hamilton JT, Brass M, et al. Methane emissions from terrestrial plants under aerobic conditions[J]. Nature, 2006, 439(7073): 187–191. |
| [63] | Bohn S, Brunke P, Gebert J, et al. Improving the aeration of critical fine-grained landfill top cover material by vegetation to increase the microbial methane oxidation efficiency[J]. Waste Management, 2011, 31(5): 854–863. |
| [64] | Gebert J, Singh BK, Pan Y, et al. Activity and structure of methanotrophic communities in landfill cover soils[J]. Environmental Microbiology Reports, 2009, 1(5): 414–423. |
| [65] | Stralis-Pavese N, Bodrossy L, Reichenauer TG, et al. 16S rRNA based T-RFLP analysis of methane oxidising bacteria:assessment, critical evalultion of methodology performance and application for landfill site cover soils[J]. Applied Soil Ecology, 2006, 31(3): 251–266. |
| [66] | Hery M, Singer AC, Kumaresan D, et al. Effect of earthworms on the community structure of active methanotrophic bacteria in a landfill cover soil[J]. The ISME Journal, 2008, 2(1): 92–104. |
| [67] | Chang CY, Tung HH, Tseng IC, et al. Dynamics of methanotrophic communities in tropical alkaline landfill upland soil[J]. Applied Soil Ecology, 2010, 46(2): 192–199. |
| [68] | Singh BK, Tate KR, Kolipaka G, et al. Effect of afforestation and reforestation of pastures on the activity and population dynamics of methanotrophic bacteria[J]. Applied and Environmental Microbiology, 2007, 73(16): 5153–5161. |
| [69] | Lee EH, Moon KE, Kim TG, et al. Depth profiles of methane oxidation potentials and methanotrophic community in a lab-scale biocover[J]. Journal of Biotechnology, 2014, 184: 56–62. |
| [70] | Han D, Zhao YC, Xue BJ, et al. Effect of bio-column composed of aged refuse on methane abatement-A novel configuration of biological oxidation in refuse landfill[J]. Journal of Environmental Sciences, 2010, 22(5): 769–776. |
| [71] | Contin M, Goi D, Nobili MD. Land application of aerobic sewage sludge does not impair methane oxidation rates of soils[J]. Science of the Total Environment, 2012, 441: 10–18. |
| [72] | 赵由才, 赵天涛, 韩丹, 等. 生活垃圾卫生填埋场甲烷减排与控制技术研究[J]. 环境污染与防治, 2009, 31(12): 48–52. |
| [73] | Lou ZY, Wang L, Zhao YC. Consuming un-captured methane from landfill using aged refuse bio-cover[J]. Bioresource Technology, 2011, 102(3): 2328–2332. |
| [74] | Wang JM, Ban H, Teng XJ, et al. Impacts of pH and ammonia on the leaching of Cu(Ⅱ)and Cd(Ⅱ)from coal fly ash[J]. Chemosphere, 2006, 64(11): 1892–1898. |
| [75] | Reichenauer TG, Watzinger A, Riesinga J, et al. Impact of different plants on the gas profile of a landfill cover[J]. Waste Management, 2011, 31(5): 843–853. |
| [76] | 韩丹, 石峰, 柴晓利, 等. 生活垃圾填埋场甲烷自然减排的新途径:厌氧与好氧的共氧化作用[J]. 环境科学学报, 2011, 31(4): 791–797. |
| [77] | Rostkowski KH, Pfluger AR, Criddle CS. Stoichiometry and kinetics of the PHB-producing Type Ⅱ methanotrophs Methylosinus trichosporium OB3b and Methylocystis parvus OBBP[J]. Bioresource Technology, 2013, 132: 71–77. |
| [78] | Xin JY, Cui JR, Chen JB. Continuous biocatalytic synthesis of epoxypropane using a biofilm reactor[J]. Process Biochemistry, 2003, 38(12): 1739–1746. |
| [79] | van Hylckama Vlieg JE, Janssen DB. Formation and detoxification of reactive intermediates in the metabolism of chlorinated ethenes[J]. Journal of Biotechnology, 2001, 85(2): 81–102. |
| [80] | Zhu GB, Jetten MSM, Kuschk P, et al. Potential roles of anaerobic ammonium and methane oxidation in the nitrogen cycle of wetland ecosystems[J]. Appl Microbiol Biotechnol, 2010, 86: 1043–1055. |
| [81] | Zhao TT, Zhang LJ, Chen HQ, et al. Co-inhibition of methanogens for methane mitigation in biodegradable wastes[J]. Journal of Environmental Sciences, 2009, 21(6): 827–833. |
| [82] | Chong TL, Matsufuji Y, Hassan MN. Implementation of the semi-aerobic landfill system(Fukuoka method)in developing countries:A Malaysia cost analysis[J]. Waste Management, 2005, 25(7): 702–711. |