2. 浙江省庆元县水利局水产技术推广站,庆元 323800;
3. 上海海洋大学,上海 201306
2. Extension Station of Fisheries Technology,Water Resources Bureau of Qingyuan County,Zhejiang Province,Qingyuan 323800;
3. Shanghai Ocean University,Shanghai 201306
角蛋白是一种细胞骨架蛋白,属于中间纤维蛋白(intermediate filament proteins,IF蛋白),并进一步组装成中间纤维(intermediate filament,IF)。中间纤维(IF)、微丝、微管等是细胞骨架的主要成份[1, 2],对维持细胞的一定形态,保持细胞的适应稳定性起着至关重要的作用[1, 3]。IF蛋白构成了一个庞大的多基因家族,目前,已经确定在人体中包含60多个不同基因[4],70多种家族成员[4, 5]。在几乎所有细胞骨架体系中1/3是中间纤维(IF),而最突出的是角蛋白[6],约占IF的75%[5, 7],是IF蛋白中迄今最复杂的一组蛋白[8, 9]。角蛋白不仅是上皮细胞骨架的组成部分,而且还充当分化标记蛋白[8],确定上皮细胞类型以及代谢终端分化和胚胎发育过程中分化的状态;也有助于确定上皮肿瘤的来源,某些情况下,是良性和恶性上皮肿瘤的预后标志物。为此,角蛋白在分化标记、疾病预防、临床等方面具有重要意义[10]。角蛋白18(keratin 18,K18)是角蛋白的一种,目前,刘红山等[11]研究了K18的免疫特异性,Knudson等[12]研究了角蛋白的同位素分析,席庆[13]、魏飞力等[14]研究了K18单克隆抗体的制备鉴定及应用,朱汇等[15]已研究了角蛋白基因启动子,崔新洁等[16]则运用K18等抗体的表达研究上皮细胞的培养或鉴定。此外,迄今为止,斑马鱼(Danio rerio)、鲨鱼(Scyliorhinus stellaris)、虹鳟(Oncorhynchus mykiss)、金鱼(Carassius auratus)、稀有鮈鲫(Gobiocypris rarus)等多种鱼类K18基因cDNA已被成功克隆[3, 8, 17, 18],但有关泥鳅(Misgurnus anguillicaudatus)K18基因的研究尚未见报道。
泥鳅(Misgurnus anguillicaudatus),俗称:真泥鳅、本地泥鳅等,隶属鲤形目(Cypriniformes)、鳅科(Cobitidae)、泥鳅属(Misgurnus),其适应性强、分布广、数量多、杂食性,同时,营养丰富、肉质细嫩、味道鲜美,具较高的药用价值,是重要的小型经济淡水鱼类。目前,泥鳅的研究基本集中在繁殖、育苗、养殖、生长、饵料、发育等领域[19, 20],有关分子生物学的研究较少。该研究采用3'-和5'-RACE法,克隆泥鳅K18基因cDNA全长序列,并对其序列特征进行分析,同时运用实时荧光定量PCR(RTQ-PCR)技术研究其在不同组织中的表达,旨在了解K18基因序列特征及其表达,为泥鳅的分子生物学研究提供参考和依据。
1 材料与方法 1.1 材料 1.1.1 实验动物所用健康泥鳅,采自浙江省淡水水产研究所的综合实验基地,取泥鳅的肠、肌肉、心脏、胃、肝脏、精巢、卵巢、脾脏等组织,迅速放入液氮冷冻,随后保存于-80℃备用。
1.1.2 主要试剂TRIzol ® Reagent购自美国Invitrogen公司。RACE试剂盒购自Clontech公司。DEPC购自上海生工生物技术有限公司。M-MLV RT-ase cDNA反转录试剂盒、10×Ex Taq Buffer(Mg2+ Plus)、dNTP Mixture(2.5 mmol/L)、Ex Taq(5 U/μL)、DNA回收试剂盒、pMD18-T载体试剂盒、SYBR ® Premix Ex TaqTM Ⅱ试剂盒均购自TaKaRa公司。
1.2 方法 1.2.1 总RNA的提取及反转录合成cDNA按照TRIzol® Reagent一步法提取各组织总RNA,用核酸蛋白测定仪检测总RNA的浓度和质量,同时用琼脂糖凝胶电泳检测其完整性。之后,取各组织总RNA 500 ng按照M-MLV RTase cDNA反转录试剂盒说明,反转录合成cDNA,保存于-20℃备用。
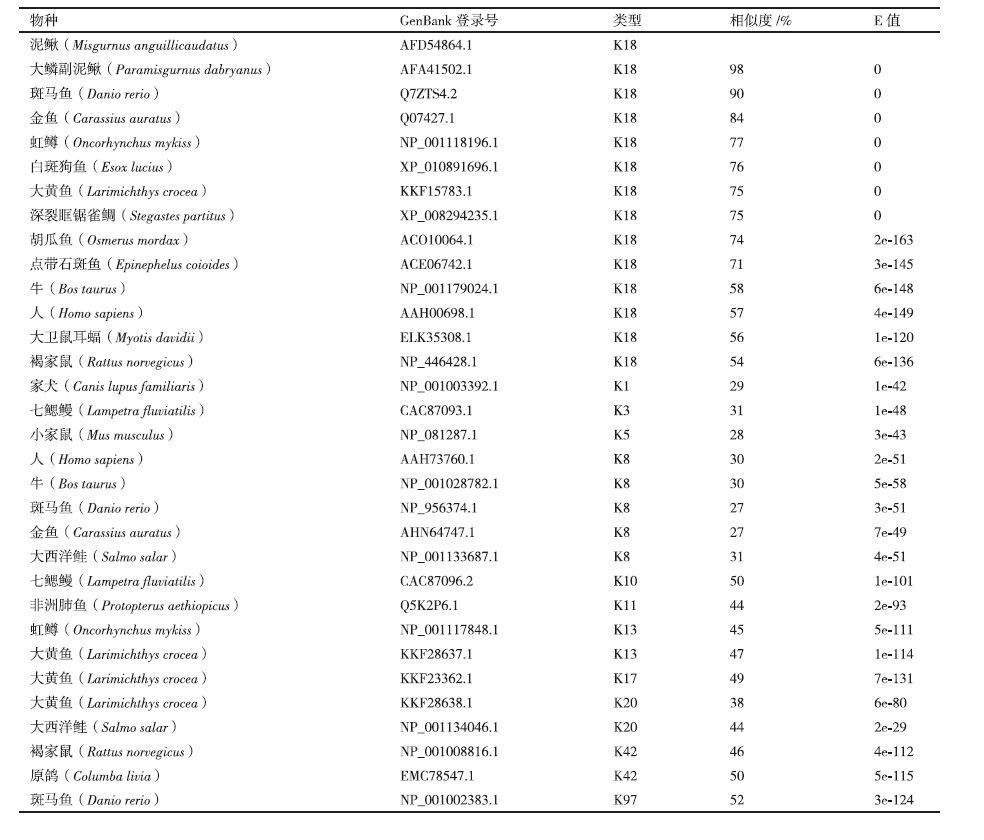
1.2.2 泥鳅K18基因部分cDNA序列扩增根据斑马鱼(GenBank登录号:NM_178437.2),虹鳟(GenBank登录号:NM_001124724.1),和稀有鮈鲫(上海海洋大学蔡生力教授提供)K18基因序列,设计兼并引物cDNA-F和cDNA-R(表 1,图 1),进行PCR扩增泥鳅K18部分cDNA序列,模板为总RNA浓度和质量检测最高的卵巢cDNA。反应体系50 μL,PCR程序:95 ℃预变性3 min进入循环;95 ℃变性30 s,53 ℃退火30 s,72 ℃延伸1 min 30 s,共30个循环;最后72 ℃延伸8 min,4 ℃结束程序。PCR产物按照DNA回收试剂盒纯化回收。之后,按照pMD18-T载体试剂盒说明,连接pMD18-T载体,再转化入DH5α感受态细胞,经培养,PCR检测后筛选阳性克隆,送上海桑尼生物技术有限公司测序。测序所得序列由NCBI的BLAST比对检验,以初步确定是否为K18基因部分cDNA序列。
|
| 图 1 引物在序列中的位置 |
根据已确定得到K18基因部分cDNA序列设计RACE引物GSP-K18-5'和GSP-K18-3'(表 1,图 1),按照RACE试剂盒说明,反转录合成总RNA浓度和质量检测最高的卵巢cDNA,作为模板,分别进行3'-和5'-RACE扩增。反应体系25 μL,扩增程序:94℃ 30 s,72℃ 3 min,共5个循环;94℃ 30 s,70℃ 30 s,72℃ 3 min,共5个循环;94℃ 30 s,68℃ 30 s,72℃ 3 min,共30个循环。扩增产物经电泳分离检测,按照1.2.2方法,进行回收纯化,连接载体,转化感受态细胞,筛选阳性克隆,送上海桑尼生物科技有限公司测序。
1.2.4 泥鳅K18序列分析根据测序结果进行拼接得到泥鳅K18基因的cDNA全长序列,应用ORF Finder 程序(http://www.ncbi.nlm.nih.gov/projects/gorf/),确定开放阅读框,翻译氨基酸序列,并进行NCBI的BLAST比对(http://blast.ncbi.nlm.nih.gov/)。预测氨基酸的物理参数用ExPASy在线Protparam 程序(http://web.expasy.org/protparam/),预测二硫键用Scratch程序(http://www.ics.uci.edu/~baldig/scratch/index.html),分析跨膜结构为TMHMM程序(http://www.cbs.dtu.dk/services/TMHMM/),预测信号肽用SignalP程序(http://www.cbs.dtu.dk/services/SignalP/),预测蛋白质二级结构用bioinf程序(http://bioinf.cs.ucl.ac.uk)分析结合Predictprotein程序(https://www.predictprotein.org/),分析蛋白质结构域用SMART程序(http://smart.embl-heidelberg.de/)分析结合NCBI的Conserved Domains(CD-Search)程序(http://www.ncbi.nlm.nih.gov/cdd/),预测蛋白质三级结构用SWISS-MODEL程序(http://www.swissmodel.expasy.org/)[21-23]。用ClustalX 和DNAMAN软件对经BLAST比对的泥鳅K18与其他物种角蛋白氨基酸序列(表 2)进行多重序列比对分析。然后用MEGA软件,采取邻位相接法(neighbor-joining,NJ),以Bootstrap重复1000次计算各分枝的置信度,构建泥鳅K18与其他物种角蛋白(表 2)的NJ系统进 化树。
根据上述已克隆的泥鳅K18基因cDNA全长序列,设计定量PCR特异性引物K18-F和K18-R(表 1,图 1)。以泥鳅β-actin基因为内参,设计内参引物β-actin-F和β-actin-R(表 1)。用于校正。以各组织合成的cDNA为模板,按照SYBR® Premix Ex TaqTM Ⅱ试剂盒说明,进行RTQ-PCR检测K18基因组织差异,PCR程序:95℃ 4 min进入循环;95℃ 30 s,55℃ 30 s,72℃ 30 s,共40个循环;72℃延伸8 min。采用2-△△Ct方法计算每个样品组织目的基因的相对表达量。
1.2.6 统计分析运用软件SPSS对泥鳅K18基因相对表达量的计算结果,以单因素方差分析法(one-way ANOVA)的多重比较等分析各组织基因表达的差异显著性。统计学上以P<0.05为显著性差异,P<0.01为极显著性差异。
2 结果 2.1 泥鳅K18基因的cDNA全长序列克隆得到泥鳅K18基因cDNA全长序列1 694 bp,其中,包含5'-非翻译区(5'-untranslated region, 5'-UTR)81 bp和3'-非翻译区(3'-untranslated reg-ion,3'-UTR)314 bp,开放阅读框1 299 bp。3'-UTR有1个多聚腺苷酸加尾信号位点(AATAAA)和1个多聚A尾巴(poly A tail)(图 2)。所得泥鳅K18基因cDNA全长序列提交于NCBI的GenBank中,登录号为JQ269652.1。

|
| 图 2 泥鳅K18基因cDNA全长序列及其推导的氨基酸序列 小写字母代表3'、5'非翻译区;大写字母代表开放阅读框编码区,其中,上方是核苷酸序列,下方是氨基酸序列;方框黑体代表多聚腺苷酸加尾信号位点AATAAA;*表示终止密码子;虚下划线表示N-端重复的甘氨酸(G);方框表示C-端富含的丝氨酸(S)、苏氨酸(T)和缬氨酸(V);下划线表示3个螺旋卷曲区域;阴影表示保守的IF蛋白结构域 |
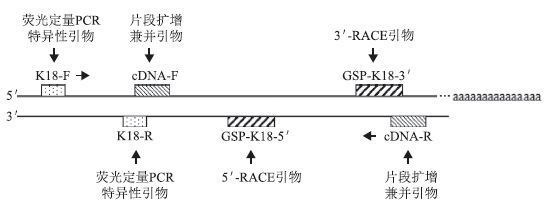
泥鳅K18基因cDNA全长序列开放阅读框共编码432个氨基酸,该K18氨基酸序列具有角蛋白特征,N-端含重复的甘氨酸(G);C-端富含丝氨酸(S)、苏氨酸(T)和缬氨酸(V)(图 2)。另外,还具有I型角蛋白共有序列DGKVVS,以及I型角蛋白非常相似的序列DNA(R/K)LAADDF(图 3)。通过预测,蛋白质分子式:C2088H3447N613O692S16,相对分子质量为48.7239 kD,理论等电点为5.42。其中,亮氨酸(L)、谷氨酸(E)、丝氨酸(S)的含量较高,分别为10.6%、10.0%、8.8%,色氨酸(W)的含量最少为0.2%。同时,66个负电荷氨基酸残基(Asp + Glu),57个正电荷氨基酸残基(Arg + Lys)。脂肪族氨基酸指数为87.13。泥鳅K18基因氨基酸预测无二硫键,也无跨膜结构及信号肽。所得泥鳅K18基因氨基酸序列提交于NCBI的GenBank中,登录号为AFD54864.1。
|
| 图 3 泥鳅与其他物种I型角蛋白基因氨基酸序列比对 实框表示I型角蛋白序列DGKVVS;虚框表示I型角蛋白非常相似的序列DNA(R/K)LAADDF |
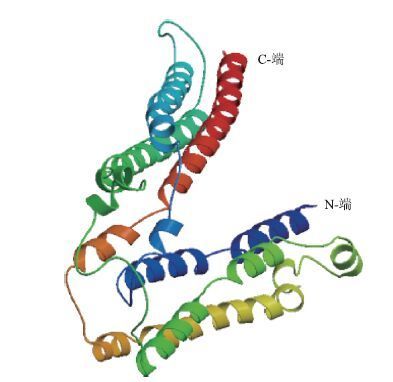
通过结构域分析(图 2)表明,泥鳅K18的结构域含:3个卷曲螺旋区域(82 aa-125 aa、189 aa-233 aa、286 aa-391 aa)和1个保守的IF蛋白结构域(84 aa-395 aa)。通过蛋白质二级结构预测,结果(图 4)表明,泥鳅K18的二级结构含:13个(α-)螺旋、7个(β-)折叠股及20个其他无规则卷曲,分别占二级结构的32.5%、17.5%及50.0%。并通过SWISS-MODEL程序,构建了K18的蛋白三级结构 (图 5)。

|
| 图 4 泥鳅K18氨基酸序列及预测的蛋白质二级结构 上方字母表示氨基酸序列,下方图形表示蛋白质二级结构;虚线表示其他无规则卷曲;黑色箭头表示(β-)折叠股;灰色圆柱表示(α-)螺旋 |
|
| 图 5 SWISS-MODEL程序预测的泥鳅K18的蛋白质三级结构 |
通过NCBI经BLAST蛋白质相似度比对分析,泥鳅K18基因氨基酸序列与大鳞副泥鳅的相似度最高为98%,与斑马鱼、金鱼、虹鳟、白斑狗鱼(Esox lucius)的K18相似度也较高,分别为90%、84%、77%、76%,此外同其他一些物种角蛋白基因氨基酸序列也存在不同程度的相似度(表 2)。
2.3 泥鳅K18基因的系统进化分析利用软件ClustalX多重序列比对和软件MEGA构建NJ系统进化树。结果(图 6)表明,不同物种及不同类型的角蛋白,分别独立占据系统进化树的不同分枝。其中,角蛋白K1、K3、K5、K8与其他类型角蛋白(K10、K11、K13、K17、K18、K20、K42、K97)独立占据一个大分枝,同时,K18又独立占据一个分枝,并分为两个分枝:一枝为人(Homo sapiens)、牛(Bos taurus)、褐家鼠(Rattus norvegicus)等脊椎动物;另一枝为鱼类,包括该研究所得泥鳅K18,表明泥鳅与其他物种的K18系统发育同源,进一步证实此基因属于K18类型家族。此外,泥鳅K18与在分类地位上也同属鳅科的大鳞副泥鳅聚在一起,系统发育关系最为密切。
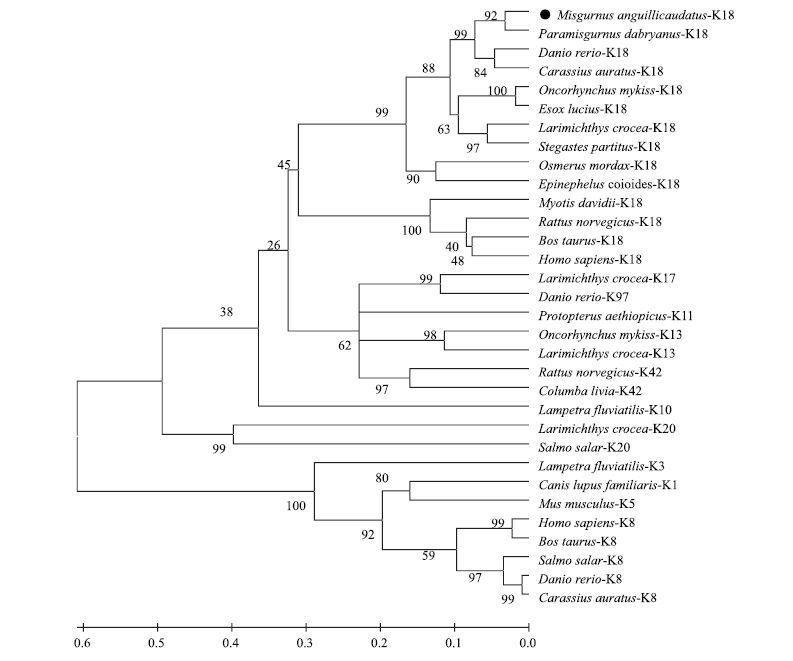
|
| 图 6 泥鳅与其他物种角蛋白的系统进化树 各节点的数字表示进化分枝的支持率。下方数字坐标轴为整个树枝长度,并代表遗传距离 |
以β-actin为内参,用RTQ-PCR检测泥鳅K18基因在肠、肌肉、心脏、胃、肝脏、精巢、卵巢、脾脏等8种组织的表达情况,结果(图 7)表明,K18基因在泥鳅8种不同组织中都有表达,但表达量存在明显的组织差异性,其中,在肠表达最高,心脏最低,在胃、肝脏、精巢、脾脏等组织也有较高表达,肠的相对表达量是心脏的22.4倍。通过统计学分析表明,K18基因的表达在不同组织间存在显著性差异,其中,肠同其他组织间的差异极显著(P<0.01);心脏除与卵巢外,同其他组织存在显著性差异(P<0.05),并与肠、胃、肝脏、脾脏的差异极显著(P<0.01)。
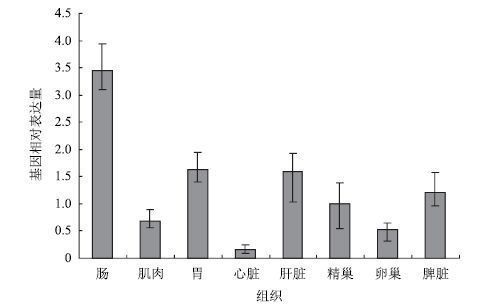
|
| 图 7 K18基因在泥鳅不同组织中的表达水平 |
IF蛋白可分为:Ⅰ型(角蛋白);Ⅱ型(角蛋白);Ⅲ型(包含波形蛋白、外周蛋白、结蛋白等);Ⅳ型(包含α-介连蛋白、神经纤维蛋白等);Ⅴ型(核纤层蛋白);Ⅵ型(联丝蛋白、平行蛋白、微管卷曲蛋白等)等类型[8]。通过研究,首次克隆得到泥鳅K18基因cDNA全长序列,经NCBI的BLAST分析为K18基因cDNA序列,属于IF蛋白。预测的分子量为48.7239 kD,在40 kD-64 kD范围内,理论等电点为5.42,在4-6范围内,符合Ⅰ型角蛋白(酸性角蛋白)特征,这与Infante等[5]的观点一致。同时,泥鳅K18基因氨基酸带66个负电荷氨基酸残基(Asp + Glu),多于57个正电荷氨基酸残基(Arg + Lys),也呈现酸性角蛋白。另外,研究所得泥鳅K18基因氨基酸序列中,N-端也发现了所有角蛋白共有特征,如富含重复的甘氨酸(G)。C-端富含丝氨酸(S)、苏氨酸(T)和缬氨酸(V),这同在其他角蛋白中也发现类似的氨基酸[24]。在C-端中还发现了其他许多IF蛋白的I型角蛋白序列DGKVVS,以及许多I型角蛋白非常相似的序列DNA(R/K)LAADDF[24]。但研究也发现,个别物种在这两个序列存在一些差异:大黄鱼(Larimichthys crocea)K17的I型角蛋白序列却为DGRVVS,I型角蛋白非常相似的序列却为DNARLAAEDF;大黄鱼K20的I型角蛋白非常相似的序列却为DNAQLAADDF,这可能是由于物种在进化过程中发生了一定的突变,尚待研究。该研究还对K18的蛋白质结构域、二级结构、三级结构进行了预测分析,但目前对这方面研究较少,其结构与功能等关系及机理也尚待研究。
3.2 泥鳅K18基因的系统进化按照表达不同,角蛋白又可细分为若干表达类型:E型、S型、H型、IRS型、ET型等[6, 10]。 K18属于S型角蛋白,存在于目前研究的所有脊椎动物中[3],且迄今为止,在已知的E型和S型系统发育分析中,这对经典的角蛋白被认为是所有脊椎动物类中唯一的角蛋白[25]。在人类中,经典的S型角蛋白K8和K18同陆生脊椎动物和硬骨鱼类有真正的同源基因[5, 18]。研究也发现,通过系统进化分析,泥鳅与其他物种的K18在进化树中占据一个分枝,在这K18分枝中又分为两枝:一分枝为人、牛、褐家鼠等陆生脊椎动物;另一分枝为泥鳅、大鳞副泥鳅、斑马鱼等硬骨鱼类,进一步表明K18的系统发育同源,且同陆生脊椎动物和硬骨鱼类同源。此外,在进化树中,K8也独立占据一个分枝,并也分为两枝:一分枝为人、牛等陆生脊椎动物;另一分枝为大西洋鲑(Salmo salar)、斑马鱼、金鱼等硬骨鱼类,也表明K8同陆生脊椎动物和硬骨鱼类同源。通过NCBI经BLAST蛋白质相似度分析,泥鳅K18基因氨基酸序列与大鳞副泥鳅具有98%的相似度,与斑马鱼、金鱼、虹鳟、白斑狗鱼等的K18相似度也较高,表明K18在进化中相对比较保守。
3.3 泥鳅K18基因的组织差异表达IF蛋白显示组织特异性[26],K18是在皮肤内主要表达于汗腺和顶泌腺分泌细胞的一种广泛分布的角蛋白[10]。在人类和小鼠的肝脏来源的细胞中,K18是唯一表达的角蛋白。该研究也发现,K18基因在泥鳅所检测的8种组织中均有表达,但肠表达最高,心脏最低,在胃、肝脏、精巢、脾脏等组织也有较高表达,表明K18基因表达水平存在明显的组织差异。K18是细胞的结构蛋白,是细胞的重要组成部分[1],在各组织器官细胞中广泛存在,该研究中K18基因在泥鳅肠、胃、肝脏、脾脏等上皮组织中有较高表达,进一步表明角蛋白通常表达于上皮组织,这与Schaffeld等[3, 6]的观点一致。此外,有研究表明,角蛋白的表达不限定于上皮组织[2, 24], 还被发现在几种卵母细胞和精子中[2]。同时,角蛋白还存在于多种间质细胞和组织中[18]。Druger 等[2, 24]研究表明,角蛋白还在金鱼等视神经中表达,S型角蛋白在硬骨鱼中还表达在间质细胞中,这可能有助于抵御渗透压,并在视神经中可能再生这种抵御渗透压能力[5, 6]。研究结果也显示,K18既在上皮组织表达外,还在肌肉、精巢、卵巢等组织表达。另外,心脏也有小部分表达,这可能是由于覆盖于心脏腔面的上皮所致,但由于心脏的独特结构和功能,心脏腔面的上皮仅是心脏的一小部分,为此,通常表达于简单上皮的S型角蛋白——K18在心脏中表达较低。总而言之,K18基因的表达显示组织特异性,其组织表达的差异及机理有待进一步研究。K18基因的组织表达存在差异性,在各组织器官细胞中广泛存在,体现了K18在细胞骨架中承担重要作用,同时,揭示了泥鳅K18的蛋白质二级结构、结构域以及三级结构等特征,并为了解泥鳅细胞骨架结构系统的研究奠定了一定基础。
4 结论研究首次克隆得到泥鳅K18基因cDNA全长序列1 694 bp,其中,包含开放阅读框1 299 bp,编码432个氨基酸,其序列与大鳞副泥鳅的相似度最高为98%,与其它物种K18也存在不同程度的相似度,并与其他物种的K18系统发育同源。通过定量统计分析,K18基因在泥鳅肠、肌肉、心脏、胃、肝脏、精巢、卵巢、脾脏等8种组织都有表达,其中,肠表达最高,心脏最低,且不同组织间存在显著性差异。
| [1] | Liu YH, Ho CC, Cheng CC, et al. Cytokeratin 18-mediated disorganization of intermediate filaments is induced by degradation of plectin in human liver cells[J]. Biochemical and Biophysical Research Communications, 2011, 407: 575–580. |
| [2] | Druger RK, Glasgow E, Fuchs C, et al. Complex expression of keratins in goldfish Optic nerve[J]. The Journal of Comparative Neurology, 1994, 340(2): 269–280. |
| [3] | Schaffeld M, Knappe M, Hunzinger C, et al. cDNA sequences of the authentic keratins 8 and 18 in zebrafish[J]. Differentiation, 2003, 71(1): 73–82. |
| [4] | Hesse M, Magin TM, Weber K. Genes for intermediate filament proteins and the draft sequence of the human genome:novel keratin genes and a surprisingly high number of pseudogenes related to keratin genes 8 and 18[J]. Journal of Cell Science, 2001, 114: 2569–2575. |
| [5] | Infante C, Ponce M, Asensio E, et al. Molecular characterization of a novel type Ⅱkeratin gene(sseKer3)in the Senegalese sole(Solea senegalensis):Differential expression of keratin genes by salinity[J]. Comparative Biochemistry and Physiology, Part B, 2011, 160(1): 15–23. |
| [6] | Schaffeld M, Bremer M, Hunzinger C, et al. Evolution of tissue-specific keratins as deduced from novel cDNA sequences of the lungfish Protopterus aethiopicus[J]. European Journal of Cell Biology, 2005, 84(2-3): 363–377. |
| [7] | Padhi BK, Akimenko MA, Ekker M. Independent expansion of the keratin gene family in teleostean fish and mammals:An insight from phylogenetic analysis and radiation hybrid mapping of keratin genes in zebrafish[J]. Gene, 2006, 368: 37–45. |
| [8] | Wang YH, Chen YH, Lin YJ, et al. Spatiotemporal expression of zebrafish keratin 18 during early embryogenesis and the establishment of a keratin 18:RFP transgenic line[J]. Gene Expression Patterns, 2006, 6(4): 335–339. |
| [9] | Schaffeld M, Haberkamp M, Schätzlein S, et al. A novel and ancient group of type I keratins with members in bichir, sturgeon and gar[J]. Frontiers in Zoology, 2007, 4: 16. |
| [10] | Kurokawa I, Takahashi K, Moll I, et al. Expression of keratins in cutaneous epithelial tumors and related disorders - distribution and clinical significance[J]. Experimental Dermatology, 2011, 20(3): 217–228. |
| [11] | 刘红山, 潘承恩, 苏国强, 等. 单克隆抗角蛋白8和18抗体免疫组化检测胆囊癌淋巴结微转移的临床意义[J]. 中华普通外科杂志, 2000, 15(10): 57–589. |
| [12] | Knudson KJ, Peters AH, Cagigao ET. Paleodiet in the Paracas Necropolis of Wari Kayan:carbon and nitrogen isotope analysis of keratin samples from the south coast of Peru[J]. Journal of Archaeological Science, 2015, 55: 231–243. |
| [13] | 席庆. 抗舌癌特异性角蛋白单克隆抗体的制备[D]. 北京:中国人民解放军军医进修学院, 2005. |
| [14] | 魏飞力, 石英, 陈杰, 等. 细胞角蛋白18凋亡片段M30单克隆抗体的制备及其在慢性乙型肝炎肝损伤检测中的初步应用[J]. 中华临床医师杂志, 2013, 7(10): 4333–4337. |
| [15] | 朱汇, 张红琳, 代蓉, 等. 绵羊高硫角蛋白KAP1.3基因启动子活性分[J]. 生物技术通报, 2012, 9: 74–79. |
| [16] | 崔新洁, 王婷, 刘秉春, 等. 牛奶中乳腺上皮细胞的分离培养及鉴定[J]. 生物技术通报, 2013, (3): 107–113. |
| [17] | 叶莹, 蔡生力, 刘红. 基因组步移技术克隆稀有鮈鲫角蛋白18基因序列及序列分析[J]. 上海海洋大学学报, 2011, 20(5): 661–668. |
| [18] | Schaffeld M, Höffling S, Markl J. Sequence, evolution and tissue expression patterns of an epidermal type I keratin from the shark Scyliorhinus stellaris[J]. European Journal of Cell Biology, 2004, 83(7): 359–368. |
| [19] | 徐如卫, 杨福生, 吴翩, 等. 泥鳅循环水集约化养殖的技术要点[J]. 安徽农学通报, 2014, 20(Z1): 128–129. |
| [20] | 胡廷尖, 刘士力, 练青平, 等. 泥鳅早期形态发育的研究[J]. 中国农学通报, 2012, 28(17): 132–138. |
| [21] | Biasini M, Bienert S, Waterhouse A, et al. SWISS-MODEL:modelling protein tertiary and quaternary structure using evolutionary information[J]. Nucleic Acids Research, 2014, 42(W1): 252–258. |
| [22] | Arnold K, Bordoli L, Kopp J, et al. The SWISS-MODEL workspace:a web-based environment for protein structure homology modeling[J]. Bioinformatics, 2006, 22(2): 195–201. |
| [23] | Benkert P, Biasini M, Schwede T. Toward the estimation of the absolute quality of individual protein structare models[J]. Bioinformatics, 2011, 27(3): 343–350. |
| [24] | Druger RK, Levine EM, Glasgow E, et al. Cloning of a type I keratin from goldfish optic nerve:differential expression of keratins during regeneration[J]. Differentiation, 1992, 52(1): 33–43. |
| [25] | Schaffeld M, Schultess J. Genes coding for intermediate filament proteins closely related to the hagfish “thread keratins(TK)” α and γ also exist in lamprey, teleosts and amphibians[J]. Experimental Cell Research, 2006, 312(9): 1447–1462. |
| [26] | García DM, Bauer H, Dietz T, et al. Identification of keratins and analysis of their expression in carp and goldfish:comparison with the zebrafish and trout keratin catalog[J]. Cell and Tissue Research, 2005, 322(2): 245–256. |