2. 中国农业科学院棉花研究所棉花生物学国家重点实验室,安阳 455000
2. Institute of Cotton Research,Chinese Academy of Agricultural Sciences,State Key Laboratory of Cotton Biology,Anyang 45500
棉花是世界上最重要的纤维作物之一,棉纤维品质优劣直接影响纺织品的市场竞争力[1, 2]。棉花为锦葵科棉属,棉属有52 个种,包括4 个栽培棉种、47 个野生棉种。现广泛种植的陆地棉和海岛棉为异源四倍体栽培种[3]。 陆地棉适应性广且产量高,占我国所产原棉的99%。海岛棉在我国目前只有新疆生产,海岛棉纤维的长度、细度和强力等方面优于陆地棉[4, 5]。棉花育种家一直致力于培育具有产量和品质兼备的棉花品种,利用基因工程技术将海岛棉中纤维品质形成相关基因转入陆地棉中来改良其纤维品质是一种有效的方法。从分子水平上研究海岛棉优质纤维形成的机理,克隆纤维发育相关基因尤为重要。最近研究表明苯丙烷代谢途径及其产物在棉花纤维品质形成过程中具有重要的作用[6, 7, 8, 9],对参与苯丙烷代谢途径的基因功能及作用机理开展研究,有望为改良纤维品质提供新的候选基因。莽草酸/ 奎宁酸羟基肉桂酰转移酶(shikimate /quinatehydroxycinnamoyl transferase,HCT)在苯丙烷C3 羟基化的上、下游起着双重调节作用,是苯丙烷代谢途径中的关键酶之一[10]。在本生烟[11](Nicotianabenthamiana)和紫花苜蓿[12](Medicago sativa)中,沉默HCT 基因导致减少转基因植株愈创木基木质素(guaiacyl lignin,G-lignin)和紫丁香基木质素(syringyllignin,S-lignin)的含量,增加对- 羟苯基木质素(phydroxyphenyllignin,H-lignin)的含量。在辐射松[13](Pinus radiata)中,沉默HCT 基因导致转基因植株木质素含量减少42%,这些研究表明通过改变HCT的活性能够调控木质素代谢。迄今,已经从本生烟[11]、紫花苜蓿[12]、棕色棉(Gossypium hirsutum L.)[14]、茶树(Camellia sinensis L.)[15, 16, 17, 18, 19]等植物中克隆了HCT 基因并鉴定了基因的功能,但海岛棉中HCT 基因的克隆研究尚未报道。本研究前期采用转录组测序方法对海岛棉品种新海21 胚珠,以及不同发育时期的纤维的转录本进行分析,从转录组数据中获得HCT 的序列,根据HCT 序列设计引物,从海岛棉品种新海21 中克隆了1 个GbHCT 基因,分析了其序列和蛋白质的亲疏水性,二级和三级结构,亚细胞定位、跨膜域和信号肽,并利用转录组数据分析该基因的表达模式,旨为进一步研究海岛棉GbHCT 基因的功能奠定基础。
1 材料和方法 1.1 植物材料收集海岛棉品种新海21(Gossypium barbadense L cv. Xinhai 21)生长至三叶期的叶片,液氮迅速冷冻,于-80℃保存备用。
1.2 方法 1.2.1 RNA 的提取及cDNA 第一链的合成按Trizol 试剂盒(Invitrogen)使用说明,提取叶片总RNA。利用first strand cDNA Synthesis 试剂盒(ThermoFisher)反转录合成cDNA 第一链。按说明书配制反转录第一链合成反应体系。
1.2.2 GbHCT 基因的克隆根据海岛棉胚珠转录组数据中的HCT 基因序列设计一对引物,nHCT-F :5 ' - A T G G A G A T T A C T A T A A A G G A G T C T G C - 3 '和n H C T - R :5 ' - T T A A A A C T C A T A A ATAAGTTTTTCAAAAA-3',以合成海岛棉品种新海21 叶片的cDNA 第一链为模板,扩增基因的开放阅读框(ORF)。按TransStart Taq DNA 酶说明书体系进行PCR 反应液的配制。PCR 程序为:94℃预变性5 min,94℃变性30 s,56℃复性45 s,72℃延伸 1min 30 s,共35 个循环;72℃延伸10 min。PCR 产物连接到pMD18-T(TaKaRa)载体,菌液PCR 检测后的阳性克隆送上海美季公司测序。
1.2.3 生物信息学分析利用DNAMAN 软件和Blast 检索GenBank 进行多重序列比较和同源性分析。利用Clustalx 1.83 软件和MEGA 4.1 软件构建系统发生树。用Protparam(http://www.expasy.org/tools/protparam)在线预测蛋白的分子质量、等电点及基本性质;利用GOR IV 程序预测蛋白质的二级结构(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)。利用Swiss-Model(http://swissmodel.expasy.org)程序预测蛋白质的三级结构。用ProtScale(http://web.expasy.org/protscale/)分析蛋白质的亲水/ 疏水性。利用PSORT 程序(http://www.psort.org/)对蛋白的亚细胞定位进行预测分析。利用TMpred(http://www.ch.embnet.org/software/TMPRED_form.html)预测跨膜域。利用SignalP 程序(http://www.cbs.dtu.dk/services/SignalP-3.0/)预测信号肽。
1.2.4 GbHCT 表达模式分析查找本实验室海岛棉纤维发育不同时期的转录组数据,获得在0、5、10、15 和25 DPA 中GbHCT 对应转录组中unigene的reads 数,用RPKM 值表示GbHCT 基因的相对表达量。
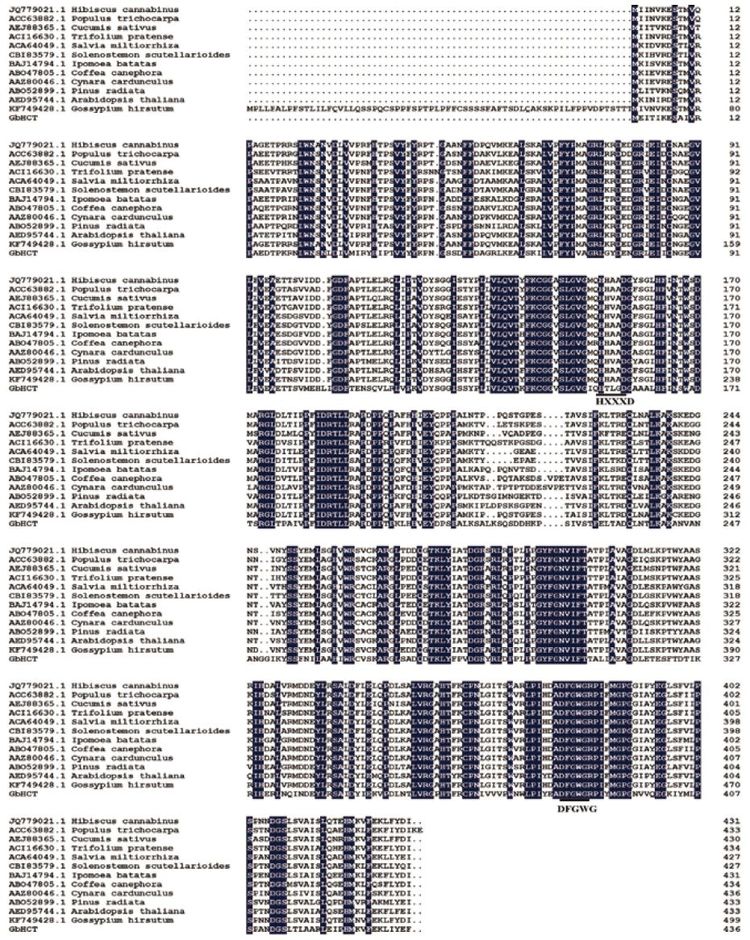
2 结果 2.1 GbHCT基因的克隆及序列分析根据海岛棉转录组数据中的HCT 序列设计引物,从海岛棉中克隆了一个编码HCT 的基因GbHCT(图 1)。GbHCT 基因开放阅读框长度为1 311 bp,编码436 个氨基酸,GenBank 登录号为KT378286。使用Protparam 在线预测GbHCT 蛋白质分子量为48.58kD,等电点为6.03,分子式为C2206H3425N581O636S10。GbHCT 与其他植物的HCT 氨基酸序列进行比对发现其氨基酸序列中含有HTLGD 和DFGWG 2 个保守基序(图 2)。BLAST 陆地棉基因组数据发现GbHCT对应陆地棉CotAD-76176,定位于第11 号染色体28871744-28873146(+),含有1 个内含子。

|
| 图 1 GbHCT 基因的扩增 |
|
| 图 2 GbHCT 与其他植物的HCT 氨基酸序列比对分析 |
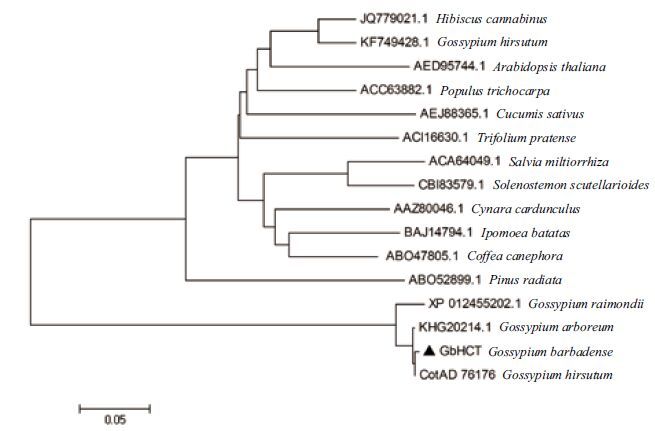
同源性分析表明,GbHCT 氨基酸与陆地棉CotAD-76176 的氨基酸序列仅相差1 个氨基酸,一致性达到99.77%。与亚洲棉GaHCT(KHG20214.1),雷蒙德氏棉GbHCT(XP_012455202.1) 和棕色棉GhHCT(KF749428.1) 的一致性分别为99.54%,97.02% 和60.7%。与其他植物中的HCT 氨基酸的一致性为55.7%-60%(图 2)。系统发生树(图 3)分析发现GbHCT 和陆地棉CotAD-76176,亚洲棉和雷蒙德氏棉HCT 成为一个分支,而棕色棉HCT(KF749428.1)和其他植物的HCT 聚在一个分支,说明GbHCT 和其他植物的HCT 蛋白亲缘关系较远。
|
| 图 3 GbHCT 蛋白与其他植物的HCT 蛋白的系统发生树分析 |
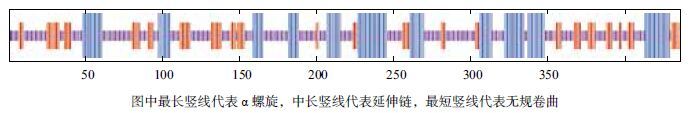
利用GOR IV 程序预测蛋白质的二级结构,如图 4 所示GbHCT 蛋白由436 个氨基酸残基组成,其中27.52%(120 个)氨基酸残基可能形成α 螺旋,21.10%(92 个)氨基酸残基可能形成延伸链,51.38%(224 个)氨基酸残基可能形成无规卷曲。
|
| 图 4 GbHCT 蛋白的二级结构 |
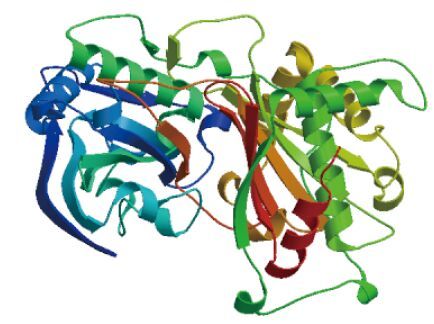
利用SWISS-MODEL 数据库进行GbHCT 蛋白质的三级结构分析,采用同源建模的方法,选择比对率最高的中粒咖啡Coffea canephora HCT 蛋白(SMTLid 4g0b.1)作为参照,得到GbHCT 蛋白的三级结构图(图 5)。
|
| 图 5 GbHCT 蛋白的三级结构同源建模 |
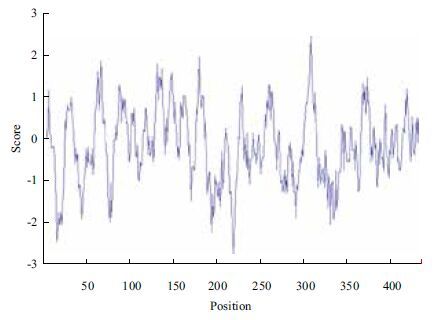
利用ProtScale 对GbHCT 蛋白进行亲水/ 疏水性分析。结果如图 6 所示,GbHCT 蛋白436 个氨基酸当中疏水性氨基酸最大值为2.444,亲水性氨基酸最小值为-2.744,亲水性总平均值为-0.151,推测GbHCT 蛋白是亲水性蛋白。
|
| 图 6 GbHCT 蛋白的亲/ 疏水性分析 |
利用 Psport 在线预测GbHCT 蛋白的亚细胞定位,结果表明定位于叶绿体基质的可能性为47.9%,定位于细胞质的可能性为45.0%,定位于微体(过氧物酶体)的可能性为44.3%,定位于叶绿体类囊体膜的可能性为13.1%。因此推测GbHCT 蛋白可能定位于叶绿体基质。TMpred 在线预测表明GbHCT蛋白存在一个跨膜域,位于301-319 个氨基酸。SignalP 预测表明GbHCT 蛋白没有信号肽。
2.4 GbHCT基因的表达模式检索海岛棉纤维转录组数据发现,GbHCT 基因在5 DPA 的纤维中表达量最高,RPKM 值为145 4.89。在10,15 DPA 的纤维中表达量逐渐降低,在25DPA 表达量最低,RPKM 值为22(图 7)。根据Gb-HCT 的表达量推测该基因可能参与棉纤维发育进程。

|
| 图 7 GbHCT 基因的表达分析 |
木质素代谢是苯丙烷代谢途径重要分支代谢途径。最近一些研究表明,木质素的生物合成及在成熟纤维中的含量与棉花纤维的品质有一定的关系。Fan 等[7]发现酚类化合物结合成熟棉花纤维的细胞壁,而且在棉花纤维次生壁形成阶段编码催化木质素代谢途径中最后一步反应的酶基因GhCAD6 为上调表达。Al-Ghazi 等[6]发现在陆地棉和海岛棉中编码催化从苯丙烷到对香豆酸途径的酶基因PAL,C4H和4CL 具有相似的表达水平。而且在纤维发育早期这些基因是非常高的表达,C4H 表达与纤维长度性状正相关。Wang 等[9]发现GhmiR397 可能在棉纤维起始阶段起正调控子作用,在野生型棉花胚珠中,GhmiR397 介导的基因沉默下调控漆酶基因的表达,漆酶的减少导致木质素产物减少,结果细胞壁表皮细胞松弛,促进纤维膨胀和起始。Han 等[8]研究发现WLIM1a 在棉花次生壁增厚期可以作为一个转录因子通过结合4CL1,CCR1 和CAD6 启动子中PALbox顺式作用元件上调这些基因的表达,从而使得超表达转基因棉花株系的木质素含量相对于野生型含量增加,因而影响纤维次生壁成分,使得纤维变细。这表明木质素或木质素类似酚类化合物可能影响纤维发育质量。对本课题组海岛棉转录组测序数据分析发现苯丙烷代谢途径中包括HCT 基因在内的多个基因在纤维发育的不同时期表达,本研究利用转录组数据从海岛棉中克隆了GbHCT 基因,对其序列进行了生物信息学分析。肖向文等[15]从棕色棉中克隆了一个HCT 基因发现,该基因在16 DPA 的胚珠中表达最高,在花瓣、茎和纤维中表达量次之,在叶片中的表达量最低。本研究发现海岛棉GbHCT 基因在5 DPA 的纤维中表达量最高,HCT 基因在棕色棉和海岛棉中表达时期的差异可能与两者品质的优劣有一定的关系,下一步将通过转化海岛棉来分析GbHCT 基因的生物学功能。
4 结论从海岛棉中克隆了1 个编码HCT 的基因GbHCT,开放阅读框长度为1 311 bp,编码436 个氨基酸,蛋白质分子量为48.58 kD,等电点为6.03。GbHCT 氨基酸序列中含有HTLGD 和DFGWG 2 个保守基序。预测了GbHCT 蛋白的二级、三级结构、亲疏水性、跨膜域和信号肽。GbHCT 氨基酸与陆地棉CotAD-76176 仅相差1 个氨基酸,与其他植物中的HCT 氨基酸的一致性为55.7%-60.7%。GbHCT 在纤维不同发育阶段都有表达,在5 DPA 的纤维中表达量最高,表明该基因可能参与棉花纤维的发育。
| [1] | 喻树迅, 王子胜. 中国棉花科技未来发展战略构想[J]. 沈阳农业大学学报:社会科学版, 2012, 14(1):3-10. |
| [2] | 喻树迅. 我国棉花生产现状与发展趋势[J]. 中国工程科学, 2013, 15(4):9-13. |
| [3] | 李付广, 袁有禄. 棉花分子育种进展与展望[J]. 中国农业科技导报, 2011, 13(5):1-8. |
| [4] | Li F, Fan G, Wang K, et al. Genome sequence of the cultivated cotton Gossypium arboreum[J]. Nature Genetics, 2014, 46(6): 567-572. |
| [5] | Wang K, Wang Z, Li F, et al. The draft genome of a diploid cotton Gossypium raimondii[J]. Nature Genetics, 2012, 44(10): 1098-1103. |
| [6] | Al-ghazi Y, Bourot S, Arioli T, et al. Transcript profiling during fiber development identifies pathways in secondary metabolism and cell wall structure that may contribute to cotton fiber quality[J]. Plant and Cell Physiology, 2009, 50(7):1364-1381. |
| [7] | Fan L, Shi WJ, Hu WR, et al. Molecular and biochemical evidence for phenylpropanoid synthesis and presence of wall-linked phenolics in cotton fibers[J]. Journal of Integrative Plant Biology, 2009, 51 (7):626-637. |
| [8] | Han LB, Li YB, Wang H Y, et al. The dual functions of WLIM1a in cell elongation and secondary wall formation in developing cotton fibers[J]. The Plant Cell, 2013, 25(11):4421-4438. |
| [9] | Wang ZM, Xue W, Dong CJ, et al. A comparative miRNAome analysis reveals seven fiber initiation-related and 36 novel miRNAs in developing cotton ovules[J]. Molecular Plant, 2012, 5(4): 889-900. |
| [10] | Hoffmann L, Maury S, Martz F, et al. Purification, cloning, and properties of an acyltransferase controlling shikimate and quinate ester intermediates in phenylpropanoid metabolism[J]. Journal of Biological Chemistry, 2003, 278(1):95-103. |
| [11] | Hoffmann L, Besseau S, Geoffroy P, et al. Silencing of hydroxycinnamoyl- Coenzyme A shikimate/quinate hydroxycinnamoyl- transferase affects phenylpropanoid biosynthesis[J]. The Plant Cell, 2004, 16(6):1446-1465. |
| [12] | Chen F, Reddy MSS, Temple S, et al. Multi-site genetic modulation of monolignol biosynthesis suggests new routes for formation of syringyl lignin and wall-bound ferulic acid in alfalfa(Medicago sativa L. )[J]. Plant Journal, 2006, 48 :113-124. |
| [13] | Wagner A, Ralph J, Akiyama T, et al. Exploring lignification in conifers by silencing hydroxylcinnamoyl-CoA :shikimate hydroxycinnamoyltransferase in Pinus radiate[J]. Proceedings of the National Academy of Sciences of the United States of America. 2007, 104(28):11856-11861. |
| [14] | 谢素霞, 程琳, 曾威, 等. 茶树HCT 基因的克隆及表达[J]. 东北林业大学学报, 2013, 41(6):19-22, 37. |
| [15] | 肖向文, 朱奇朗, 刘海峰, 等. 棉花莽草酸/ 奎宁酸羟基肉桂酰转移酶基因(GhHCT)的克隆、生物信息学分析及表达特性[J]. 农业生物技术学报, 2014, 22(5):572-579. |
| [16] | 何柳, 徐晓兰, 王振中, 等. 金银花莽草酸/ 奎宁酸香豆酰转移酶(LjHCT)基因克隆与序列分析[J]. 世界科学技术- 中医药现代化, 2014, 16(2):263-268. |
| [17] | 王雪霞, 曹方, 薛永常. 杨树木质素合成酶hct 基因的克隆及核苷酸序列分析[J]. 辽宁林业科技, 2009, 6 :4-7. |
| [18] | 葛文雅, 惠伟, 闫洪波, 等. 鸭梨PbHCT3 基因的克隆及表达分析[J]. 西北植物学报, 2012, 32(5):871-875. |
| [19] | 刘伟, 李高, 杨杞, 等. 柠条锦鸡儿HCT 基因克隆及序列分析[J]. 内蒙古农业大学学报, 2014, 35(1):77-82. |




