MicroRNA(miRNA) 是一类内源性的非编码RNA,具有调控功能,广泛存在于真核生物中,其大小长约20-25 个核苷酸。多数miRNA 还具有高度保守性、时序性和组织特异性[1, 2, 3]。到目前为止,在人类基因组中已发现超过1 000 种miRNAs[4],因其具有抑制靶mRNA 转录、翻译或者能够剪切靶mRNA 并促进其降解的功能,所以在人类多种生物过程中起着至关重要的作用,如细胞生长、分化、凋亡和增殖等过程[5]。现在许多研究表明,miRNAs的异常表达与许多癌症相关,如肺癌、肝癌、胃癌、乳腺癌、结肠癌和卵巢癌等[6]。这些miRNAs 所起的作用类似于抑癌基因和癌基因的功能[7],因此有研究人员将miRNA 命名为“oncomirs”。miRNAs 被用于有价值的标志物在早期的癌症诊断和预后中,研究miRNA 的表达水平及其生物学功能,对探索生物进化及防治人类疾病具有重要意义[8]。
在过去的一段时间,由于其特殊的生物学功能,miRNAs 检测技术的研究受到了广泛关注,同时亦有大量相关文章发表,从印迹杂交、微阵列芯片检测到最近发展起来的一些基于信号扩增技术在内的等一系列miRNA 检测方法被大量报道。印迹杂交技术(northern blotting)是用于miRNAs 检测分析的最为经典的方法之一[9, 10, 11, 12]。很多专家学者对其进行了发展优化,在一定程度上解决了miRNA 在传统northern blotting 时因其序列短,所引起杂交效率与检测灵敏度的问题[13, 14, 15]。但是该方法存在一些缺点,如灵敏度较低、操作过程繁琐、检测时间长、检测范围窄、容易污染等。微阵列基因芯片技术[16, 17]是20 世纪90 年代发展起来的高通量检测技术,虽然具有高通量、高灵敏度、高特异性等优点,但检测所用试剂和设备费用高,且该方法对多个miRNAs的量化存在困难。聚合酶链式反应(polymerasechain reaction,PCR)技术[18]是体外快速扩增特定基因最经典和最广泛应用的方法,其中反转录聚合酶链式反应(reverse transcription PCR,RT-PCR)在miRNA 的检测中亦被广泛的研究及应用。目前主要采用引物延伸RT-PCR、茎环引物RT-PCR[19, 20]和miRNA 加尾RT-PCR[21, 22]等方法进行miRNA 的定性和定量检测。尽管这些传统的检测技术基本能够进行miRNA 的检测,而且许多研究者从不同角度进行了这样那样的优化研究,以此弥补传统检测技术带来的缺陷。但是考虑到miRNA 检测的特异性和灵敏度的问题,这些已有的方法仍然需要进一步优化。由于miRNA 具有序列短、低丰度及家族成员间的高度保守性等性质,致使其检测难度增加,同时也增加了人们对于其检测方法进行深入研究的兴趣。由于miRNA 的检测对于医学及分子生物学都有着非常重要的意义,在很多临床研究中,需要针对一些与癌症相关的基因进行检测。由于miRNA 在临床研究中获取难度很大,而且miRNA 的稳定性较差,放置时间不宜过长,在一定程度上增加了其检测难度,所以提高检测特异性和灵敏度是miRNA 检测面临的一大挑战[23]。同时,miRNA 的表达调控比较精细,同一基因可能受不同miRNA 的调节,对应的同一miRNA 也可能控制着多种基因的表达,如若想更好的将miRNA 应用于临床研究中,则需要精确地区分miRNA 的结构和长度等信息,这也成为了当前miRNA 检测的另一大技术难题。
近年来已有大量的文章涉及了这方面的工作,也有学者对miRNA 的检测方法做了综述,但是关于高选择性和高灵敏度的miRNA 检测技术的概括还不甚全面。本文主要从基于信号放大的等温扩增技术和电化学检测技术用于高选择性和高灵敏度的miRNA 的检测着手进行了系统性总结,以便于人们对于其检测技术有更深层次的认识和了解,从而明确miRNA 的研究进展及研究方向,为更加充分的研究和利用miRNA奠定基础。
1 高选择性和高灵敏度的microRNA 检测技术 1.1 基于信号放大的等温扩增技术用于高选择性和高灵敏度的microRNA的检测
由于miRNA 检测的传统技术在选择性和灵敏度上需要进一步提高,所以一些学者在提高miRNA的选择性和灵敏度上做了大量的工作,也报道了一些相关文献,如表 1 所示。这些新的检测技术将miRNA 的检测提高到了一种新的层次,从而推动了miRNA 在临床中的应用。
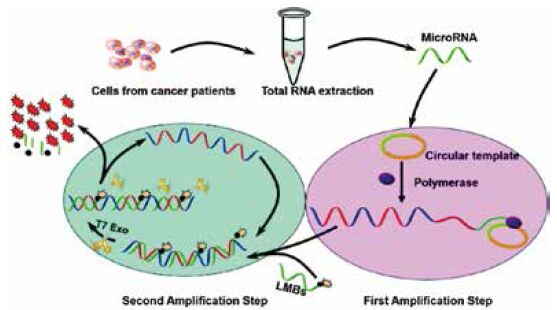
一些学者在已有的miRNA 检测技术上做了改进,从而大大提高了miRNA 检测的特异性和灵敏度。Cui 等[24]提出了在T7 核酸外切酶协助下利用滚环-循环酶双扩增法(RCA-CEAM)检测miRNAs,如图 1 所示。RCA-CEAM 法是将RCA(滚环扩增)与CEAM(循环酶扩增)结合起来的一种高选择性和高灵敏度的miRNA 检测方法。该方法为了减少背景信号,在CEAM 环节中采用特定设计的线性分子信标(linear molecular beacon,LMB)作为报告探针。其检测限低至12 fM,而传统的RCA 法的检测限为10 pM。与此同时,这种方法成功的应用于肝癌细胞和正常肝细胞的let-7a 的表达水平的检测,很好地证明了其在早期癌症诊断中的潜在应用。Ge 等[25]提出基于靶细胞引发的滚环扩增- 荧光原位检测法(TPRCA-FISH)。该方法采用circular DNA 作为探针与miRNA 靶细胞进行原位杂交,随后miRNA游离的3' 末端引发了原位RCA 反应,扩增产物可以与荧光探针进行杂交,以此来高灵敏度的指征靶 miRNA 的存在。该技术中RCA 反应只能由成熟miRNA 的游离的3' 末端引发,从而消除了miRNA前体或 mRNA 的干扰作用。这种方法被用于在人类肝癌细胞SMMC-7721 和肝细胞L02 中的miR-222 表达水平的检测,显示其高灵敏性和强选择性的检测。此外,该方法中RCA 产物与荧光探针杂交,产生的信号很强,能够很容易的从背景信号中区分出来,可利用普通的荧光显微镜检测,能够很好地检测低丰度的miRNA。
恒温指数扩增模式(IEXPAR)是利用线性扩增的寡核苷酸产物来产生更多与引物序列相同的寡核苷酸序列,从而产生指数放大效果,该方法扩增效率高。在此基础上Yin 等[26]提出了采用分子信标的方法用于miRNA 的检测,这是一种新发展起来的简单且可一步完成的反应。这种方法是运用荧光分子信标(MB)固有的信号传导机理,构成一个“生物回路”,包含两个反应:由Nicking 核酸内切酶协助的等温扩增反应和用于引发MB 扩增反应及信号传导的链置换聚合反应。该反应的检测动态限为1 fM-100 nM,具有较高的灵敏性和特异性。Liu等[27]提出了基于锁式探针的恒温指数滚环扩增技术(P-ERCA)用于miRNA 的检测,其中该锁式探针由与miRNA 的杂交序列和Nicking 核酸内切酶的Nicking 位点组成,大大提高了反应的特异性和灵敏度,其检测限降至0.24 zmol。
1.2 电化学检测技术用于高选择性和高灵敏度的microRNA的检测生物传感器利用生物物质作为识别元件,将特异性的生化反应转换成可识别的信号,从而能够进行生命物质和化学物质检测和监控的器件[37]。电化学生物传感器具有高效、灵敏快速、简便等特点,因此基于电化学技术的miRNA 生物传感器具有独特的优势。电化学生物传感器是将肽核酸固定在硅纳米线(SiNWs)的表面上作为检测miRNA 的探针,当与miRNA 进行杂交时,将会引起SiNWs 界面电阻的变化,其变化值与miRNA 浓度相关[38]。对于电化学生物传感器,控制识别探针在表面的密度和位置是一大挑战;信号扩增的效率和灵敏度是电化学检测技术的另一大挑战。表 2 列出了近年来研究者提出并发展的一些基于电化学技术的miRNA 检测方法。
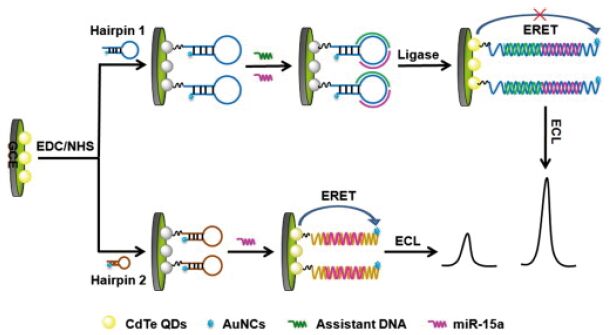
Cheng等[39]提出了一种基于CdTe 纳米晶体和Au 纳米簇(AuNCs)的电化学能量共振转移技术,在连接酶的作用下进行miRNA 的检测,如图 2 所示。其中CdTe 纳米晶体作为电化学发光的供体,而AuNCs作为电化学发光的受体。其基本原理为:首先合成AuNCs 功能化的发夹结构的DNA,然后在玻璃碳电极上通过酰胺反应结合到羧酸化的CdTe 纳米晶体上。当只有miRNA 存在时,杂交作用可直接将发夹结构的DNA 打开,这时由于CdTe 纳米晶体和AuNCs 之间的距离很近,使得能量传递受到影响,进而致使电化学发光信号很弱。而当miRNA 和协助DNA 同时存在时,连接酶能够选择性连接两者到发夹结构的DNA 链上,以形成长的DNA-RNA 异源杂交双链。较长的链抑制了CdTe 纳米晶体和AuNCs之间的能量传递,从而大大增强了电化学发光信号,很好的提高了miRNA 的检测灵敏度。该检测方法的线性范围达到6 个数量级,检测限低至21.7 fM,而且具有很高的特异性。
Dong 等[40]提出了采用耦合的Hg2+ 介导的构象的分子信标(MB),用Ag 纳米簇(AgNCs)作为荧光团,在核酸内切酶辅助下进行的扩增反应用于miRNA 的检测。其中Hg2+ 能够有效地淬灭AgNCs 的荧光信号,在特定的温度下,Hg2+ 介导的特定构象的MB 不能与靶标分子杂交。在协助探针分子存在的情况下,靶标分子和MB 探针退火,形成Y 型结构,随即通过释放Hg2+ 对AgNCs 产生高的荧光淬灭效率。该技术的检测限低至1 fM,同时消除了复杂的温度控制和复杂的标记,在临床诊断中具有重要的应用前景。Ge 等[41]提出了运用四面体Au 纳米结构探针与杂交链反应相结合的方法进行miRNA 的检测,其中三维四面体DNA 纳米结构作为支架用于固定DNA 识别探针,检测信号通过杂交链反应进行放大。因为超级三明治结构的分子虽然被广泛用于信号扩增,但是在构建该结构的过程中,其表面修饰及目标分子消耗难以精确控制,从而影响了扩增效率。而该检测方法通过特定的引发剂或目标分子来引发DNA 杂交链扩增反应,其产物为双螺旋结构,能够捕获大量的信号分子,从而大大增加了扩增效率,与传统的超级三明治扩增法相比,提高了3 个数量级,使miRNA 的检测限降低至10 aM。
1.2.2 基于酶的电化学检测技术最近,基于酶的电化学检测技术用于miRNA 的检测研究越来越广泛,由于在其催化作用下可将单个杂交分子转化成大量的检测分子,这也大大的提高了检测的灵敏性和特异性,同时为电化学生物传感器用于miRNA 的检测开辟了一条新的途径。近年来,关于这方面的报道也日益增多。
基于此,Cai 等[42]曾设计了非标记的特殊结构的分子信标用于的miRNA 检测的电化学生物传感器。该生物传感器所采用的非标记的功能化变构分子信标(aMBs)在目标物存在的情况下,能够高效的与链霉亲和素过氧化物酶聚合物结合,进而通过催化反应产生可检测的电信号。该传感器消除抗原抗体相互作用需要复杂的DNA 修饰这一缺点。该aMB 电化学生物传感器用于miRNA 的检测具有高选择性和高特异性,其检测限低至3.4 fM。另外该传感器可成功用于10% 的血清样本的检测。
Lin 等[43] 报道了第三代E-DNA 传感器用于miRNA 的检测,该设计主要采用DNA 四面体来确保茎环结构能够被很好地控制。茎环结构DNA 的热力学稳定性降低了反应的背景信号,增加了反应的特异性。随后用生化酶进行电化学信号扩增。该E-DNA 传感器对于miRNA 的检测低至1 fM,能够很好地区分高相似的miRNA 家族成员。
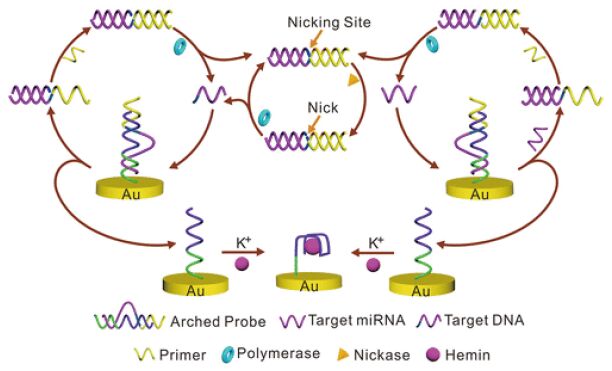
Yu 等[44]提出了基于拱形探针(arched probe)所介导的等温指数扩增反应的一种简单且非标记的电化学生物传感器的miRNA 检测技术,如图 3 所示。该探针是在电极表面的由两条在两端彼此部分互补的特殊的探针分子。目标miRNA 可以与拱形结构的互补序列进行杂交,产生的不完整的双链DNA 分子可以与引物结合,通过置换反应或切割来获得更多的miRNA 分子,以实现指数循环扩增。反应过程中产生的单链DNA 分子在钾离子存在的情况下与血红素结合形成G-四联体DNA,产生的电化学信号可被高效的检测。释放到溶液中的另一条链触发EXPAR循环和再生目标分子。该技术的检测限低至5.36fM,其检测线性范围为3 个数量级。
Deng 等[45]提出了一种新的miRNA 检测技术,该技术首先是靶标分子miRNA 分子与固定在磁珠上的特异性探针(DZ-CPs)进行杂交,然后利用双链特异性核酸酶对其进行切割,通过磁铁去除未反应的DZ-CPs 和磁珠,溶液中获得的DNA 分子能够作为催化剂在过氧化氢存在的情况下催化3,3',5,5'-联苯胺发生反应,可以通过比色法进行高灵敏度检测。这种方法的检测限低至0.8 aM。
2 结语miRNA 的检测对疾病的分子诊断和新型生物医药制剂的研发具有重要意义,近年来,有研究表明miRNA 可作为一些癌症治疗的潜在靶向药物用于临床治疗[62]。尽管目前为止,只有一部分miRNA 的生物学功能得到了解释,但是人们对于miRNA 的研究仍在不断努力。 miRNA 的研究方法正在不断发展,越来越多的报道阐明了检测miRNA 在不同组织、不同时期、不同疾病状态下的表达水平。miRNA 检测技术也越来越受到人们的关注,与此同时各种各样的新发展起来的检测技术已被运用到miRNA 的检测之中,基于以前一些传统方法的基础上,miRNA 的检测选择性和灵敏度有了很大的提高。随着新技术新方法的迅速发展,科研人员在定量检测 miRNA 方面有了更多的选择,但仍需要专家学者不断努力,争取提出更完善的技术方法。
| [1] | Bentwich I, Avniel A, Karov Y, et al. Identification of hundreds of conserved and nonconserved human microRNAs[J]. Nat Genet, 2005, 37(7):766-770. |
| [2] | Drummond MJ, McCarthy JJ, Sinha M, et al. Aging and microRNA expression in human skeletal mussscle :a microarray and bioinformatics analysis[J]. Physiol Genomics, 2011, 43(10): 595-603. |
| [3] | Liang Y, Ridzon D, Wong L, et al. Characterization of microRNA expression profiles in normal human tissues[J]. BMC Genomics, 2007, 8 :166-186. |
| [4] | Li R, Li X, Ning S, et al. Identification of a core miRNA-pathway regulatory network in glioma by therapeutically targeting miR-181d, miR-23b, β-Catenin, BP and STAT[J]. PLoS One, 2014, 9(7): e101903. |
| [5] | Bartel DP. MicroRNAs :genomics, biogenesis, mechanism, and function[J]. Cell, 2004, 116(2):281-297. |
| [6] | Yanaihara N, Caplen N, Bowman E, et al. Unique microRNA molecular profiles in lung cancer diagnosis and prognosis[J]. Cancer Cell, 2006, 9(3):189-198. |
| [7] | Tong F, Cao P, Yin Y, et al. MicroRNAs in gastric cancer :from benchtop to bedside[J]. Dig Dis Sci, 2014, 59(1):24-30. |
| [8] | Lukiw WJ, Zhao Y, Cui JG. An NF-kappaB-sensitive microRNA- 146a-mediated inflammatory circuit in Alzheimer disease and stressed human brain cells[J]. J Biol Chem, 2008, 283(46): 31315-31322. |
| [9] | Reinhart BJ, Slack FJ, Basson M, et al. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans[J]. Nature, 2000, 403(6772):901-906. |
| [10] | Lee RC, Ambros V. An extensive class of small RNAs in Caenorhabditis elegans[J]. Science, 2001, 294(5543):862- 864. |
| [11] | Lagos-Quintana M, Rauhut R, Lendeckel W, et al. Identification of novel genes coding for small expressed RNAs[J]. Science, 2001, 294(5543):853-858. |
| [12] | Lim LP, Lau NC, Weinstein EG. The microRNAs of Caenorhabditis elegans[J]. Genes Dev, 2003, 17(8):991-1008. |
| [13] | Válóczi A, Hornyik C, Varga N, et al. Sensiti- ve and specific detection of microRNAs by northern blot analysis using LNAmodified oligonucleotide probes[J]. Nucleic Acids Res, 2004, 32(22):e175. |
| [14] | Pall GS, Codony-Servat C, Byrne J, et al. Carbodiimide-media- ted cross-linking of RNA to nylon membranes improves the detection of siRNA, miRNA and piRNA by northern blot[J]. Nucleic Acids Research, 2007, 35(8):e60. |
| [15] | Nilsen TW. An alternative method for processing northern blots after capillary transfer[J]. Cold Spring Harbor Laboratory Press, 2015, 3 :314-318. |
| [16] | Thomson JM, Parker J, Perou CM, et al. A custom microarray platform for analysis of microRNA gene expression[J]. Nat Methods, 2004, 1(1):47-53. |
| [17] | Miska EA, Alvarez-Saavedra E, Townsend M, et al. Microarray analysis of microRNA expression in the developing mammalian brain[J]. Genome Biology, 2004, 5(9):R68. |
| [18] | 朱玉贤, 李毅, 郑晓峰. 现代分子生物学[M]. 第3 版. 北京: 高等教育出版社, 2007, 11 :165-166. |
| [19] | Mestdagh P, Feys T, Bernard N, et al. High-throughput stem-loop RT-qPCR miRNA expression profiling using minute amounts of input RNA[J]. Nucleic Acids Research, 2008, 36(21):e143. |
| [20] | Kramer MF. Stem-loop RT-qPCR for miRNAS[J]. Curr Protoc Mol Biol, 2011, Chapter 15 :Unit 15. 10. DOI :10. 1002/0471142727.mb1510s95. |
| [21] | Shi R, Chiang VL. Facile means for quantifying microRNA expression by realtime PCR[J]. Biotechniques, 2005, 39(4): 519-525. |
| [22] | Zhang J, Du YY, Lin YF, et al. The cell growth suppressor, mir- 126, targets IRS[J]. Biochem Biophys Res Commun, 2008, 377 (1):136-140. |
| [23] | Koshiol J, Wang E, Zhao Y, et al. Strengths and limitations of laboratory procedures for microRNA detection Cancer Epidemiol[J]. Biomarkers Prev, 2010, 19(4):907-911. |
| [24] | Cui L, Zhu Z, Lin N, et al. A T7 exonuclease-assisted cyclic enzymatic amplification method coupled with rolling circleamplification :a dual-ampliction strategy forsensitive and selective microRNA detection[J]. Chem Commun, 2014, 50(13): 1576-1578. |
| [25] | Ge J, Zhang LL, Liu SJ, et al. A highly sensitive target-primed rolling circle amplificationTPRCA method for fluorescent in situ hybridization detection of microRNA in tumor cells[J]. Anal Chem, 2014, 86(3):1808-1814. |
| [26] | Yin BC, Liu YQ, Ye BC. Sensitive detection of microRNA in complex biological samples via enzymatic signal amplification using DNA polyme- rase coupled with nicking endonuclease[J]. Anal Chem, 2013, 85(23):11487-11492. |
| [27] | Liu HY, Li L, Duan LL, et al. High specific and ultrasensitive isothermal detection of microRNA by Padlock probe-based exponential rolling circle amplification[J]. Anal Chem, 2013, 85 (16):7941-7947. |
| [28] | Oishi M. Enzyme-free and isothermal detection of microRNA based on click-chemical ligation-assisted hybridization coupled with hybridization chain reaction signal amplification[J]. Anal Bioanal Chem, 2015, 407(14):4165-4172. |
| [29] | Zhang Q, Chen F, Xu F, et al. Target-triggered three-way junction structure and polymerase/nicking enzyme synergetic isothermal quadratic DNA machine for highly specific, one-step, and rapid microRNA detection at attomolar level[J]. Anal Chem, 2014, 86 (16):8098-8105. |
| [30] | Zhou DM, Du WF, Xi Q, et al. Isothermal nucleic acid amplification strategy by cyclic enzymatic repairing for highly sensitive microRNA detection[J]. Anal Chem, 2014, 86(14):6763- 6767. |
| [31] | Ma C, Liu S, Shi C. Ultrasensitive detection of microRNAs based on hairpin fluorescence probe assisted isothermal amplification[J]. Biosensors and Bioelectronics, 2014, 58 :57-60. |
| [32] | Ma CP, Han D, Shi C. A new isothermal nucleic acid detection strategy mediated by a double-nicked beacon[J]. Chem Commun, 2014, 50(29):3799-3801. |
| [33] | Zhu X, Zhou X, Xing D. Label-free detection of microRNA :twostep signal enhancement with a hairpin-probe-based graphene fluorescence switch and isothermal amplification[J]. Chemistry, 2013, 19(17):5487-5494. |
| [34] | Wang XP, Yin BC, Wang P, et al. Highly sensitive detection of microRNAs based on isothermal exponential amplification-assisted generation of catalytic G-quadruplex DNAzyme[J]. Biosensors and Bioelectronics, 2013, 42 :131-135. |
| [35] | Bi S, Cui Y, Li L. Dumbbell probe-mediated cascade isothermal amplification :A novel strategy for label-free detection of microRNAs and its application to real sample assay[J]. Analytica Chimica Acta, 2013, 760 :69-74. |
| [36] | Ogawa A. Isothermal sensitive detection of microRNA using an autonomous DNA machine recycling output as input[J]. Bioorganic & Medicinal Chemistry Letters, 2010, 20(20):6056- 6060. |
| [37] | Thévenot DR, Toth K, Durst RA, et al. Electrochemical biosensors: recommended definitions and classification Biosens[J]. Bioelectron, 2011, 16(1-2):121-131. |
| [38] | Zhang GJ, Chua JH, Chee RE, et al. Label-free direct detection of MiRNAs with silicon nanowire biosensors[J]. Biosens Bioelectron, 2009, 24(8):2504-2508. |
| [39] | Cheng Y, Lei J, Chen Y, et al. Highly selective detection of microRNA based on distance-dependent electrochemiluminescence resonance energy transfer between CdTe nanocrystals and Au nanoclusters[J]. Biosensors and Bioelectronics, 2014, 51 :431- 436. |
| [40] | Dong H, Hao K, Tian Y, et al. Label-free and ultrasensitive microRNA detection based on novel molecular beacon binding readout and target recycling amplification[J]. Biosensors and Bioelectronics, 2014, 53 :377-383. |
| [41] | Ge Z, Lin M, Wang P, et al. Hybridization chain reaction amplification of microRNA detection with a tetrahedral DNA nanostructure-based electrochemical biosensor[J]. Anal Chem, 2014, 86(4):2124-2130. |
| [42] | Cai Z, Song Y, Wu Y, et al. An electrochemical sensor based on label-free functional allosteric molecular beacons for detection target DNA/miRNA[J]. Biosens Bioelectron, 2013, 41 :783- 788. |
| [43] | Lin M, Wen Y, Li L, et al. Target-Responsive, DNA Nanostructure- Based E?DNA Sensor for microRNA Analysis[J]. Anal Chem, 2014, 86(5):2285-2288. |
| [44] | Yu Y, Chen Z, Shi L, et al. Ultrasensitive electrochemical detection of microRNA based on an arched probe mediated isothermal exponential amplification[J]. Anal Chem, 2014, 86(16): 8200-8205. |
| [45] | Deng H, Shen W, Ren Y, et al. A highly sensitive and selective homogenous assay for profiling microRNA expression[J]. Biosensors and Bioelectronics, 2014, 54 :650-655. |
| [46] | Cai Z, Song Y, Wu Y, et al. An electrochemical sensor based on label-free functional allosteric molecular beacons for detection target DNA/miRNA[J]. Biosensors and Bioelectronics, 2013, 41 :783-788. |
| [47] | Yang J, Strickler JR, Gunasekaran S. Indium tin oxide-coated glass modified with reduced graphene oxide sheets and gold nanoparticles as disposable working electrodes for dopamine sensing in meat samples[J]. Nanoscale, 2012, 4(15):4594-4602. |
| [48] | Gao Z, Deng H, Shen W, et al. A label-free biosensor for electrochemical detection of femtomolar microRNAs[J]. Anal Chem, 2013, 85(30):1624-1630. |
| [49] | Shen W, Deng H, Ren Y, et al. A label-free microRNA biosensor based on DNAzyme-catalyzed and microRNA-guided formation of a thin insulating polymer film[J]. Biosensors Bioelectronics, 2013, 44 :171-176. |
| [50] | Wang Z, Zhang J, Guo Y, et al. A novel electrically magneticcontrollable electrochemical biosensor for the ultra sensitive and specific detection of attomolar level oral cancer-related microRNA[J]. Biosensors and Bioelectronics, 2013, 45 :108- 113. |
| [51] | Ren Y, Deng H, Shen W, et al. A highly sensitive and selective electrochemical biosensor for direct detection of microRNAs in serum[J]. Anal Chem, 2013, 85(9):4784-4789. |
| [52] | Meng X, Zhou Y, Liang Q, et al. Electrochemical determination of microRNA-21 based on bio bar code and hemin/G-quadruplet DNAenzyme[J]. Analyst, 2013, 138(12):3409-3415. |
| [53] | Tran HV, Piro B, Reisberg S, et al. Label-free and reagentless electrochemical detection of microRNAs using a conducting polymer nanostructured by carbon nanotubes :application to prostate cancer biomarker miR-141[J]. Biosensors and Bioelectronics, 2013, 49 :164-169. |
| [54] | Hong CY, Chen X, Liu T, et al. Ultrasensitive electrochemical detection of cancer-associated circulating microRNA in serum samples based on DNA concatamers[J]. Biosensors and Bioelectronics, 2013, 50 :132-136. |
| [55] | Ramnani P, Gao Y, Ozsoz M, et al. Electronic detection of microRNA at ettomolar level with high specificity[J]. Anal Chem, 2013, 85(17):8061-8064. |
| [56] | Labib M, Ghobadloo SM, Khan N, et al. Four-way junction formation promoting ultrasensitive electrochemical detection of microRNA[J]. Anal Chem, 2013, 85(20):9422-9427. |
| [57] | Zhu W, Su X, Gao X, et al. A label-free and PCR-free electrochemical assay for multiplexed microRNA profiles by ligase chain reaction coupling with quantum dots barcodes[J]. Biosensors and Bioelectronics, 2014, 53 :414-419. |
| [58] | Liu L, Xia N, Liu H, et al. Highly sensitive and label-free electrochemical detection of microRNAs based on triple signal amplification of multifunctional gold nanoparticles, enzymes and redox-cycling reaction[J]. Biosensors and Bioelectronics, 2014, 53 :399-405. |
| [59] | Zhou Y, Wang M, Xu Z, et al. Investigation of the effect of phytohormone on the expression of microRNA-159a in Arabidopsis thaliana seedlings based on mimic enzyme catalysis systematic electrochemical biosensor[J]. Biosensors Bioelectronics, 2014, 54 :244-250. |
| [60] | Bartosik M, Hrstkaa R, Palecek E, et al. Magnetic beadbased hybridization assay for electrochemical detection of microRNA[J]. Analytica Chim Acta, 2014, 813 :35-40. |
| [61] | Deng H, Shen W, Ren Y, et al. A highly sensitive microRNA biosensor based on hybridized microRNA-guided deposition of polyaniline[J]. Biosensors and Bioelectronics, 2014, 60 :195- 200. |
| [62] | Jafri MA, Zaidi SK, Ansari SA, et al. MicroRNAs as potential drug targets for therapeutic intervention in colorectal cancer[J]. Expert Opin Ther Targets, 2015, 18 :1-19. |