2. 中国农业科学院生物技术研究所,北京 100081
2. Biotechnology Research Institute, Chinese Academy of Agricultural Sciences, Beijing 100081
生物固氮是微生物在某些特定的条件下将空气中的氮气还原为铵的过程[1],是自然生态系统中氮的主要来源,在氮素的生态平衡和农、林生产业中发挥重要作用,而对于生物固氮调控过程的研究是目前微生物研究领域的热点之一。
氮源是自然环境中影响生物固氮效率的一个重要的限制性因素,固氮微生物能否高效利用环境中的氮源,使生物固氮仅发生在有利和必要的条件下是关键性问题。目前已有研究表明在大肠杆菌(Escherichia coli)、鼠伤寒沙门氏菌(Salmonellatyphimurium)、克雷伯氏菌(Klebsiella)和肺炎克氏杆菌(Klebsiella pneumoniae)中氮素吸收和氮源代谢由NtrBC(Nitrogen regulation,ntr)双组份系统全局调控[2]。NtrBC 双组份系统(亦称GlnLG 双组份系统)在革兰氏阴性菌中广泛存在,由组氨酸激酶NtrB 和转录调控因子NtrC 构成。在细菌所处生长环境缺乏氮源时,NtrB 能够自身磷酸化,并且发挥激酶作用将NtrC 磷酸化,磷酸化以后的NtrC 转录激活氮源转运吸收和代谢相关基因的表达[3]。其中NtrC 蛋白(在固氮施氏假单胞菌A1501 中由ntrC 基因编码)是固氮施氏假单胞菌A1501 中固氮岛基因表达激活的关键调节蛋白。在大肠杆菌中,GlnG 蛋白(在DH10B 菌中由glnG 基因编码)是氮限制条件下氮源选择性同化途径的全局激活子[4]。研究表明假单胞菌中的NtrC 蛋白是激活一系列编码参与氮源吸收和利用的蛋白基因表达所必需的因素[5],包括自身操纵子glnA-ntrBC 及atzDEF 操纵子。同时NtrC 在施氏假单胞菌中也参与固氮调控过程[6],也有许多研究表明肠杆菌中的氮调节系统与假单胞菌很多相同之处[6, 7]。大肠杆菌的代谢调控网络目前研究较为透彻,具有可进行厌氧生长、易于培养以及遗传操作方便等显著优点。而作为第一株完成测序的联合固氮菌,施氏假单胞菌A1501 含有一全长49 kb 的固氮岛,含有59 个编码基因,其中传统的20 多个固氮相关基因是其实现生物固氮的遗传基础[8]。本实验室已经成功将A1501 的固氮岛基因转入大肠杆菌DH10B 获得了具有固氮酶活性的重组大肠杆菌,使得联合固氮菌摆脱了遗传背景不清楚的局限。然而重组大肠杆菌的固氮酶活性只有野生型A1501 的1/10,因此,研究固氮条件下大肠杆菌内的氮代谢相关基因与施氏假单胞菌固氮岛内相关基因的表达调控是否具有相关性,是提高重组大肠杆菌固氮能力的关键。
本研究通过构建DH10B glnG 回补A1501 ntrC突变株的功能互补株和A1501 ntrC 回补DH10B glnG突变株的功能互补株,对突变株和功能互补株在唯一氮源条件下生长能力进行分析,旨在为揭示DH10B GlnG 与A1501 NtrC 两蛋白在细菌一般氮代谢过程中是否具有调控功能的相关性奠定理论基础,也为进一步提高重组菌固氮酶活性提供理论依据。
1 材料与方法 1.1 材料 1.1.1本研究中所涉及的细菌菌株主要是固氮施氏假单胞菌野生型及相关突变株和互补株(表 1)。
LB 培养基(固体培养基含1.5% 琼脂粉):0.5% 酵母提取物,1.0% 胰蛋白胨,1.0% 氯化钠,pH7.0。改良K 培养基(固体培养基含1.5% 琼脂粉):0.04% KH2PO4,0.01%K2HPO4,0.01% NaCl,0.02% MgSO4·7H2O,0.001%MnSO4·H2O,0.001% Fe2(SO4)3·H2O,0.001%Na2MoO4·H2O,0.6% C3H5NaO3,0.04%(NH4)2SO4,pH6.8 。M9 培养基(固体培养基含1.5% 琼脂粉):20% 5×M9 盐溶液,0.2% 1 mol/L MgSO4,20%葡萄糖,0.01% 1 mol/L CaCl2。5×M9 盐溶液:6.4%Na2HPO4,1.5% KH2PO4,0.25% NaCl,0.5% NH4Cl。
1.1.3 引物合成和测序由北京擎科生物技术有限公司及六合华大基因有限公司完成。
1.2 方法 1.2.1 三亲结合实验活化供体菌( 大肠杆菌JM109)、受体菌(A1501) 以及含有助质粒pRK2013 的大肠杆菌,转接至相应液体培养基中。过夜培养后接至新鲜的液体培养基中,30℃,220r/min 荡培养至OD600=0.6-0.8。吸取1.5 mL 液离心收集菌体沉淀。用0.85% 的生理盐水洗涤菌体2 次,并将3 管相同的菌液合为一管,离心收集菌体沉淀后加入100 μL 0.85% 的生理盐水将3 种菌液稀释成同一浓度,按供体菌∶受体菌∶助质粒为2∶2∶1的比例将3 种混合均匀离心收集菌体沉淀。用50-100 μL 0.85% 的生理盐水重悬菌体并点于LB(无任何抗性)平板中央,风干后放于30℃培养箱。培养24-48 h 后将所有菌苔刮下,用0.85% 的生理盐水倍比稀释各取100 μL 涂在加相应抗生素的K 培养基平板上,30℃培养至单菌落长到合适大小。
1.2.2 以10 mmol/L 硝酸钾和10 mmol/L 尿素为唯一氮源条件下的生长曲线测定活化菌株A1501、ΔntrC、ntrC-comp、glnG-comp,接种至新鲜的K 培养基中,30℃过夜培养,离心收集菌体沉淀。用0.85%的生理盐水洗涤菌体2 次。用以10 mmol/L 硝酸钾和10 mmol/L 尿素为唯一氮源的液体培养基重悬菌体,并调节菌液初始OD600 为1.0。将2 mL 菌液接种于18 mL 以10 mmol/L 硝酸钾和10 mmol/L 尿素为唯一氮源的液体培养基,30℃,220 r/min 震荡培养,每个体系3 个重复。分别于2、4、6、8、10、12、14、16、18、20、22 和24 h 取样,测定各菌液的OD600。
1.2.3 施氏假单胞菌固氮酶活测定活化菌株A1501、ΔntrC、ntrC-comp、glnG-comp,接种至新鲜无氮培养基中,30℃过夜培养,离心收集菌体沉淀。用0.85% 的生理盐水洗涤菌体2 次。用液体无氮培养基K 重悬菌体,并调节菌液初始OD600 为1.0。将1 mL 菌液接种于9 mL 无氮K 培养基中,封口后每瓶充5 min 氩气并注入0.5% 的氧气和10% 的乙炔,30℃,220 r/min 震荡培养,每个体系3 个重复。分别于4、6、8、10 h 取样250 μL 气体进行气谱检测,并记录乙烯的峰面积。
利用公式固氮酶活= 乙烯峰面积×(摇瓶中气相总体积/ 进样量)/(1 nmol 标准乙烯峰面积×反应时间× 蛋白量)计算固氮酶活性。其中摇瓶中气相总体积为70 mL,1 nmol 标准乙烯峰面积为1 962.9,蛋白量为0.68 mg。
1.2.4 以精氨酸为唯一氮源条件下的生长曲线测定活化菌株DH10B、ΔglnG、ntrC-comp-E,接种至液体培养基M9 中,37℃过夜培养。离心收集菌体。用0.85% 的生理盐水洗涤菌体2 次。用以精氨酸为唯一氮源的液体培养基重悬菌体,并调节菌液初始OD600 为1.0。接种200 μL 菌液于19.8 mL 以精氨酸为唯一氮源的液体培养基,37℃,220 r/min 震荡培养,每个体系3 个重复。分别于2、4、6、8、10、12、14、16、18、20、22 和24 h 取样,测定各菌液的OD600。
1.2.5 引物设计根据NCBI 数据库的GenBank 中获得大肠杆菌DH10B 及施氏假单胞菌A1501 的基因组序列后分别找到目标基因glnG 和ntrC,用Primer premier 5.0 设计引物glnG(F)/glnG(R)和ntrC(F)/ntrC(R)。glnG(F):5'-CAAAGCTTC C A C T C G A T A C C A G A T T A - 3 ' 和gln G (R)5'-CGGGATCC AGGAAATAAAGGTGACG-3',下划线分别为Hind Ⅲ 和BamH I 酶切位点。ntrC(F):5'-TTTAAGCTTTGTCGCAGGTCGGAT-3',和ntrC(R):5'-TTGGATCCAGAACATCATCAGTCAG-3',下划线分别为Hind Ⅲ 和BamH I 酶切位点。以基因组DNA为模板用引物glnG(F)/glnG(R)和ntrC(F)/ntrC(R)扩增glnG 和ntrC 基因片段。PCR 扩增条件:95℃10 min ;95℃ 30 s,54-62℃ 1.5 min,72℃ 30 s,30个循环;72℃ 10 min。
2 结果 2.1 DH10B GlnG和A1501 NtrC的生物信息学及理化性质分析在NCBI 数据库中(http://blast.ncbi.nlm.nih.gov/Blast.cgi) 分别检索DH10B GlnG 及A1501 NtrC两蛋白质的氨基酸序列,获得序列后利用DNAMAN软件对两个序列进行序列比对分析,比对结果(图 1)显示两个蛋白的一级结构相似,氨基酸序列相似性为63.5%。
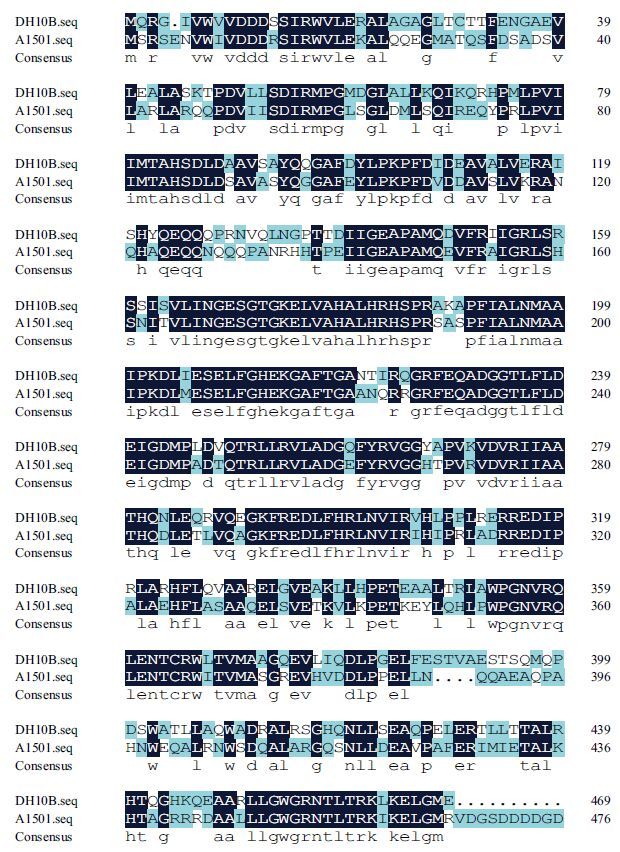
|
| 图 1 DH10B GlnG 与A1501 NtrC 蛋白质氨基酸序列比对 |
利用ExPASy(Expert Protein Analysis System)数据库中进行氨基酸理化参数计算的工具ProtParam(http://expasy.org/tools/protparam.html)对两个蛋白质进行了比较分析。结果(表 2)显示两个蛋白的分子量、等电点等都非常接近。以上结果表明DH10BGlnG 蛋白与A1501 NtrC 蛋白在结构上具有一定相似性,两蛋白可能具有功能相关性。
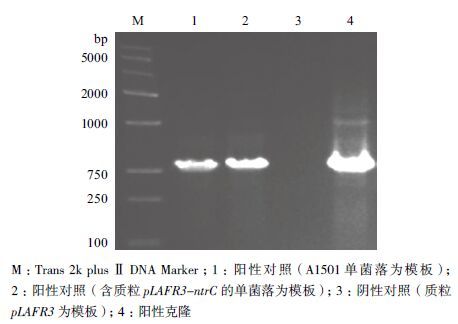
本实验室已经采用广宿质粒pLAFR3 通过双酶切成功构建了pLAFR3-glnG 重组质粒,本研究通过三亲结合的方法将重组质粒 pLAFR3-glnG 导入ΔntrC 中。在含有四环素(Tc)和氯霉素(Cm)双抗性的固体限制性培养基K 上进行阳性克隆筛选,再利用引物glnG(F)/glnG(R)进行菌落PCR 验证(图 2),得到glnG 互补ΔntrC 的功能互补株(glnG-comp)。

|
| 图 2 菌落PCR 验证glnG 基因 |
为了研究GlnG 能否替代NtrC 在ΔntrC 一般氮代谢过程中行使功能,我们构建了glnG 互补ΔntrC的功能互补株并进行了相应功能表型测定。因本实验室已经证实ΔntrC 不能在以硝酸钾或尿素为唯一氮源的培养基上生长,所以本研究测定了A1501、A1501ΔntrC、ntrC-comp 以及glnG-comp 在培养基K中以硝酸钾和尿素为唯一氮源的生长曲线(图 3)。从图中可以看到ΔntrC 能够利用硝酸钾和尿素为唯一氮源进行生长,野生型A1501 在该条件下生长良好。而不论是用ntrC 还是用glnG 回补ΔntrC 都能使其恢复一定的生长能力。结果表明,glnG 已在ΔntrC中成功表达,并能够替代ntrC 实现对硝酸钾和尿素功能的利用,这一结果表明GlnG 蛋白与NtrC 蛋白在氮代谢过程中具有功能相关性。
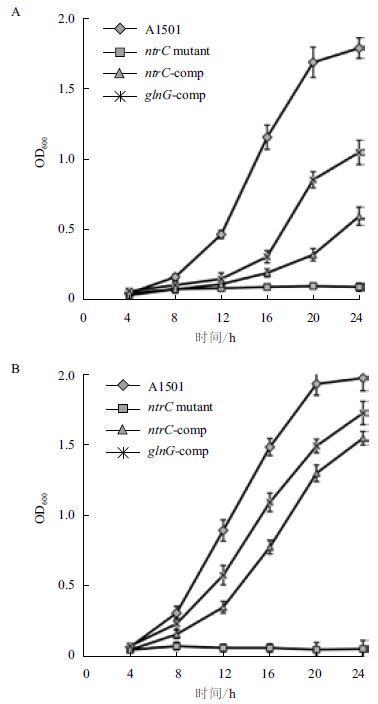
|
| 图 3 四种菌株在以10 mmol/L 硝酸钾(A)及10 mmol/L尿素(B)为唯一氮源条件下的生长曲线 |
为了进一步验证 GlnG 蛋白与NtrC 蛋白在固氮调控过程中是否具有功能相关性,我们对A1501、ΔntrC、ntrC-comp 及glnG-comp 的固氮酶活性进行了测定。结果(图 4)发现ntrC 突变以后使菌株的固氮酶活性基本丧失,而glnG-comp 具有一定的固氮酶活性,约为野生型A1501 的45%。说明在固氮过程中,glnG 能够部分互补ntrC 的调节功能,使菌株表现出固氮酶活性。结果表明,在重组大肠杆菌中GlnG 参与了固氮表达调控,与NtrC 蛋白具有功能相关性。
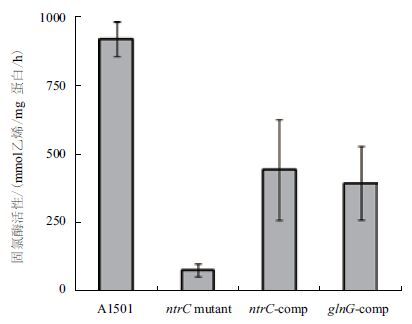
|
| 图 4 四种菌株固氮酶活性的测定 |
本实验室已成功地将A1501ntrC 片段克隆到广宿主质粒载体pLAFR3 上,构建了A1501 NtrC 蛋白的表达载体 pLAFR3-ntrC。本研究首先提取了重组质粒 pLAFR3-ntrC,然后通过电击转化将重组质粒pLAFR3-ntrC 导入到ΔglnG 中,再利用含有四环素(Tc)和卡那霉素(Km)双抗性的固体LB 平板进行阳性克隆筛选。最后利用ntrC(F)/ntrC(R)引物进行菌落PCR 验证(图 5),得到ntrC 回补ΔglnG的功能互补株(ntrC-comp-E)。
|
| 图 5 菌落PCR 验证ntrC 基因 |
为了研究NtrC 蛋白能否在 E.coli DH10B 中代替GlnG 在一般氮代谢过程中行使功能,我们对ΔglnG和ntrC-comp-E 进行了相应表型测定。因为有研究表明ΔglnG 不能在以精氨酸为唯一氮源的条件下进行生长[9],所以本研究测定了E.coli DH10B、ΔglnG以及ntrC-comp-E 在培养基M9 中以精氨酸为唯一氮源条件下的生长曲线。结果(图 6)显示 ΔglnG 在M9 培养基中不能够以精氨酸为唯一氮源进行生长,野生型E.coli DH10B 在该条件下生长良好。而当用ntrC 互补ΔglnG 突变株时,功能互补菌株基本恢复了生长能力。结果表明,ntrC 已在ΔglnG 中成功表达,并能够替代glnG 在E.coli DH10B 菌的一般氮代谢过程中行使功能,实现对精氨酸利用功能的互补。
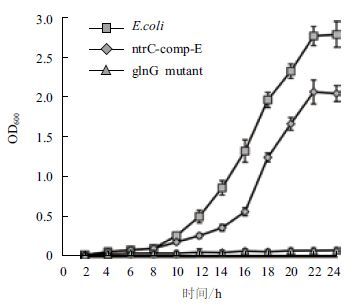
|
| 图 6 三种菌株在以精氨酸为唯一氮源条件下的生长曲线 |
氮调节是涉及多个信号转导和效应蛋白的复杂调控网络,该领域在肠道菌中已经进行了深层研究。假单胞菌的氮代谢系统与肠细菌相比有很多共同特点,均含有典型的氮代谢调节系统即NRⅡ-NRⅠ 双组分调节系统[3],该系统由尿苷转移酶(GlnD)、PⅡ(GlnB、GlnK)、NRⅡ(GlnL/NtrB)和 NRⅠ(GlnG/NtrC)等具有双功能的蛋白组成。然而,施氏假单胞菌的氮调节与大肠杆菌也有很多不同之处。首先,施氏假单胞菌中Ntr 系统的功能仅由glnK 基因编码的PⅡ 蛋白调控,其产物直接编码高亲和力转运蛋白的amtB 基因相互作用。而在大肠杆菌中,glnB 和glnK 都编码PⅡ 蛋白,其中组成型表达的GlnB 蛋白由glnB 编码,由于GlnK 的转录依赖于NRⅠ,因此glnK 编码的GlnK 蛋白只在低氮条件下表达,GlnB和 GlnK 的功能也部分重叠[10]。
Schumacher 等[5]研究表明,NRⅠ(NtrC)的存在导致恶臭假单胞菌中glnK 和amtB 的表达受氮源调控,其功能仅在氮限制的条件下由NtrC 激活[5]。体外纯化蛋白实验表明,glnK 的转录直接被 NtrC 激活[5]。通过对施氏假单胞菌A1501 中glnK 基因的序列分析,发现在glnK 基因的启动子上游-12/-24处具有与σ54 结合位点相似的序列。在低氮的条件下,依赖于σ54 的启动区开始转录后glnK 基因开始表达,该表达受NtrC 蛋白调控;在高氮条件下,glnK 基因可能在一个不依赖于σ54 和NtrC 蛋白调控的启动子区开始转录并进行低水平表达[11]。
在大肠杆菌中,glnK 基因上游区域也含有一个 σ54 结合位点,一个 NRⅠ(GlnG)结合位点,操纵子表达也依赖GlnG 同时也受到氮素调控[12, 13]。另外,与大肠杆菌glnKp 不同的是恶臭假单胞菌glnKp开放复合物的形成强烈依赖于IHF,这在体内才能观察到。并且IHF 的参与,加上其螺旋的同一侧存在两个连续的NtrC 结合位点,表明恶臭假单胞菌glnKp 的转录激活对低浓度的NtrC 是敏感的。这两个不同点可能与缺乏组成型GlnB 蛋白进行高效氮抑制有关[14]。其次,大肠杆菌氮调节的操纵子也包含能被 σ70 RNA 聚合酶全酶识别的启动子[15],这些操纵子的转录受到氮同化控制蛋白(Nac)-LysR 型调节子的调控。在大肠杆菌中受Nac 激活的基因和操纵子涉及氮化分子的转运[16],这也可能作为氮的来源。然而施氏假单胞菌A1501 全基因组数据表明其不含有nac 基因[17],并且已测序的假单胞菌基因组表明该属菌中不含有Nac 同源物[18]。有研究证实在恶臭假单胞菌中,由NtrC 直接激活的基因同时含有σ54 依赖型启动子和Nac 激活基因同源物,这避免了需要一个适配器来共同调节σ54 和σ70 依赖型基因[19]。进而推测通过Nac 介导的依赖σ70 的级联氮调控不是肠杆菌所必须的,因为许多菌中不含有nac 基因。在沙门氏菌中,最近发现似乎Nac 也丢失了,因为在响应氮的情况下它不能控制依赖σ70 基因的表达。然而沙门氏菌中hutC 基因的启动子仍易受克雷伯氏菌Nac 的激活[20, 21]。相反在恶臭假单胞菌中,这些基因有的仍然响应氮的调控,但是它们的启动子依赖σ54,直接受到NtrC 的调控,从而避免了额外的调控[22]。由此可知施氏假单胞菌A1501 中由氮介导的调控网络也是大肠杆菌中氮调控网络的简化模式:只含有一个PⅡ 蛋白,没有Nac 参与的级联调控[23],这可能是造成DH10B GlnG 和A1501 NtrC 调控功能差异的因素。
本研究证实了DH10B GlnG 和A1501 NtrC 蛋白在一级结构、理化性质以及功能上都具有很大的相似性。这些数据表明,在大肠杆菌和施氏假单孢菌中的NRⅠ 蛋白的功能是高度保守的,两个蛋白可以进行相互替换。同时也发现,DH10B glnG 互补A150 ntrC 突变株时,在一般氮代谢方面的功能互补性高于调控固氮过程的功能互补性,推测这可能与大肠杆菌和施氏假单胞菌中氮介导的调节网络的不同有关,也可能是在进化过程中,由于大肠杆菌所处的环境营养条件优越,造成其一般氮代谢调节蛋白GlnG 在功能上产生了分化。
4 结论本研究成功构建了DH10B glnG 回补A1501ΔntrC 的功能互补株和A1501ntrC 回补DH10B ΔglnG的功能互补株,并对互补株和突变株与野生型的氮源利用和生物固氮能力等方面进行了比较分析,结果显示DH10B glnG 能够互补A1501 ntrC 在氮源利用和氮限制条件下的调节功能,而A1501 ntrC 能够互补DH10B glnG 在氮源条件下的生长能力,这一结果表明DH10B 菌中的GlnG 可能通过互补A1501 菌中 NtrC 蛋白在氮限制条件下的调节功能进而参与到一般氮代谢调控网络过程中。
| [1] | Halbleib CM, Ludden PW. Regulation of biological nitrogen fixation[J]. J Nutr, 2000, 130(5):1081-1084. |
| [2] | Li W, Lu CD. Regulation of carbon and nitrogen utilization by CbrAB and NtrBC two-component systems in Pseudomonas aeruginosa[J]. J Bacteriol, 2014, 59(4):524-592. |
| [3] | Arcondeguy T, Jack R, Merrick M. P(Ⅱ)signal transduction proteins, pivotal players in microbial nitrogen control[J]. Microbiol Mol Biol Rev, 2001, 65(1):80-105. |
| [4] | Vidangos NK, Heideker J. DNA recognition by a σ(54)transcriptional activator from Aquifex aeolicus[J]. J Mol Biol, 2014, 175(19):667-684. |
| [5] | Schumacher J, Behrends V, Pan ZS, et al. Nitrogen and carbon status are integrated at the transciptional level by the nitogen regulator BtrC in vivo[J]. Microbiology, 2013, 149(Pt 8):251-262. |
| [6] | Hervas AB, Canosa I, Santero E. Transcriptome analysis of Pseudomonas putida in response to nitrogen availability[J]. J Bacteriol, 2008, 190(1):416-420. |
| [7] | Desnoues N, Lin M, Guo X, et al. Nitrogen fixation genetics and regulation in a Pseudomonas stutzeri strain associated with rice[J]. Microbiology, 2003, 149(Pt 8):2251-2262. |
| [8] | Yan Y, Yang J, Dou Y, et al. Nitrogen fixation island and rhizophere competence traits in the genome of root-associated Pseudomonas stutzeri A1501[J]. Proc Natl Acad Sci USA, 2008, 105(21): 7564-7569. |
| [9] | Mcfarland N, Mccarter L, Artz S, et al. Nitrogen regulatory locus "glnR" of enteric bacteria is composed of cistrons ntrB and ntrC : identification of their protein products[J]. Proc Natl Acad Sci USA, 1981, 78(4):2135-2139. |
| [10] | Williams KJ, Bennett MH, Barton GR, et al. Adenylylation of mycobacterial GlnK(PⅡ)protein is induced by nitrogen limitation[J]. Tuberculosis(Edinb), 2013, 49(Pt 8):1251- 1262. |
| [11] | Chen B, Doucleff M, Wemmer DE, et al. ATP ground- and transition states of bacterial enhancer binding AAA+ ATPases support complex formation with their target protein, sigma54[J]. Structure, 2007, 15(4):429-440. |
| [12] | Benkert P, Biasini M, Schwede T. Toward the estimation of the absolute quality of individual protein structure models[J]. Bioinformatics, 2010, 27(3):343-350. |
| [13] | Jiang H, Shang L, Yoon SH, et al. Optima production of polygamma- glutamic acid by metabolically engineered Escherichia coli[J]. Biotechnol Lett, 2006, 29(2):631-647. |
| [14] | Hervas AB, Canosa I, Little R, et al. NtrC-dependent regulatory network for nitrogen assimilation in Pseudomonas putida[J]. J Bacteriol, 2009, 181(4):1056-1062. |
| [15] | Abbas MM, Mohie-Eldin MM, EI-Manzalawy Y. Assessing the effects of data selection and representation on the development of reliable E. coli sigma 70 promoter region predictors[J]. PLoS One, 2015, 162(19):3028-3039. |
| [16] | Zeng J, Spiro S. Finely tuned regulation of the aromatic amine degradation pathway in Escherichia coli[J]. J Bacteriol, 2013, 195(22):5141-5150. |
| [17] | Babaer P, Marashi SA, Asad S. Genome-scale reconstruction of the metabolic network in Pseudomonas stutzeri A1501[J]. RSC Publishing, 2015, 41(13):3082-3088. |
| [18] | Yan Y, Ping S, Peng J, et al. Global transcriptional analysis of nitrogen fixation and ammonium repression in root-associated Pseudomonas stutzeri A1501[J]. BioMed Central, 2010, 115(21): 7543-7565. |
| [19] | Melchiorsen CR, Jokumsen KV, Villadsen J, et al. The level of pyruvate-formate lyase controls the shift from homolactic to mixedacid product formation in Lactococcus lactis[J]. Appl Microbiol Biotechnol, 2002, 58(3):338-344. |
| [20] | Utrilla J, Gosset G, Martinez A. ATP limitation in a pyruvate formate lyase mutant of Escherichia coli MG1655 increases glycolytic flux to D-lactate[J]. J Ind Microbiol Biotechnol, 2009, 36(8):1057-1062. |
| [21] | Sieira R, Arocena GM, Zorreguieta A, et al. A MarR-Type regulator directly activates transcription from the Brucella abortus virB promoter by sharing a redundant role with HutC[J]. J Bacteriol, 2012, 73(13):6143-6158. |
| [22] | Vidangos NK, Heidiker J, Lyubimov A, et al. DNA recognition by a σ(54)transcriptional activator from Aquifex aeolicus[J]. J Mol Biol, 2014, 114(3):1162-1169. |
| [23] | Hasona A, Kim Y, Healy FG, et al. Pyruvate formate lyase and acetate kinase are essential for anaerobic growth of Escherichia coli on xylose[J]. J Bacteriol, 2004, 186(22):7593-7600. |






