表观遗传学(epigenetics)是1942 年由Waddingtong[1]首次提出,主要研究在DNA 序列没有发生改变的情况下,基因功能发生改变,并且这种改变可以随着细胞的有丝分裂及减数分裂遗传下去的现象,其中以DNA 甲基化最为常见。
DNA 甲基化在植物体中有两个关键作用:一是,保护基因组免受外来插入序列的侵害;二是调节基因表达[2, 3]。大量实验研究表明,植物DNA 甲基化与基因表达存在一定的关联性。本文首先就植物DNA 甲基化的形式,建立与维持机制进行综述,进而阐述植物DNA 甲基化影响基因表达的机制,对逆境胁迫下植物单个基因DNA 甲基化对基因表达的影响,以及当前利用高通量测序技术分析植物全基因组范围内DNA 甲基化与基因表达的关联分析进行归纳,以更深入理解DNA 甲基化效应机制。
1 植物基因组中的DNA 甲基化 1.1 植物DNA甲基化的模式DNA 复制完成后,甲基转移酶(DNMT)催化S-腺苷酰-L-甲硫氨酸(SAM)上的甲基基团连接到DNA 分子的胞嘧啶碱基上。
植物基因组DNA 甲基化主要发生在对称序列CG 富含区以及高度重复序列,主要集中在着丝粒重复区、核糖体RNA 编码重复区以及转座子元件序列等区域[4]。甲基化有3 种主要修饰形式,其中5'-5mCG-3' 是最普遍也是稳定性最佳的形式;其次是5'-5mCNG-3'(N 代表任何核苷酸)和5'-5mCHH-3' 位点(不对称位点,H 代表A、C 或T)修饰形式,后两者发生频率较低[5]。
植物基因组DNA 甲基化产生方式主要有两种:一是维持甲基化:在DNA 复制过程中,DNA 甲基化酶识别半甲基化的DNA 子链,催化新生链相应位置发生甲基化修饰;二是从头甲基化:DNA 甲基化酶催化母链上未发生甲基化的胞嘧啶位点在子链上进行甲基化修饰[6]。
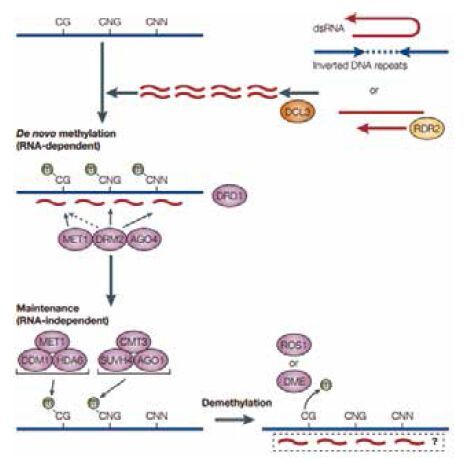
1.2 植物DNA甲基化的建立及维持机制在对植物体的研究中发现,DNA 甲基可以被小分子RNA 介导,该过程被称之为RNA 介导的DNA甲基化(RNA-depended DNA methylation,RdDM)[7, 8]。参与RdDM 过程的小RNA 由RNAi 过程产生。植物细胞核内,在RNA 依赖的RNA 聚合酶的作用下使单链RNA 形成双链RNA 或插入重复序列的转录产物形成双链RNA,随后在DCL3(Dicer-like3)的作用下,这些双链RNA 被加工成24 nt 的RNA 信号分子,通过作用于不同的甲基转移酶导致基因不同序列位点发生DNA 甲基化(图 1)。RdDM 导致RNADNA序列同源区域几乎所有的胞嘧啶高度从头甲基化。
植物中鉴定到3 类结构不同的胞嘧啶甲基转移酶,分别是MET,CMT 和DRM,这3 类酶负责维持DNA 甲基化[8, 9]( 图 1)。甲基转移酶MET(methyltransferase Ⅰ)主要功能是在重复及单拷贝的DNA 序列中维持CG 双核苷酸中的胞嘧啶甲基化。DNA 甲基转移酶基因DDM Ⅰ(Decrease in DNA MethylationⅠ)突变也会导致CG 位点DNA 去甲基化,说明MET Ⅰ和DDM Ⅰ对维持CG 位点完整的DNA甲基化状态是必需的;植物特异的染色质甲基转移酶CMT3(chromomethylation 3) 主要维持CNG 以及CHH 核苷酸序列中胞嘧啶的甲基化;结构域重排甲基转移酶(domain rearranged methylation,DRM)维持失活转座子和转基因沉默位点的胞嘧啶甲基化修饰。
1.3 植物DNA甲基化的特征植物DNA 甲基化具有种属差异性。所有植物基因组中都存在不同程度的DNA 甲基化,高等植物基因组中约有6%-30% 的胞嘧啶发生甲基化,DNA 甲基化的发生比例因植物种类不同而有差异,以CG甲基化为例,拟南芥比例为22.3 %、杨树为41.9%、水稻为59.4 %[10]。
植物DNA 甲基化具有组织特异性。研究报道,在番茄叶片中CG、CNG 和CHH 的甲基化水平分别为85.51%、56.15% 及8.63%,但是在番茄果实中3 种甲基化水平分别为73.97%-79.16%、51.99-53.88% 及13.52-14.20%[11],表明同一物种不同组织的DNA 甲基化水平有差异。
植物DNA 甲基化具有发育阶段特异性。植物同一组织的不同发育时期或同一类型细胞不同发育阶段,基因组DNA 甲基化比例有所不同。报道显示,在白杨树中,春天生长及冬天冬眠时,植株分别出现整体范围内的高甲基化及低甲基化水平[12];在番茄果实成熟过程中,CG 甲基化水平降低[11]。这些结果说明DNA 甲基化在植物发育过程中是一个动态的过程。
1.4 植物与哺乳动物DNA甲基化的区别
植物和哺乳动物有着不同的序列特异性,不同的甲基化酶和迥异的甲基化遗传特性,具体区别如表 1 所示[13, 14, 15, 16]。在植物中,DNA 甲基化状态和甲基化的变换往往能忠实地被后代继承[17, 18],而在哺乳动物中,DNA 甲基化在胚胎发育的早期需要一个全部抹除和重新建立的过程[19]。
2 DNA 甲基化调控基因表达的机制近年来,随着对DNA 甲基化的深入研究,发现DNA 甲基化对于调节基因表达有3 种可能机制[21],详细介绍如下。
2.1 DNA甲基化干扰转录因子结合在基因组中,含有CpG 位点的启动子序列位于DNA 双螺旋结构的大沟内,此处也是众多蛋白转录因子与DNA 结合的部位。该处胞嘧啶发生甲基化后,对甲基化位点敏感的转录因子如碱性磷酸酶(AP2)、周期性AMP 依赖启动子(CAREB)、转录因子2(E2F2)、反式作用因子(NFκB)无法与相应的识别位点正常结合,导致转录无法启动,从而干扰相关基因的表达[22]。
2.2 甲基CpG结合蛋白阻碍转录因子结合MBPs 是特异的转录抑制物,通常以蛋白复合体的形式发挥作用。甲基CpG 结合蛋白复合体包括MeCP1 和MeCP2,后者对单一的甲基化CpG 位点有较高的亲和性。当转录因子的启动子识别序列中含有的CpG 位点发生甲基化时,MBPs 与转录因子竞争甲基化的结合位点[23]。这种竞争关系导致基因的转录表达受到阻碍。
2.3 染色体结构改变调控转录活性DNA 甲基化可以改变染色质的结构,抑制基因表达。染色质构型的改变伴随着组蛋白的乙酰化与去乙酰化过程。DNA 甲基化不会引起异染色质凝聚,但是可以导致组蛋白H3,H4 氨基端赖氨酸残基去乙酰化,影响核小体的结构,同时DNA 甲基化改变组蛋白H1 的连接作用从而调节基因活性[24]。此外,DNA 甲基化通过阻止转录因子的进入以诱导染色质失活的方式防止染色质活化。
3 植物DNA 甲基化与基因表达的关联性研究目前,对于植物DNA 甲基化与基因表达的关联性研究主要集中在两个方面:外界环境胁迫下单个基因的甲基化状态改变及对应的基因表达改变的分析;通过高通量测序技术进行全基因组甲基化测序和表达测序,从而进行两者关联性分析。
3.1 胁迫下植物特定基因位点DNA甲基化改变影响基因表达目前,大量实验结果证实了胁迫环境引起的单个基因的DNA 甲基化状态或程度的改变影响了对应基因的表达。相关的胁迫环境研究主要包括温度胁迫,化学试剂胁迫、金属胁迫、病原菌胁迫及辐射胁迫等。现对相关研究进展进行综述。
3.1.1 温度胁迫研究发现,在低温处理玉米时分离到一个低温特异表达基因ZmM Ⅰ 1,该基因片段含有类似逆转座子的基因序列,并且DNA 甲基化程度下降且在低温下特异转录表达。推测胁迫下基因编码区发生去甲基化,导致染色质结构较为松散,进而促进转录水平上调[25]。另有研究发现,离体烟草叶片在低温处理后,编码类甘油磷酸酯酶蛋白的基因NtGPDL 发生去甲基化,基因表达上调[26]。华扬等的研究证实在5℃下处理水稻48 h,利用甲基化敏感扩增多态性技术(methylation sensitiveamplification polymorphism,MSAP)找到了一系列甲基化差异片段,其中发现CIDM7 片段在冷胁迫后去甲基化,并且利用Northern 技术证明CIDM7 在冷处理后表达增加[27]。温度升高,拟南芥中编码基因At3g50770 甲基化水平降低,同时基因表达水平增高。遗憾的是,在编码基因At5g43260 中并未观察到甲基化水平与基因表达的相关性[28]。这表明环境刺激下DNA 甲基化的改变可以作为控制许多基因表达的开关,通过去除基因DNA 甲基化而对逆境做出响应。
3.1.2 化学试剂胁迫5-氮胞苷(5-azacytidine)甲基化抑制剂处理水稻后,获得了一个遗传稳定的矮化突变株Line-2,利用MSAP 技术分析发现Line-2突变系较其野生型具有特异的类似于水稻白叶枯病抗性基因Xa21 的克隆Xa21G,该基因启动子区在野生型中胞嘧啶完全甲基化,但是在Line-2 突变系中完全去甲基化,同时Xa21G 在Line-2 突变系中高表达,分析可能是由于基因启动子区去甲基化促进了Xa21G 的高表达[17]。Finnegan 等[29]利用去甲基化试剂处理水稻种子使其DNA 甲基化水平降低,使植株组织中正常情况下不表达的基因启动表达,植株出现矮化、叶片变小等一系列改变。另有报道称利用5-氮胞苷处理紫苏,紫苏在未经春化处理后开花,同时发现控制开花的基因有去甲基化现象[30]。拟南芥中的At1g13470 是一个通过RdDM 抑制基因表达的类似转座子沉默的基因,水杨酸激素(salicylicacid)处理之后,呈现出明显的去甲基化现象,并且基因表达也随之上调[31]。拟南芥受到二氧化硫胁迫之后,基因组胞嘧啶出现高甲基化现象,1-氨基环丙烷-1-羧酸合成酶6 和腈水解酶2 基因的启动子区甲基化水平降低,并且转录水平升高[32]。有研究表明,SO2 胁迫后拟南芥植株地上组织细胞中NIT2基因的编码区甲基化状态未变,而启动子区的多个CG 和CHH 位点去甲基化,胞嘧啶总的甲基化水平降低,同时NIT2 基因转录上调,证实了逆境胁迫响应基因的转录应答与启动子序列的甲基化特征有关,表明DNA 甲基化修饰在植物胁迫应答过程中发挥了重要作用[33]。以上研究结果都进一步证实了甲基化的去除对相应调控基因表达的促进作用。
李雪林等(Li)[34]研究发现高盐胁迫诱导棉花根中抗逆相关基因陆地棉cDNA 克隆GH_TMO 和海岛棉gypsy 反转座子反转录酶基因DNA 序列去甲基化,编码基因表达水平升高,作者认为NaCl 胁迫诱导上述基因发生去甲基化而活化基因表达以适应逆境胁迫。另有研究报道利用盐胁迫处理水稻,细胞周期进程受到干扰,推测可能是基因通过升高DNA甲基化水平而关闭了DNA 复制启动与DNA 损伤修复基因的表达,使细胞生长与分裂延迟[35]。
3.1.3 金属离子胁迫目前研究多集中在金属离子胁迫对植物DNA 甲基化水平的影响上,有报道研究铝离子胁迫离体烟草叶片,重亚硫酸盐处理DNA 后测序发现NtGPDL 基因启动子区DNA 甲基化状态未发生改变,基因编码区部分CG 位点迅速发生去甲基化,且基因表达量随之上调[26]。推测NtGPDL 基因去甲基化可能影响了染色质构造,也说明胁迫环境下基因表达调控不局限于依赖启动子区甲基化状态改变,也可以通过改变编码区的甲基化程度从而使染色质结构松弛进行调控基因表达。有研究发现利用3 mmol/L Pb(NO3)2 处理玉米12 h、24 h 和48h 与对照组比较,在基因区域上游2 000 bp 和下游2 000 bp 的临近启动子区的基因表达水平与DNA 甲基化水平呈现负相关。其中,AP2/ERF、zinc finger和leucine-rich repeat 基因发生去甲基化,同时基因表达水平上升[36]。
3.1.4 生物胁迫病原菌应答基因NtAlix1 在正常烟草叶片上不表达,编码区基因高度甲基化。在人工接种烟草花叶病毒(TMV)之后,NtAlix1 基因发生去甲基化,并且随时间呈现动态变化的特点,并且基因表达量也随之上调,作者认为在外部胁迫作用下DNA 去甲基化可活化染色质并且促进基因高表达,NtAlix1 基因甲基化状态可能作为一个基因表达的开关存在[37]。
3.1.5 辐射胁迫本课题组长期致力于研究空间辐射环境下植物DNA 甲基化与基因表达的关联性。经第20 颗返回式科学与技术卫星搭载的松粳6 号水稻种子,运用MSAP 技术以及双向电泳技术对水稻叶片DNA 位点甲基化状态及蛋白表达水平进行分析发现,尽管单株水稻存在个体差异,但是DNA 位点甲基化状态与蛋白表达结果表现出一致性:在以蛋白质表达量下调为主的单株材料中,其DNA 甲基化水平以甲基化水平上调为主;在以蛋白表达量上调为主的单株材料中,其DNA 甲基化水平上以DNA 去甲基化变化为主;在蛋白表达量没有主要变化趋势的单株材料中,其DNA 甲基化水平上甲基化变化和去甲基化变化也基本持平[38]。本课题组在前期研究中也发现,空间飞行和模拟重离子辐射都能引起基因编码区的DNA 甲基化变化,而且部分编码区发生甲基化变化的对应基因的转录水平也发生了变化:发现热休克蛋白DNAJ 以及分子伴侣家族蛋白对应基因位点去甲基化,两者基因表达均上调;与核酸代谢相关的蛋白DNA 胞嘧啶脱氨基酶对应基因位点发生甲基化,其基因表达下调。推测空间环境可能是通过改变DNA 甲基化状态改变了基因组的稳定性进而影响基因和蛋白质的表达状况[20]。
逆境胁迫下,DNA 甲基化作为一种重要的表观遗传现象,核苷酸序列未发生改变,而基因表达改变,是与基因活性开启和关闭密切相关的动态过程,参与了应答逆境胁迫的生命活动[2, 39]。逆境胁迫下,植物DNA 甲基化水平上调抑制基因表达,推测可能会降低常规新陈代谢的速率;特定抗性基因DNA 去甲基化活化基因表达,推测可能是活化染色质,使抗性基因表达上调,协助植物渡过逆境。
3.2 基于全基因组测序的植物DNA甲基化与基因表达的关联性研究全基因组甲基化分析采用的方法主要包括甲基化DNA 免疫共沉淀技术(methylated DNAimmunoprecipitation sequencing,MeDIP-seq)或者全基因组重亚硫酸盐测序技术(whole genome bisulfitesequencing,BS-seq),同时采用数字基因表达谱技术(digital gene expression profiling,DGE)或者转录组测序技术(transcriptome sequencing,RNA-seq)分析基因表达情况[40, 41],将两者联合分析讨论,分析基因组甲基化水平与基因表达水平的关联性[42]。
3.2.1 拟南芥DNA 甲基化与基因表达的关联性利用上述方法,在拟南芥中的研究发现,中度表达基因最有可能被甲基化,而高表达及低表达基因最不易被甲基化;且DNA 甲基化阻碍转录延伸[4]。另一研究进行的更细致分析表明,基因编码区甲基化的基因表达水平明显高于未甲基化的基因,但是启动子区甲基化的基因表达水平普遍低于未甲基化的基因[43]。
拟南芥met1 突变体中,CG 位点的甲基化水平降低,特异性诱导31 个基因高表达,突变体对病原菌的抗性提高,表明这31 个基因很可能与植物抗逆相关。在进一步实验中用病原菌对拟南芥进行处理并分析了基因组的甲基化状态变化,全基因组甲基化测序图谱显示有大量胁迫诱导的甲基化多态性位点出现,包括抗逆相关基因、转座子和一些重复序列,其中很多与mRNA 测序得到的表达差异基因密切相关,作者推测在一定程度上,拟南芥植株为了响应环境刺激,可以通过DNA 甲基化状态的改变调节基因表达[31]。
3.2.2 水稻DNA 甲基化与基因表达的关联性有课题组研究了两种水稻(耕种水稻Oryza sativa spp.japonica、indica 及野生稻Oryza rufipogon、Oryzanivara)孕穗期的基因组甲基化水平及其对基因表达的影响。采用了全基因组重亚硫酸盐测序技术,测序深度为4.54-13.5 倍,基因表达分析采用数字基因表达谱技术。结果与拟南芥的研究类似,即启动子区甲基化抑制基因表达,并且编码区甲基化与基因表达呈正相关,同时在基因转录末端(transcriptionaltermination region,TTRs)DNA 甲基化可以明显抑制基因表达,甚至对于基因表达的影响比启动子区甲基化更强烈[44]。
3.2.3 杨树DNA 甲基化与基因表达的关联性Liang等[45]将杨树(populus trichocarpa)基因按照表达水平分为了4 类:高表达、中度表达、低表达和沉默基因。研究发现,沉默基因与表达基因相比较,有明显的高甲基化水平,说明基因沉默可能是由于高甲基化引起的。在表达基因中,基因编码区域与上游区域甲基化水平和基因表达呈现明显的正相关;然而,在基因转录起始位置(transcriptional startregion,TSSs)和TTRs 以及下游区域甲基化水平和基因表达呈现负相关。基因编码区域甲基化基因比未甲基化基因有明显的高表达说明在基因编码区域2016,32(4) 赵倩等 :植物 DNA 甲基化与基因表达的关联性研究进展 21基因表达和甲基化呈现正相关性。在TSS 上游100bp,DNA 甲基化阻碍基因表达;在TSS 上游100-2 000 bp 及基因编码区域,DNA 甲基化与基因表达呈现正相关。用干旱胁迫处理杨树,沉默基因的甲基化水平明显上升。TSSs 上游100 bp 基因甲基化水平上升,上游2 000 bp 未甲基化的基因的基因表达水平降低。下游2 000 bp 基因在干旱处理后基因表达水平上升。基因编码区域甲基化及未甲基化基因的表达都没有明显改变。
3.2.4 大豆DNA 甲基化与基因表达的关联性研究将大豆(glycine max,cultivar Heinong44)基因按照表达水平分为了7 类,大豆的低表达基因启动子区CG、CHG 的甲基化水平较高,CHH 的甲基化水平相对较低。在基因编码区域,中等程度表达的基因有更高的CG 甲基化水平,低表达的基因上CHG、CHH 的甲基化水平高。这些结果暗示基因编码区域的CG 甲基化与中等程度表达的基因有关联,同时CHG、CHH 甲基化会导致基因沉默[46]。
3.2.5 玉米DNA 甲基化与基因表达的关联性有研究分析玉米(Zea mays)自交系478 和自交系58 的幼苗DNA 甲基化改变与基因表达变化的关系。两种玉米品系在同源相同基因中有119 个差异表达基因,差异甲基化区域在这119 个基因的上游2 000 bp 区域及基因内部有富集,在基因下游2 000 bp 区域未发现差异甲基化区域富集现象。这一结果说明育种过程中启动子区域和基因内部的基因甲基化变异可以影响基因表达水平[47]。在相同区域中,有2 211个差异甲基化区域,在11 个其上游2 000 bp 区域有差异甲基化区域的差异表达基因中,8 个基因表达与甲基化变化呈现负相关性;在10 个其基因内部有差异甲基化区域的差异表达基因中,6 个基因表达与甲基化变化呈现负相关性;在其中11 个上述基因中,有8 个基因表达上升,DNA 甲基化水平下降。同时,Eichten 等[48]的研究也证实了上述结果,发现差异基因的表达模式与DNA 甲基化改变负相关。
基于全基因组测序的植物DNA 甲基化与基因表达的研究发现,在基因不同区域上,两者关联性存在差异,植物基因启动子区甲基化与基因表达呈现负相关性;但是编码区基因甲基化与基因表达的关系在不同物种间有差异,在玉米中,DNA 甲基化与基因表达呈现负相关性;在水稻、拟南芥和杨树中DNA 甲基化与基因表达呈现正相关性,在大豆中中等程度表达的基因有更高的CG 甲基化水平,而低表达的基因上CHG、CHH 的甲基化水平高;在基因转录末端,水稻中DNA 甲基化与基因表达呈现负相关性。这一结果体现了植物DNA 甲基化与基因表达之间的复杂关系。
同时,这一结果与上述胁迫下单个基因甲基化与表达呈现负相关的结论并不完全一致,造成这一现象的原因是,研究单个基因甲基化状态所选取的基因位置比较随机(各个实验选取启动子区或者编码区并不统一[49])。随着高通量测序技术的应用,我们可以更加细致得观察基因各个区域甲基化与基因表达的关系,从而可以更加准确翔实地总结基因甲基化与基因表达的关联性。
4 结语在目前研究中,对植物DNA 甲基化的建立和维持机制以及对植物生命活动的调节已经很清楚。但是,针对植物DNA 甲基化的状态对基因表达的影响的认识还非常少,尤其是胁迫处理下,植物DNA 甲基化的改变对于基因表达的影响机制尚不完全清楚。DNA 甲基化是有机体极为重要的调控方式[15, 50],植物DNA 甲基化影响基因表达的研究进展揭示了两者之间可能存在对应关系,但是植物如何通过DNA甲基化的改变影响基因表达的研究尚处于起步阶段,亟待深入研究。
| [1] | Waddington CH. Canalization of development and the inheritance of acquired characters[J]. Nature, 1942, 150(3811):563-565. |
| [2] | Kumar S, Kumari R, Sharma V, et al. Roles, and establishment, maintenance and erasing of the epigenetic cytosine methylation marks in plants[J]. Journal of genetics, 2013, 92(3):629-666. |
| [3] | Bird A. DNA methylation patterns and epigenetic memory[J].Genes Dev, 2002, 16(1):6-21. |
| [4] | Zilberman D, Gehring M, Tran RK, et al. Genome-wide analysis of Arabidopsis thaliana DNA methylation uncovers an interdependence between methylation and transcription[J]. Nat Genet, 2007, 39(1):61-69. |
| [5] | Vanyushin BF, Ashapkin VV. DNA methylation in higher plants :past, present and future[J]. Biochim Biophys Acta, 2011, 1809(8):360-368. |
| [6] | Saze H, Tsugane K, Kanno T, et al. DNA methylation in plants :relationship to small RNAs and histone modifications, and functions in transposon inactivation[J]. Plant Cell Physiol, 2012, 53(5):766-784. |
| [7] | Mahfouz MM. RNA-directed DNA methylation :mechanisms and functions[J]. Plant Signaling & Behavior, 2010, 5(7):806-816. |
| [8] | Matzke MA, Birchler JA. RNAi-mediated pathways in the nucleus[J]. Nat Rev Genet, 2005, 6(1):24-35. |
| [9] | Finnegan E, Kovac K. Plant DNA methyltransferases[J]. Plant Molecular Biology, 2000, 43(2-3):189-201. |
| [10] | Feng S, Cokus SJ, Zhang X, et al. Conservation and divergence of methylation patterning in plants and animals[J]. Proc Natl Acad Sci USA, 2010, 107(19):8689-8694. |
| [11] | Zhong S, Fei Z, Chen YR, et al. Single-base resolution methylomes of tomato fruit development reveal epigenome modifications associated with ripening[J]. Nat Biotechnol, 2013, 31(2):154-159. |
| [12] | Conde D, González-Melendi P, Allona I. Poplar stems show opposite epigenetic patterns during winter dormancy and vegetative growth[J]. Trees, 2012, 27(1):311-320. |
| [13] | Lister R, Pelizzola M, Dowen RH, et al. Human DNA methylomes at base resolution show widespread epigenomic differences[J]. Nature, 2009, 462(7271):315-322. |
| [14] | Laurent L, Wong E, Li G, et al. Dynamic changes in the human methylome during differentiation[J]. Genome Res, 2010, 20(3): 320-331. |
| [15] | Law JA, Jacobsen SE. Establishing, maintaining and modifying DNA methylation patterns in plants and animals[J]. Nat Rev Genet, 2010, 11(3):204-220. |
| [16] | Zemach A, McDaniel IE, Silva P, et al. Genome-wide evolutionary analysis of eukaryotic DNA methylation[J]. Science, 2010, 328 (5980):916-919. |
| [17] | Akimoto K, Katakami H, Kim HJ, et al. Epigenetic inheritance in rice plants[J]. Ann Bot, 2007, 100(2):205-217. |
| [18] | Boyko A, Kovalchuk I. Genome instability and epigenetic modification--heritable responses to environmental stress?[J]. Curr Opin Plant Biol, 2011, 14(3):260-266. |
| [19] | Reik W, Dean W, Walter J. Epigenetic reprogramming in mammalian development[J]. Science, 2001, 293(5532): 1089-1093. |
| [20] | 史金铭. 空间和重离子辐射环境的诱变效应与DNA 甲基化变化的关联[D]:哈尔滨:哈尔滨工业大学, 2010. |
| [21] | Attwood J, Yung R, Richardson B. DNA methylation and the regulation of gene transcription[J]. Cellular and Molecular Life Sciences CMLS, 2002, 59(2):241-257. |
| [22] | Goren A, Simchen G, Fibach E, et al. Fine tuning of globin gene expression by DNA methylation[J]. PLoS One, 2006, 1(1): e46. |
| [23] | Das PM, Singal R. DNA methylation and cancer[J]. J Clin Oncol, 2004, 22(22):4632-4642. |
| [24] | Tariq M, Paszkowski J. DNA and histone methylation in plants[J]. Trends Genet, 2004, 20(6):244-251. |
| [25] | Steward N, Ito M, Yamaguchi Y, et al. Periodic DNA methylation in maize nucleosomes and demethylation by environmental stress[J]. J Biol Chem, 2002, 277(40):37741-37746. |
| [26] | Choi CS, Sano H. Abiotic-stress induces demethylation and transcriptional activation of a gene encoding a glycerophosphodiesteraselike protein in tobacco plants[J]. Mol Genet Genomics, 2007, 277(5):589-600. |
| [27] | 华扬, 陈学峰, 熊建华, 等. 水稻冷胁迫诱导的甲基化差异片段 CIDM7 的分离和分析[J]. 遗传, 2005, 27(4):595-600. |
| [28] | Naydenov M, Baev V, Apostolova E, et al. High-temperature effect on genes engaged in DNA methylation and affected by DNA methylation in Arabidopsis[J]. Plant Physiol Biochem, 2015, 87102-87108. |
| [29] | Finnegan EJ, Genger RK, Peacock WJ, et al. DNA methylation in plants[J]. Annual Review of Plant Biology, 1998, 49(1):223- 247. |
| [30] | Takeno K. Epigenetic regulation of photoperiodic flowering[J]. Plant Signaling & Behavior, 2014, 5(7):788-791. |
| [31] | Dowen RH, Pelizzola M, Schmitz RJ, et al. Widespread dynamic DNA methylation in response to biotic stress[J]. Proc Natl Acad Sci USA, 2012, 109(32):E2183-E2191. |
| [32] | Yi H, Li L. DNA methylation changes in response to sulfur dioxide stress in Arabidopsis plants[J]. Procedia Environmental Sciences, 2013, 1837-42. |
| [33] | 李利红, 仪慧兰, 王艺雯, 等. 二氧化硫胁迫诱导拟南芥NIT2基因DNA 甲基化修饰[J]. 农业环境科学学报, 2012(4):685-690. |
| [34] | Li XL, Lin ZX, Nie YC, Zhang XL. MSAP analysis of epigenetic changes in cotton(Gossypium hirsutum L. )under salt stress[J]. Acta Agronomica Sinica, 2009, 35(4):588-596. |
| [35] | 曹聪. 水稻早期盐胁迫生长调控中的DNA 甲基化研究[D].北京:中国农业大学, 2014. |
| [36] | Ding H, Gao J, Qin C, et al. The dynamics of DNA methylation in maize roots under Pb stress[J]. Int J Mol Sci, 2014, 15(12): 23537-23554. |
| [37] | Wada Y, Miyamoto K, Kusano T, et al. Association between upregulation of stress-responsive genes and hypomethylation of genomic DNA in tobacco plants[J]. Mol Genet Genomics, 2004, 271(6):658-666. |
| [38] | 王连嵋. 空间环境引起水稻DNA 甲基化与蛋白质组变化及其关联分析[D]. 哈尔滨:哈尔滨工业大学, 2007. |
| [39] | Grativol C, Hemerly AS, Ferreira PC. Genetic and epigenetic regulation of stress responses in natural plant populations[J]. Biochim Biophys Acta, 2012, 1819(2):176-185. |
| [40] | Schmitz RJ, Zhang X. High-throughput approaches for plant epigenomic studies[J]. Curr Opin Plant Biol, 2011, 14(2): 130-136. |
| [41] | Stirzaker C, Taberlay PC, Statham AL, et al. Mining cancer methylomes :prospects and challenges[J]. Trends Genet, 2014, 30(2):75-84. |
| [42] | Moreno-Risueno MA, Busch W, Benfey PN. Omics meet networks - using systems approaches to infer regulatory networks in plants[J]. Curr Opin Plant Biol, 2010, 13(2):126-131. |
| [43] | Zhang X, Yazaki J, Sundaresan A, et al. Genome-wide highresolution mapping and functional analysis of DNA methylation in Arabidopsis[J]. Cell, 2006, 126(6):1189-1201. |
| [44] | Li X, Zhu J, Hu F, et al. Single-base resolution maps of cultivated and wild rice methylomes and regulatory roles of DNA methylation in plant gene expression[J]. BMC Genomics, 2012, 13 :300. |
| [45] | Liang D, Zhang Z, Wu H, et al. Single-base-resolution methylomes of Populus trichocarpa reveal the association between DNA methylation and drought stress[J]. BMC Genet, 2014, 15(Suppl1) 1S9. |
| [46] | Song QX, Lu X, Li QT, et al. Genome-wide analysis of DNA methylation in soybean[J]. Mol Plant, 2013, 6(6):1961- 1974. |
| [47] | 解少俊. 玉米表观遗传组的研究[D]. 北京:中国农业大学,2014. |
| [48] | Eichten SR, Briskine R, Song J, et al. Epigenetic and genetic influences on DNA methylation variation in maize populations[J]. Plant Cell, 2013, 25(8):2783-2797. |
| [49] | Karan R, DeLeon T, Biradar H, et al. Salt stress induced variation in DNA methylation pattern and its influence on gene expression in contrasting rice genotypes[J]. PLoS One, 2012, 7(6): e40203. |
| [50] | Gutzat R, Mittelsten Scheid O. Epigenetic responses to stress : triple defense?[J]. Curr Opin Plant Biol, 2012, 15(5):568- 573. |