2. 西南林业大学 西南地区生物多样性保育国家林业局重点实验室,昆明 650224 ;
3. 西南林业大学 云南生物多样性研究院,昆明 650224 ;
4. 西南林业大学 西南山地森林资源保育与利用省部共建教育部重点实验室,昆明 650224
2. Key Laboratory of Biodiversity Conservation in Southwest China,State Forestry Administration,Southwest Forestry University,Kunming 650224 ;
3. Yunnan Academy of Biodiversity,Southwest Forestry University,Kunming 650224 ;
4. Key Laboratory for Forest Resources Conservation and Use in the Southwest Mountains of China,Ministry of Education,Southwest Forestry University, Kunming 650224
滇杨(Populus yunnanensis Dode)是我国西南地区特有的杨属青杨派树种,分布于云南中部、北部及南部的开远、蒙自和文山,贵州威宁,四川凉山州的美姑、布拖等地,生长于海拔1 300-3 200 m, 部分地区可达到3 700 m,多沿山涧河溪生长,在海拔2 000 m 以上地带有纯林存在,而在海拔较低地区(<1 900 m)主要以混交林或散生形式存在[1]。 由于滇杨具有生长速度快、耐寒、易无性繁殖、抗叶锈病和叶斑病等优良性状[1, 2],兼有较高的观赏价值[3],被应用于环境保护、城市绿化并在林业生产中扮演着重要角色[4],具有独特的培养价值。目前关于滇杨的研究主要集中于生长特性[5, 6, 7, 8]及栽培繁育[9, 10, 11, 12]方面,通过与美洲黑杨(P. deltoides)、欧洲黑杨(P. nigra)的种间杂交对其遗传改良做出了尝试[13, 14]。但有关其分布区域内的遗传多样性研究还未见报道。
在用于分析植物遗传多样性及种质资源的多种分子标记中,SRAP(Sequence-Related Amplified Polymorphism)针对开放阅读框进行标记,在分析种间、种内杂交调查基因多态性与发现新的变异位点等方面有不可估量的价值[15]。该方法已被应用于遗传多样性分析[16]、遗传图谱构建[17]及差异表达基因分离[18]等实验研究。杨树SRAP 标记体系的建立和优化已表明SRAP 标记是一种适于杨树遗传多样性研究的分子标记技术[19, 20]。基于此,本研究采用SRAP 标记分析采集于四川省凉山州、云南省丽江市、曲靖市、昆明市、大理市、楚雄市和昭通市7 个滇杨居群的208 个样本,探讨滇杨遗传多样性与遗传结构,以期为其种质资源的保护、开发和利用提供科学理论依据。
1 材料与方法 1.1 材料以行政区划的地级市(自治州)为居群单位, 按照随机取样的原则,从四川省凉山州、云南省丽江市、曲靖市、昆明市、大理市、楚雄市和昭通市共计7 个滇杨居群中选取样株,样株间相隔100 m 以上,采集嫩叶用硅胶干燥,带回实验室保存备用。
1.2 方法 1.2.1 DNA 提取和SRAP 分析干燥后的叶片用冷冻混合球磨仪进行研磨,采用改良的SDS 法依照标准酚/ 氯仿流程[21]提取分析样本基因组DNA,利用0.8% 的琼脂糖凝胶电泳和核酸蛋白检测仪共同检测基因组DNA 的纯度和浓度。
1.2.2 SRAP 分析根据SRAP 引物设计原理[22, 23], 在已有标准引物序列的基础上自行设计正反引物各15 条,共计225 对引物组合。从中筛选出重复性好、 多态性高、分辨率高的7 对引物组合用于后续实验。
PCR 反应体系(10 μL):10×buffer( 含25 mmol/L Mg2+)1 μL,dNTP(2.5 mmol/L)0.7 μL,正反引物(10 μmol/L)各1.5 μL,Taq 酶(2.5 U/μL)0.2 μL,DNA 模板2.5 μL,ddH2O 补足(2.6 μL) 至10 μL。PCR 扩增程序为:94℃预变性5 min ;前5 个循环,94℃变性50 s,36℃退火50 s,72℃延伸90 s ; 后30 个循环,94℃变性50 s,50℃退火50 s,72℃ 延伸90 s ;72℃延伸10 min。
PCR 扩增产物经94℃变性10 min 后在6% 变性聚丙烯酰胺上进行电泳分离,恒定功率为70 W,电泳产物采用银染法[24]进行谱带的显色反应。
1.2.3 数据分析根据电泳图谱中相同片段位置谱带的“有”与“无”构建0/1 矩阵。采用POPGENE version 1.32 软件[25]计算滇杨每个居群的多态性条带数、多态带百分率、观测等位基因数(Na)、有效等位基因数(Ne)、Nei’s 基因多样性指数(H)、 Shannon’s 信息指数(I),以及滇杨总遗传多样性(Ht)、居群内遗传多样性(Hs)、居群间遗传分化系数(Gst)、基因流(Nm)、Nei’s 遗传距离和遗传相似系数。应用AMOVA 1.55 软件[26]进行分子变异方差分析。利用GENALEX v6.41 软件[27]对滇杨居群地理距离和遗传距离的相关性进行Mantel test 分析,依据遗传距离进行主坐标轴分析(PCoA)。 在NTSYS2.1e 软件[28]中运用非加权配对算术平均法(UPGMA)进行基于遗传相似系数的聚类分析。 采用Structure 2.3.1[29]进行Bayesian 聚类分析,选择混合模型(Admixture model)和相关等位基因模型(Correlated alleles frequencies model),设定参数“burn-in-period”为104,MCMC 重复运算105 次,K 值检测范围设定为1-11,每个K 值独立运行20 次。 应用Clumpp 1.1.2[30]对数据进行整理和分析,参照Evanno 等[31]的方法确定最佳K 值,利用Distruct 1.1[32]绘制Bayesian 聚类图。
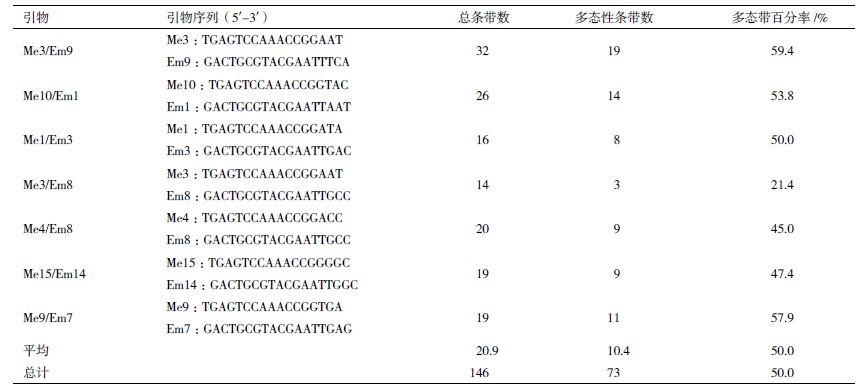
2 结果 2.1 SRAP多态性分析筛选出的7 对引物组合共扩增出条带146 条, 多态性条带73 条,多态带百分率为50%。引物组合Me3/Em9 获得的总条带数(32 条)、多态性条带数(19 条)、多态带百分率(59.4%)均最高,引物组合Me3/Em8 的最少,总条带数、多态性条带数和多态带百分率分别为14 条、3 条和21.4%(图 1 和表 1)。
|
| 图 1 PCR 扩增产物的6% 变性聚丙烯酰胺凝胶电泳银染图 |
7 个居群的遗传多样性(表 2)显示,平均多态带百分率为22.70%,平均观测等位基因数为1.227 0, 平均有效等位基因数为1.107 6,Nei’s 基因多样性指数与Shannon’s 信息指数分别为0.064 4 与0.098 9。 其中丽江居群的有效等位基因数(1.148 2)、Nei’s 基因多样性指数(0.088 7)和Shannon’s 信息指数(0.134 8)均最高,略高于曲靖居群(Ne=1.134 3, H=0.081 2,I=0.125 7);而多态带百分率与观测等位基因数则是曲靖居群最高,分别为30.82% 和1.308 2 ;昭通居群的多态带百分率(14.38%)、观测等位基因数(1.143 8)、有效等位基因数(1.070 1)、 图 1 PCR 扩增产物的6% 变性聚丙烯酰胺凝胶电泳银染图表 1 SRAP 引物及其扩增结果引物引物序列(5'-3') 总条带数多态性条带数多态带百分率/% Nei’s 基因多样性指数(0.042 6)和Shannon’s 信息指数(0.065 2)均最低。
滇杨总遗传多样性(Ht)为0.136 9,居群内平均遗传多样性(Hs)为0.064 4,7 个居群总遗传分化系数(Gst)为0.529 4,基因流(Nm)为0.444 4, 表明居群间的遗传变异大于居群内,其基因交流处于中等水平。分子方差分析结果(表 3)显示,居群间的变异分量为6.71,占总变异的55.61%,差异达极显著水平(P<0.001)。而居群内个体的变异分量为5.36,占总变异的44.39%,低于居群间的变异程度。由此可知,滇杨的遗传差异主要来自于不同居群之间。
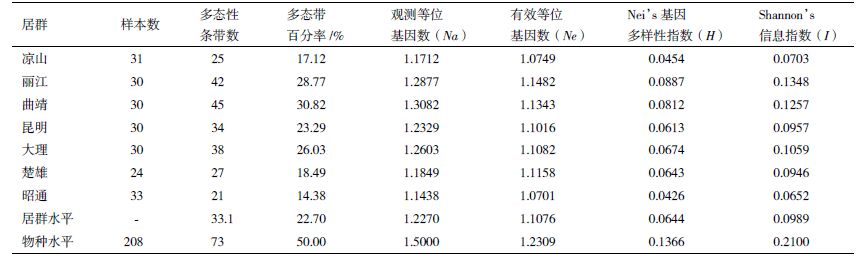
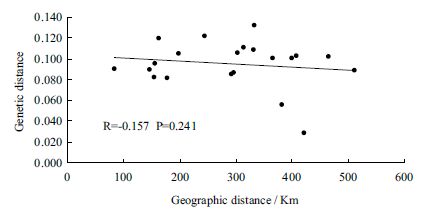
两两居群间的最高遗传分化系数出现在昆明与凉山居群之间,其Gst 值为0.523 3 ;其次为昭通与凉山居群Gst 值为0.509 2 ;而楚雄与昭通居群(Nm=1.987 8)、丽江与曲靖居群(Nm=1.704 9)基因交流最多,其Nm 值均大于1(表 4)。经Mantel 检验,滇杨7 个居群的地理距离与遗传距离的相关系数为-0.157(P=0.241),表明两者间无相关性(图 2)
|
| 图 2 滇杨7 个居群的地理距离和遗传距离相关性 |
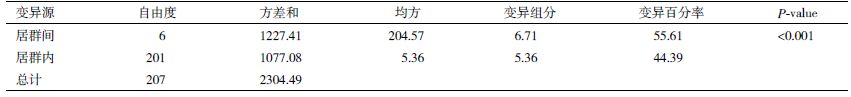
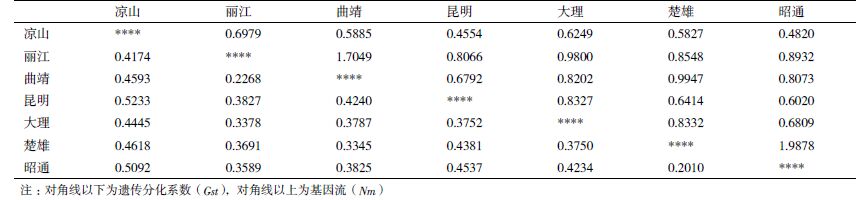
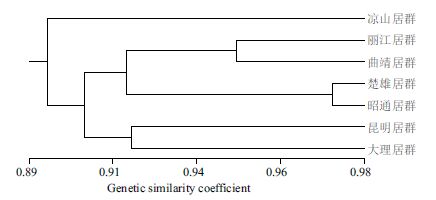
居群间的平均遗传相似系数为0.909 7,变幅为0.876 2-0.971 6,楚雄与昭通居群之间遗传相似系数最高,为0.971 6,其次为丽江与曲靖居群(0.945 6), 凉山与昆明居群最低,为0.876 2。如图 3 所示,在阈值为0.91 时可分为3 个组,凉山居群为第1 组, 昆明与大理居群构成第2 组,其余4 个居群为第3 组。主坐标轴分析(PCoA)中,前3 个特征向量的累计贡献率为72%,基于PCoA 的结果与UPGMA 聚类结果基本一致,昭通与楚雄居群聚类,丽江与曲靖居群聚类,而凉山、大理、昆明3 个居群相对分散(图 4)。
|
| 图 3 滇杨7 个居群之间的UPGMA 聚类图 |
|
| 图 4 滇杨7 个居群的主坐标轴分析 |
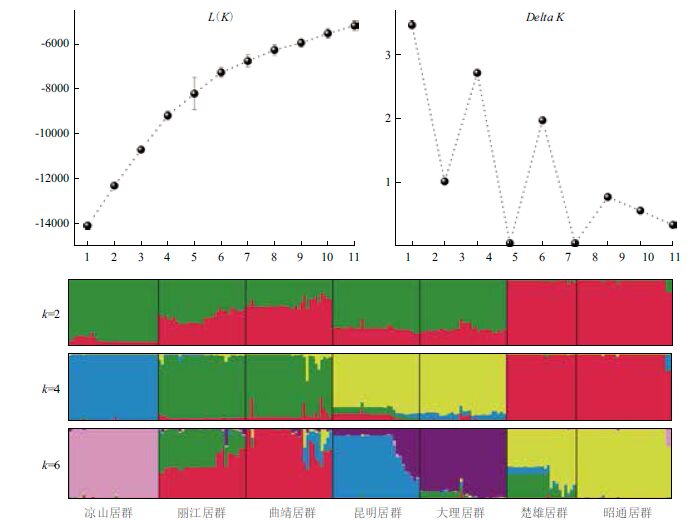
根据Evanno 等[31]的方法确定最佳K 值,滇杨7 个居群的Delta K 在K=2 时最大(3.47),K=4 (2.71)和K=6(1.97)时次之,分别对其进行聚类(图 5),3 种K 值的聚类结果基本一致,也与PCoA和UPGMA 的聚类结果类似。如当K=4 时,凉山居群单独构成第1 组,丽江和曲靖居群为第2 组,昆明和大理居群组成第3 组,昭通和楚雄居群被分到第4 组。
|
| 图 5 滇杨7 个居群的Bayesian 分析 |
SRAP 标记多态性高,产率中等,重复性好, 在基因组中分布均匀;较易对扩增得到的目的片段进行测序;操作简单,其正向引物可以与反向引物两两搭配组合,用少量的引物可以得到多个引物对,引物的使用效率高,可降低实验成本,研究表明,SRAP 标记比AFLP、RAPD 等标记方式更能体现表型的多样性及进化史[33, 34],其聚类结果要比AFLP 更相似于形态学分析结果[19]。SRAP 标记已广泛应用于遗传多样性分析,但在不同树种中扩增得到的多态性各异,如紫铆( Butea monosperma)、 桑树(Morus)和石榴(Punica granatum)的多态带百分率分别为91.90%、78.41% 和53.20%[35, 36, 37]。而SRAP 标记在杨树遗传变异分析中的应用较少,还处于起步阶段,郭丽琴等[19]、陈罡等[20]通过建立和优化SRAP-PCR 体系,为SRAP 标记在杨树中的应用奠定了科学基础,随后郭娟等[38]对比了SRAP 和EST-SSR 标记在美洲黑杨品种间遗传差异分析中的结果,并认为SRAP 标记更适合用于杨树亲缘关系较近材料的分析。
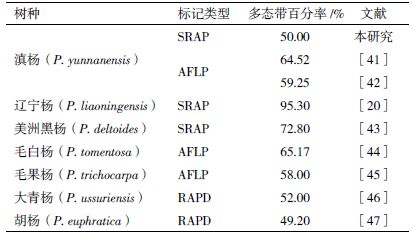
遗传多样性是生物长期生存、进化和适应的结果[39],研究遗传多样性,有利于合理保存和利用基因资源[40]。在滇杨的研究中,纵丹等[41]采用AFLP 分析52 株滇杨优树的遗传多样性,其多态带百分率为64.52%,李里等[42]同样用AFLP 标记对采集于四川省和云南省的56 株滇杨优树的遗传多样性进行了分析,多态带百分率为59.25%。本研究中,滇杨的SRAP 多态带百分率为50%,略低于AFLP 分析结果。与杨属其他树种相比,辽宁杨为95.3%(SRAP)[20],美洲黑杨为72.8%(SRAP)[43], 毛白杨为65.17%(AFLP)[44],毛果杨为58% (AFLP)[45],大青杨为52%(RAPD)[46],胡杨为49.2%(RAPD)[47],表明滇杨的遗传多样性水平中等。同时,由于无性繁殖群体与近缘有性繁殖种相比遗传多样性较低[39],滇杨居群的Nei’s 基因多样性指数与Shannon’s 信息指数(H=0.1366, I=0.2100)均低于无性繁殖与有性繁殖同时存在的苦杨(P. laurifoli,SSR,H=0.3924,I=0.9185)和欧洲黑杨(SSR,H=0.2187,I=0.3348)[48](表 5)。
遗传分化系数Gst 为居群间变异与总变异的比值,Nm 为居群间和居群内的遗传物质交流[49],二者均为分析居群遗传结构的重要指标。Govindajaru 认为,Nm>1 则基因交流程度高,0.250 <Nm<0.999 则基因交流适中[50]。本研究中,丽江与曲靖居群(Gst=0.226 8)、楚雄与昭通居群(Gst=0.201 0)遗传分化系数低且Nm 值大于1,表明其变异主要来自于居群内部且居群间存在较大的基因交流。然而,上述各居群的地理位置相互间隔并不利于基因交流,滇杨自身的无性繁殖方式和绿化造林及木材生产等人为活动很可能是造成这种现象的主要原因。 滇杨是雌雄异株植物,但由于雄株比雌株更能适应恶劣的环境条件[4, 51, 52, 53],使滇杨林分基本由雄株组成,雌株极为少见,在自然状态下多以根蘖方式繁衍。生殖方式上的差异可以造成遗传分化系数Gst 上近40% 的变异,一般而言,自交种总的遗传变异中有51% 的变异存在于居群之间[54],其平均基因流为0.43[55]。本研究中滇杨居群间差异占总变异的55.61%,基因流为0.444 4,仅略高于自交种,而当前滇杨的繁殖方式使其更接近于无性系植物,无性系植物种群间基因流存在较大障碍时,居群间遗传变异比例会大为增加[56]。由此推断,滇杨居群间差异有继续增大的可能,即使居群相邻也会因基因交流受阻而继续分化。滇杨现存林分以人工林为主, 人工繁殖主要通过大枝扦插来实现,致使人为因素发挥了重要作用。丽江与曲靖居群、楚雄与昭通居群,两对居群的地理距离较远而遗传相似度较高可能也与此有关。
滇杨主要被用作行道树或民间自发地用于四旁零散种植,栽种地域内的百姓根据自己的判断在有限范围内选取本地优良单株采枝扦插繁殖[1],促使滇杨在选取一定种源后仅在小范围内传播,缺乏居群间的交流。同时,不科学的人工种植会造成遗传上的高度同质化[57],降低滇杨的遗传多样性。因此, 保护滇杨的遗传多样性,构建核心种质并完善遗传改良策略,是开发滇杨资源的必要环节。
4 结论本研究利用7 对SRAP 引物组合从7 个不同居群的208 个滇杨样本中共扩增出146 条条带,多态带百分率为50%。滇杨居群间的遗传分化系数(Gst) 为0.529 4,基因流(Nm)为0.444 4,AMOVA 分析结果表明居群间的变异占总变异的55.61%,表明滇杨的遗传变异主要存在于居群间,居群间基因交流处于中等水平。UPGMA、PCoA 和Bayesian 聚类分析结果均显示丽江与曲靖居群、楚雄与昭通居群的亲缘关系较近,Mantel test 结果表明滇杨居群间的遗传距离与地理距离不相关。
| [1] | 何承忠, 车鹏燕, 周修涛, 等. 滇杨基因资源及其研究概况[J]. 西南林学院学报, 2010, 30(1):83-88, 94. |
| [2] | 何承忠, 张志毅, 陈宝昆, 等. 滇杨遗传改良策略初论[J]. 西部林业科学, 2004, 33(1):44-48. |
| [3] | Lin TT, Zhu XQ, Zhang F, et al. The detoxification effect of nitrogen on cadmium stress in Populus yunnanensis[J]. Botany Research Journal, 2011, 4(1):13-19. |
| [4] | Jiang H, Korpelainen H, Li C. Populus yunnanensis males adopt more efficient protective strategies than females to cope with excess zinc and acid rain[J]. Chemosphere, 2013, 91(8):1213-1220. |
| [5] | 周安佩, 刘东玉, 纵丹, 等. 滇杨侧芽不同季节内源激素含量变化动态[J]. 林业科学研究, 2014, 27(1):113-119. |
| [6] | 吴海, 周安佩, 刘东玉, 等. 滇杨落叶期侧芽内源激素含量与分枝特性的关系[J]. 西南林业大学学报, 2013, 33(2):35-41, 47. |
| [7] | 王德新, 张晏, 段安安, 等. 滇杨优树无性系物候期观测[J]. 西南林学院学报, 2009, 29(6):20-23, 27. |
| [8] | 罗建勋, 郑文, 辜云杰, 等. 滇杨生长特性研究[J]. 西南林学院学报, 2006, 26(6):22-25. |
| [9] | 张春霞, 樊军锋, 黄建, 等. 滇杨的组织培养和植株再生[J]. 植物生理学通讯, 2006, 42(6):1131. |
| [10] | 辛培尧, 刘岩, 李根前, 等. 滇杨组培苗的移栽技术研究[J]. 中南林业科技大学学报, 2012, 32(2):23-25, 30. |
| [11] | 刘东玉, 周安佩, 纵丹, 等. 不同处理措施对滇杨根萌苗形成与生长的影响[J]. 西南林业大学学报, 2014, 34(1):31- 35, 39. |
| [12] | 韩燕, 段安安, 张晏, 等. 滇杨扦插繁殖试验初报[J]. 西南林学院学报, 2009, 29(2):86-88. |
| [13] | 罗建勋, 索铁阿罗, 辜云杰, 等. 滇杨与美洲黑杨派间杂交育种试验初报[J]. 四川林业科技, 2010, 31(2):57-59. |
| [14] | 张春霞, 樊军锋, 高建社. 欧洲黑杨分别与川杨和滇杨杂交及其F1 代苗期性状[J]. 中南林业科技大学学报, 2007, 27(3): 37-40. |
| [15] | Robarts DWH, Wolfe AD. Sequence-related amplified polymorphism( SRAP)markers :a potential resource for studies in plant molecular biology[J]. Applications in Plant Sciences, 2014, 2(7): 1400017. |
| [16] | Li XY, Li J, Zhao ZJ, et al. Sequence-related amplified polymorphism( SRAP)for studying genetic diversity and population structure of plants and other living organisms :a protocol[J]. The Journal of Animal & Plant Sciences, 2014, 24(5):1478-1486. |
| [17] | Venkat SK, Bommisetty P, Patil MS, et al. The genetic linkage maps of Anthurium species based on RAPD, ISSR and SRAP markers[J]. Scientia Horticulturae, 2014, 178 :132-137. |
| [18] | Liu C, Yuan D, Zhang X, et al. Isolation, characterization and mapping of genes differentially expressed during fibre development between Gossypium hirsutum and G. barbadense by cDNASRAP [J]. Journal of Genetics, 2013, 92(2):175-181. |
| [19] | 郭丽琴, 卫尊征, 张金凤, 等. 均匀设计优化杨属的SRAPPCR 反应体系[J]. 北京林业大学学报, 2010, 32(2):34- 38. |
| [20] | 陈罡, 关明东, 叶景丰, 等. 杨树SRAP-PCR 反应体系的建立与优化[J]. 北方园艺, 2010(16):132-134. |
| [21] | Murray M, Thompson WF. Rapid isolation of high molecular weight plant DNA[J]. Nucleic Acids Research, 1980, 8(19):4321- 4326. |
| [22] | Li G, Quiros CF. Sequence-related amplified polymorphism (SRAP), a new marker system based on a simple PCR reaction : its application to mapping and gene tagging in Brassica[J]. Theor Appl Genet, 2001, 103 :455-461. |
| [23] | Bharti A, Neelam R, Yadav VC, et al. Sequence-related amplified polymorphism(SRAP)molecular marker system and its applications in crop improvement[J]. Mol Breading, 2012, 30 : 1635-1648. |
| [24] | Tixier MH, Sourdille P, Rder M, et al. Detection of wheat microsatellites using a non radioactive silver-nitrate staining method[J]. Journal of Genetic Breeding, 1997, 51(2):175- 177. |
| [25] | Yeh FC, Yang R, Boyle TJ. PopGene32, microsoft windows-based freeware for population genetic analysis(version 1. 32)[M]. Edmonton :Molecular Biology and Biotechnology Centre, University of Alberta, 2000. |
| [26] | Excoffier L. Analysis of molecular variance(AMOVA)version 1. 55[M]. Switzerland :Genetics and Biometry Laboratory, University of Geneva, 1993. |
| [27] | Peakall R, Smouse P. Genalex6 :genetic analysis in excel population genetic software for teaching and research[J]. Mol Ecol Notes, 2006, 6 :288-295. |
| [28] | Rohlf EJ. NTSYS-pc :numerical taxonomy and multivariate analysis system. version 2. 1[M]. New York :Exeter Software, 2000. |
| [29] | Pritchard JK, Stephens M, Donnelly P. Inference of population structure using multilocus genotype data[J]. Genetics, 2000, 155 :945-959. |
| [30] | Jakobsson M, Rosenberg NA. CLUMPP :a cluster matching and permutation program for dealing with label switching and multimodality in analysis of population structure[J]. Bioinformatics, 2007, 23 :1801-1806. |
| [31] | Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE :a simulation study[J]. Molecular Ecology, 2005, 14 :2611-2620. |
| [32] | Rosenberg NA. DISTRUCT :a program for the graphical display of population structure[J]. Molecular Ecology Notes, 2004, 4 : 137-138. |
| [33] | Ferriol M, Pico B, Nuez F. Genetic diversity of a germplasm collection of Cucurbita pepo using SRAP and AFLP markers[J]. Theor Appl Genet, 2003, 107(2):271-282. |
| [34] | Ferriol M, Pico B, Nuez F. Genetic diversity of some accession of Cucurbita maxima from Spain using RAPD and SBAP markers [J]. Genetic Resources and Crop Evolution, 2003, 50(3):227- 238. |
| [35] | Vashishtha A, Jehan T, Lakhanpaul S. Genetic diversity and population structure of Butea monosperma(Lam. )Taub. -a potential medicinal legume tree[J]. Physiology and Molecular Biology of Plants, 2013, 19(3):389-397. |
| [36] | Hu DC, Zhang P, Hao W, et al. SRAP analysis on the genetic relationships of 24 mulberry(Morus L. )accessions in the lower regions of the yellow river[J]. Genetic Resources and Crop Evolution, 2015, 62(1):13-19. |
| [37] | Soleimani MH, Talebi M, Sayed-Tabatabaei BE. Use of SRAP markers to assess genetic diversity and population structure of wild, cultivated, and ornamental pomegranates(Punica granatum L. ) in different regions of Iran[J]. Plant Systematics and Evolution, 2012, 298(6):1141-1149. |
| [38] | 郭娟, 樊军锋, 梁军. 美洲黑杨遗传差异的SRAP 和EST-SSR 分析[J]. 西北植物学报, 2013, 33(9):1762-1767. |
| [39] | 文亚峰, 韩文军, 吴顺. 植物遗传多样性及其影响因素[J]. 中南林业科技大学学报, 2010, 30(12):80-87. |
| [40] | 葛颂, 洪德元. 生物多样性研究的原理与方法[M]. 北京: 中国科学技术出版社, 1994. |
| [41] | 纵丹, 员涛, 周安佩, 等. 滇杨优树遗传多样性的AFLP 分析[J]. 西北林学院学报, 2014, 29(4):103-108. |
| [42] | 李里, 江涛, 王滨蔚, 等. 滇杨优树基因组及其无性系落叶期侧芽cDNA 的AFLP 分析[J]. 分子植物育种, 2013, 11(3): 393-402. |
| [43] | 郭娟, 樊军锋, 梁军, 等. 利用SRAP 标记鉴别美洲黑杨及指纹图谱构建[J]. 西北林学院学报, 2014, 29(2):98-102. |
| [44] | 何承忠. 毛白杨遗传多样性及起源研究[D]. 北京:北京林业大学, 2005. |
| [45] | Gervera MT, Storme V, Ivens B, et al. Dense genetic linkage maps of three Populus species(Populus detoides, P. nigra and P. trichocarpa)based on AFLP and Microsatellite marker[J]. Genetics, 2001, 158 :787-809. |
| [46] | 苏晓华, 张绮纹, 郑先武, 等. 用RAPD 分析大青杨天然群体的遗传结构[J]. 林业科学, 1997, 33(6):504-512. |
| [47] | Saito Y, Shiraishi S, Tanimoto T, et al. Genetic diversity of Populus euphratica populations in northwestern China determined by RAPD DNA analysis[J]. New Forests, 2002, 23 :97-103. |
| [48] | 郑书星, 张建国, 何彩云, 等. 新疆额尔齐斯河流域苦杨与欧洲黑杨遗传多样性分析[J]. 林业科学研究, 2014, 27(3): 295-301. |
| [49] | Grant V. The evolutionary process :a critical study of evolutionary theory[M]. New York :Columbia University Press, 1991. |
| [50] | Buso GSC, Rangel PH, Ferreira ME. Analysis of genetic variability of south American wild rice populations(Oryza glumaepatula) with isozymes and RAPD markers[J]. Molecular Ecology, 1998, 7(1):107-118. |
| [51] | Jiang H, Peng SM, Zhang S, et al. Transcriptional profiling analysis in Populus yunnanensis provides insights into molecular mechanisms of sexual differences in salinity tolerance[J]. Journal of Experimental Botany, 2012, 63(10):3709-3726. |
| [52] | Chen LH, Han Y, Jiang H, et al. Nitrogen nutrient status induces sexual differences in responses to cadmium in Populus yunnanensis[J]. Journal of Experimental Botany, 2011, 62(14): 5037-5050. |
| [53] | Chen L, Zhang S, Zhao H, et al. Sex-related adaptive responses to interaction of drought and salinity in Populus yunnanensis[J]. Plant, Cell and Environment, 2010, 33 :1767-1778. |
| [54] | 张大勇, 姜新华. 植物交配系统的进化、资源分配对策与遗传多样性[J]. 物生态学报, 2001, 25(2):130-143. |
| [55] | Morjan CL, Rieseberg LH. How species evolve collectively : implications of gene flow and selection for the spread of advantageous alleles[J]. Molecular Ecology, 2004, 13 :1341- 1356. |
| [56] | Ellstrand NC, Roose ML. Patterns of genotypic diversity in clonal plant species[J]. American Journal of Botany, 1987, 74(1): 123-131. |
| [57] | Soltis PS, Solos DE. Genetic variation in endemic and widespread plant species examples from Saxifragaceae and Polystichum (Dryopteridaceae)[J]. Aliso, 1991, 13(1):215-224. |