2. 中国热带农业科学院热带作物品种资源研究所,儋州 571737
2. Tropic Crops Genetic Resources Institute,Chinese Academy of Tropic Agricultural Sciences,Danzhou 571737
油梨(Persea americana Mill),又名鳄梨、酪梨、牛油果等,原产于热带美洲,为樟科(Lauraceae)油梨属(Persea)乔木果树,是一种著名的热带水果,也是木本油料树种之一[1],同时,因其四季常绿、树姿优美,还可作为一种优良的绿化树种[2]。近年来,随着世界油梨生产发展迅速,产量与消费量与日俱增,已成为引人瞩目的热带亚热带新兴水果,现已广泛应用于食用、医药和化妆工业[3],具有较高的经济效益,经济潜力巨大。我国于1918 年就已引进油梨品种[2, 4],在广东、广西、云南、福建、四川、台湾、海南等省(区)试种成功[5],但由于我国油梨资源研究匮乏、适栽品种较少及消费市场的有限性导致油梨生产发展十分缓慢,尚未形成大规模商品性生产。故研究油梨树种的多样性,深入开发利用油梨资源成为目前研究的热点[4]。
简单序列重复(Simple sequence repeats,SSR),又名微卫星DNA,是以1-6 个碱基为基本单元的串联重复序列,广泛分布于植物基因组中[6, 7]。SSR标记具有通用性、高可变性、共显性遗传、重复性好[6, 7, 8, 9]等优点,且数量丰富,分布广,覆盖整个基因组[10, 11, 12, 13, 14],并能检测出丰富多样性。目前,该技术已广泛用于遗传多样性检测[15, 16, 17, 18, 19]、遗传图谱构建[20]、目标基因的标定[21]、指纹图的绘制[19, 22]、种质鉴定[17, 23]、分子标记辅助选育[24]、品种纯度鉴定[25]及杂种优势利用[26]等研究中。在国外,分子标记已被证明在阐明油梨种质个体间的遗传关系方面非常有用,其中小卫星技术[27, 28, 29, 30, 31]和微卫星技术[32, 33, 34]已经被应用于油梨品种的指纹分析、鉴定和分类;此外,种质资源鉴定、遗传图谱已经利用分子标记构建[35, 36, 37],但与其他作物相比,分子标记技术在油梨方面的应用仍然不足,仍需要学者继续利用分子标记对油梨种质进行研究。目前,在中国RAPD、SSR 已分别用于油梨的品种分类及授粉研究[38, 39],但油梨的发展仍然处于不成熟阶段,尤其是油梨分子标记的研究相对滞后,在一定程度上限制了油梨遗传改良和育种工作的进行。
SSR 标记虽然有许多优点,但其反应条件易受各种因素干扰,从而影响整个实验结果。因此,建立适合的SSR 检测体系至关重要,而油梨SSR-PCR体系优化尚未见报道。本研究对油梨基因组DNA 提取方法、SSR 反应体系形成及引物筛选进行摸索,以期建立一套适合油梨的SSR 反应体系,为油梨种质资源遗传多样性及亲缘关系的分析提供参考。
1 材料与方法 1.1 材料实验所用的油梨叶片取自于海南大学园艺园林学院油梨种质资源圃。DNA 提取的实验材料选取10个品种;引物筛选的实验材料选取差异明显的哈斯、大岭7 号、福尔特及巴康品种为DNA 模板;体系优化选用哈斯品种。
引物由英潍捷基(上海)贸易有限公司合成,PCR 所用dNTPs、Taq 酶、DNA Marker 和植物基因组DNA 提取试剂盒均购自TaKaRa 公司。PCR 仪为Eppendorf Mastercycler Pro S,琼脂糖凝胶电泳仪为Biometro Standard Power Pack P25,变性聚丙烯酰胺凝胶电泳仪为北京六一仪器厂DYY-10C 型。
1.2 方法 1.2.1 基因组DNA的提取与检测以油梨叶片为材料,比较2×CTAB 法、改良2×CTAB 法、植物DNA 提取试剂盒法等3 种方法的优劣;采用0.8%琼脂糖凝胶电泳,以DL2000 Marker 为标准,在琼脂糖凝胶电泳仪120 V 恒压下检查DNA 的完整性检测DNA 的纯度和浓度;提取原液 -20℃保存。
1.2.1.1 2×CTAB 法取0.1 g 新鲜叶片放入研钵中,在液氮中研磨成粉末,转入预冷的2 mL 离心管,加入850 μL 65℃预热的2×CTAB 提取缓冲液(2% CTAB,100 mmol/L Tris-HCl,50 mmol/L EDTA,2 mol/L NaCl,2% β-巯基乙醇,pH8.0),摇匀,65℃水浴1 h,期间翻动几次;取出,加入等体积的氯仿∶异戊醇(24∶1,V/V),混匀并上下颠倒数次,12 000 r/min,4℃离心10 min,将上清液转入另一干净的2 mL 离心管中,重复此操作1 或2 次;再将上清液转到另一个1.5 mL 的离心管中,加入80 μL 的NaAc(5 mol/L)和2/3 体积的异丙醇,缓慢翻转,混匀放置-20℃冰箱 20 min 或以上;12 000 r/min,4℃离心10 min,弃上清,用500 μL 的70% 乙醇洗涤沉淀两次,放于超净工作台吹干,然后加100 μL的TE 缓冲液(10 mmol/L Tris-HCl,1 mmol/L EDTA)充分溶解;再加RNaseA 酶37℃ 水浴30 min,取出,-20℃保存待用。
1.2.1.2 改良2×CTAB法取0.1 g 新鲜叶片放入研钵中,加少许PVP 在液氮中研磨成粉末,转入预冷的2 mL 离心管,加入850 μL STE 缓冲液(200mmol/L Tris-HCl,100 mmol/L EDTA,1.5 mol/L NaCl,2% PVP,1% β-巯基乙醇,pH8.0),摇匀,置于冰上5 min ;4℃条件下5 000 r/min 离心8 min,弃上清后继续加入850 μL STE 缓冲液清洗,再次4℃条件下5 000 r/min 离心8 min,弃上清;加入850 μL 65℃预热的2×CTAB 提取缓冲液,用振荡器振荡1 min,充分混匀,放入水浴锅中水浴约1 h,期间来回轻缓颠倒几次;取出至于冰上,加入150 μL NH4AC 混匀,静置5 min,4℃下12 000 r/min 离心10 min ;取上清,加入等体积的氯仿∶异戊醇(24∶1,V/V),放入脱色摇床上来回摇荡10 min,放入离心机,4℃条件下12 000 r/min 离心10 min ;将上清液转入另一干净的2 mL 离心管中,加入等体积的氯仿∶异戊醇(24∶1,V/V),放入脱色摇床上来回摇荡5 min,放入离心机,4℃下12 000 r/min 离心10 min,根据上清液情况,选择是否继续重复抽提一次;将上清液转到另一个1.5 mL 的离心管中,加入80 μL 的NaAc(5 mol/L)和2/3 体积的异丙醇沉淀,缓慢翻转,混匀放置-20℃冰箱 20 min 或以上;12 000 r/min,4℃离心10 min,弃上清,用500 μL 的70% 乙醇洗涤沉淀两次,放于超净工作台吹干,然后加100 μL 的TE 缓冲液充分溶解;再加1 μL RNaseA 酶37℃水浴30 min,取出,-20℃保存待用。
1.2.1.3 植物基因组DNA 提取试剂盒法TaKaRa MiniBEST Plant Genomic DNA Extraction Kit 步骤参照 说明书去多糖多酚的Protocol- Ⅱ步骤进行。
1.2.2 PCR 扩增及体系优化PCR 扩增结果会因Mg2+、dNTP、Taq 酶、引物浓度及模板DNA 量等因素而受到影响,为了得到清晰、客观、准确的结果,需对反应条件进行优化后再进行大量样本扩增。针对影响PCR 扩增反应体系的5 个因素(Mg2+、dNTP、Taq DNA 聚合酶、引物及模板DNA 量),结合相关文献报道,选用L16(45)正交实验设计,共16 个处理组合,每个组合3 次重复,探讨正交实验因素水平和正交设计见表 1 和表 2。
体系优化所用引物AVAG21,反应体积为20μL。PCR 反应程序为:94℃预变性3 min ;94℃变性30 s,50℃退火30 s,72℃延伸1 min,35 个循环;72℃延伸10 min。检测用2% 琼脂糖凝胶,120 V 恒电压,电泳约40 min,凝胶成像系统观察并拍照。1.2.3 引物筛选 实验选用的SSR 引物共73 对,其中48 对参考Sharon 等[35]的油梨SSR 引物序列,25对参考Ashworth 等[40]的油梨SSR 引物序列。根据公式ƞ=1-(1-1/2)n(n 为样品数,ƞ 为有多态性引物的选中概率)计算选中的有多态性引物的概率,本研究利用4 个DNA 样品进行测试,则有多态性引物的选中概率为93.75%。选取稳定性高、重复性好、有多态性产物的引物对模板DNA 进行PCR 扩增。
由于引物的最佳退火温度不同,利用TD-PCR进行大量引物多态性筛选,反应程序为:94℃预变性3 min ;94℃变性30 s,58℃退火30 s,72℃延伸1 min,其后每个循环退火温度下降1℃,13 个循环;94℃变性30 s,45℃退火30 s,72℃延伸1 min,27个循环;72℃延伸10 min。PCR 产物检测用2% 琼脂糖凝胶,120 V 恒电压电泳约40 min ;PCR 产物分离利用6% 变性聚丙烯酰胺凝胶,60 W 恒定功率电泳1.5-2 h,参考杨珺的银染法[41]进行银染检测。1.2.4 油梨SSR-PCR 的多态性验证 选取较好的方法大量提取的油梨基因组DNA,选用筛选出的5对多态性引物、优化的20 μL SSR-PCR 反应体系及TD-PCR 程序对提取的DNA 进行PCR 扩增验证,检测DNA、优化体系及程序是否满足分子研究要求。
2 结果 2.1 基因组DNA的提取与检测3 种方法提取的DNA 比较:A260/A280 的比值常用来表示DNA的纯度,核酸蛋白检测仪结果显示,改良2×CTAB法产率最高;常规的2×CTAB 法次之,试剂盒法最低;常规的2×CTAB 法A260/A280 值在1.5-1.7 之间,部分DNA 纯度符合SSR 的要求,改良2×CTAB 法和DNA 试剂盒法的该值在1.7-2.0之间,说明DNA 的纯度都符合SSR 的要求。DNA电泳检测结果显示,常规2×CTAB 法大部分泳道点样孔有杂质,DNA 条带较暗,部分拖尾,有蛋白质或者糖类杂质以及RNA 污染(图 1-A);改良2×CTAB 法提取的油梨叶片DNA 条带亮度最大且各条带较均匀清晰,稳定性高,杂质较少(图 1-B);DNA 试剂盒法提取的DNA 条带最暗,亮度均匀几乎无杂质,DNA 较纯,但成本高,不利于大批量样品DNA 的提取(图 1-C)。由此选用改良2×CTAB法提取大批量油梨植物基因组DNA。
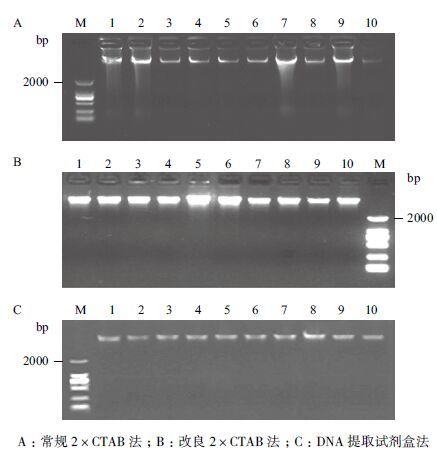
|
| 图 1 三种方法提取的DNA 凝胶电泳图 |
由图 2 可见,正交设计SSR-PCR 反应体系的扩增结果存在明显差异。在实验设计的16 个组合、3次重复中:组合1、2、3、4、7、12、14 扩增条带较弱或未扩增出条带,稳定性和重复性低;组合5、9、10、11、13、15、16 主带明显,但存在非特异性带;组合6、8 主带明显,无非特异性带,但两者相对而言第6 组合扩增条带亮度大、清晰,且稳定性好、重复性高。综合比较分析,第6 组合为油梨的最优SSR-PCR 反应体系,即20 μL 总反应体系中,含约40 ng DNA 模板、1.5 mmol/L Mg2+、0.15 mmol/L dNTP、0.5 U Taq DNA 聚合酶、0.5 μmol/L 引物。
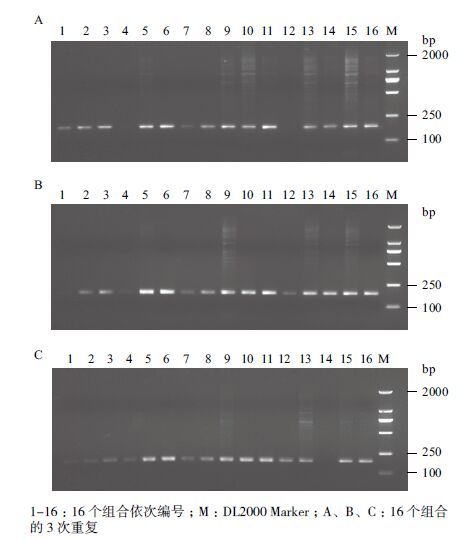
|
| 图 2 SSR-PCR 正交实验结果 |
采用优化的SSR-PCR 反应体系和TD-PCR 程序进行引物多态性筛选,扩增得到清晰且特异性强的谱带(图 3);结果表明该体系扩增不同引物时,条带依然清晰稳定,重复性好,因此该体系适用于油梨品种进行SSR-PCR 扩增;对于大量引物的多态性筛选,TD-PCR 省去了众多引物退火温度的摸索,可以减少非特异性带的产生,有效提高筛选的特异性和效率。73 对引物中,初步筛选出30 条扩增较好的多态性引物,多态性的引物达到41%。

|
| 图 3 油梨多态性引物筛选 |
选用已筛选出的5 对多态性引物,利用优化的SSR-PCR 反应体系和TD-PCR 程序对45 份油梨种质的DNA 进行扩增,所选用的5 对多态性引物均得到很好的扩增效果,获得了清晰稳定的谱带(图 4)。电泳结果显示该反应体系进行大量PCR 扩增时,条带依然清晰稳定,重复性好,表明建立的SSR 反应体系稳定可靠,且筛选的引物多态性明显,适用于油梨种质进行SSR-PCR 扩增。

|
| 图 4 引物AUCR418(A)和AVD017(B)对45 份油梨种质的扩增验证结果 |
20 世纪80 年代,CTAB 法开始在国内外广泛应用,CTAB 是一种去污剂,既能裂解细胞又能有效沉淀多糖,因此有其独到优点[42]。在30 多年的发展中,许多学者对CTAB 法不断改进,除在技术上的改进,大多是在DNA 提取液成分配比上发生变化,并针对不同物种形成独特的优化提取方法。许多报道利用高盐缓冲条件下去除植物DNA 中多糖[43, 44, 45, 46],广泛采取β-巯基乙醇、抗坏血酸或PVP(聚乙烯毗咯烷酮)等抗氧化剂来去除植物组织中的多酚类物质[47, 48]。樟科植物含有大量的多糖、鞣质、多酚、单宁等次生代谢物,在DNA 提取过程中经裂解液处理后,细胞破碎,释放出次生代谢物,使提取液变得异常黏稠,操作困难,导致提取的DNA 质量差,溶解困难[49],对PCR 产生影响,导致实验结果不稳定和不可靠。本研究改良2×CTAB 法所提DNA 浓度及纯度均符合SSR-PCR 扩增的要求,且该法在DNA 获得量上优于常规2×CTAB 法和DNA 试剂盒法,原因主要在于其解决了黏稠、易氧化褐变及DNA 纯度低等问题。常规方法提取会使这些物质与DNA 发生不可逆的结合使 DNA 呈褐色、黏稠,影响DNA 质量,不能用于PCR 扩增和酶切等分子生物学研究;经多次实验发现与常规2×CTAB 法相比,改良2×CTAB 法中样品中加入少许PVP 研磨,反复用STE 缓冲液进行洗脱,同时在65℃水浴核裂解后加入150 μL 7.5 mol/L 醋酸铵溶液可在很大程度上解决黏稠问题,同时发现裂解后加入醋酸铵溶液冰上静置10 min 可以大量去除蛋白质杂质。抽提时,与上下颠倒数次相比,放入脱色摇床上反复摇荡几分钟,可获得较透明的上清液。在木本植物DNA 提取中,提取液加入β-琉基乙醇、PVP(聚乙烯毗咯烷酮)等这几种抗氧化剂去除酚类物质是必须的,这与闫桂琴等[50]的研究结果一致。故选用改良2×CTAB 法作为后期分子研究实验的油梨基因组DNA 提取方法对油梨大量样本进行提取。
SSR-PCR 反应涉及诸多因素,每个因素的反应参数对反应体系有很大影响,确定合适的反应参数是SSR 分析的前提。SSR-PCR 反应体系中各组分均可能影响扩增的特异性、敏感性和产量,采用多因素联合优化的正交实验设计,借助合适的正交表,利用正交表的均衡分散性和整齐可比性,可有效解决理论与实际可行的实验次数的矛盾,以及实际所做的有限量实验与要求全面掌握事物内在规律之间的矛盾[51]。影响PCR 扩增效率的因子主要有5 个,Mg2+、dNTP、Taq DNA 聚合酶、DNA 和引物的浓度对PCR 扩增效率都起着抑制或促进的作用。利用正交设计优化油梨SSR 反应体系是一种有效、适用而且简便的方法,能较好地识别SSR-PCR 反应体系中的关键影响因素,并优化反应条件。本研究利用正交实验设计方法建立了适合油梨SSR-PCR 反应体系的优化组合,即20 μL 总反应体系中,含约40 ngDNA 模板、1.5 mmol/L Mg2+、0.15 mmol/L dNTPs、0.5U Taq 酶、0.5 μmol/L 引物,这与油松[52]、鸭茅[53]、大豆[54]、东兴金花茶[55]、油葵[56]、冬瓜[57]、菊花[58]等植物已报道的SSR-PCR 优化体系都有所不同,说明不同物种的SSR-PCR 优化反应体系存在一定差异。
SSR-PCR 反应体系的优化、建立及引物的筛选是SSR 多态性标记应用的基础。影响SSR 反应的主要因子除了Mg2+、dNTPs、Taq 酶、模板等外,SSRPCR扩增时的退火温度也是一个关键要素,对扩增条带有明显影响。本研究选用TD-PCR 扩增程序进行大量SSR 引物多态性筛选,发现TD-PCR 可以有效避免退火温度过高或过低对SSR-PCR 扩增反应造成的影响,省去了众多引物退火温度的摸索,加快了实验进程;可以减少非特异性带的产生,有效提高筛选的特异性和效率;选用的TD-PCR 引物筛选程序为今后油梨SSR 引物筛选提供一定的参考。
4 结论本研究结果表明改良2×CTAB 法对油梨基因组DNA 的提取效果最佳;20 μL SSR-PCR 最优反应体系含约40 ng DNA 模板、1.5 mmol/L Mg2+、0.15 mmol/L dNTPs、0.5 U Taq DNA 聚合酶、0.5 μmol/L 引物;以此体系为基础进行引物筛选,从73 对油梨SSR 引物中筛选出了30 对扩增条带清晰的多态性引物。
| [1] | 郑淑娟, 白净. 世界油梨产销发展概况及前景[J]. 世界热带农业信息, 2011(11):6-9. |
| [2] | 欧珍贵. 油梨的研究现状及在贵州地区的发展前景[J]. 林业科技开发, 2006, 20(3):11-13. |
| [3] | 中国科学院中国植物志编辑委员会. 中国植物志:第31 卷[M].北京:科学出版社, 1982. |
| [4] | 钱学射, 张卫明, 顾龚平, 等. 鳄梨资源的开发利用[J]. 中国野生植物资源, 2010, 29(5):23-25. |
| [5] | 陈海红. 油梨新品种的区域化表现及栽培技术研究[D]. 南宁:广西大学, 2006. |
| [6] | Litt M, Luty JA. A hypervariable microsatellite revealed by in vitro amplification of a dinucleotide repeat within the cardiac muscle actin gene[J]. Am J Hum Genet, 1989, 44 :397-401. |
| [7] | Tautz D. Hyper variability of simple sequence as a general source for polymorphic DNA marker[J]. Nucleic Acids Search, 1989, 17 (16):6463-6471. |
| [8] | Smeets AJM, Brunner HG, Ropers HH, et al. Use of variable simple sequence motifs as genetic markers :application to study of myotonic dystrophy[J]. Human Genetics, 1989, 83 :245-251. |
| [9] | Weber JL, May PE. Abundant class of human DNA polymorphisms which can be typed using the polymerase chain reaction[J]. American Journal of Human Genetics, 1989, 44 :388-396. |
| [10] | Hamada H, Petrino MG, Kakunaga T. A novel repeated element with Z-DNA-forming potential is widely found in evolutionarily diverse eukaryotic genomes[J]. Proc Nat Acad Sci USA, 1982,79 :6465-6469. |
| [11] | Stallings RL, Ford AF, Nelson D, et al. Evolution and distribution of(GT)n repetitive sequences in mammalian genomes[J]. Genomics, 1991, 10 :807-815. |
| [12] | Weissenbach J, Gyapay G, et al. A second generation linkage map of the human genome[J]. Nature, 1992, 359 :794-801. |
| [13] | Schmidt E, Heslop-Harrison JS. The physical and genomic organization of microsatellites in sugar beet[J]. Proceedings of the National Academy of Sciences of the United States of America, 1996,93:8761-8765. |
| [14] | Roots EH, Baker RJ. Distribution and characterization of microsatellites in the emu(Dromaius novaehollandiae)genome[J]. Journal of Heredity, 2002, 93 :100-106. |
| [15] | Song BH, Thomas MO. High genetic diversity and population differentiation in Boechera fecunda, a rare relative of Arabidopsis[J]. Molecular Ecology, 2007, 16 :4079-4088. |
| [16] | Gong W, Gu L, Zhang DX. Low genetic diversity and high genetic divergence caused by inbreeding and geographical isolation in the populations of endangered species Loropetalum suhcordatum (Hamamelidaceae)endemic to China[J]. Conservation Genetics, 2010, 11 :2281-2288. |
| [17] | 张萌. 基于SSR 分子标记的葡萄种质资源的遗传多样性分析及品种鉴定[D]. 南京:南京农业大学, 2012. |
| [18] | 李丽, 徐立, 李志英, 等. 不同地理来源美丽鸡血藤遗传多样性的SSR 分析[J]. 广东农业科学, 2013(22):156-160. |
| [19] | 杨勇. 甘蓝型油菜遗传多样性分析及核心亲本的构建[D].武汉:华中农业大学, 2013. |
| [20] | Zhang Y, Mary KS, Bouton JH. Genome mapping of white clover (Trifolium repens L. )and comparative analysis within the Trifolieae using cross-species SSR markers[J]. Theoretical and Applied Genetics, 2007, 114 :1367-1378. |
| [21] | Zraidia A, Stiftg G, Pachner M, et al. A consensus map for Cucurbita pepo[J]. Mol Breeding, 2007, 20(4):375-388. |
| [22] | 文雁成, 王汉中, 沈金雄, 等. SRAP 和SSR 标记构建的甘蓝型油菜品种指纹图谱比较[J]. 中国油料作物学报, 2006, 28(3):233-239. |
| [23] | 李鸿雁, 李志勇, 米福贵, 等. 利用微星标记鉴定扁蓿豆种质资源[J]. 华北农学报, 2008, 23(3):67-71. |
| [24] | 王永强, 刘建光, 赵俊丽, 等. 利用SSR 分子标记辅助棉花提纯选育的研究[J]. 分子植物育种, 2014, 12(3):492-498. |
| [25] | 周志成, 王惠林, 王贤磊, 等. SSR 标记鉴定甜瓜品种“红月亮”种子纯度[J]. 中国瓜菜, 2014, 27(1):21-24. |
| [26] | 曾莉娟, 郑成木. SSR 技术及其应用[J]. 热带农业科学,2001(3):56-59. |
| [27] | Furnier GR, Cummings MP, Clegg MT. Evolution of the avocados as revealed by DNA restriction fragment variation[J]. Journal of Heredity, 1990, 81(3):183-188. |
| [28] | Lavi U, Hillel J, Vainstein A, et al. Application of DNA fingerprints for identification and genetic analysis of avocado[J]. J Amer Soc Hort Sci, 1991, 116(6):1078-1081. |
| [29] | Mhameed S, Sharon D, Hillel J, et al. Level of heterozygosity and mode of inheritance of variable number of tandem repeat loci in avocado[J]. Journal of the American Society for Horticulturalence, 1996, 121(5):768-772. |
| [30] | Davis J, Henderson D, Kobayashi M, et al. Genealogical relationships among cultivated avocado as revealed through RFLP analysis[J]. Journal of Heredity, 1998, 89(4):319-323. |
| [31] | Fiedler J, Bufler G, Bangerth F. Genetic relationships of avocado (Persea americana Mill. )using RAPD markers[J]. Euphytica, 1998, 101 :249-255. |
| [32] | Schnell RJ, Brown JS, Olano CT, et al. Evaluation of avocado germplasm using microsatellite markers[J]. Journal of the American Society for Horticulturalence, 2003, 128(6):881-889. |
| [33] | Rodriguez NN, Fuentes JL, Coto O, et al. Proc VI World Avocado Congress :Comparative study of polymorphism level, discrimination capacity and informativeness of AFLP, ISTR, SSR and Isoenzymes markers and agro-morphological traits in avocado[C]. Viña del Mar, Chile, 2007 :76. |
| [34] | Alcaraz ML, Hormaza JI. Molecular characterization and genetic diversity in an avocado collection of cultivars and local Spanish genotypes using SSRs[J]. Hereditas, 2007, 144 :244-253. |
| [35] | Sharon D, Cregan PB, Mhameed S, et al. An integrated genetic linkage map of avocado[J]. Theoretical & Applied Genetics, 1997, 95 :911-921. |
| [36] | Viruel MA, Gross E, Barcelo-munoz A. Proc VI World Avocado Congress :Development of a linkage map with SSR and AFLP markers in avocado[C]. Viña del Mar, Chile, 2007 :52. |
| [37] | Borrone JW, Brown JS, Tondo CL, et al. An EST-SSR-based linkage map for Persea Americana Mill.( avocado)[J]. Tree Genetics & Genomes, 2009, 5 :553-560. |
| [38] | 刘康德, 李建国, 彭世清, 等. 油梨基因组DNA 的提取及RAPD 分析[J]. 热带作物学报, 1999, 20(4):58-61. |
| [39] | 张泰芳. 油梨(Persea americana)授粉的研究——微卫星技术(SSR)在油梨种群父本鉴定上的应用[D]. 儋州:华南热带农业大学, 2007. |
| [40] | Ashworth VETM, Kobayashi MC, De La Cruz M, et al. Microsatellite markers in avocado(Persea americana Mill. ):development of dinucleotide and trinucleotide markers[J]. Scientia Horticulturae, 2004, 101(3):255-267. |
| [41] | 杨珺. 蝴蝶兰种植资源鉴定与评价[D]. 海口:海南大学,2010. |
| [42] | 安娜, 郭宏波, 周铜水, 等. 党参基因组DNA 提取、ISSRPCR反应体系优化及引物筛选[J]. 植物研究, 2009, 29(3):346-351. |
| [43] | Dellaporta SL, Wood J, Hicks JB. A plant DNA minipreparation : version II[J]. Plant Mol Biol Rep, 1983, 1(4):19-21. |
| [44] | Moller EM, Bahnweg G, Sandermann H, et al. A simple and efficient protocol for isolation of high molecular weight DNA from filamentous fungi, fruit bodies, and infected plant tissues[J]. Nucleic Acids Research, 1992, 20(22):6115-6116. |
| [45] | Porebski S, Bailey LG, Bernard R. Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components[J]. Plant Molecular Biology Reporter, 1997, 15(1):8-15. |
| [46] | 郑云柯, 胡翔宇, 宋希强, 等. 石解属植物基因组DNA 提取方法的对比[J]. 热带生物学报, 2015, 6(2):148-152. |
| [47] | 谭晓风, 漆龙霖, 黄晓光, 等. 山茶属植物叶片的DNA 抽提[J]. 中南林学院学报, 1999, 19(4):75-79. |
| [48] | 陈析丰, 查笑君, 范文杰, 等. 山茶花叶片DNA 提取及RAPD反应体系的研究[J]. 植物研究, 2007, 27(2):218-223. |
| [49] | 徐虹, 郑敏, 章军, 等. 三种樟科植物的细胞总DNA 提取[J].云南植物研究, 2004, 26(4):451-457. |
| [50] | 闫桂琴, 任鹰, 张变红, 等. 三种木本植物基因组DNA 的提取及纯度检测[J]. 山西师范大学学报:自然科学版, 2004, 18(l):72-77. |
| [51] | 姜同川. 正交试验设计[M]. 济南:山东科学技术出版社,1985 :1-71. |
| [52] | 张冬梅, 杨娅, 沈熙环, 等. 油松SSR-PCR 引物筛选及反应体系的建立[J]. 北京林业大学学报, 2007, 29(2):13-17. |
| [53] | 谢文刚, 张新全, 彭燕, 等. 鸭茅SSR-PCR 反应体系优化及引物筛选[J]. 分子植物育种, 2008, 6(2):381-386. |
| [54] | 苏辉, 李志刚, 宋书宏. 正交设计优化大豆SSR-PCR 反应体系及引物筛选[J]. 华北农学报, 2009, 24(2):99-102. |
| [55] | 唐健民, 陈宗游, 韦霄, 等. 东兴金花茶SSR-PCR 反应体系的优化及引物筛选[J]. 基因组学与应用生物学, 2014, 33(2):398-404. |
| [56] | 房冬梅, 吕品, 侯建华. 油葵SSR-PCR 反应体系的优化及引物筛选[J]. 中国农学通报, 2015, 31(12):205-209. |
| [57] | 潘珍珍, 吴才君, 刘文睿, 等. 冬瓜SSR-PCR 体系优化及引物筛选[J]. 分子植物育种, 2015, 13(4):898-902. |
| [58] | 李亚慧, 黄丛林, 董然. 菊花SSR-PCR 反应体系的建立和优化[J]. 北方园艺, 2012(13):127-131. |






