2.北京市落叶果树工程技术研究中心,北京 100093 ;
3.农业部华北地区园艺作物生物学与种质创制重点实验室,北京 100093
2. Beijing Engineering Research Center for Deciduous Fruit Tree,Beijing 100093 ;
3. Key Laboratory of Biology and Genetic Improvement of Horticultural Crops (North China),Ministry of Agriculture of P. R. China,Beijing 100093
葡萄属葡萄科(Vitaceae) 葡萄属(Vitis L.),是全球性果树之一,品种繁多,分布广泛。我国是世界上葡萄栽培面积较大的国家之一,目前全国入圃保存的葡萄种质资源也有2 000 份左右[1],并且每年都会有多个新品种出现,因此进行品种保护、品种鉴定及谱系分析等工作不仅对苗木繁育具有重要意义,也是保护这些品种知识产权的需要。
SSR(Simple sequence repeat)分子标记技术具有重复性好、多态性丰富、稳定性高及简便快速等特点[2],近年来被广泛应用于葡萄种质鉴别[3]、遗传多样性分析[4, 5]、亲缘关系鉴定[6, 7]及分子遗传图谱构建[8]等方面,同时也成为葡萄指纹图谱构建的有效工具[9]。尹玲等[10]对我国近年来新育成的24 个鲜食葡萄品种,利用6 对国际通用的SSR荧光标记引物对其进行扩增,并利用毛细管电泳技术对产物进行分离,建立了这些葡萄品种的指纹图谱。近几年,研究者在遗传图谱基础上提出了“分子身份证”的概念,并将它赋予品种本身作为识别品种的一个标准。分子身份证是在指纹图谱的基础上将种质的特征数字化,即每个种质都拥有一个属于自己的字符串形式的代码,达到更加简单明了区分种质的目的。在果树方面,研究者利用SSR 标记分别构建了甜樱桃、香蕉和桃等的分子身份证[11, 12, 13]。2013 年,杜晶晶等[14]以国家葡萄品种资源圃内保存的80 份葡萄种质为试材,对利用SSR 标记构建葡萄种质分子身份证的方法进行了探讨,证明SSR标记可以用于进行葡萄的分子身份证构建。
本研究选取北京市农林科学院林业果树研究所选育的13 个葡萄优新品种为试材,通过SSR 标记分析技术,分析它们的亲缘关系,尝试构建13 个品种的分子身份证,旨在为保护中国葡萄优新品种的知识产权,并为育成品种的分子鉴定奠定理论基础,对中国葡萄遗传育种及品种保护提供帮助。
1 材料与方法 1.1 材料本研究所用材料均为北京市农林科学院林业果树研究所选育的优新品种,共13 份(表 1),分别种植于所内资源圃和温室。在四月中旬采集各个葡萄品种幼叶液氮速冻,保存于-80℃冰箱,用于基因组DNA 的提取。
基因组DNA 提取参照Marsal 等[15]的方法。从葡萄幼叶中提取基因组DNA,1% 琼脂糖凝胶电泳检测,利用Eppendorf 公司生产的Bio-Photometer 核酸检测仪检测DNA 溶液的浓度与纯度,并将浓度稀释至50 ng/μL。
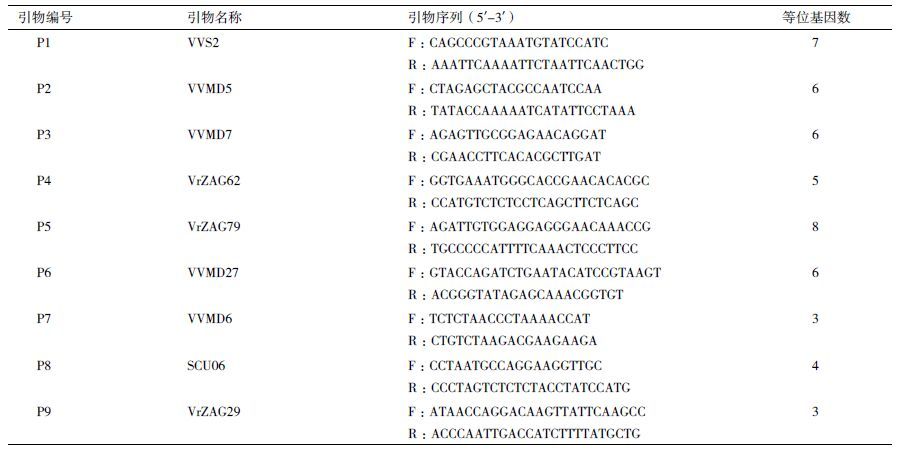
1.2.2 DNA 扩增和电泳本研究选取9 对国际通用的SSR 引物进行扩增(表 2),其中6 个标记(VVMD5、VVMD7[16]、VVMD27、VVS2[17]、VRZAG62及VRZAG79[18])曾被This 等[19]推荐为葡萄品种筛选的核心标记。引物由上海生工合成,每对引物合成2OD,-20℃冰箱保存备用。
DNA 扩增采用20 μL 体系。其中含:10×PCRbuffer,引物0.25 μmol/L,模板DNA 50 ng,dNTP 0.2mmol/L,rTaq DNA 聚合酶0.5 U。用于SSR-PCR 反应的dNTP、Taq 酶、DNA Marker 购自TaKaRa 公司。
PCR 扩增程序:95℃预变性5 min ;95℃变性15 s,65℃-55℃退火15 s,72℃延伸1 min,10 个循环;95℃变性15 s,55℃退火15 s,72℃延伸1 min,25 个循环;4℃保存。PCR 反应在美国Bio-Rad 公司生产的9600 Thermal Cycler 型PCR 仪上进行。然后取PCR 产物4 μL 进行点样,6% 聚丙烯酰胺凝胶电泳,银染显色,胶干后在清华紫光e100 扫描仪上成像保存。
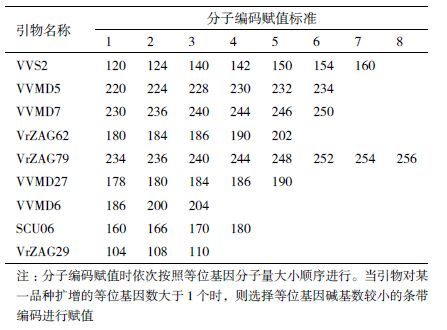
1.2.3 数据分析采用Powermarker 软件(Version 3.25)[20]对9 个SSR 标记的遗传多样性指数进行分析。数据标准化处理方法参考陈昌文等[11]的方法。根据每对引物对不同种质扩增片段对应的分子量大小,按从小到大依次编码为1-8。同时参考桃分子身份证的构建方法,当引物扩增的某一品种等位基因有2 个时,按等位基因碱基数较小的条带编码进行赋值。并通过非加权配对算术平均法(UPGMA)进行聚类分析,建立亲缘关系图。
2 结果 2.1 SSR引物扩增多态性通过改进的CTAB 方法提取全部实验材料的基因组DNA,通过电泳和紫外分光光度计检测,基因组DNA 的浓度和纯度均符合SSR 实验要求。聚丙烯酰胺凝胶电泳结果显示,SSR 扩增条带清晰。本实验所选用的9 对SSR 引物在供试葡萄品种中共检测到48 个等位基因,平均每对引物扩增的等位基因数是5.3 个。最少的VVMD6 和VrZAG29 引物分别扩增出3 个等位基因,VrZAG79 扩增的等位基因数最多为8 个,其中有5 对引物的等位基因数超过平均数(表 2)。等位基因数越多,表明该引物更能反应品种之间的差异。其中VVMD5 和VVMD7 对13 个品种扩增结果(图 1)显示,13 个品种分别有6 个等位基因。尽管9 对引物的多态性不高,但是足以区分这13 个葡萄品种。
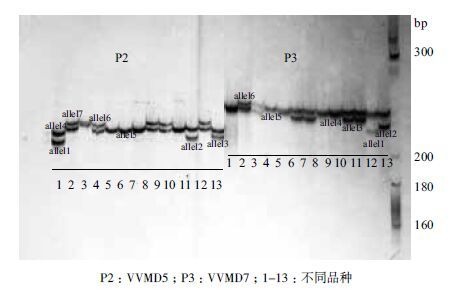
|
| 图 1 VVMD5 和VVMD7 扩增的等位基因特征 |
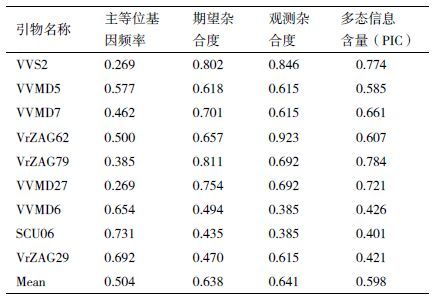
利用Powermaker 软件对9 个SSR 标记在13 个葡萄品种中的主等位基因频率、期望杂合度、观测杂合度及扩增位点的多态信息含量等进行了计算分析。结果(表 3)表明,扩增位点的观测杂合度在0.385-0.846 之间,平均值为0.641。期望杂合度的范围在0.435-0.811 之间,平均值为0.638。多态性信息含量PIC 变幅为0.401-0.784,平均为0.598。VrZAG79 引物的PIC 值最高,结合期望杂合度值及等位基因数值,该引物对本研究供试葡萄品种的区分能力最强。
根据电泳结果,对9 对引物在供试种质扩增的等位基因谱带大小分布范围进行赋值。因为本实验中引物扩增最多的等位基因数为8,所以赋值范围为1-8(表 4)。为了保证分子身份证字符串的每位上只有1 个字符,对同一引物下的扩增条带只选择位点较小的进行分子编码。
根据表 4 中等位基因的赋值标准和各引物对品种的扩增结果,对13 份供试葡萄品种进行分子编码,结果(表 1)显示,用选取的9 对引物可以将13 个品种进行有效区分。但是因为品种亲缘关系较近,进行分子编码最少也需要8 对引物才能够将所有品种区分开,如瑞都红玫和瑞都早红的分子编码比较相似,在第8 对引物时才区分开。另外在所构建的13 个品种分子身份证中,有7 个品种具有至少一个特异等位基因。紫珍珠、早玫瑰香和瑞都脆霞各具有一个特异等位基因,艳红、早玛瑙和峰后各具有2 个特异等位基因,瑞都无核怡则具有3 个特异等位基因。
2.3 供试葡萄品种亲缘关系分析根据本研究所用的9 对SSR 引物扩增结果进行数据统计,利用POWERMAKER 软件,采用UPGMA 法对所有品种进行聚类分析,聚类结果(图 2)显示,亲缘关系近的多聚为一类,如父母本相同的瑞都红玫、瑞都早红、瑞都脆霞和瑞都香玉聚在一起,而瑞都红玉为瑞都香玉的芽变,也聚为一类。紫珍珠和早玫瑰香亲本相同聚为一类。艳红、早玛瑙爱神玫瑰与香妃聚为一个大类,他们都有相同的亲本玫瑰香。该聚类结果清楚地反应了品种间亲缘关系的远近。

|
| 图 2 13 个葡萄品种基于SSR 的聚类分析 |
采用9 对SSR 引物对13 个葡萄品种进行了分子身份证构建和亲缘关系分析。平均等位基因数(5.3)、期望杂合度(0.435-0.811)和引物多态性信息含量(0.401-0.784)方面显示,本实验结果都较低(表 3)。Guo 等[21]利用相同引物在32 个中国葡萄品种扩增平均位点数11.5,期望杂合度0.740-0.915,多态性信息含量0.716-0.908。本研究结果也低于Moreno-Sanz 等[22]采用相同引物在西班牙品种中的扩增结果。可能是由于13 个供试葡萄品种来源于相同的育种单位,许多品种父母本相同,亲缘关系较近,选育品种的性状相似,导致品种遗传多样性较低。但是通过这9 个分子标记,也可以将13 个葡萄品种完全区分开。同时我们发现标记VrZAG79在本研究所用的13 个中国葡萄品种中扩增等位基因数最多,多态信息含量最高,这与Guo 等[21]在32个中国葡萄品种的实验结果一致,表明VrZAG79 可以作为区分中国葡萄品种的核心标记之一。
在引物扩增结果的基础上,我们采取最小等位基因编码法[13]对13 个葡萄品种进行分子编码。得到了13 个葡萄品种编码各异的分子身份证。在桃编码中发现亲缘关系较近的品种很难区分[13],而本研究中,对于亲缘关系较近的品种通过增加引物也得到了有效区分,如瑞都红玫和瑞都早红的区分等。本研究对来自同一育种单位亲缘关系相同或较近的品种均可被有效区分,充分说明我们所选取的引物足以区分亲缘关系很近的不同材料,亦即利用SSR标记构建葡萄分子身份证具有较强的可靠性,可作为鉴别葡萄品种的一种有效方法。本实验得出的结果在一定程度上可以作为供试品种鉴定的依据,但在后期的大量鉴定中的适用性还需进一步的验证。如果能将分子标记和品种来源及表型性状相结合来研究制定它们的分子身份证将会得到更加科学客观的实验结果。
4 结论本研究采用国际通用的9 对SSR 引物,对13份中国葡萄优新品种进行了亲缘关系分析,构建了它们的有效分子身份证,再一次表明SSR 分析技术可以作为葡萄品种分子身份证构建的有效技术手段。
| [1] | 刘崇怀, 马小河, 武岗. 中国葡萄栽培品种[M]. 北京:中国农业出版社, 2014. |
| [2] | Morgante M, Olivieri A. PCR-amplified microsatellites as markers in plant genetics[J]. The Plant Journal, 1993, 3(1):175-182. |
| [3] | 吴子龙, 王军, 沈育杰, 路文鹏. 8 个山葡萄及山欧杂种葡萄品种的SSR 分析[J]. 植物资源学报, 2008, 9(1):105-109. |
| [4] | 邹瑜, 杨柳, 黄大辉, 等. 70 份毛葡萄种质资源遗传多样性的SSR分析[J]. 南方农业学报, 2013, 44(12):1943-1948. |
| [5] | Emanuelli F, Lorenzi S, Grzeskowiak L, et al. Genetic diversity and population structure assessed by SSR and SNP markers in a large germplasm collection of grape[J]. BMC Plant Biology, 2013, 13 : 39 |
| [6] | 李慧, 罗正荣, 张青林. 基于SSR 和IRAP 标记的‘关口葡萄’亲缘关系分析[J]. 果树学报, 2014, 31(6):1024-1027. |
| [7] | 温景辉, 申海林, 邹利人, 等. 20 份葡萄种质亲缘关系的SSR分析[J]. 果树学报, 2011, 28(5):782-786. |
| [8] | Vezzulli S, Troggio M, Coppola G, et al. A reference integrated map for cultivated grapevine(Vitis vinifera L.)from three crosses, based on 283 SSR and 501 SNP-based markers[J]. Theoretical and Applied Genetics, 2008, 117 :499-511. |
| [9] | Karata H, Değirmenci D, Velasco R, et al. Microsatellite fingerprinting of homonymous grapevine(Vitis vinifera L. ) varieties in neighboring regions of South-East Turkey[J]. Scientia Horticulturae, 2007, 114 :164-169. |
| [10] | 尹玲, 张晨, 向江, 等. 我国新育成葡萄品种SSR 指纹图谱的建立[J]. 果树学报, 2015, 32(3):366-373. |
| [11] | 艾呈祥, 张力思, 魏海蓉, 等. 甜樱桃品种SSR 指纹图谱数据库的建立[J]. 中国农学通报, 2007, 23(5):55-58. |
| [12] | 王静毅, 陈业渊, 黄秉智, 等. 部分香蕉品种SSR 指纹图谱的构建[J]. 果树学报, 2009, 26(5):733-738. |
| [13] | 陈昌文, 曹珂, 王力荣, 等. 中国桃主要品种资源及其野生近缘种的分子身份证构建[J]. 中国农业科学, 2011, 44(10):2081-2093. |
| [14] | 杜晶晶, 刘国银, 魏军亚, 等. 基于SSR 标记构建葡萄种质资源分子身份证[J]. 植物研究, 2013, 33(2):232-237. |
| [15] | Marsal G, Baiges I, Canals JM, et al. A fast, efficient method for extracting DNA from leaves, stems, and seeds of Vitis vinifera L.[J]. American Journal of Enology and Viticulture, 2011, 62 :376-381. |
| [16] | Bowers JE, Dangl GS, Vignani R, Meredith CP. Isolation and characterization of new polymorphic simple sequence repeat loci in grape(Vitis vinifera L).[J]. Genome, 1996, 39 :628-633. |
| [17] | Thomas MR, Scott NS. Microsatellite repeats in grapevine reveal DNA polymorphisms when analyzed as sequence-tagged sites (STSs) [J]. Theoretical Applied Genetics, 1993, 86 :985-990. |
| [18] | Sefc KM, Regner F, Turetschek E, et al. Identification of microsatellite sequences in Vitis riparia and their applicability for genotyping of different Vitis species[J]. Genome, 1999, 42(3):367-373. |
| [19] | This P, Jung A, Boccacci P, et al. Development of a standard set of microsatellite reference alleles for identification of grape cultivars [J]. Theoretical and Applied Genetics, 2004, 109(7):1448-1458. |
| [20] | Liu K, Muse SV. PowerMarker :an integrated analysis environment for genetic marker analysis[J]. Bioinformatics, 2005, 21 :2128-2129. |
| [21] | Guo DL, Zhang Q, Zhang GH. Characterization of grape cultivars from China using microsatellite markers[J]. Czech Journal of Genetics and Plant Breeding, 2013, 49 :164-170. |
| [22] | Moreno-Sanz P, Loureiro MD, Suárez B. Microsatellite characterization of grapevine(Vitis vinifera L. )genetic diversity in Asturias (Northern Spain)[J]. Scientia Horticulturae, 2011, 129 :433-440. |