藏山羊主要分布在青藏高原牧区,其繁殖性能较低,多数一年产一羔[1]。金堂黑山羊产于四川省成都市金堂县,是经过长期自然选择和人工重点选育的多胎地方山羊品种,其繁殖性能高、产羔率高(一胎 2-3羔)[2]。山羊的产羔率是一个经济价值巨大的数量性状,其遗传力低,只有 0.05-0.15[3],表明采用传统的育种方法难以在较短时间内提高山羊的繁殖力。国内外研究者从生殖激素基因、绵羊多胎基因等方面开展了山羊多胎机制研究,但目前对山羊排卵数性状形成的生理学和遗传学机理还不清楚[4, 5]。减数分裂是一种高度保守的有性生殖细胞分裂方式,是生殖细胞成熟的重要阶段,为染色体数目的恒定以及生物体的变异奠定了物质基础。减数分裂的完成需要依赖一系列重要复杂的过程,任何一个阶段出现问题都会导致生殖细胞异常,使机体出现生殖障碍。若人的同源染色体在减数分裂时未分离,会导致不孕和先天缺陷(如唐氏综合症)[6]。错配修复蛋白除了参与 DNA的错配修复、细胞周期的调控外,还可以影响生殖细胞减数分裂过程中DNA重组的忠实性和效率[7],其缺陷可导致哺乳动物的不孕。对于减数分裂和错配修复蛋白之间关系的研究是从 MSH4、MSH5基因的研究开始的,现已确定参与减数分裂的错配修复蛋白包括:MLH1、MLH3、MSH4、MSH5和 PMS2[8]。MSH4和 MSH5是 MutS同源蛋白 DNA错配修复蛋白家族中的 2个主要成员,二者可以形成异源二聚体,形成的异源二聚体在双链 DNA分子上形成“夹子”型结构,称之为 Holliday连接体,参与减数分裂过程中的重组过程,并在减数分裂Ⅰ期促进染色体互换,可影响减数分裂中交换位点的数量与分布[9, 10, 11, 12, 13, 14]。MSH5蛋白是雄性和雌性小鼠减数分裂过程中染色体配对和联会所必需的,参与 DNA损伤应答以及在免疫球蛋白多样性的形成中起重要作用,具有识别并特异性结合错配碱基的特殊功能[15, 16, 17, 18]。研究发现,Msh4和 /或Msh5基因被敲除后会造成小鼠减数分裂Ⅰ前期染色体联会失败,使得小鼠卵巢功能退化,最终导致其不育[18, 19, 20, 21, 22]。现阶段,根据 Shenker等[23]和 Kazma等[24]研究团队进行全基因组关联研究,结果均发现,MSH5可能是肺癌的一个重要相关基因。
目前,有关 MSH4和 MSH5基因在人、小鼠、酵母等物种的研究中均有报道,但在对山羊中的研究尚未见报道。本研究通过对单胎山羊品种藏山羊与多胎山羊品种金堂黑山羊 MSH4和 MSH5基因的cDNA克隆、序列分析、组织表达差异分析,探索MSH4和 MSH5基因在山羊卵母细胞减数分裂中可能发挥的作用,以期为研究山羊多胎机制提供理论依据。
1 材料与方法 1.1 材料 1.1.1实验动物及样品采集分别选取处于同一发情周期的健康藏山羊、金堂黑山羊各 5只作为研究对象。藏山羊选自四川省理县,连续 3胎产单羔的母羊;金堂黑山羊选自四川省金堂县,连续 3胎产3羔的母羊。对选取的 10只母羊进行同期发情处理:将孕酮阴道栓(CIDR)放入山羊阴道内,经 11 d后取出,并在取出 CIDR前 1天肌肉注射氯前列醇钠(Merck Animal Health,Canada)2 mL,在取出 CIDR后 40 h左右将山羊进行屠宰,屠宰后立即采集垂体、输卵管、卵巢、子宫等组织放入冻存管中,并做好标记投入到液氮罐中保存备用。
1.1.2 主要试剂反转录试剂盒购自Fermentas(M -BI)公司;DNA Marker DL2000、2× Taq PCR Maste-rMix、感受态细胞均购自上海天根生物科技有限公司;DNA凝胶回收试剂盒购自爱思进生物技术(杭州)有限公司;氨苄青霉素、克隆载体 pMD19 -T Vector购自大连宝生物工程有限公司; E.coli DH5α感受态细胞购自康迪生物技术有限公司;SsoAdvan -ced™SYBR®Green Supermix、八连管、盖子均购自伯乐公司;引物合成与测序由成都擎科梓熙生物技术有限公司完成。
1.2 方法 1.2.1 总RNA的提取及 cDNA的合成在同一条件下,采用 Trizol法分别提取藏山羊、金堂黑山羊 4种组织(垂体、输卵管、卵巢、子宫)的总 RNA。使用反转录试剂盒 Thermo Scientific RevertAid First Strand cDNA Synthesis Kit进行 cDNA的合成。
1.2.2 引物设计与 PCR扩增目的片段根据 NCBI上已发表的 MSH4、MSH5的基因序列,利用引物设计软件 Primer Premier 5设计克隆引物,引物序列见表 1。以合成的卵巢 cDNA第一链为模板,扩增MSH4、MSH5基因,PCR扩增程序:95℃预变性 4 min;95℃变性 45 s,MSH4、MSH5退火温度分别为55℃/40 s、53.2℃/40 s,72℃延伸120 s,30个循环;最后72℃延伸 5 min;4℃保存。
用1%琼脂糖凝胶电泳对 PCR产物进行检测,并用爱思进 DNA凝胶回收试剂盒对其进行回收纯化。将回收产物与 pMD19-T Vector于 16℃连接过夜,再将连接产物转化宿主菌 E.coli DH5α感受态细胞,并涂布于含有氨苄青霉素的 LB固体培养基上,37℃恒温培养 12h。挑选白色单克隆菌落于含有苄青霉素的 LB液体培养基中,37℃震荡培养 12 h,再对菌液做 PCR鉴定,产物经 1%琼脂糖凝胶电泳检测后,挑选藏山羊、金堂黑山羊的阳性克隆菌液各 2 mL送至成都擎科梓熙生物技术有限公司测序。
1.2.4 山羊MSH4、MSH5基因的生物信息学分析通过DNAman、DNAstar等生物分析软件对基因序列进行同源性比对,利用生物软件 MEGA 5.1构建核苷酸系统进化树。
1.2.5实时荧光定量 PCR根据已克隆出的藏山羊和金堂黑山羊 MSH4、MSH5的序列设计基因的定量引物,引物序列及扩增长度见表 2。
对藏山羊、金堂黑山羊的垂体,输卵管,卵巢,子宫中的MSH4、MSH5基因及内参 β-actin基因 mRNA表达量进行实时荧光定量 PCR检测。反应体系为 10 μL:上下游引物各 0.8 μL,cDNA模板 0.5 μL,ddH2O 2.9 μL,SsoAdvanced ™ SYBR®Green Supermix 5 μL。反应条件为:95℃预变性 3 min;95℃变性 10 s,MSH4、MSH5退火温度分别为61℃/20 s、60℃/20 s,65℃延伸 5 s,40个循环;65℃延伸 5 min;4℃保存。每个样品设置 3个重复,并设置阴性对照。实验结果采用 Pfaffl法[25]分析MSH4、MSH5基因分别在两种山羊不同组织中的相对表达量,计算公式为:

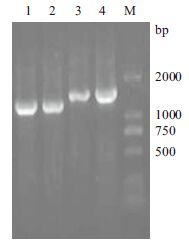
吸取藏山羊和金堂黑山羊 MSH4、MSH5基因PCR扩增产物各 5 μL,进行电泳检测。经 1%琼脂糖凝胶电泳检测条带后,结果(图 1和图 2)显示,其中 MSH4上半段、下半段和 MSH5上半段、下半段条带与预期大小基本一致,且目的条带明亮清晰,可用于后续实验。

|
| 1,2 :分别为藏山羊和金堂黑山羊MSH4 基因上半段;3,4 :分别为藏山羊和金堂黑山羊MSH4 基因下半段;M :DNA Marker DL2000 图 1 MSH4 基因PCR 产物电泳检测 |
|
| 1,2 :分别为藏山羊和金堂黑山羊MSH5 基因上半段;3,4 :分别为藏山羊和金堂黑山羊MSH5 基因下半段;M :DNA Marker DL2000 图 2 MSH5 基因PCR 产物电泳检测 |
获得的藏山羊、金堂黑山羊的 MSH4和 MSH5基因测序已提交到 NCBI中(MSH4登录号分别为 KR864751和KR864752,MSH5登录号分别为KR864753和KR864754)。藏山羊和金堂黑山羊MSH4基因编码区均长2 499 bp,编码 832个氨基酸,两品种基因编码区有 5处碱基不同,并导致了 3处氨基酸的差异。藏山羊和金堂黑山羊 MSH5基因编码区均长 2 496 bp,编码 831个氨基酸,两品种基因编码区有 9处碱基不同,并导致了 5处氨基酸的差异(表 3)。
通过 DNAstar分析藏山羊和金堂黑山羊与其他物种 MSH4、MSH5基因编码区核苷酸序列同源性,结果显示,藏山羊 MSH4基因编码区核苷酸序列与金堂黑山羊、山羊(XM_005678235)、绵羊(XM_004002088)、牛(NM_001206255)、马(XM_001918273)、小鼠(AF178957)、褐家鼠(NM_001 -106477)和人(HSU89293)的同源性分别为:99.8%、99.8%、99.4%、98.1%、94.4%、85.1%、 84.7%和 93.5%;藏山羊 MSH5基因编码区核苷酸序列与金堂黑山羊、山羊(XM_005696572)、牛(NM_ 001205-499)、家犬(XM_005627746)、小鼠(AF -107352)、褐家鼠(NM_212536)和人(AF070071)的同源性分别为:99.6%、99.6%、97.3%、88.0%、85.8%、85.3%和90.2%。
2.4 MSH4、MSH5基因系统发生树的构建利用 Clustal X、MAGA5.1对相应物种的MSH4、MSH5基因编码区进行多序列对比,并构建基因 Neighbor-Joining(NJ)系统发生树(图 3和图 4)。图 3显示,藏山羊首先与金堂黑山羊、山羊聚为一类,再依次与绵羊、牛、马、人聚为一类,最后与小鼠、褐家鼠聚为一类。图 4显示,藏山羊首先与山羊聚为一类,再依次与金堂黑山羊、牛、家犬、人聚为一类,最后与小鼠、褐家鼠聚为一类。系统发生树的构建结果与核苷酸同源性比对结果一致,与哺乳动物进化程度相符合。

|
| 图 3 MSH4 基因的核苷酸系统发生树 |

|
| 图 4 MSH5 基因的核苷酸系统发生树 |
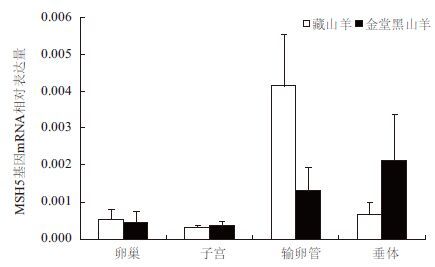
本实验通过实时荧光定量 PCR技术,以 β-actin基因为参照,检测 MSH4、MSH5基因在单胎品种藏山羊和多胎品种金堂黑山羊不同组织间 mRNA表达量,结果表明:MSH4和 MSH5的 mRNA在山羊卵巢、子宫、输卵管、垂体中均有表达,但两个品种间 mRNA表达量无显著性差异( P>0.05,图 5和图 6)。

|
| 图 5 MSH4 基因在藏山羊和金堂黑山羊不同组织中的相对 表达量(x±s) |
|
| 图 6 MSH5 基因在藏山羊和金堂黑山羊不同组织中的相对 表达量(x±s) |
山羊的产羔率是一个具有巨大经济价值的数量性状,提高山羊的产羔率已成为提高养羊业经济效益的重要途径。山羊的产羔率主要取决于一个发情周期的排卵数,所以研究者对山羊开展了大量有关生殖激素基因、绵羊多胎基因等方面的研究,以期揭示其排卵数性状形成的分子调控机制,但未取得突破性进展[3, 26]。减数分裂是生物体有性生殖的基础,是生物遗传、进化和多样性的重要保证,受精和减数分裂一同确保了物种世代染色体数目的稳定不变。研究报道,在生殖细胞减数分裂过程中,若减数分裂相关基因遭到破坏,则后期卵巢内生殖细胞迅速凋亡,机体出现生殖障碍[20],雌性动物主要表现为卵巢功能早衰,雄性动物主要表现为少精、无精等生精缺陷。错配修复蛋白主要参与了减数分裂粗线期的细胞同源重组,在一定程度上保证生殖细胞在减数分裂过程中的高效率与准确性。MSH4、MSH5是 MutS同源蛋白 DNA错配修复蛋白中的两个重要成员,二者以 MSH4 -MSH5异源二聚体的形式通过重组参与减数分裂,并且 MSH4、MSH5蛋白的表达大部分在睾丸跟卵巢,说明它们对生殖细胞的发育与减数分裂有直接作用[27, 28, 29]。Ross-Macdonald等[30]报道,敲除 MSH4基因的小鼠的精子与卵母细胞出现了受损的联会复合体结构以及染色体错配现象。MSH4和 /或 MSH5基因敲除的小鼠生长发育正常,但生殖器官出现异常,雄性小鼠表现为睾丸缩小,雌性小鼠则表现为卵巢退化,最终导致不育[19, 20, 21]。研究结果表明MSH4、MSH5基因在小鼠的减数分裂Ⅰ期同源染色体配对中起到至关重要的作用[20, 31]。
本实验选取连续 3胎产单羔的藏山羊和连续 3胎产 3羔的金堂黑山羊为研究对象,克隆得到各自的MSH4、MSH5基因序列。藏山羊 MSH4基因编码区序列与金堂黑山羊有 5处碱基不同,导致 3处氨基酸的差异;MSH5基因编码区序列有 9处碱基不同,导致 5处氨基酸的差异。藏山羊与金堂黑山羊 MSH4、MSH5氨基酸序列的差异是否会引起生殖细胞减数分裂的变化,进而使得山羊排卵数发生改变而影响山羊的繁殖性状还有待进一步研究。藏山羊和金堂黑山羊 MSH4、MSH5基因编码区序列与牛、小鼠、人等都具有较高的同源性,说明 MSH4和 MSH5基因在哺乳动物进化中均具有较高的保守性。根据 MSH4和 MSH5基因编码区核苷酸序列构建的分子系统发生树,藏山羊、金堂黑山羊跟山羊亲缘关系最近,各分支置信度高,表明系统发生树具有较高的可信度。MSH4、MSH5的 mRNA分别在藏山羊和金堂黑山羊的卵巢、子宫、输卵管和垂体中均有表达,但两个品种各组织间 mRNA表达量均无显著性差异(P>0.05),说明两个基因在山羊卵巢、子宫等组织中发挥着重要的作用,但是这两个基因mRNA表达量的差异可能不是引起排卵数差异的主要原因。
4 结论采用 RT -PCR技术,首次克隆了藏山羊、金堂黑山羊 MSH4和 MSH5基因,两个山羊品种的MSH4基因编码区序列有 5处碱基不同,导致 3处氨基酸的差异;MSH5基因编码区序列有 9处碱基不同,导致 5处氨基酸的差异。MSH4和 MSH5基因在两个山羊品种的卵巢、子宫、输卵管和垂体中均有表达,但品种间 mRNA表达量无显著性差异。
| [1] | 王杰,王永,欧阳熙,等 .藏山羊研究[J].中国畜牧杂志,1993, 29(1):10-13. |
| [2] | 许德贵, 张家明, 唐诗军, 等 . 金堂黑山羊生产性能研究[J]. 四川畜牧兽医, 2006, 33(11):25-26. |
| [3] | Notter DR. Genetic improvement of reproductive efficiency of sheep and goats[J]. Anim Reprod Sci, 2012, 130(3-4):147-151. |
| [4] | 胡亮, 龙石太, 吴宪红, 等 . 川中黑山羊 OPN基因 cDNA克隆及生物信息学分析[J]. 江苏农业科学, 2014, 42(5):34-36. |
| [5] | 龙石太, 胡亮, 吴宪红, 等 . 川中黑山羊BMP2、BMP4基因cDNA克隆及生物信息学分析[J]. 江苏农业科学, 2014, 42(3): 19-23. |
| [6] | Nagaoka SI, Hassold TJ, Hunt PA. Human aneuploidy:mechanisms and new insights into an age-old problem[J]. Nat Rev Genet,2012, 13:493-504. |
| [7] | Schroering AG, Edelbrock MA, Richards TJ, et al. The cell cycle andDNA mismatch repair[J]. Exper Cell Res, 2007, 313(2):292-304. |
| [8] | Jirieny J. The multifaceted mismatch-repair system[J]. Nat RevMol Cell Biol, 2006, 7(5):335-346. |
| [9] | Eisen JA. A phylogenomic study of the MutS family of proteins[J]. Nucleic Acids Res, 1998, 26(18):4291-4300. |
| [10] | Fishel R, Wilson T. MutS homologs in mammalian cells[J]. Curr Opin Genet Dev, 1997, 7(1):105-113. |
| [11] | Karen VM, Cassandra J, Yashna T, et al. Separable crossover-promoting and crossover-constraining aspects of Zip1 activityduring budding yeast meiosis[J]. PLoS Genet, 2015, 11(6): e1005335. |
| [12] | 骆骅 .黄牛和犏牛睾丸组织中减数分裂同源重组基因表达、克隆与启动子区甲基化分析[D].南京:南京农业大学, 2013. |
| [13] | 禄婷婷 .错配修复蛋白基因多态性与少精、无精症的相关研究[D]. 长沙:中南大学, 2009. |
| [14] | Ramaswamy R, Narayanaswamy S, Koodali TN. Structural insights into saccharomyces cerevisiae Msh4-Msh5 complex function using homology modeling[J]. PLoS One, 2013, 8(11):e78753. |
| [15] | Snowden T, Acharya S, Butz C, et al. hMSH4-hMSH5 recognizes Holliday Junctions and forms a meiosis-specific sliding clamp that embraces homologous chromosomes[J]. Mol Cell, 2004, 15(3): 437-451. |
| [16] | 禄婷婷, 徐克前 . 一组多功能的错配修复蛋白[J]. 生命的化学, 2008, 165(6):696-700. |
| [17] | Wu XL, Xu KQ, Her CT. The Role of muS homologues MSH4 and MSH5 in DNA metabolism and damage response[M]// Kusic-Tisma J. DNA replication and related cellular processes. Rijeka,Croatia:In Tech Press, 2001:87-110. |
| [18] | Her C, Zhao N, Wu X, et al. MutS homologues hMSH4 andhMSH5:diverse functional implications in humans[J]. FrontBiosci, 2007, 12(3):905-911. |
| [19] | Edelmann W, Cohen PE, Kneitz B, et al. Mammalian MutShomologue 5 is required for chromosome pairing in meiosis[J].Nature Genetics, 1999, 21:123-127. |
| [20] | Kneitz B, Cohen PE, Avdievich E, et al. MutS homolog 4 localization to meiotic chromosomes is required for chromosomepairing during meiosis in male and female mice[J]. Genes Dev, 2000, 14(9):1085-1097. |
| [21] | De Vries SS, Baart EB, Dekker M, et al. Mouse MutS-like protein Msh5 is required for proper chromosome synapsis in male andfemale meiosis[J]. Genes Dev, 1999, 13(5):523-531. |
| [22] | Ehrenstein MR, Neuberger MS. Deficiency in Msh2 affects the efficiency and local sequence specificity of immunoglobulin class-switch recombination:parallels with somatic hypermutation[J]. EMBO J, 1999, 18(12):3484-3490. |
| [23] | Shenker NS, Polidoro S, van Veldhoven K, et al. Epigenome-wide association study in the European Prospective Investigation intoCancer and Nutrition(EPIC-Turin)identifies novel genetic lociassociated with smoking[J]. Hum Mol Genet, 2013, 22(5): 843-851. |
| [24] | Kazma R, Babron MC, Gaborieau V, et al. Lung cancer and DNArepair genes:multilevel association analysis from the International Lung Cancer Consortium[J]. Carcinogenesis, 2012, 33(5): 1059-1064. |
| [25] | Pfaffl MW. A new mathematical model for relative quantification in real-time RT-PCR[J]. Nucleic Acids Res, 2001, 29(9):e45. |
| [26] | 胡亮, 字向东, 卢建远, 等 . 多胎和单胎山羊品种 Bcl-2和 Bax基因的克隆及组织表达研究[J]. 畜牧与兽医, 2015, 47(1): 28-32. |
| [27] | Bocker T, Barusevicius A, Snowden T, et al. hMSH5: a human MutS homologue that forms a novel heterodimer with hMSH4 and is expressed during spermatogenesis[J]. Cancer Res, 1999, 59(4): 816-822. |
| [28] | Her C, Doggett NA. Cloning, structural characterization, and chromosomal localization of the humanorthologue of Saccharomyces cerevisiae MSH5 gene[J]. Genomics, 1998, 52(1):50-61. |
| [29] | Moens PB, Kolas NK, Tarsounas M, et al. The time course and chromosomal localization of recombination-related proteins at meiosis in the mouse are compatible with models that can resolve the early DNA-DNA interactions without reciprocalrecombination[J]. J Cell Sci, 2002, 115(8):1611-1622. |
| [30] | Ross-Macdonald P, Roeder GS. Mutation of a meiosis-specific MutShomolog decreases crossing over but not mismatch correction[J]. Cell, 1994, 79(6):1069-1080. |
| [31] | Her C, Wu X, Griswold MD, et al. Human MutS homologue MSH4 physically interacts with von Hippel-Lindau tumor suppressor-binding protein 1[J]. Cancer Res, 2003, 63(4):865-872. |