2. 湖南省农业科学院植物保护研究所,长沙 410125
2. Plant Protection Institute, Hunan Academy of Agricultural Sciences,Changsha
乙烯应答转录因子(Ethylene-response factor,ERF)属于AP2/ERF转录因子超家族,共同特征是都含有由57-66个氨基酸残基组成的保守AP2/ERF结构域,在调控生物和非生物胁迫应答基因表达中发挥重要的转录调控作用[1, 2, 3, 4, 5]。根据AP2/ERF保守结构域的数量差异和是否含有其他结构域,可将ERF转录因子超家族划分为3个主要的亚族:AP2亚族、ERF亚族和RAV亚族[6]。AP2亚族含有两个重复的AP2/ERF结构域,ERF亚族只含有一个AP2/ERF结构域,RAV亚族除了含有一个AP2/ERF结构域,还含有一个植物特异性转录因子中的保守B3结构域[6]。另外,依据AP2/ERF氨基酸序列的保守性序列特征,通常又将ERF超家族分成两个亚家族:CBF/DREB和ERF[6]。
ERF转录因子在植物生长发育,如花和种子发育、果实成熟,植物非生物胁迫(如低温、干旱和高盐胁迫等[7]),以及植物抗病反应中起着重要的作用[8, 9]。水稻乙烯转录因子OsEATB通过下调赤霉素生物合成酶基因的表达控制节间的生长[10];ERF是植物非生物胁迫的关键调节子[11, 12],AtERF73应对低氧胁迫[13]、AtRAP2应对低钾胁迫[14];DREB类型转录因子参与植物的高温和干旱胁迫[15, 16, 17];ERF是植物抗病信号转导路径的重要调控基因[18],超表达GmERF5的大豆植株能显著增强对大豆疫霉菌的抗性[19],超表达小麦ERF基因TaPIE1能显著增强小麦植株对丝核菌(Rhizoctonia cerealis)的抗性[20],辣椒的ERF基因CaERF1、CaJERF1在植物的抗病应答以及非生物胁迫中发挥重要作用[21, 22]。因此,筛选、鉴定、分离新的ERF转录因子,将为利用生物技术改良植物的抗病、抗逆、生长发育调控提供有效的基因资源。
本研究在辣椒与根结线虫互作后的转录组分析基础上,依据一系列备选上调表达的基因序列结合ERF基因的保守性,设计序列特异性引物,采用RT-PCR方法,从南方根结线虫侵染辣椒后的根部组织中分离出一个乙烯应答转录因子基因的核心序列,命名为CaERF18,研究这一基因在南方根结线虫诱导后不同时间的表达特性,初步了解CaERF18基因的结构和诱导特征,为深入阐明CaERF18基因的结构和功能、揭示CaERF18基因介导的辣椒抗根结线虫机制,最终为利用CaERF18基因改良植物的抗病性和抗逆性鉴定基础。
1 材料与方法 1.1 材料辣椒(Capsicum annuum L.)双单倍体材料HDA149,含有单显性抗根结线虫基因Me3,由中国农业科学院蔬菜花卉研究所茆振川博士赠送(最初从法国农业科学院Alain Palloxin 博士处引进),在湖南省蔬菜研究所示范基地扩繁。南方根结线虫(Meloidogyne incognita)从湖南省蔬菜研究所示范基地采集,经形态学、特异引物PCR鉴定后进行致病性测定,最后采集单卵块用茄门甜椒扩繁。
1.2 方法 1.2.1 总RNA的提取和cDNA的合成辣椒种子消毒后播种于育苗钵内,幼苗长至4-5片真叶时移栽,移栽10 d后接种南方根结线虫二龄幼虫,每株接种1 000条,分别在取样之后的第12、36和72 h取1.5-2.0 cm长的根尖组织,液氮冷冻后置于-80℃的冰箱中,取用时混合磨样。总RNA采用天根生化科技有限公司的“TRNzol A+总RNA提取试剂盒”,总RNA的质量和完整性用1%的琼脂糖凝胶电泳鉴定。第1链cDNA的合成采用SuperScript®Ⅲ First-Strand Synthesis System(Invitrogen)试剂盒。
1.2.2 CaERF18部分序列的克隆与分析根据南方根结线虫侵染辣椒后的转录组数据分析结果,发现一条含有约900 bp的序列显著上调表达,初步预测是一个具有特定结构的转录因子,根据此序列的结构特征设计一对特异引物CaERF18-F(5'-AAGAAT-CGGTTCTCGCAAAAG-3')和CaERF18-R(5'-ATTA-TCATCCTCAGTTTCACAG-3'),以1.2.1合成的cDNA模板进行PCR扩增,20 µL扩增体系(1 µL cDNA、1 µL CaERF18-F、1 µL CaERF18-R、1.6 µL dNTP(2.5 mmol/L)、2 µL 10×buffer、Taq DNA polymerase 0.2 µL(5 U/µL)、13.2 µL ddH2O)。扩增程序为:94℃预变性3 min;94℃ 30 s,58℃ 30 s,72℃ 50 s,35个循环;72℃延伸10 min。电泳后回收目的扩增产物,连接、克隆到T 载体后送上海生工测序。测序结果用Primer、DNAman软件及NCBI网站的Nucleoide Blast、Protein Blast等程序进行分析。
1.2.3 根结线虫侵染对CaERF18基因表达的影响参照1.2.1的方法用南方根结线虫二龄幼虫接种辣椒幼苗,以不接种南方根结线虫的辣椒幼苗为对照,分别在接种后的6、12、24、36和72 h取根尖组织样品,用液氮速冻、-80℃保存。总RNA提取及cDNA的合成参照1.2.1。
根据CaERF18基因cDNA序列特征,用Primer Premier5.0和Oligo7.0软件设计实时荧光定量PCR引物,引物为CaERF18-qF(Primer:5'-CGATGAT-TCTGAGAGCCCAC-3')和CaERF18-qR(Primer:5'-TGCCCTATTACCTGGAAGTTGC-3')。辣椒β-actin为 参照基因[23],扩增引物为β-actin-qF(5'-TGCAGG-AATCCACGAGACTAC-3')和β-actin-qR(5'-TACCA-CCACTGAGCACAATGTT-3')。qRT-PCR以各取样时间点的根部组织cDNA为模板,20 µL扩增体系中包含10 µL 2×SYBR®Premix Ex TaqTMⅡ(TaKaRa),上、下游引物(10 µmol/L)各0.8 µL,cDNA(第一链cDNA合成后稀释40倍)1.6 µL,加ddH2O至总体积20 µL。PCR反应程序为:94℃预变性30 s;94℃ 30 s,58℃ 30 s,72℃ 10 s,40个循环;反应完成后分析熔解曲线以及荧光值变化曲线,实验重复3次。
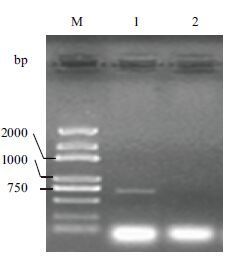
2 结果 2.1 CaERF18的序列结构将辣椒植株接种南方根结线虫,接种后的12、36和72 h分别取根尖组织样品,混合磨样提取总RNA,用设计的扩增引物对提取的RNA进行RT-PCR扩增,扩增产物经电泳检测后扩增出 694 bp的专一性片段(图 1)。对扩增产物进行回收、连接、克隆和测序,将测序序列在GenBank中进行Blastn比对和保守结构预测,发现这一序列含有一典型的保守AP2/ERF基序,与烟草(Nicotiana tomentosiformis)NtERF118的相似性达46%,因此将其暂命名为CaERF18。进一步分析推定的231个氨基酸序列发现,序列中含有一个长度为59个氨基酸残基组成的AP2结构域,以及一段富含R、K氨基酸残基的类似核定位信号区(图 2),同时将这一推定的氨基酸序列在BaCeILo(http://gpcr2.biocomp.unibo.it/bacello/index.htm)上分析,发现定位于细胞核,说明CaERF18基因是在细胞核中起转录调控作用的转录因子。
|
| M :Trans 2K DNA marker ;1 :12 、36 和72 h 的混合样品扩增;2 :对照 图 1 特异引物RT-PCR 扩增电泳图 |

|
| Nt :烟草(Nicotiana tomentosiformis);Sl :番茄(Solanum lycopersicum);Ca :辣椒(Capsicum annuum);Br :芜菁(Brassica rapal); At :拟南芥(Arabidopsis thaliana);St :马铃薯(Solanum tuberosum)。下划线为推定的核定位信号域;方框为推定的AP2/ERF 结构域 图 2 CaERF18 cDNA 核心序列及推定的氨基酸序列 |
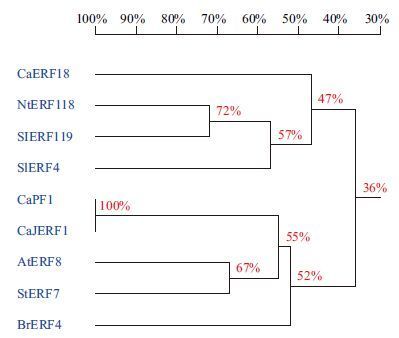
为进一步分析比较CaERF18基因与其他种类植物ERF基因的相似性,利用NCBI数据库中的BlastTP同源搜索,选择了同源性较高的烟草(Nicotiana tomentosiformis L.)、番茄(Solanum lycop-ersicum L.)、辣椒(C. annuum L.)、芫菁(Brassica rapal L.)、马铃薯(Solanum tuberosum L.)和拟南芥(Arabidopsis thaliana)6个物种中的8个ERF蛋白进行多重氨基酸序列比对,比对采用DNAMAN软件进行,比对结果(图 3)显示,8个基因在保守结构域上都具有共同的氨基酸序列。相似性和进化关系(图 4)表明,CaERF18与同科的烟草NtERF118和SlERF119的相似性最高。

|
| 图 3 部分ERF 转录因子与 CaERF18 蛋白AP2/ERF 结构域的比对 |
|
| 图 4 CaERF18 与其他作物ERF 类型转录因子同源性比对 |
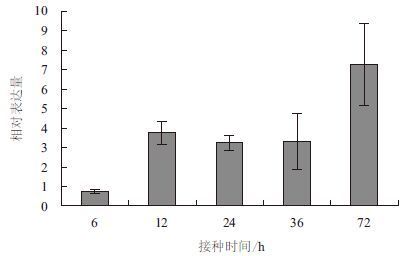
用南方根结线虫二龄幼虫侵染辣椒材料HDA149,依据线虫侵染的前期、初期、中期和后期4个时间段,取侵染后不同时间段的根尖组织,提取总RNA,实时荧光定量PCR分析了CaERF18在病原线虫侵染后的表达特征。结果(图 5)显示,线虫接种6 h的相对表达量为0.8倍,12 h后上升到了3.8倍,之后在24 h和36 h的表达量相对较为稳定,分别为3.3倍和3.4倍,到72 h后,相对表达量达到监测的最高值,为7.2倍。根结线虫侵染的时间段来看,CaERF18的相对表达水平呈上升的趋势,具有被病原物诱导而显著上调表达的特征。
|
| 图 5 CaERF18 在南方根结线虫接种的辣椒中表达的实时荧光定量PCR 分析 |
ERF类型转录因子是植物中特有的一类参与植物应对生物和非生物胁迫的转录因子[7, 19, 24]。许多研究认为ERF转录因子主要与植物抗逆,如水胁迫、盐胁迫、耐旱、耐冷、耐热等相关[7, 12, 17, 25],但越来越多的研究显示,ERF类型转录因子是植物抗病应答反应的关键调控因子,在植物的抗病反应中发挥重要作用。超表达GmERF5可增强大豆对大豆疫霉病的抗性[19],TaPIE1通过激活乙烯信号转导途径中下游防卫基因和胁迫基因的表达正调控小麦对病原物立枯丝核菌侵染和冷害的防卫反应[20],ERF类型转录因子是油菜(B. napus)应对生物胁迫信号转导中的重要参与者[26]。而很多ERF类型的转录因子既参与生物胁迫又参与逆境胁迫[20, 26],本实验通过RT-PCR技术分离到一个辣椒ERF类型转录因子的核心cDNA序列,将为下一步从转录水平上同时研究辣椒的抗病和抗逆机制提供一个新的候选基因。
多数ERF类型转录因子都具有诱导表达的特性,但具有一定的时空特性。根结线虫侵染辣椒是一个动态的过程,茆振川等[27]研究显示,用根结线虫接种辣椒品种HDA149后的0-6 h根结线虫侵染率为0,12 h有少量线虫开始侵入,24 h和36 h有大量线虫侵入,且在线虫周围有许多坏死细胞。本研究结果显示,在线虫接种后的6 h,CaERF18的相对表达量为0.8倍,此时线虫还尚未游动到根尖组织附近,没有触动辣椒的抗病性反应,而当接种后的12、24和36 h,CaERF18的相对表达量上升到了3.3-3.8倍,说明大量线虫侵入后诱导的辣椒抗病性反应中抗病相关基因的表达被激活,特别是在线虫大规模侵入后的72 h,CaERF18的相对表达量达到了7.2倍,而辣椒HDA149含有单显性抗线虫基因Me3,推测CaERF18可能参与了这一由Me3基因介导的抗病信号转导过程,在这一过程中CaERF18由于根结线虫的大量侵入而被诱导激活和上调表达,可能具有重要的调控作用。在拟南芥中乙烯信号转导对北方根结线虫的侵入有强烈的吸引或趋避作用[28],那么辣椒中的乙烯信号转导对南方根结线虫而言,是否具有如拟南芥一样的吸引或趋避作用,需要进一步的实验进行验证。此外CaERF18与NtERF118、SlERF119、SlERF4等具有较高的同源性,还具有典型的核定位信号区,推测CaERF18可能同时参与了抗病和抗逆信号转导。因此分离CaERF18的全长cDNA,详细了解多种病原物和多种逆境胁迫的表达特性、组织表达特性、调控的下游靶标基因、参与的信号调控网络等,将为阐明辣椒的抗病和抗逆分子机制提供新的基因资源,为今后的抗病和抗逆育种提供新思路。
4 结论通过RT-PCR方法从辣椒中分离到一个新的ERF类型转录因子核心序列,序列分析显示该基因具有一个由59个氨基酸残基组成的典型AP2/ERF类型转录因子保守结构域和一个核定位信号区。qRT-PCR分析表明,CaERF18受病原物南方根结线虫的侵染而上调表达,接种南方根结线虫6、12、24、36和72 h的相对表达量分别为0.8、3.8、3.3、3.4和7.2倍。
| [1] | Zhang G, Chen M, Chen X, et al. Phylogeny, gene structures, and expression patterns of the ERF gene family in soybean(Glycine max L. )[J]. J Exp Bot, 2008, 59(15):4095-4107. |
| [2] | Zhuang J, Cai B, Peng RH, et al. Genome-wide analysis of the AP2/ERF gene family in Populus trichocarpa[J]. Biochem Biophys Res Commun, 2008, 371(3):468-474. |
| [3] | Hu L, Liu S. Genome-wide identification and phylogenetic analysis of the ERF gene family in cucumbers[J]. Genetics and Molecular Biology, 2011, 34(4):624-633. |
| [4] | Yan HW, Hong L, Zhou YQ, et al. A genome-wide analysis of the ERF gene family in sorghum[J]. Genetics and Molecular Research:GMR, 2013, 12(2):2038-2055. |
| [5] | Rao G, Sui J, Zeng Y, et al. Genome-wide analysis of the AP2/ERF gene family in Salix arbutifolia[J]. FEBS Open Bio, 2015(5):132-137. |
| [6] | Nakano T, Suzuki K, Fujimura T, et al. Genome-wide analysis of the ERF gene family in Arabidopsis and rice[J]. Plant Physiol, 2006, 140(2):411-432. |
| [7] | Li MY, Xu ZS, Huang Y, et al. Genome-wide analysis of AP2/ERF transcription factors in carrot(Daucus carota L. )reveals evolution and expression profiles under abiotic stress[J]. Mol Genet Genomics, 2015, 290(6):2049-2061. |
| [8] | Xu ZS, Chen M, Li LC, et al. Functions and application of the AP2/ERF transcription factor family in crop improvement[J]. J Integr Plant Biol, 2011, 53(7):570-585. |
| [9] | Zhang JY, Wang QJ, Guo ZR. Progresses on plant AP2/ERF transcription factors[J]. Hereditas, 2012, 34(7):835-847. |
| [10] | Qi W, Sun F, Wang Q, et al. Rice ethylene-response AP2/ERF factor OsEATB restricts internode elongation by down-regulating a gibberellin biosynthetic gene[J]. Plant Physiol, 2011, 157(1):216-228. |
| [11] | Mizoi J, Shinozaki K, Yamaguchi-Shinozaki K. AP2/ERF family transcription factors in plant abiotic stress responses[J]. Biochim Biophys Acta, 2012, 1819(2):86-96. |
| [12] | Yang H, Yu C, Yan J, et al. Overexpression of the Jatropha curcas JcERF1 gene coding an AP2/ERF-type transcription factor increases tolerance to salt in transgenic tobacco[J]. Biochemistry Biokhimiia, 2014, 79(11):1226-1236. |
| [13] | Yang CY, Hsu FC, Li JP, et al. The AP2/ERF transcription factor AtERF73/HRE1 modulates ethylene responses during hypoxia in Arabidopsis[J]. Plant Physiol, 2011, 156(1):202-212. |
| [14] | Kim MJ, Ruzicka D, Shin R, et al. The Arabidopsis AP2/ERF transcription factor RAP2. 11 modulates plant response to low-potassium conditions[J]. Mol Plant, 2012, 5(5):1042-1057. |
| [15] | Sakuma Y, Maruyama K, Osakabe Y, et al. Functional analysis of an Arabidopsis transcription factor, DREB2A, involved in drought-responsive gene expression[J]. Plant Cell, 2006, 18(5):1292-1309. |
| [16] | Qin F, Sakuma Y, Tran LS, et al. Arabidopsis DREB2A-interacting proteins function as RING E3 ligases and negatively regulate plant drought stress-responsive gene expression[J]. Plant Cell, 2008, 20(6):1693-1707. |
| [17] | Sato H, Mizoi J, Tanaka H, et al. Arabidopsis DPB3-1, a DREB2A interactor, specifically enhances heat stress-induced gene expression by forming a heat stress-specific transcriptional complex with NF-Y subunits[J]. Plant Cell, 2014, 26(12):4954-4973. |
| [18] | Gutterson N, Reuber TL. Regulation of disease resistance pathways by AP2/ERF transcription factors[J]. Curr Opin Plant Biol, 2004, 7(4):465-471. |
| [19] | Dong L, Cheng Y, Wu J, et al. Overexpression of GmERF5, a new member of the soybean EAR motif-containing ERF transcription factor, enhances resistance to Phytophthora sojae in soybean[J]. J Exp Bot, 2015, 66(9):2635-2647. |
| [20] | Zhu X, Qi L, Liu X, et al. The wheat ethylene response factor transcription factor pathogen-induced ERF1 mediates host responses to both the necrotrophic pathogen Rhizoctonia cerealis and freezing stresses[J]. Plant Physiol, 2014, 164(3):1499-1514. |
| [21] | 蔡汉阳, 肖卓丽, 严雁, 等. 辣椒CaERF4的全长cDNA克隆及其表达分析[J]. 热带作物学报, 2014(6):1102-1107. |
| [22] | 张秋平, 杨宇红, 茆振川, 等. 辣椒乙烯反应转录因子基因CaJERF1的克隆及诱导表达[J]. 园艺学报, 2012, 39(4):705-712. |
| [23] | Chen R, Li H, Zhang L, et al. CaMi, a root-knot nematode resistance gene from hot pepper(Capsium annuum L.)confers nematode resistance in tomato[J]. Plant Cell Reports, 2007, 26(7):895-905. |
| [24] | Lata C, Mishra AK, Muthamilarasan M, et al. Genome-wide investigation and expression profiling of AP2/ERF transcription factor superfamily in foxtail millet(Setaria italica L. )[J]. PLoS One, 2014, 9(11):e113092. |
| [25] | Wang X, Han H, Yan J, et al. A new AP2/ERF transcription factor from the oil plant Jatropha curcas confers salt and drought tolerance to transgenic tobacco[J]. Applied Biochemistry and Biotechnology, 2015, 176(2):582-597. |
| [26] | Zhuang J, Zhu B. Analysis of Brassica napus ESTs:gene discovery and expression patterns of AP2/ERF-family transcription factors[J]. Mol Biol Rep, 2014, 41(1):45-56. |
| [27] | 茆振川, 谢丙炎, 杨宇红, 等. 辣椒 Me3 基因介导抗根结线虫相关基因的SSH分析[J]. 园艺学报, 2007, 34(6):1453-1458. |
| [28] | Fudali SL, Wang C, Williamson VM. Ethylene signaling pathway modulates attractiveness of host roots to the root-knot nematode Meloidogyne hapla[J]. Mol Plant Microbe Interact, 2013, 26(1):75-86. |



