2. 上海申漕特种水产开发公司,上海 201516
2. Shanghai Shencao Special Fisheries Development Corps,Shanghai 201516
罗氏沼虾是一种养殖范围较广的淡水经济虾类,现已成为我国主要的养殖虾类之一,具有重要的经济价值[1]。但近几年,罗氏沼虾在养殖过程中病害频发,苗种质量也时好时坏,给我国罗氏沼虾养殖发展造成了重大困扰,多数学者提出是高密度养殖、过多使用抗菌药物、虾苗抗逆性差及近亲交配导致的种质退化等原因所致[2, 3, 4]。苗种质量的好坏主要取决于种质、亲本质量、育苗工艺、药物使用情况等,且很大程度上取决于幼体的质量[5]。凡纳滨对虾幼体发育至仔虾需12 d左右,中国明对虾一般需21 d,发育速度快慢相差一般不超过3 d[6, 7, 8, 9]。不同于凡纳滨对虾(Litopenaeus vannamei)、中国明对虾(Fenneropenaeus chinensis)等虾类,罗氏沼虾的无节幼体期(Nauplius,N)是在胚胎中度过的,而其溞状幼体(Zoea,Z)需经11次蜕皮才能发育成仔虾[10]。经过多年生产实践和研究,罗氏沼虾幼体发育具有明显的不同步性。相同培育条件下,同窝同天孵出的溞状幼体发育至仔虾所需时间约13-40 d[11],即使改善饵料和环境因子,部分幼体发育滞缓仍明显,幼体发育速度显著不一致。造成罗氏沼虾幼体发育速度不同步的原因现阶段尚未见报道。
前期研究表明,罗氏沼虾的幼体发育速度不同对其生长、存活等有显著影响。罗氏沼虾在仔虾期与幼虾期的生长速度、成活率、规格整齐度、性别比均随幼体期发育速度的减缓而呈下降趋势。本实验基于前期研究基础,通过人工培育同窝同日幼体,分析幼体发育不同步变态的罗氏沼虾仔虾间的遗传多样性和遗传变异,以期从分子生物学角度探讨造成幼体期发育速度不同步和生长差异的原因,为罗氏沼虾的人工繁殖、遗传育种与制种方面提供理论基础和实践指导。
1 材料与方法 1.1 材料 1.1.1 实验用亲虾实验用亲虾取自上海申漕特种水产开发公司养殖的罗氏沼虾群体。随机挑选数十对附肢完整、规格一致的健康成虾,雌雄分开饲养并营养强化,待雌虾性腺发育至近成熟时,雌雄虾两两随机组合配对,交配抱卵,从中挑选4尾同日抱卵雌虾作为试验用繁殖亲虾,L=(9.226±0.330)cm,W=(23.376±4.082)g,同池饲养。
1.1.2 实验用仔虾与分级待抱卵亲虾腹部卵块呈浅灰色时,将4尾抱卵虾分别随机放入盛满育苗用水的水槽中暂养,溞状幼体全部孵出后移走亲虾。每日观察并记录幼体发育情况,从仔虾出现开始,每隔3日,将3日内连续变态发育的仔虾全部移出,幼体在原育苗桶中继续培育,重复此过程5次,每批仔虾移出后均采用间断添加淡水方式淡化4 d至盐度小于1,每批取30尾入水泥池中饲养。根据幼体发育至仔虾的不同培育天数共获得幼体期发育速度不同的PⅠ、PⅡ、PⅢ、PⅣ、PⅤ(PⅠ表示第1日出现的仔虾日,PⅡ为出现仔虾第2日-第4日出现的仔虾,PⅢ为出现仔虾第5日-第7日出现的仔虾,PⅣ为出现仔虾第8日-第10日出现的仔虾,PⅤ为出现仔虾第11日-第13日出现的仔虾)5批仔虾,即为试验用仔虾。采用循环水系统育苗,水温、水质保持一致,育苗期间投喂卤虫,投喂密度相似。因仔虾偏小,提取DNA后DNA浓度偏低,故将实验用仔虾在相同规格的水泥池中饲养后取肌肉提取DNA。饲养期间投喂量、温度和密度等基本保持一致,30 d后取仔虾肌肉浸没于无水乙醇中,4℃保存备用。
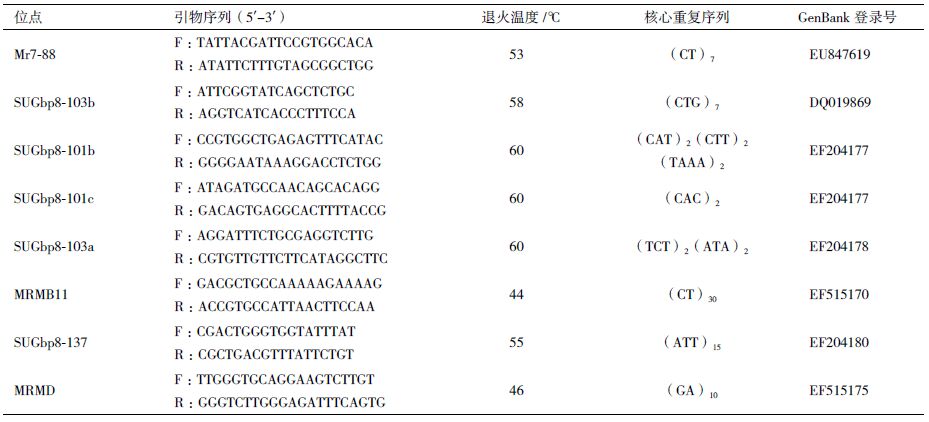
1.1.3 微卫星引物本实验使用的微卫星标记引物共8对,Mr7-88、SUGbp8-101b、SUGbp8-101c、SU-Gbp8-103a、MRMB11、MRMC2六对引物由已发表的外文文献中筛选而来,SUGbp8-103b、SUGbp8-137两对引物则由本实验室自主开发。引物合成由上海生工生物工程服务有限公司完成,引物信息见表 1。
组织DNA提取主要过程参照Strauss[12]提取方法,取肌肉组织100 mg左右洗净、剪碎、烘干后经蛋白酶消化,酚-氯仿法抽提、纯化,无水乙醇沉淀,最后加入适量TE缓冲液(10 mmol/L Tris-HCl,pH8.0;1 mmol/L EDTA,pH8.0)溶解DNA,制成终浓度为50 ng/μL的模板DNA,4℃保存备用。
1.2.2 PCR反应扩增及PCR产物电泳检测PCR反应体系主要参考戴习林等[13]方法,6对国外引物的循环次数和退火温度参考文献[14, 15, 16, 17, 18]。采用Eppendorf公司PCR仪(22331HAMBURG)对5组幼体发育速度不同的罗氏沼虾群体DNA进行PCR扩增,8%非变性聚丙烯酰胺对PCR产物进行电泳分离,常规银染法监测。
1.2.3 数据分析采用Gelpro analyzer4.5[19]软件分析电泳谱带,确定扩增片段大小并判定个体基因型;采用PopGene(Version 3.2)[20]和Excle统计软件分析微卫星基因座位的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、遗传分化系数(Gst)、基因流(Nm)以及Nei’s遗传距离和遗传一致度[21]。
根据Botstein等[22]公式计算群体的多态信息含量(Polymorphism information content,PIC):

利用8对微卫星引物对5组幼体发育速度不同的罗氏沼虾群体进行检测,结果(表 2)显示,共检测到57个等位基因,各微卫星的等位基因数(Na)在5-10个之间,其中微卫星座位SUGbp8-103a检测到的等位基因最多(10个),微卫星座位Mr7-88、SUGbp8-101b、MRMC2发现最少(5个),平均等位基因数为7.125;有效等位基因数(Ne)为1.820 8-5.085 5,平均有效等位基因数为3.782 1;观测杂合度(Ho)为0.416 7-0.833 3;期望杂合度(He)为0.451 2-0.804 0。8个微卫星座位的PIC为0.409 3-0.777 9,平均PIC值为0.678 1,大于0.50[22],属于高度多态位点,表明8个微卫星座位适用于本试验群体的遗传多样性分析。其中微卫星座位MRMB11和SUGbp8-137的PIC非常接近,分别为0.683 7和0.685 1,说明两个标记来自父本(或母本)的同一个等位标记的可能性相似。
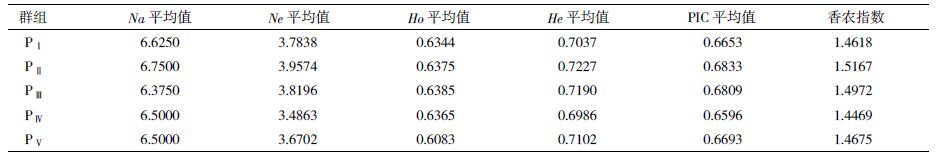
5组幼体发育速度不同的罗氏沼虾群体在8个微卫星座位上的遗传参数(表 3)显示,5组罗氏沼虾群体的平均等位基因数、平均有效等位基因数、观测杂合度、期望杂合度、香农指数和遗传多样性指数均比较接近,无明显差异。5组罗氏沼虾群体的多态信息含量(PIC)均大于0.5,分别为 0.665 3(PⅠ)、0.683 3(PⅡ)、0.680 9(PⅢ)、0.659 6(PⅣ)、0.669 3(PⅤ),说明5组群体均表现为高度多态。
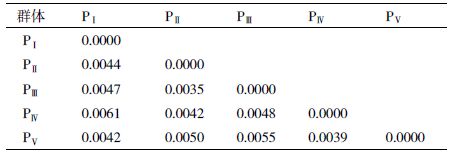
遗传分化系数(Gst)是表征群体间遗传分化程度的重要参数。5组幼体发育速度不同的罗氏沼虾群体的遗传分化系数(表 4)显示,两两群体间的遗传分化系数均小于0.05,介于0.003 5-0.006 1之间,说明各组群体间的遗传分化都很微弱,或不存在遗传分化。且5组群体在8个位点的平均遗传分化系数为0.007 4(表 2),表明5组群体的遗传分化,仅0.74%来自幼体发育速度不同,99.26%来自幼体发育速度不同的个体间。无论是两两群体间的遗传分化系数,还是5组群体在8个位点的平均遗传分化系数,均说明5组幼体发育速度不同的罗氏沼虾群体的遗传分化主要来自于个体间的遗传差异。
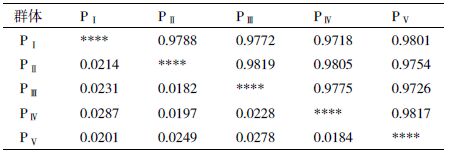
Nei’s遗传距离和遗传一致度可以反应各样本组间亲缘关系的远近,根据其计算方法,计算得到5组幼体发育速度不同的群体间的Nei’s遗传距离和遗传相似系数。结果(表 5)显示,5组幼体发育速度不同的罗氏沼虾群体间遗传距离和遗传相似系数均非常接近,彼此间并无明显的差异。其中PⅠ群体和PⅣ群体间遗传距离最大,为0.028 7,遗传相似系数最小,为0.971 8;PⅡ群体和PⅢ群体间遗传距离最小为0.018 2,遗传相似系数最大,为0.981 9。
遗传分化系数(Gst)和遗传距离均可有效表示物种间的遗传变异和遗传分化程度,用微卫星标记计算出的遗传距离更能客观地反映出群体间的遗传变异和系统分化[24, 25]。实验中5组罗氏沼虾群体两两群体间的遗传分化系数和遗传距离均非常接近,且无明显差异,说明5组罗氏沼虾群体间的遗传分化主要来自于幼体发育速度不同的个体间;同时5组罗氏沼虾群体的杂合度、多态信息含量和香农指数亦无明显差异,说明5组幼体发育速度不同的群体间的遗传变异很微弱或不存在遗传变异[26]。结合前期研究表明基因对同窝罗氏沼虾幼体发育速度的不同步和生长差异的影响微弱,或不存在影响。
生物性状表现是基因效应和环境效应共同影响的结果。实验中,同窝同日产出的罗氏沼虾幼体,在水温、水质和投喂量基本一致的情况下,蚤状幼体的变态依然表现出不同步性。同时前期研究中,幼体发育速度不同步的罗氏沼虾群体在饲养条件基本相同的条件下,其生长速度、成活率等均有差异,并与幼体发育速度显著相关,表明罗氏沼虾幼体发育速度不同步和生长差异亦非环境因素引起的,或者影响较小。
虾类胚胎发育和开口前仔体发育都属于卵黄营养阶段,卵黄主要成分是卵黄蛋白,卵黄蛋白的数量和质量对于维持虾类早期生命至关重要[27]。罗氏沼虾的胚胎发育和溞状幼体孵化后的第一期幼体都是由卵巢中卵黄蛋白来提供物质和能量,所以胚胎和幼体中卵黄蛋白的消耗状况可直接影响罗氏沼虾发育进程[28]。罗氏沼虾卵黄物质主要在初级卵母细胞的细胞器上形成,并随卵母细胞发育而逐渐积累[29, 30]。同一卵巢内不同卵细胞发育区的初级卵母细胞,其分化时间和发育阶段不同,周围卵母细胞较中央卵母细胞发育快,先转入卵母细胞的大生长期[9]。因此,罗氏沼虾亲虾卵巢发育的不同步可能导致卵黄蛋白积累不同,从而间接导致其幼体发育速度不同步和生长差异。同时,罗氏沼虾胚胎发育过程中利用卵黄物质所需的消化酶类来源于母本,受精时已经存在于卵内,在卵母细胞形成过程中合成并储存于卵黄内[28]。因此增强母体营养,使受精卵带有更多卵黄蛋白和消化酶类,提高卵黄物质的利用,对提高幼体质量和幼体变态具有重要作用。此外卤虫(生物饵料)体内不饱和脂肪酸含量变化、温度剧变和惊吓等对罗氏沼虾的幼体变态、生长和存活都有一定影响[31, 32, 33, 34]。
4 结论通过对5组幼体发育速度不同的罗氏沼虾群体进行遗传多样性和遗传变异分析发现,基因对同窝幼体发育速度不同步和生长差异的影响非常微弱,具体原因尚需继续探讨。
| [1] | 刘恩生, 万全. 罗氏沼虾的养殖现状与发展前景[J]. 安徽农业大学学报:自然科学版, 1997(2):85-88. |
| [2] | 陈马康. 罗氏沼虾养殖的现状和前景[J]. 科学养鱼, 2000(8):10-13. |
| [3] | 林治术, 曹志海, 王琴勋. 加快水产优良品种选育促进苗种的健康生产[J]. 齐鲁渔业, 2006(11):47-48. |
| [4] | 史建华, 肖雨, 徐琴英. 罗氏沼虾引种复壮技术的研究[J]. 水产科技情报, 2001(2):64-67. |
| [5] | 卢小花, 江林源, 黄光华, 等. 罗氏沼虾幼体质量对育苗影响的研究[J]. 科学养鱼, 2011(1):7-8. |
| [6] | 刘永. 南美白对虾人工育苗技术[J]. 水产养殖, 2002(5):13-16. |
| [7] | 姚新华. 南美白对虾人工育苗技术[J]. 天津水产, 2004(1):45-47. |
| [8] | 周志明. 南美白对虾的生物学特性及淡养技术[J]. 内陆水产, 2001(6):20-22. |
| [9] | 王克行. 虾类健康养殖原理与技术[M]. 北京:科学出版社, 2008:144. |
| [10] | 陈文静, 熊国根, 邓勇辉, 等. 罗氏沼虾生物学研究[J]. 江西水产科技, 2000(4):15-25. |
| [11] | New MB. Farming freshwater prawns:a manual for the culture of the giant river prawn(Macrobrachium rosenbergii)[M]. Rome:Food and Agriculture Organization of the United Nations, 2002. |
| [12] | Strauss WM. Preparation of genomic DNA from mammalian tissue[M]. Current Protocols in Neuroscience / Editorial Board, Jacqu-eline N. Crawley, 2001. |
| [13] | 戴习林, 邓平平, 施永海, 等. 罗氏沼虾SSR标记再开发及其影响因素初探[J]. 生物技术通报, 2012(10):142-149. |
| [14] | Divu D, Karunasagar I, Karunasagar I. Microsatellite DNA markers in the giant freshwater prawn, Macrobrachium rosenbergii:a tool for genetic analysis[J]. Molecular Ecology Resources, 2008, 8(5):1040-1042. |
| [15] | Divu D, Khushiramani R, Malathi S, et al. Isolation, characterization and evaluation of microsatellite DNA markers in giant freshwater prawn Macrobrachium rosenbergii, from South India[J]. Aquaculture, 2008, 284(1-4):281-284. |
| [16] | Bhassu S, See LM, Hassan R, et al. Isolation and characterization of microsatellite loci in the Malaysian giant freshwater prawn, Macrobrachium rosenbergii[J]. Molecular Ecology Resources, 2008, 8(5):983-985. |
| [17] | Bhat S, Patel A, Das P, et al. Isolation and characterization of microsatellite loci in giant freshwater prawn, Macrobrachium rosenbergii[J]. Conserv Genent, 2009, 10(5):1473-1475. |
| [18] | Chareontaweea K, Poompuangb S, Na-nakornb U, et al. Genetic di-versity of hatchery stocks of giant freshwater prawn(Macrobrach-ium rosenbergii)in Thailand[J]. Aquaculture, 2007, 271(1-4):121-129. |
| [19] | Han Z, Wang C, Song X, et al. Characteristics, development and mapping of Gossypium hirsutum derived EST-SSRs in allotetraploid cotton[J]. Theor Appl Genet, 2006, 112(3):430-439. |
| [20] | Yeh FC, Yang R, Boyle T, et al. The user-friendly shareware for population genetic analysis[M]. Edmonton:Biology and Biotechnology Center, University of Alberta, 1997. |
| [21] | Nei M, Li WH. Mathematical model for studying genetic variation in terms of restriction endonucleases[J]. Proc Natl Acad Sci USA, 1979, 76(10):5269-5273. |
| [22] | Bostein D, White RL, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Proc Natl Acad Sci USA, 1980, 32(3):314-331. |
| [23] | Tamura K, Peterson D, Peterson N, et al. MEGA5:Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10):2731-2739. |
| [24] | 张红玉, 何毛贤, 管云雁. 马氏珠母贝红色壳家系不同世代遗传变异的SRAP分析[J]. 水产学报, 2009(5):727-733. |
| [25] | Crawford A, Littlejohn R. The use of DNA markers in deciding conservation priorities in sheep and other livestock[J]. Animal Genetic Resources Information, 1998, 23:21-26. |
| [26] | 田燚. 中国对虾育种方法研究与遗传连锁图谱的构建[D]. 青岛:中国科学院研究生院, 2007. |
| [27] | 高祥刚, 刘红, 徐佳念, 等. 日本沼虾卵黄蛋白原合成部位的初步研究[J]. 生物技术通报, 2006(1):438-444. |
| [28] | 姚俊杰. 罗氏沼虾胚胎营养与形态发生相关性的研究[D]. 上海:华东师范大学, 2006. |
| [29] | 姜乃澄, 卢建平, 袁保京. 罗氏沼虾初级卵母细胞在卵黄形成期超微结构的变化[J]. 东海海洋, 2001, 19(1):35-43. |
| [30] | 王玉凤, 堵南山. 罗氏沼虾卵母细胞细胞器与卵黄发生的关系[J]. 水生生物学报, 1999, 23(1):24-28. |
| [31] | 陈宗永. 罗氏沼虾抱卵亲虾质量差和数量不足的成因与对策探讨[J]. 广西水产科技, 2000(4):26-28. |
| [32] | Brunson MW, Griffin JL. Comparison of rice-crayfish and grain sorghum-crayfish double cropping systems[J]. Aquaculture, 1988, 72(3):265-272. |
| [33] | Kuftina N, Novikov G. Embryonic growth and utilisation of yolk storage proteins during early ontogeny of cod, Gadus morhua, at different temperatures[J]. J Ichthyol(USSR), 1986, 26:75-88. |
| [34] | 戴子坚, 蔡春芳. 卤虫的营养强化及其对罗氏沼虾幼体培育的影响[J]. 水利渔业, 2002, 22(5):1-3. |