转座子是一类有效的转基因工具,它可以将外源基因导入受体基因组,同时还可通过随机的插入诱变使内源基因失活,在插入的DNA中可以加入一些功能元件,从而实现基因捕获(Gene trap)、启动子捕获(Promoter trap)和染色体重组(Chromosome rearrangement)等遗传操作。转座子作为正向遗传学研究的重要工具受到越来越多人的关注[1, 2]。
PiggyBac转座子属于DNA转座子,最初的序列在粉纹夜蛾(Trichoplusia ni)细胞系(IFP2)中分离出来的[3]。PiggyBac转座子长2 472 bp,中间带有一个2.1 kb的转录区域,可编码一个预测大小为64 kD的转座酶;其两端最外侧各有长13 bp的对称的反向末端重复(Inverted terminal repeat,ITR)[4]。PB转座子的转座遵循“切离-粘贴”机制[5]。研究发现,PB特异地插入到TTAA四碱基位点,插入的同时在其两侧形成TTAA四碱基重复。它可以从插入位点处精确地切离,不留下任何痕迹,使插入位点完全回复到插入以前的状态[6]。
PiggyBac转座子具有转座效率高、删除精确、负载量大及半随机插入等优点,已经开发成为转基因昆虫中应用最为广泛的载体之一。它已被证明能够在5个目(鳞翅目、双翅目、膜翅目、鞘翅目和直翅目)的20多种昆虫中转座,其主要应用于转基因和基因插入诱变两方面[7],近年来的研究表明 PB转座系统在哺乳动物中同样也具有高效转座活性以及其他特性[8],使得该系统在基因组研究、基因治疗、干细胞诱导和诱导后分化等研究领域获得了广泛的应用[9, 10, 11, 12, 13, 14]。研究证明PB转座子在斑马鱼原代培养细胞及胚胎中均能进行转座[15],但是,PiggyBac转座子结合基因捕获技术用于斑马鱼突变体筛选及基因功能鉴定则未见报道。本研究以斑马鱼为实验材料,利用PiggyBac转座子自主转座的特性,构建无外源启动子、带有PiggyBac转座子左右臂同时具有剪接受体(Splicing acceptor,SA)以及红色荧光蛋白报告基因(mCherry)的基因捕获载体供体质粒,将供体质粒与编码PiggyBac转座酶mRNA通过显微注射共注射到斑马鱼单细胞期受精卵中,以期获得具有红色荧光蛋白mCherry表达的转基因斑马鱼,为利用PiggyBac转座子进行斑马鱼突变体筛选及基因功能鉴定奠定基础。
1 材料与方法 1.1 材料 1.1.1 实验动物实验所用AB系野生型斑马鱼由清华大学生命科学学院孟安明教授惠赠,本实验室培育繁殖,28.5℃恒温鱼房;光暗时间按14 h∶10 h交替。
1.1.2 主要试剂与仪器编码PiggyBac转座酶的辅助质粒pCMV-hyPBase,PiggyBac转座子供体质粒5'-PTK-3'由英国Sanger研究所构建与保存;编码PiggyBac转座酶的辅助质粒pCS2+-CMV-hyPBase由本实验室保构建并保存;pmCherry-N1质粒由本实验室保存。5'-PTK-3'质粒图谱见图 1-A,pCS2+-CMV-hyPBase体外转录辅助质粒包含有原核SP6启动子、1 804 bp的PiggyBac转座酶基因。

|
| A :5'-PTK-3' ;B :PTK-mCherry 图 1 改造前后的PiggyBac 转座子供体质粒结构示意图 |
E.coli DH5α感受态细胞购自TaKaRa公司;PCR用的DNA聚合酶为TransTaq HiFi,与T4 DNA连接酶均购自Transgene公司;限制性内切酶Bcl I、Pst I、Nco I均购自TaKaRa公司,Acc65I 为Ferme-ntas公司产品;琼脂糖凝胶回收试剂盒为Tiangen公司产品;其他试剂均为国产分析纯。显微注射系统为Eppendorf产品,拉针仪及毛细管玻璃均为NARI-SHIGE产品;倒置荧光显微镜产自奥林巴斯。
1.2 方法 1.2.1 PiggyBac转座子供体质粒PTK-mCherry的构建对5'-PTK-3'进行Bcl I和Nco I双酶切,酶切产物经1%琼脂糖凝胶,割胶并回收纯化目的片段。为了将mCherry基因定向插入5'-PTK-3'载体上,设计1对引物(F:5'-CATGCCATGGTGACGCAAATG-CGCGGTAGG-3',R:5'-GGTGATCATGATTATGAT-CTAGAGTCGCGG-3'),(下划线分别为Nco I和Bcl I酶切位点)。使用F、R引物,以pmCherry-N1质粒为模板,对其进行PCR扩增,反应条件为:94℃预变性5 min;94℃变性30 s,55℃复性30 s,72℃延伸1 min,共35个循环;72℃延伸10 min。扩增产物经Bcl I和Nco I限制性内切酶双酶切,酶切后进行胶回收、纯化,并与经相同的限制性内切酶酶切获得的5'-PTK-3'的双酶切产物大片段连接,转化E.coli DH5α感受态细胞后涂于含氨苄青霉素(Amp+)的LB平板上,挑选阳性克隆,经酶切鉴定后使用T3测序引物对构建好的PTK-mCherry重组质粒进行了测序。具体操作方法参照Sambrook等的《分子克隆实验手册》以及相关产品推荐的使用说明书。
1.2.2 PiggyBac转座酶mRNA的体外转录用限制性内切酶Acc65I酶切辅助质粒pCS2+-CMV-hyPBase,酶切产物经Cycle-Pure kit(OMEGA,USA)纯化,并进行电泳检测。以该回收纯化产物作为模板,采用SP6 mMessage mMachine kit(Ambion,USA)试剂盒进行体外转录合成,该试剂盒可对体外转录出的mRNA进行5'加帽反应,mRNA 5'端的“帽子”结构可使mRNA免遭核酸酶的破坏,且更有利于被蛋白合成的起始因子所识别,从而促进蛋白质的合成。转录出的mRNA经0.8%的琼脂糖凝胶电泳检测质量。
1.2.3 显微注射在进行显微注射的前一天晚上,选择性成熟的亲鱼,按雌雄数量2∶1的比例放入交配盒。第2天注射前抽去交配盒中间隔板,雌雄鱼追尾并产卵,收集、清洗、分选,等待10 min后观察到卵膜隆起即可进行注射。显微注射仪装好显微注射针,吸取发育时期处于1-2细胞期的斑马鱼胚胎,轻轻压挤进入注射皿的凝胶凹槽中,上面再滴几滴胚胎孵育液,保证胚胎湿润,把注射皿置于立体解剖镜下,调节好注射压力,每个胚胎注射PiggyBac转座子供体质粒PTK-mCherry与PiggyBac转座酶mRNA混合液2 nL,PiggyBac转座子供体质粒PTK-mCherry DNA浓度为50 ng/μL,PiggyBac转座酶mRNA的浓度为100 ng/μL。注射后的胚胎28℃培养于胚胎孵育液中,使其恢复发育;对照组斑马鱼受精卵仅注射生理盐水。注射后的斑马鱼受精卵经孵化、培育,荧光显微镜下进行荧光检测。
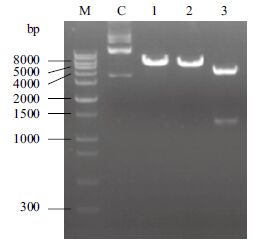
2 结果 2.1 基因捕获载体转座子供体质粒PTK-mCherry的构建与鉴定实验设计构建的基因捕获载体转座子供体质粒PTK-mCherry包含PiggyBac转座子左臂(313 bp)、右臂(241 bp)以及内部核糖体进入位点(IRES)和mCherry基因(图 1-B)。用引物F和R从pmCherry-N1扩增到编码mCherry的DNA片段,经测序鉴定后插入到5'-PTK-3'的Bcl I和Nco I位点,获得PTK-mCherry。酶切方法鉴定获得的PTK-mCherry,琼脂糖凝胶电泳(图 2)显示所获得的酶切产物与预期大小一致,测序结果进一步证实所构建的载体序列正确。
|
| C :control,PTK-mCherry 重组质粒;1 :Pst I 单酶切PTK-mCherry 重组质粒;2 :Xho I 单酶切PTK-mCherry 重组质粒;3 :Pst I 及Xho I 双酶切PTK-mCherry 重组质粒;M :1 kb plus DNA ladder 图 2 PTK-mCherry 重组表达质粒的酶切鉴定 |
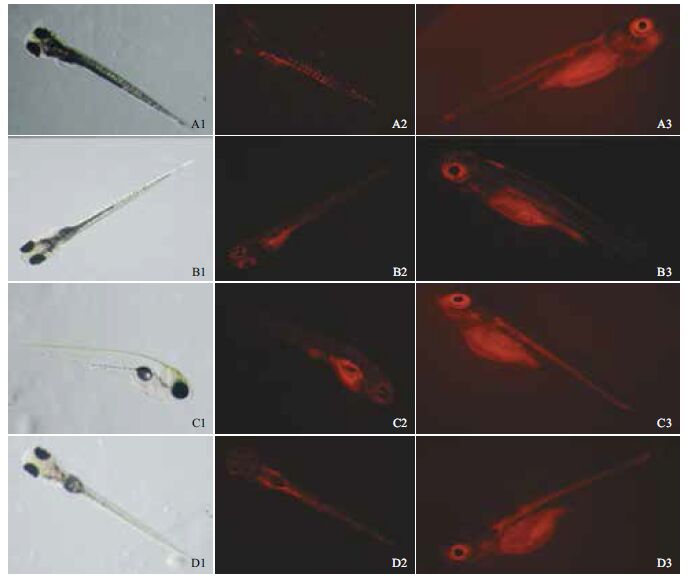
在立体解剖镜下挑选高质量1-2细胞期受精卵,对其注射PiggyBac转座子供体质粒PTK-mCherry与PiggyBac转座酶mRNA混合液。注射后的受精卵置于胚胎孵育液水体中,孵化培育,定期在荧光显微镜下观察胚胎荧光表达情况。注射5天(5 dpf)后的胚胎中首次发现有红色荧光表达,且红色荧光的表达部位在不同斑马鱼中也不尽相同。图 3-A2为一嵌合体鱼(5 dpf),红色荧光主要在背部的肌肉和神经管呈镶嵌分布,在眼睛周围也有表达;而此鱼发育到第15天(15 dpf)时红色荧光在身体几乎所有部位均有表达(图 3-A3)。如图 3-B2所示,此嵌合体鱼(5 dpf)的红色荧光主要分布在腹部的肠道及眼睛周围,发育到第15天时红色荧光仍主要在这两处表达(图 3-B3)。图 3-C2所示为另一不同表型的斑马鱼(5 dpf),在第5天时红色荧光主要在腹部表达,而到第15天时则在整个头部、腹部以及背部脊柱中均有表达(图 3-C3)。图 3-D2为一红色荧光呈泛表达的斑马鱼(5 dpf),在第15天时仍呈泛表达(图 3-D3)。
|
| A、B、C、D 为四种不同表型斑马鱼;A1-D1 :4 种鱼在普通光镜下的图片(5 dpf);A2-D2 :4 种鱼在587 nm 激发光下的图片(5 dpf);A3-D3 :4 种鱼在587 nm 激发光下的图片(15 dpf) 图 3 共注射hyPBase mRNA 及质粒PTK-mCherry 进行基因捕获得到的不同表型Donor 品系红色荧光鱼 |
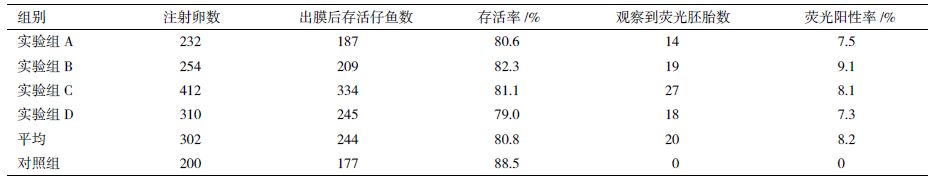
本研究进行了4批斑马鱼受精卵的显微注射,共注射受精卵1 208颗,出膜后存活仔鱼975 尾。转mCherry基因斑马鱼在出膜时的平均存活率和荧光蛋白表达阳性率分别为80.8%和8.2%(表 1)。目前转mCherry基因阳性斑马鱼F0代培育工作正在进行中,mCherry的表达稳定,未发现红色荧光减弱的现象。
斑马鱼具有繁殖力强,性成熟期短,受精卵透明,诱变效率高等特点,适用于大规模的诱变和突变体筛选[16]。迄今为止,已证明在斑马鱼中具有转座活性的转座子包括TC3转座子、Mariner转座子、Tgf2转座子、Tol2转座子、SB(Sleeping beauty)转座子等[17, 18, 19, 20, 21],这些活性转座子的发现极大方便了斑马鱼功能基因的研究。在以上几种转座子中,Tol2转座子和SB转座子系统在斑马鱼转基因及插入突变研究中采用较多。从1998年Kawakami等[22]在硬骨鱼类青鳉中克隆出Tol2转座子开始,科学家们利用这一转座子进行了斑马鱼转基因、增强子捕获及基因捕获等研究,筛选得到了一些和斑马鱼个体发育、神经通路相关的基因[23, 24, 25]。自2003年开始,Stephen[26]研究小组将SB转座子应用到斑马鱼转基因研究中,证明SB在斑马鱼中具有较高的基因转移和表达效率,随后他们采用SB转座子在斑马鱼中开展增强子捕获及转座子介导的插入突变筛选,筛选得到一个斑马鱼胚胎发育相关基因[27]。Clark等[28]运用SB转座子在脊椎动物中开展基因捕获研究,并证明了SB转座子进行基因捕获的有效性。Mátés等[29, 30]对SB转座系统中的转座酶进行了改良,得到了高活性SB转座酶SB100X,成功运用到斑马鱼等脊椎动物的转基因研究中。
与Tol2转座子系统、SB转座子系统等诱变工具相比较,PB转座系统有其独特的优势:可以在生殖细胞中高效转座,PB转座子插入在基因组中分布广泛而且偏好基因区,携带外源基因能力更强,在转座酶存在的情况下可以进行自发转座。目前,PB转座系统已经广泛应用于脊椎动物功能基因的筛选研究,并且在使用过程中不断被进行各种改造,以具备更强的转座能力。Landrette等[31]使用PB转座系统进行体细胞突变筛选,得到了小儿肾癌相关基因 TFE3、TFEB、MITF;Thomas等[32]利用PB转座子插入突变,筛选到了黑素瘤相关基因Arhgef3、Map3k1、Map3k2、Mitf、Rapgef2;Rad等[33]在小鼠中使用PB转座系统进行基因筛选,发现了胰腺癌发生中的原癌基因相互作用网络;Gayle等[34]利用PB转座子插入突变筛选发现了RhoA在人胚胎干细胞分化过程中的作用机制;Sato和Pettitt[35, 36]两个研究小组分别对PB转座系统进行了改造,使之在包括小鼠单倍体胚胎干细胞在内的非人类哺乳动物细胞中能够更加高效进行转座;Cooney等[37]使用腺病毒作为PB转座系统的运载工具,对体细胞进行在体(in vivo)外源基因导入,极大提高了PB转座系统的导入效率。
在基因筛选过程中,使用含有PB转座子的转基因动物,与含有PB转座酶基因的异性同种转基因动物交配,即可改变转座子插入位点[38, 39],极大地方便了大规模功能基因筛选工作的进行。
本实验利用斑马鱼为研究材料,探索PiggyBac转座子结合基因捕获技术用于斑马鱼突变体筛选及基因功能鉴定的可行性,同时尝试在斑马鱼中实现使用PiggyBac转座系统进行在体(in vivo)转座,为斑马鱼突变体的大规模筛选和遗传分析提供新的插入诱变工具。在此工作基础上,一方面,可以建立自己的斑马鱼突变体资源库,另一方面,还可以通过与已知的斑马鱼突变品系进行杂交,研究基因与基因之间的相互作用网络,应用于基因功能的进一步研究。
为了达到上述目的,首先需要筛选得到含有PB转座子的转基因斑马鱼,我们对PiggyBac二元转座系统的供体质粒-转座子载体质粒5'-PTK-3'进行改造,构建了一个无外源启动子,包含PiggyBac转座子的5'-TR及3'-TR、剪接受体(the mouse engrail-ed-2 gene splicing acceptor,En-2SA)、内部核糖体进入位点(Internal ribosome entry site,IRES)、报告基因(mCherry)以及polyA信号序列(bGH polyA)的基因捕获载体质粒:PTK-mCherry。理论上,mCherry将在两种情况下表达:第一,当mCherry插入在5'非翻译区(5' UTR)的一个外显子之后并在该基因的起始密码子之前,而且mCherry的表达方向与该基因一致,则mCherry的第一个ATG将被当作起始密码子被转录;第二,当mCherry插入到一个基因编码区内的某个内含子当中且表达方向与该基因一致,则mCherry将被融合到插入位点前的编码区,转录成融合mRNA。
参考Tol2转座子进行基因捕获时所用的条件[40, 41],使用环状基因捕获载体质粒PTK-mCherry与转座酶hyPBase mRNA共注射导入斑马鱼受精卵中,成功筛选到了红色荧光表达斑马鱼,且红色荧光表达率约为8.2%,与使用Tol2进行基因捕获时效率相似。
本研究筛选到多种红色荧光表型的斑马鱼,且发现随着发育的进行同一条斑马鱼的荧光表达谱也会有变化。推测斑马鱼红色荧光表型不同是因为捕获到了不同的基因,由于红色荧光的表达受捕获到的内源基因启动子控制,反映内源基因的表达,捕获到的基因不同也就造成了不同斑马鱼中红色荧光表达不尽相同。在同一条斑马鱼中可能同时捕获到多个不同基因,这些基因的表达时期不尽相同,即使同一个基因随着发育的进行表达强度和范围也有可能发生改变,这也就解释了为什么随着发育的进行同一条斑马鱼的荧光表达谱也会有变化。
4 结论本研究证明PiggyBac转座子能够在斑马鱼中进行有效转座且捕获到不同基因,因此PiggyBac转座系统在斑马鱼中可以作为一种新的技术手段,有效实现突变体的筛选。
| [1] | Ni J, Clark KJ, Fahrenkrug SC, et al. Transposon tools hopping in vertebrates[J]. Brief Funct Genomic Proteomic, 2008, 7(6):444-453. |
| [2] | Xue YL, Xiao A, Wen L, et al. Generation and characterization of blood vessel specific EGFP transgenic zebrafish via Tol2 transposon mediated enhancer trap screen[J]. Progress in Biochemistry and Biophysics, 2010, 37(7):720-727 . |
| [3] | Fraser MJ, Smith GE, Summers MD. Acquisition of host cell DNA sequences by baculoviruses:relationship between host DNA insertions and FP mutants of Autographa californica and Galleria mellonella nuclear polyhedrosis viruses[J]. J Virol, 1983, 47(2):287-300. |
| [4] | Handler, AM. Use of the PiggyBac transposon for germ-line transformation of insects[J]. Journal of Insect Biochemistry and Molecular Biology, 2002, 32(10):1211-1220. |
| [5] | Lobo N, Li X, Fraser MJ. Transposition of the PiggyBac element in embryos of Drosophila melanogaster, Aedes aegypti and Trichoplusia ni[J]. Mol Gen Genet, 1999, 261(4-5):803-810. |
| [6] | Fraser MJ, Cary L, Boonvisudhi K, et al. Assay for movement of Lepidopteran Transposon IFP2 in insect cells using a baculovirus genome as a target DNA[J]. Virology, 1995, 211(2):397-407. |
| [7] | Wimmer EA. Innovations:Applications of insect transgenesis[J]. Nat Rev Genet, 2003, 4(3):225-232. |
| [8] | Ding S, Wu XH, Li G, et al. Efficient transposition of the PiggyBac(PB)transposon in mammalian cells and mice[J]. Cell, 2005, 122(3):473-483. |
| [9] | Wu SCY, Meir YJJ, Coates CJ, et al. PiggyBac is a flexible and highly active transposon as compared to Sleeping Beauty, Tol2, and Mos1 in mammalian cells[J]. Proc Natl Acad Sci USA, 2006, 103(41):15008-15013. |
| [10] | Woltjen K, Michael IP, Mohseni P, et al. PiggyBac transposition reprograms fibroblasts to induced pluripotent stem cells[J]. Nature, 2009, 458(7239):766-770. |
| [11] | Nakanishi H, Higuchi Y, Kawakami S, et al. PiggyBac transposon-mediated long-term gene expression in mice[J]. Mol Ther, 2010, 18(4):707-714. |
| [12] | 杨欢欢, 魏峰, 刘全. PiggyBac转座子应用研究进展[J]. 动物医学进展, 2010, 31(12):91-94. |
| [13] | 刘琳, 张美丽, 黄粤. DNA转座子在小鼠基因功能研究中的应用[J]. 遗传, 2011, 33(5):485-493. |
| [14] | 钱秋杰, 车家倩, 叶露鹏. PiggyBac转座系统的功能改进及在哺乳动物中的应用[J]. 遗传, 2014, 36(10):965-973. |
| [15] | Lobo N, Fraser TS, Adams JA, et al. Interplasmid transposition demonstrates PiggyBac mobility in vertebrate species[J]. Genetica, 2006, 128(13):347-357. |
| [16] | 桂建芳. 分子发育生物学研究的理想模式──斑马鱼[J]. 中国生物工程杂志, 1995, 15(3):30-33. |
| [17] | Balciunas D, Davidson AE, Sivasubbu S, et al. Enhancer trapping in zebrafish using the Sleeping Beauty transposon[J]. BMC Genomics, 2004, 5:62. |
| [18] | Raz E, Luenen HG, Schaerringer B, et al. Transposition of the nematode Caenorhabditis elegans Tc3 element in the zebrafish Danio rerio[J]. Curr Biol, 1998 , 8(2):82-88. |
| [19] | 吴芳, 叶星, 邹曙明, 等. Tgf2 转座系统在转RFP基因斑马鱼上的应用[J]. 中国水产科学, 2014, 21(4):647-654. |
| [20] | Fadool JM, Hartl DL, Dowling JE, et al. Transposition of the marin-er element from Drosophila mauritiana in zebrafish[J]. Proc Natl Acad Sci USA, 1998, 95(9):5182-5186. |
| [21] | Kawakami K, Koga A, Hori H, et al. Excision of the tol2 transpos-able element of the medaka fish, Oryzias latipes, in zebrafish, Danio rerio[J]. Gene, 1998, 225(1-2):17-22. |
| [22] | Kawakami K, Takeda H, Kawakami N, et al. A transposon-mediated gene trap approach identifies developmentally regulated genes in zebrafish[J]. Dev Cell, 2004, 7(1):133-144. |
| [23] | Parinov S, Kondrichin I, Korzh V, et al. Tol2 transposon-mediated enhancer trap to identify developmentally regulated zebrafish genes in vivo[J]. Dev Dyn, 2004, 231(2):449-459. |
| [24] | Kotani T, Kawakami K. Misty somites, a maternal effect gene iden-tified by transposon-mediated insertional mutagenesis in zebrafish that is essential for the somite boundary maintenance[J]. Dev Biol, 2008, 316(2):383-396. |
| [25] | Nagayoshi S, Hayashi E, Abe G, et al. Insertional mutagenesis by the Tol2 transposon-mediated enhancer trap approach generated mutations in two developmental genes:tcf7 and synembryn-like[J]. Development, 2008, 135(1):159-169. |
| [26] | Davidson AE, Balciunas D, Mohn D, et al. Efficient gene delivery and gene expression in zebrafish using the Sleeping Beauty transposon[J]. Dev Biol, 2003, 263(2):191-202. |
| [27] | Sivasubbu S, Balciunas D, Davidson AE, et al. Gene-breaking tran-sposon mutagenesis reveals an essential role for histone H2afza in zebrafish larval development[J]. Mech Dev, 2006, 123(7):513-529. |
| [28] | Clark KJ, Geurts AM, Bell JB, et al. Transposon vectors for gene-trap insertional mutagenesis in vertebrates[J]. Genesis, 2004, 39(4):225-233. |
| [29] | Mátés L, Chuah MK, Belay E, et al. Molecular evolution of a novel hyperactive Sleeping Beauty transposase enables robust stable gene transfer in vertebrates[J]. Nat Genet, 2009, 41(6):753-761. |
| [30] | Newman M, Lardelli M. A hyperactive sleeping beauty transposase enhances transgenesis in zebrafish embryos[J]. BMC Res Notes, 2010, 282(3):121-125. |
| [31] | Landrette SF, Cornett JC, Ni TK, et al. PiggyBac transposon somatic mutagenesis with an activated reporter and tracker(PB-SMART)for genetic screens in mice[J]. PLoS One, 2011, 6(10):1-12. |
| [32] | Ni TK, Landrette SF, Bjornson RD, et al. Low-copy piggyBac tran-sposon mutagenesis in mice identifies genes driving melanoma[J]. Proc Natl Acad Sci USA, 2013, 110(38):3640-3649. |
| [33] | Rad R, Ten Hoeve J, Wessels L, et al. A conditional piggyBac tran-sposition system for genetic screening in mice identifies oncogenic networks in pancreatic cancer[J]. Nat Genet, 2015, 47(1):47-56. |
| [34] | Gayle S, Pan Y, Landrette S, et al. PiggyBac insertional mutagenesis screen identifies a role for nuclear RHOA in human ES Cell differentiation[J]. Stem Cell Reports, 2015, 4(1):926-938. |
| [35] | Sato M. A combination of targeted toxin technology and the piggyBac-mediated gene transfer system enables efficient isolation of stable transfectants in nonhuman mammalian cells[J]. Biotechnol J, 2014, 10(1):143-153. |
| [36] | Pettitt SJ, Tan EP, Yusa KH. PiggyBac transposon-based insertional mutagenesis in mouse haploid embryonic stem cells[J]. Methods Mol Biol, 2015, 1239(1):15-28. |
| [37] | Cooney AL, Singh BK, Sinn PL. Hybrid Non-viral/Viral vector systems for improved piggyBac DNA transposon in vivo delivery[J]. Mol Ther, 2015, 23(4):667-674. |
| [38] | Bonin CP, Mann RS. A piggyBac transposon gene trap for the analysis of gene expression and function in Drosophila[J]. Genetics, 2004, 167(4):1801-1811. |
| [39] | Furushima K, Jang CW, Chen DW, et al. Insertional mutagenesis by a hybrid piggyBac and Sleeping Beauty transposon in the rat[J]. Genetics, 2012, 192(4):1235-1248. |
| [40] | Balciunas D, Ekker SC. Trapping fish genes with transposons[J]. Zebrafish, 2005, 1(4):335-341. |
| [41] | Kotani T, Nagayoshi S, Urasaki A, et al. Transposon-mediated gene trapping in zebrafish[J]. Methods, 2010, 39(3):199-206. |