纤维二糖酶又称为β-葡萄糖苷酶[1],能够水解纤维二糖产生两分子的葡萄糖[2],并能极大地促进内切葡聚糖酶和外切葡聚糖酶的水解作用,加快纤维素酶系的整体水解速度,促使纤维素的水解更完全[3]。现在对真菌中纤维二糖酶产生菌研究较多的是丝状真菌,主要为曲霉属和木霉属[4],而细菌中研究较多的是芽孢杆菌属[5, 6]。
黑曲霉(Aspergillus niger)是纤维二糖酶的高产菌株[7, 8],但是关于黑曲霉纤维二糖酶基因的研究报道仍然较少[9, 10]。2000年,美国Dan等[11]研究者在GenBank上登录了第一个黑曲霉纤维二糖酶的全基因序列。2007年,荷兰工业化学公司首次完成了对一株黑曲霉的全基因组测序,该黑曲霉基因组有3 390万个碱基对,并且构成超过14 000个独特的基因,其中编码纤维二糖酶的基因已被完全确定[12]。国内2006年首次出现关于黑曲霉纤维二糖酶基因克隆研究的报道。
本研究通过参照GenBank上已发表的黑曲霉纤维二糖酶基因序列(登录号:AJ132386)设计引物,实验菌株是经过紫外诱变筛选获得的高产纤维素酶黑曲霉菌,以此菌株总RNA为模板克隆了纤维二糖酶的基因,进行基因序列测序。国内外对该基因生物学信息还未见报道,因此本研究对该基因进行初步的生物信息学分析,以更好地了解纤维二糖酶的功能性质,并且实现该基因在大肠杆菌BL21中胞内融合表达。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒黑曲霉(Aspergillus niger)C112[13],保存于中南林业科技大学发酵工程实验室。大肠杆菌DH5α、BL21(DE3),pEASY-T1购自北京全式金生物技术有限公司。
1.1.2 酶与试剂总RNA提取试剂(Trigol)、焦碳酸二乙酯(DEPC),北京鼎国昌盛生物技术有限公司;普通琼脂糖凝胶DNA回收试剂盒、质粒小提试剂盒,北京天根公司;M-MLV逆转录试剂、EasyTaq PCR SuperMix,北京全式金生物公司;其它试剂均为国产分析纯。
1.1.3 培养基PDA培养基:马铃薯200 g/L(将马铃薯切成方块,置于1 000 mL水中煮沸,过滤收集液体,即得),葡萄糖20 g/L,琼脂粉15 g/L。纤维素酶诱导培养基:蛋白胨10 g/L,麦芽糖4 g/L,MgSO4 0.5 g/L,KH2PO4 1 g/L。LB液体培养基:胰蛋白胨10 g,酵母提取物5 g,NaCl 10 g,加入去离子水950 mL,用10 mol/L NaOH调pH值至7.0,加去离子水定容1 L。LB固体培养基:配方和液体培养基一样,并加入15 g琼脂粉。所配的培养基均在1×105 Pa灭菌20 min,4℃保存备用。
1.2 方法 1.2.1 引物设计参照GenBank上已发表的黑曲霉纤维二糖酶(bgl1)基因序列(GenBank登录号为AJ132386),利用Primer premier 5.0设计引物,引物序列为上游引物P1:5'-CGGAATTCATGAGGTTCACTTTGATCGAGGCGG-3',下游引物P2:5'-ATGCGGCCGC TTAGTGAACAGTAGGCAGAGACG-3',由苏州金唯智生物科技有限公司合成。为了便于与载体连接,在引物中加入酶识别位点,bgl1的引物中加入了EcoR I和Not I位点(引物中的划线部分)。
1.2.2 黑曲霉C112的复苏和保藏从真空冻干菌管中用接种环刮下少量细胞接于PD液体培养基,30℃培养24 h,用接种环从培养液中蘸取少量菌液画线于PDA斜面,30℃静置培养到长出孢子,于 4℃保存。
1.2.3 黑曲霉C112总RNA提取及cDNA的合成总RNA的提取方法参照北京鼎国昌盛生物技术有限公司的Trigol试剂盒的说明。cDNA的合成按照北京全式金生物公司的RT-PCR试剂盒说明书操作。
1.2.4 纤维二糖酶基因的测序及系统发生分析将PCR产物回收纯化后,克隆到pEASY-T1载体中,转化大肠杆菌DH5α,经蓝白斑筛选重组子,挑取经菌落PCR验证的阳性克隆子进行序列测定。利用Blast工具进行序列相似性分析,选取部分同源序列,利用Clustal X 软件包进行序列同源进化比对,形成一个多重序列匹配排列矩阵,构建系统发育进化树。
1.2.5 测序及序列的生物信息学分析测序结果采用在线软件对纤维二糖酶基因氨基酸序列进行物理性质(http://web.expasy.org/protparam/)、二级结构(http://npsa-pbil.Ibcp.fr/cgi-bin/npsa-auomat.pl/page=/NPSA/npsa-hnn.html)、三级结构(http://swissmodel.expasy.org/SWISS-MODEL.html)及信号肽(http://www.cbs.dtu.dk/services/SignalP-3.0)等信息的预测分析,氨基酸序列同源比对用Blast在线工具和DNAMAN8.0软件完成。
1.2.6 纤维二糖酶的诱导表达验证将阳性重组子接种到新鲜含有相应抗体的液体LB培养基中,37℃、200 r/min过夜培养,以1%的比例接种到30 mL抗性LB液体培养基,37℃、200 r/min培养细菌生长到对数生长期后(OD600=0.4-0.5),加入终浓度为1 mmol/L的IPTG诱导目的基因表达,30℃诱导3 h,取1 mL菌液,4℃、12 000 r/min离心2 min,收集菌体,用PBS缓冲液(pH7.0)洗涤沉淀,并溶于500 µL相同缓冲液中。超声破碎细胞:功率200 W,超声2 s间隔4 s,共60次。4℃、12 000 r/min离心30 min,得到上清液即粗酶液。取适量菌液做诱导表达效果的检测,实验以未加IPTG诱导为对照,并运用Western blotting技术对重组蛋白进行验证。
1.2.7 纤维二糖酶的酶活检测采用国际通用的方法测定纤维二糖酶的活力[14]:取0.1 mL稀释了适当倍数的粗酶液(对照管不加),加入1 mL pH4.8的0.05 mol/L柠檬酸缓冲溶液,于50℃水浴预热10 min;加入已预热10 min的0.9 mL 5 mmol/L pNPG 溶液,计时,10 min后立即加入1 mL 1 mol/L Na2CO3溶液终止反应;加入蒸馏水定容到25 mL;室温放置5 min,于410 nm处测吸光度值A。在上述条件下,1 mL酶液1 min水解产生1 µmol 的对硝基苯酚的酶活力,定义为一个酶活单位(U)。
1.2.8 重组酶学性质的初步研究 1.2.8.1 温度对酶活力的影响粗酶液在不同温度下(30、40、50、60、70、80℃)保温30 min后,按照1.2.7中的方法测定酶活。
1.2.8.2 pH对酶活力的影响粗酶液分别在不同pH值的醋酸缓冲液条件下(pH4.0、4.5、5.0、5.5、6.0、6.5、7.0)室温处理30 min后,按照1.2.7中的方法测定酶活。
2 结果 2.1 纤维二糖酶基因的克隆利用所设计的引物,以黑曲霉菌株的cDNA为模板进行PCR扩增,电泳结果(图 1)显示扩增片段约为2 900 bp左右,与预期相符。

|
| 图 1 纤维二糖酶基因的PCR扩增电泳图 |
纤维二糖酶基因片段大小为2 934 bp,将该基因命名为bgl,GenBank数据库登录号为KP307454。其中A:644个、T:666个、G:834个、C:790个,G+C含量为55.35%。该基因编码860个氨基酸和一个终止密码子,以ATG为起始密码子,含有非编码序列。BLAST序列分析表明,该片段与数据库的Aspergillus niger(GenBank登录号JX982101.1)核苷酸序列有着99%的相似性(表 1),由此可以确定PCR扩增产物确实是bgl基因。
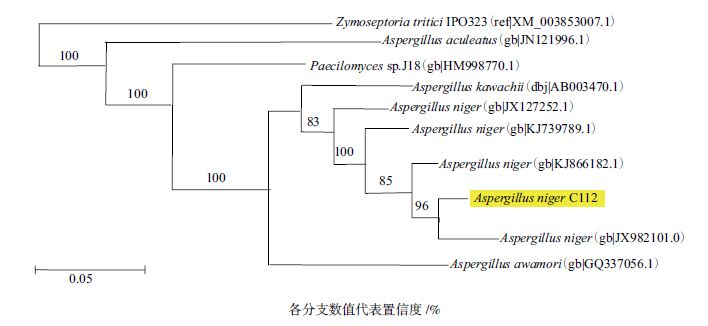
Blastp同源性检索表明,黑曲霉C112菌株纤维二糖酶基因与黑曲霉(GenBank登录号:JX982101.1)的bgl1具有很高的同源性。采用Mega 4.0软件(Neighbor-Joining法)对已报道的黑曲霉纤维二糖酶氨基酸序列进行系统发生分析(图 2),该基因与其他bgl基因聚类在一起,进一步表明克隆获得的基因确实为bgl基因。
|
| 图 2 纤维二糖酶基因的系统发生进化树 |
采用在线软件对纤维二糖酶的氨基酸序列进行分析(http://web.expasy.org/protparam/),结果显示,纤维二糖酶的蛋白质理论分子量为93.33 kD;预测等电点为4.70;分子式为C4153H6311N1117O1300S20;不稳定系数为30.65,是物理性质稳定的蛋白质;疏水性分析的GRAVY值为-0.357,该蛋白为亲水蛋白。酸性氨基酸残基总数(Asp+Glu)为99,碱性氨基酸(Arg+Lys)为64,表明其为酸性蛋白质。20种氨基酸中甘氨酸(Gly)含量最高,为10.7%;丙氨酸(Ala)次之,为9.07%。
利用SignalP-NN软件分析发现其1-20位氨基酸为该蛋白信号肽序列(图 3)。C score:酶切位点:数值越高说明其作为酶切位点的可能性越高;S score:单氨基酸数值:越高说明该氨基酸作为信号肽部分的可能性越高;Y score:C值和S值的派生值,更准确地确定酶切位点,数值最高处为酶切位点。

|
| 图 3 Bgl蛋白信号肽分析 |
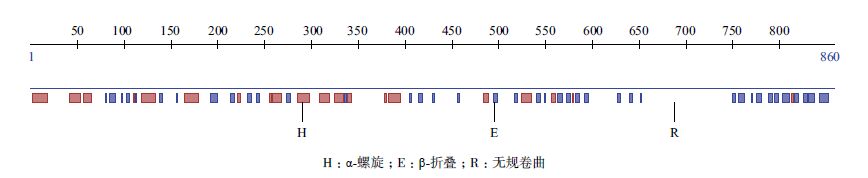
通过国际蛋白质生物学和化学研究所(PBIL)在线分析网站对该基因进行蛋白质二级结构预测。分析结果(图 4)表明,该蛋白质富含无规卷曲(Random coil),含量高达60.35%;其次为α-螺旋(Alpha helix),含量为22.09%;而β-折叠(Extended strand)含量最低,只有17.56%。
|
| 图 4 纤维二糖酶二级结构预测 |
利用自动比较蛋白建模服务器SWISS-MODEL,与蛋白质数据库中已知蛋白质的三级结构进行同源建模,从而预测该蛋白的三级结构模型(图 5)。该蛋白的三级结构与Aspergillus aculeatus的纤维二糖酶三级结构相似,相似度为83.10%,结果较可靠。

|
| 图 5 纤维二糖酶的三维结构预测图 |
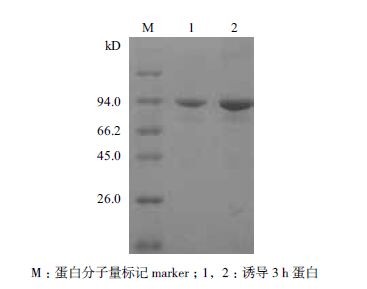
经过优化条件,最终获得目的蛋白最优诱导表达条件为:30℃下用1 mmol/L的IPTG诱导表达3 h,SDS-PAGE电泳(图 6)显示,表达产物的分子量为93.3 kD。通过Western blotting对重组蛋白进行验证,获得目的条带(图 7),再次说明表达的重组蛋白是纤维素酶Bgl。
|
| 图 6 重组质粒蛋白表达产物的SDS-PAGE鉴定 |

|
| 图 7 重组蛋白免疫印迹 |
挑选酶活最高的重组酵母菌再进行诱导培养,每隔24 h测定一次酶活,其产酶曲线见图 8。其纤维二糖酶活在连续诱导的前72 h一直呈增长趋势,72 h以后便不再增长,呈缓慢下降趋势。

|
| 图 8 重组纤维二糖酶酶活测定曲线 |
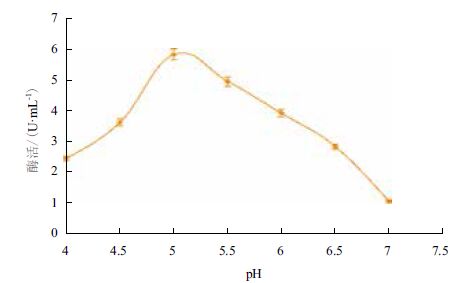
重组后的菌株诱导72 h后,在不同温度反应条件下测得酶活力。以温度值为横坐标,酶活力为纵坐标,作出温度曲线(图 9),可以看出,纤维二糖酶的最适温度为50℃。在最适温度条件下(图 10),分别在pH4.0-7.0条件下测定重组纤维二糖酶的活力,该酶最适pH为5.0。

|
| 图 9 温度对重组纤维二糖酶的影响 |
|
| 图 10 pH值对重组纤维二糖酶的影响 |
纤维二糖酶是纤维素彻底降解为单糖的一个瓶颈[15],采用基因工程与蛋白质工程手段获得酶活较高的纤维二糖酶已经成为研究热点[16]。国外许多研究机构正致力于纤维二糖酶的分子生物学研究,从基础领域研究酶的催化机制及表达调控机制,以期更好地改善纤维素酶的催化效率[17]。周晓明等[18]克隆了bgl基因,与黑曲霉已知序列同源性达到了99%;王冰冰等[19]克隆了纤维二糖酶基因,并且实现了在里氏木霉中的表达;朱龙宝等[20]在毕赤酵母中成功分泌表达了纤维二糖酶,蛋白酶活力达到38 U/mL。由于大肠杆菌表达系统遗传背景清楚、操作手段较为成熟,并且大肠杆菌繁殖快、营养要求低,表达的外源蛋白稳定、易纯化等特点,因此大肠杆菌在基因表达技术中应用广泛[21]。本研究为了获取高产量的重组纤维素酶,并进一步研究其酶学性质、功能与结构,将克隆到的纤维素酶基因与表达载体pEASY-T1构建重组表达载体pEASYT-bgl,并转化大肠杆菌BL21得到了重组工程菌,实现了bgl基因的胞内融合表达。
1974年,Bause等[22]提出纤维二糖酶活性位点中的Asp是糖苷水解酶家族3的高度保守氨基酸,作为该酶的催化亲和试剂,此位点以及周围的几个氨基酸序列GFVMSDW等在糖苷水解酶家族3中也是高度保守的。大多数微生物所产的β-葡萄糖苷酶也属于家族3的成员,糖苷水解酶家族3有A区和B区两个域构成,B区包括SDW序列,内有活性为点Asp(D)残基。在分子水平上,糖苷水解酶家族3的编码基因有5个典型的区域构成:N端区、N端催化区、非同源区、C端未知功能区和C端残基[23]。本研究所得到的bgl的氨基酸序列也具有文献提到这一特征,因此它属于糖苷水解酶家族3的成员。
本研究构建的工程菌酶活低于原菌株,可能因为构建的重组菌的表达产物为胞内酶,而且本研究仅在摇瓶做了初步实验,未对IPTG诱导浓度、金属离子、温度、pH值等影响目的基因表达的条件进行优化,也是影响重组蛋白表达和酶活力的原因之一。今后有必要对重组菌株的发酵条件进一步优化,为该基因的工业应用奠定基础。
4 结论本研究通过RT-PCR方法从菌株黑曲霉C112克隆了纤维素酶基因bgl,并测得了其核酸序列(GenBank登录号KP307454)。序列分析表明bgl基因大小为2 934 bp,含有非编码序列,编码具有860个氨基酸分子的成熟纤维素酶,分子量大约为93.3 kD;二级结构以α-螺旋、β-折叠和无规则卷曲为结构元件。该基因成功在大肠杆菌中进行了胞内融合表达,重组蛋白pNPG酶活最大为5.847 U/mL,最适反应温度为50℃,最适pH值为5.0;并且初步探索了诱导纤维素酶基因表达的条件,用1 mmol/L 的IPTG诱导重组菌3 h后能够检测到目的蛋白。
| [1] | Foreman P, Brown D, Dankmeyer L, et al. Transcriptional regulation of biomass-degrading enzymes in the filamentous fungus Trichoderma reesei[J]. The Journal of Biological Chemistry, 2003, 278:31988-31997. |
| [2] | Watanabe H, Tokuda G. Animal cellulases[J]. Cellular and Molecular Life Sciences, 2001, 58(9):1167-1178. |
| [3] | Dekker RFH. Kinetic, inhibition and stability properties of acomme-rcial β-glucosidase(cellobiase)preparation from Aspergillus niger and its suitability in the hydrolysis of lignocellulose[J]. Biotechnology and Bioengineering, 1986, 28(9):1438-1442. |
| [4] | Nina A, Marja I, Anu S, et al. ACEI of Trichoderma reesei is a repressor of cellulase and xylanase expression[J]. Applied and Environmental Microbiology, 2003, 69(1):56-65. |
| [5] | Imjongjirak C, Amparyup P, Sittipraneed S. Cloning, genomic organization and expression of two glycosyl hydrolase family 10(GHF10)genes from golden apple snail(Pomacea canaliculata)[J]. Food Technology, 2008, 19(3):224-236. |
| [6] | 钟斐, 叶秀云, 李仁宽, 等. 长梗木霉β-葡萄糖苷酶基因的克隆及表达[J]. 微生物学通报, 2012, 39(8):1102-1111. |
| [7] | Iwashita K, Nagahara T, Kimura H, et al. The bglA gene of aspergillus kawachii encodes both extracellular and cell wall-bound β-glucosidases[J]. Applied and Environmental Microbiology. 1999, 65(12):5546-5553. |
| [8] | Dharmawardhana DP, Ellis BE, Carlson JE. cDNA cloning and heterologous expression of coniferin β-glucosidase[J]. Plant Molecular Biology, 1999, 40(8):365-372. |
| [9] | Shen XL, Xia LM. Production and immobilization of cellobiase from Aspergillus niger ZU-07[J]. Process Biochemistry, 2004, 39(11):1363-1367. |
| [10] | Seidle HF, Marten I, Shoseyov O, et al. Physical and kinetic properties of the family 3 β-glucosidase from Aspergillus niger which is important for cellulose breakdown[J]. The Protein Journal, 2004, 23(1):158-163. |
| [11] | Dan S, Marton I, Dekel M, et al. Cloning, expression, characterization and nucleophile identification of family 3, Aspergillus niger β-glucosidase[J]. The Journal of Biologicai Chemistry, 2000, 275(7):4973-4980. |
| [12] | Pel HJ, de Winde JH, Archer DB, et al. Genome sequencing and analysis of the versatile cell factory Aspergillus niger CBS 513. 88[J]. Biotechnol, 2007, 25(2):221-231. |
| [13] | 先天敏, 陈介南, 张林. 黑曲霉改良株C112产β-葡萄糖苷酶的诱导及条件优化[J]. 中南林业科技大学学报, 2013, 33(11):154-161. |
| [14] | Takashima S, Nakamura A, Hidaka M, et al. Molecular cloningand expression of the novel fungal β-glucosidase genes from Hu-micola griseaand Trichoderma reesei[J]. J Biochem, 1999, 125(4):728-736. |
| [15] | Hong MR, Kim YS, Park CS, et al. Characterization of a recombin-ant β-glucosidase from the thermophilic bacterium Caldicellulosir-uptor saccharolyticus[J]. Journal of Bioscience and Bioenginee-ring, 2009, 108(1):36-40. |
| [16] | 孟宪文, 宋小红, 陈历俊, 等. β-葡萄糖苷酶的研究进展[J]. 中国乳业, 2009(10):42-44. |
| [17] | 李泰明, 庄舰峰, Jean Louis Didier MEKOO, 等. 黑曲霉β-葡萄糖苷酶基因在大肠杆菌中的克隆与表达[J]. 安徽医药, 2012, 16(5):576-578. |
| [18] | 周晓明, 莫隽颖, 陶冶, 等. 黑曲霉β-葡萄糖苷酶基因在大肠杆菌中的克隆与表达[J]. 化学与生物工程, 2010, 27(6):50-53. |
| [19] | 王冰冰, 夏黎明, 杜风光. 黑曲霉纤维二糖酶基因的克隆及其在里氏木霉中的表达[J]. 化工学报, 2011, 62(2):452-457. |
| [20] | 朱龙宝, 汤斌, 陶玉贵, 等. 黑曲霉β-葡萄糖苷酶基因克隆及在毕赤酵母中分泌表达[J]. 食品与生物技术学报, 2012, 31(9):973-977. |
| [21] | 王正祥, 刘吉泉, 诸葛健, 等. 微生物酶的分子改性和人工进化的研究进展[J]. 生物工程学报, 2000, 16:301-303. |
| [22] | Bause E, Legler G. Isolation and amino acid sequence of a hexadecapeptide from the active site of beta-glucosidase A3 from Aspergillus wentii[J]. Hoppe-Seyler’s Zeitschrift fur Physiologische Chemie, 1974, 355(4):438-442. |
| [23] | Li YK, Chir FY. Catalytic mechanism of a family 3 β-glucosidase and mutageneais study on residue Asp-247[J]. The Biochemical Journal, 2001, 355(3):835-840. |





