组蛋白修饰、染色体重塑、DNA甲基化和小RNA介导的mRNA降解等,这些表观遗传机制在调控相关基因的表达过程中具有很重要的作用和功能,而且是植物生长发育和对环境的适应过程中必不可少的。其中DNA甲基化不仅在表观途径中调控着相关基因的表达,而且还会影响基因组的稳定性[1, 2]。在原核生物中,DNA的甲基化是广泛存在的,而在植物中根据甲基化模式、甲基化的分子机制和去甲基化酶的分类,DNA甲基化具有各种不同的特性[3]。哺乳动物中的DNA甲基化大部分发生在CpG位点,而且是由DNA甲基转移酶DNMT1(DNA methyltransferase)和它的同源体催化的。但是,在植物中,DNA的甲基化不仅发生在CpG位点,还发生在CHG和CHH位点,其中H代表A、T或者C。例如,在拟南芥中,CpG、CHG和CHH位点的甲基化分别是MET1(DNA methyltransferase 1)、CMT-3(chromomethylase 3)和DRMs(domains rearranged methyltransferases)这3种酶催化产生的[4]。
含有JmjC结构域的蛋白在动物中研究得比较多,主要起着组蛋白/DNA去甲基化的作用,在植物中的研究主要集中在拟南芥和水稻上,在番茄中还很少见相关基因的研究报道。动物中JmjC蛋白分为JHDM1、PHF2/PHF8、JARID、JHDM3/JMJD2、UTX/UTY、JHDM2和JmjC domain only 7个亚家族[5]。JmjC domain only亚家族只含有JmjC结构域,其他亚家族不仅含有JmjC结构域,还含有ZF-C2H2、F-Box、PHD、TPR等结构域形成特定的酶活性。不同亚家族的JmjC蛋白作用位点存在特异性。全基因组的系统发育分析表明,水稻和拟南芥中分别有20个和21个编码JmjC蛋白的基因[6],其中拟南芥中已报道8个,具有广泛的生物学功能[7]。通过番茄的基因组序列分析,我们发现番茄中共有5个编码JmjC蛋白的基因,其功能还都不清楚。前期的研究结果表明JMJ524属于JmjC domain only亚家族,它的氨基酸序列与其他物种中具有较高的同源性,广泛存在于很多物种中,且基因受到赤霉素(GA)的诱导表达。RNAi抑制表达JMJ524导致番茄的严重矮化,叶片变小皱缩,节间变短,生殖生长期植株皱缩矮化更加严重,而且这种矮化是GA不敏感的。通过对矮化植株和野生型对照进行RNA-seq转录组分析,我们发现两个缺少N端的DELLA结构域的类DELLA蛋白SlGLD1(GRAS protein Lacking the DELLA domain)和SlGLD2基因在转基因植株中有较高的表达[8]。番茄中至少有54个GRAS基因[9],但是目前只有很少的GRAS基因的功能被鉴定出来,最近的研究表明这类基因在植物生长发育[10]和环境信号传导[11]过程中具有很重要的作用。于是分别克隆了SlGLD1和SlGLD2这两个基因并且通过转基因的方法将其在番茄中超量表达,结果表明超量表达SlGLD1也导致番茄的矮化,而SlGLD2没有明显的矮化表型[8]。但是JMJ524是如何通过调控SlGLD1调节植物的生长发育等分子机理尚不清楚。本研究在前期结果的基础上,分离和分析SlGLD1基因的启动子序列,通过生物信息学的方法对基因的全长进行分析,利用已有的JMJ524抑制表达植株,探索JMJ524是否是通过调控SlGLD1基因的甲基化程度来调控基因的表达。
1 材料与方法 1.1 材料普通番茄番茄M82(Solanum lycopersicum cv. M82)和JMJ524-RNAi转M82抑制表达株系种植于西南大学科技楼温室。
1.2 方法 1.2.1 SlGLD1 DNA甲基化位点分析和BSP(Bisulfite sequencing PCR)引物设计根据前期我们克隆得到的SlGLD1(Solyc10g086380.1.1)的基因组序列,根据序列比对,在SGN(https://solgenomics.net)中查找其启动子序列。得到-2 011 bp的SlGLD1启动子序列,并用在线软件(http://www.bioinformatics.org/sms2/cpg_islands.html)预测GC岛位置,设计引物运用在线BSP引物设计软件(http://www.urogene.org/cgi-bin/methprimer/methprimer.cgi),引物由华大基因科技服务有限公司合成,引物序列分别为SlGLD1BSP F1:5’-GTTGGAGAATTTAGAGTGGATTAAG-3’,SlGLD1BSP R1:5’-TTTCCATTTATATACAAAACCAACAAC-3’;SlGLD1BSP F2:5’-TGGTTAGTATTTTTATAGGTTTTTATTTTG-3’,SlGLD1BSP R2:5’-CCAACACTTTCTCAATTACTCCTAAA-3’。SlGLD1启动子序列和编码序列在番茄各个部位的甲基化分析使用番茄的表观遗传数据库[12](http://ted.bti.cornell.edu/epigenome/)。
1.2.2 DNA的提取和重亚硫酸氢盐修饰处理用CTAB小量法[13]提取番茄矮化植株(Ri14)和野生型(M82)叶片的DNA,DNA的重亚硫酸氢盐修饰使用QIAGEN公司的EpiTect® Bisulfite试剂盒,操作步骤和实验过程参照EpiTect® Bisulfite操作手册(2009版)。
1.2.3 BSP扩增和甲基化分析处理完成后,分别用引物(SlGLD1BSP F1+SlGLD1BSP R1;SlGLD1BSP F2+SlGLD1BSP R2)对重亚硫酸氢盐处理之后的DNA为模板进行PCR扩增,PCR程序如下:PCR反应体系20 μL:10×PCR Buffer 2.0 μL,dNTP(10 mmol/μL)0.4 μL,正反引物(100 μmol/L)各0.4 μL,DNA模板(300-500 ng/μL)1.0 μL,Taq酶(5 U/μL)0.2 μL,ddH2O补足20 μL。Buffer、dNTP和Taq酶购自Transgene公司。PCR反应程序:94℃预变性3 min;94℃变性30 s,55℃退火30 s,72℃复性30 s,35个循环;72℃延伸10 min,16℃保存1 s。
PCR产物经1%的琼脂糖凝胶电泳后,用E.Z.N.A®DNA凝胶回收试剂盒(Omega公司,美国)回收。回收纯化的PCR片段与pMD18-T载体(宝生物工程大连有限公司)进行连接反应,体系中PCR插入片段与pMD18-T载体的摩尔比为3:1连接,反应总体积是10 μL,16℃过夜连接。取10 μL连接产物,热激法参照文献[14]转化大肠杆菌DH5α,在含有100 mg/L氨苄青霉素的LB固体平板中筛选阳性克隆,每个扩增产物挑取18-24个单克隆进行测序(由华大基因公司完成)。分析得到目的片段后,将序列和目标序列(未经过重亚硫酸盐处理的DNA序列)输入QUMA(http://quma.cdb.riken.jp/)和Kism-eth(http://katahdin.mssm.edu/kismeth/revpage.pl)甲基化统计软件计算甲基化程度。
2 结果 2.1 SlGLD1的启动子和BSP甲基化位点分析利用番茄基因组序列,分离得到SlGLD1的-2 011 bp的启动子序列,我们发现启动子序列含有3个GA响应的顺式作用元件GARE(GA-responsive element),它的核心基序是TAACAAR[15],分别在启动子的-1 455、-822、-463处(图 1),说明SlGLD1可能是在GA调控途径中发挥功能。为了进一步研究SlGLD1的DNA甲基化程度,我们发现在SlGLD1的编码区有两个GC岛(图 2),但是在启动子区域未发现GC岛。因为DNA甲基化通常发生在GC岛附近或者GC岛上[16],所以我们设计BSP引物,重点分析这两个GC岛和附近的DNA的甲基化程度。

|
| 图 1 SlGLD1启动子区域GA响应的顺式作用元件(红色下划线部分为GARE元件) |

|
| 图 2 SlGLD1启动子(A)和编码序列(B)的GC岛预测(阴影部分为GC岛) |
将SlGLD1的基因序列在番茄表观组数据库比对发现,SlGLD1的启动子序列和编码区的DNA是高度甲基化的,如图 3所示,在果实成熟的时期,如17D(授粉后17 d)、MG(绿熟期)、Br(破色期)、Br+10(破色后第10天),SlGLD1启动子区域相对于编码区甲基化程度要高,相反,在叶片中,SlGLD1编码区的DNA甲基化程度较高。在CNR(colorless nonripening)突变体中,无论是在启动子区域和编码区,其DNA甲基化程度都相对在Rin(ripening-inhibitor)要高,这些都说明SlGLD1基因的DNA甲基化在番茄不同的器官中是有差异的,因此选择叶片分析DNA甲基化程度,分析相应的编码区可能会有更好的结果。
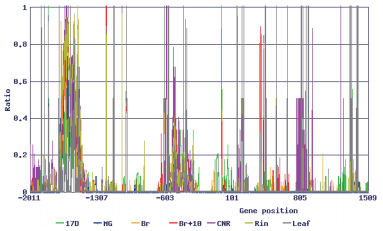
|
| 图 3 SlGLD1启动子序列和编码序列的在番茄相关组织和突变体中的DNA甲基化程度分析 不同颜色代表不同的组织发育时期或突变体的SlGLD1的甲基化程度。17D:授粉后17天;MG:绿熟期;Br:破色期;Br+10:破色后第10天;CNR:colorless nonripening;Rin:ripening-inhibitor |
基于前面的结果,选取了编码区的两个GC岛作为分析对象进行DNA甲基化分析。利用BS-PCR技术,用重亚硫酸盐对JMJ524-RNAi抑制系和WT(野生型M82)叶片的DNA进行处理,然后利用引物对这两个片段进行PCR扩增,克隆到T载体上进行测序分析。结果(图 4)显示,利用重亚硫酸盐处理后,转化效率大于99%,以WT第2个GC岛附近(1 164-1 400)为例,所得到的未甲基化的“C”经过处理扩增后全部变为“T”。我们选取那些转化效率为100%的序列进行分析,结果表明在第1个GC岛附近(728-1 039),野生型WT中CpG、CHG和CHH的总甲基化程度为77.92%、2.63%和1.51%,对应的在JMJ524-RNAi抑制系中CpG、CHG和CHH的甲基化程度分别为75.75%、1.75%和1.41%,在野生型WT中的DNA甲基化程度明显高于JMJ524-RNAi抑制系(图 5-A和B);同样的在第2个GC岛附近(1 164-1 400),野生型对照WT CpG、CHG和CHH的甲基化程度分别为63.81%、2.33%和0.40%,在JMJ524-RNAi抑制系中CpG、CHG和CHH的甲基化程度分别为59.16%、1.49%和2.30%,除了CHH在RNAi抑制系中是提高的,其他类型的甲基化都降低了,其总甲基化程度在野生型WT中也是高于JMJ524-RNAi抑制系(图 5-C和D)。特别是CpG类型的甲基化,在这两个甲基化热点都是显著降低的,如图 6所示,在第2个GC岛位置,编码区的1 189、1 284、1 303、1 355和1 365这些位点的DNA CpG甲基化程度在Ri14中都比WT低。结合以上结果,说明JMJ524可能是通过调控植物基因组的甲基化程度,使SlGLD1的DNA CpG的甲基化降低,从而激活这个基因的表达,导致番茄的矮化。

|
| 图 4 重亚硫酸盐处理SlGLD1基因序列前后测序结果分析(Target为处理前目标序列:1 164-1 400) |

|
| 图 5 SlGLD1编码区的两个甲基化热点的DNA甲基化分析 |
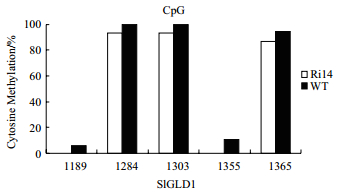
|
| 图 6 SlGLD1编码区在JMJ524抑制系(Ri14)和野生型(WT)的甲基化比较分析 |
本研究中,我们发现SlGLD1的启动子序列含有3个GA响应的顺式作用元件(图 1),印证了SlGLD1这类GRAS基因是参与GA调控途径的[17],这也与我们前期发现其受到GA诱导表达是一致的。但是通过生物信息学分析,我们并没有在其启动子区域发现甲基化热点,反而在其编码区发现两个甲基化热点(图 2),由于JmjC类基因在很多报道中已经证明参与植物的DNA甲基化或者组蛋白修饰[18-20],所以推测JMJ524调控基因的表达可能是通过表观遗传的机制进行的。表观遗传的研究已成为生命科学研究的前沿和热点,其主要研究在基因的核苷酸序列没有发生改变的情况下,基因的功能发生了可遗传性变化,并最终导致表型的变化。随着分子生物学的发展,表观遗传的研究发展也越来越快,然而总体来说,植物中的表观遗传研究远落后于动物,也主要集中在拟南芥和水稻中,番茄作为一种重要的模式作物的研究更是远远不够。随着基因组测序的完成以及丰富的遗传资源使番茄成为表观遗传学研究的重要对象。
表观遗传的机制较复杂,包括染色质重塑、基因组印迹、基因表达的重编程、DNA甲基化及RNA干扰等。染色质重塑常因染色质核心组蛋白发生甲基化、乙酰化等共价修饰,从而改变染色质的结构并影响基因的转录表达。组蛋白的甲基化发生在赖氨酸和精氨酸上,能调控一系列生物学过程,包括基因活性、染色质结构、剂量补偿效应及表观遗传学记忆[21]。DNA甲基化、组蛋白甲基化和染色体重塑之间也是相互影响,通过控制染色质的结构从而调控相关基因的表达[22]。本研究对已获得了番茄的JMJ524基因RNAi沉默的转基因植株和野生型对照,并且进一步进行BSP甲基化分析发现,JMJ524的抑制系中SlGLD1的CpG甲基化程度比野生型明显降低(图 5和6),虽然CHG和CHH的甲基化程度也有所降低,但是甲基化程度不高,所以推测JMJ524对基因的调控可能主要是控制CpG的甲基化。上述结果确定JMJ524的抑制表达影响了SlGLD1基因编码区的DNA甲基化程度,特别是CpG的甲基化程度,从而调控这个基因表达,这与我们前期通过HPLC发现抑制表达JMJ524后影响植物全基因组甲基化水平是一致的[8]。在JMJ524抑制系中,SlGLD1是上调表达的,而且超量表达SlGLD1也导致了番茄的不敏感的矮化,但是SlGLD1的矮化没有JMJ524-RNAi抑制系严重,所以推测,JMJ524可能不只是调控SlGLD1来影响植物的生长发育,还调控其他的靶基因或影响其他基因的甲基化来行使其功能。
4 结论本研究分别对JMJ524-RNAi转基因植株和野生型M82的SlGLD1基因的DNA序列进行了甲基化的分析,结果表明在SlGLD1 DNA序列中有两个GC岛,而且通过预测发现这两个片段的序列是高度甲基化的。利用BSP分析这两个片段,结果表明在JMJ524-RNAi转基因株系中,DNA的整体甲基化程度,特别是CpG的甲基化程度显著低于野生型对照M82。
| [1] | Ge X, Chang F, Ma H. Signaling and transcriptional control of reproductive development in Arabidopsis. Curr Biol, 2010, 20 : R988–997. DOI:10.1016/j.cub.2010.09.040 |
| [2] | Chang F, Wang Y, Wang S, et al. Molecular control of microsporoge-nesis in Arabidopsis. Curr Opin Plant Biol, 2011, 14 : 66–73. DOI:10.1016/j.pbi.2010.11.001 |
| [3] | Yang H, Chang F, You C, et al. Whole-genome DNA methylation patterns and complex associations with gene structure and expression during flower development in Arabidopsis. Plant J, 2015, 81 : 268–281. DOI:10.1111/tpj.2015.81.issue-2 |
| [4] | Chan SW, Henderson IR, Jacobsen SE. Gardening the genome: DNA methylation in Arabidopsis thaliana. Nat Rev Genet, 2005, 6 : 351–360. DOI:10.1038/nrg1601 |
| [5] | Klose RJ, Kallin EM, Zhang Y. JmjC-domain-containing proteins and histone demethylation. Nat Rev Genet, 2006, 7 : 715–727. |
| [6] | Lu F, Li G, Cui X, et al. Comparative analysis of JmjC domain-containing proteins reveals the potential histone demethylases in Arabidopsis and rice. J Integr Plant Biol, 2008, 50 : 886–896. DOI:10.1111/jipb.2008.50.issue-7 |
| [7] | 秦巧, 张海文, 黄荣峰. 拟南芥和水稻JMJC蛋白的研究进展. 生物技术通报, 2013(10): 1–5. |
| [8] | Li J, Yu C, Wu H, et al. Knockdown of a JmjC domain-containing gene JMJ524 confers altered gibberellin responses by transcriptional regulation of GRAS protein lacking the DELLA domain genes in tomato. J Exp Bot, 2015, 66 (5): 1413–1426. DOI:10.1093/jxb/eru493 |
| [9] | Jin J, Zhang H, Kong L, et al. PlantTFDB 3.0: a portal for the functional and evolutionary study of plant transcription factors. Nucleic Acids Res, 2014, 42 : D1182–1187. DOI:10.1093/nar/gkt1016 |
| [10] | Bolle C. The role of GRAS proteins in plant signal transduction and development. Planta, 2004, 218 : 683–692. DOI:10.1007/s00425-004-1203-z |
| [11] | Ma HS, Liang D, Shuai P, et al. The salt-and drought-inducible poplar GRAS protein SCL7 confers salt and drought tolerance in Arabidopsis thaliana. J Exp Bot, 2010, 61 : 4011–4019. DOI:10.1093/jxb/erq217 |
| [12] | Zhong S, Fei Z, Chen YR, et al. Single-base resolution methylomes of tomato fruit development reveal epigenome modifications associated with ripening. Nat Biotechnol, 2013, 31 : 154–159. DOI:10.1038/nbt.2462 |
| [13] | Murray MG, Thompson WF. Rapid isolation of high molecular weight plant DNA. Nucleic Acids Res, 1980, 8 : 4321–4325. DOI:10.1093/nar/8.19.4321 |
| [14] | J.萨姆布鲁克, D.W拉赛尔.黄培堂译.分子克隆实验指南[M].第3版.北京:科学出版社, 2002. |
| [15] | Ogawa M, Hanada A, Yamauchi Y, et al. Gibberellin biosynthesis and response during Arabidopsis seed germination. Plant Cell, 2003, 15 : 1591–1604. DOI:10.1105/tpc.011650 |
| [16] | Irizarry RA, Ladd-Acosta C, Wen B, et al. The human colon cancer methylome shows similar hypo-and hypermethylation at conserved tissue-specific CpG island shores. Nat Genet, 2009, 41 : 178–186. DOI:10.1038/ng.298 |
| [17] | Huang W, Xian Z, Kang X, et al. Genome-wide identification, phylogeny and expression analysis of GRAS gene family in tomato. BMC Plant Biol, 2015, 15 : 209. DOI:10.1186/s12870-015-0590-6 |
| [18] | Huang Y, Chen D, Liu C, et al. Evolution and conservation of JmjC domain proteins in the green lineage. Mol Genet Genomics, 2016, 291 : 33–49. DOI:10.1007/s00438-015-1089-4 |
| [19] | Miura A, Nakamura M, Inagaki S, et al. An Arabidopsis jmjC domain protein protects transcribed genes from DNA methylation at CHG sites. EMBO J, 2009, 28 : 1078–1086. DOI:10.1038/emboj.2009.59 |
| [20] | Fan D, Dai Y, Wang X, et al. IBM1, a JmjC domain-containing histone demethylase, is involved in the regulation of RNA-directed DNA methylation through the epigenetic control of RDR2 and DCL3 expression in Arabidopsis. Nucleic Acids Res, 2012, 40 : 8905–8916. DOI:10.1093/nar/gks647 |
| [21] | Martin C, Zhang Y. The diverse functions of histone lysine methylation. Nature reviews. Molecular Cell Biology, 2005, 6 : 838–849. DOI:10.1038/nrm1761 |
| [22] | Li BZ, Huang Z, Cui QY, et al. Histone tails regulate DNA methylation by allosterically activating de novo methyltransferase. Cell Res, 2011, 21 : 1172–1181. DOI:10.1038/cr.2011.92 |




