2. 成都丹凤科技有限公司,成都 611130
2. Danfeng (Chengdu) Technology Co., Ltd., Chengdu 611130
玉米是集粮饲经于一体的栽培作物,在全球范围内广泛种植。提高粮食作物的产量是满足全球人口指数增长的重要前提和基础。但在玉米生产上一直面临土壤有效磷浓度低和磷素利用率低的制约[1-3],严重影响了玉米的品质和产量,磷作为一种不可再生的资源而备受关注。在植物的生长和发育中,磷作为三大营养元素之一,在能量转移和代谢调节以及抗逆性等过程中起着至关重要的作用。而磷在土壤中极易被螯合固定且利用率低,提高作物耐低磷能力和磷高效利用水平是一个全球性的研究热点[4]。因此,发掘提高玉米低磷胁迫能力的基因,为分子育种改良玉米基因资源提供基础,对农业生产具有重要的意义。
在长期的磷饥饿胁迫下,植物进化出以根系形态结构变化为主的形态学变化[5]和代谢通路中生理生化的综合调节,以及低磷胁迫适应性相关基因分子水平表达调控[6, 7]等复杂的适应性机制来满足自身生长发育的需要。已有的研究发现,分子水平的适应性调节基因主要包括紫色酸性磷酸酶基因PAPs[8]、核糖核酸酶基因Rnase[9]、磷酸烯醇式丙酮酸羧化酶基因PEPC[10]、焦磷酸酶基因[11]、转录因子PHR[12]和ARF[13]以及磷转运蛋白基因PHT[6]等。其中磷转运蛋白对植物磷素营养的吸收和利用至关重要,按基因序列的相似性和功能的保守性,可将植物基因组中的磷转运蛋白分为五大家族,分别为Pht1、Pht2、Pht3、Pho1和Pho2[14]。
随着玉米自交系B73全基因组测序的完成,为分子水平上进行抗逆相关基因的克隆和功能研究提供了便利。本研究即是通过同源克隆的方法,在极端耐低磷玉米自交系中克隆得到一个磷转运蛋白基因ZmPht3;1,并利用荧光定量PCR的方法分析其时空表达模式,结合亚细胞定位实验初步探索其可能的生物学功能,旨在为玉米分子育种的耐低磷遗传改良奠定试验基础。
1 材料与方法 1.1 材料 1.1.1 试验材料和处理选用玉米耐低磷自交系Mo17和低磷敏感自交系B73为试验材料[15, 16],以3%的双氧水浸泡处理30 min,并用无菌水冲洗3次,置于培养皿中浸种催芽至萌发,再播入无菌蛭石中恒温光照培养。培养温度控制为白天28℃,夜间22℃,每天光照12 h。玉米幼苗长至三叶期,选取长势一致的植株移至营养液中进行水培。分别进行6、12、24和72 h的磷胁迫(缺磷营养液)处理,根和叶均3株混合取样。同时在各相应处理时段以全营养液(供磷水平为1 mmol/L)培养为对照,培养条件不变。营养液配制参考Hoagland营养液配方,胁迫处理组和对照组取材后迅速置于液氮中速冻,-80℃冰箱保存备用。
1.2 方法 1.2.1 总RNA提取以及cDNA第一链合成利用Aidlab公司(北京)的EASYspin plus植物RNA提取试剂盒分别提取Mo17和B73在低磷胁迫处理和正常对照组各个时间段的总RNA。1%琼脂糖凝胶电泳检测总RNA的完整性,-80℃保存备用。分别以总RNA为模板,按照TaKaRa公司的反转录试剂盒(Code:DRR037A)反转录合成cDNA,-20℃保存备用。
1.2.2 基因克隆参照玉米基因组网站MaizeGDB(http://www.maizegdb.org/)中玉米B73的cDNA序列和CDS序列,用Primer5.0设计特异性引物(F:5’-CACGAGAGAGACGAATCT-3’,R:5’-TGATCTCAG-GGAACACAG-3’),以Mo17的cDNA为模板进行ZmPht3;1同源序列克隆。高保真酶KOD FX Neo(Toyobo)扩增体系包含2×Buffer、200 μmol/L dNTP、0.2 μmol/L上下游引物、0.5 U DNA聚合酶、1 μL cDNA模板。扩增程序为:95℃预变性2 min;98℃ 10 s,54.5℃ 20 s,68℃ 1 min,40个循环;68℃延伸3 min,4℃保存。PCR扩增产物经1.2%的琼脂糖凝胶电泳分离、检测,将目标条带切下后,用OMEGA公司的DNA凝胶回收试剂盒回收、纯化,并连接pEASY-Blunt载体(北京全式金公司)转化大肠杆菌DH5α,将挑取的阳性克隆菌液进行ABI 3730测序,并对测序结果进行序列比对验证。
1.2.3 序列分析根据测序验证的基因序列,用NCBI Conserved Domain Database(http://www.ncbi.nlm.nih.gov/Structure/index.shtmlb)对其编码蛋白功能域进行预测。利用SOPMA软件在线预测蛋白质二级结构类型及其分子量和等电点等理化特性。利用在线工具HMMTOP、Signal P V4.0、SMART V4.0[17]和Cello v.2.5[18]等分析克隆基因是否含有跨膜区、信号肽以及功能域和亚细胞定位预测。
1.2.4 基因表达分析依据克隆基因的测序结果,在其非保守区设计特异性引物,并以玉米基因18S(F:5’-CTGAGAAACGGCTACCACA-3’,R:5’-CCCAAGGTCCAACTACGAG-3’)作为内参对照,对不同处理的样品进行qRT-PCR。将反转录合成的cDNA稀释10倍作为qRT-PCR的模板。按照SsoFastTM EvaGreen® Supermix试剂盒(Bio-Rad公司)说明配制反应体系,在Bio-Rad CFX96TM PCR仪上进行扩增,每个样品设3个技术重复。反应体系总体积为10 μL,包含2×EvaGreen Supermix 5 μL,cDNA模板1 μL,上下游引物各0.2 μL,ddH2O 3.6 μL。反应程序设定为:98℃预变性2 min;98℃ 2 s,54.6℃(内参基因18S为58℃)10 s,并在此温度收集荧光,读板(Plate Read)共40个循环。之后从65℃开始,以每步0.5℃的温度梯度升高至95℃,每步温度保持1 s并读板,绘制熔解曲线。采用2-ΔΔCT法分析数据计算基因的相对表达量。
1.2.5 亚细胞定位检测依据克隆基因的测序结果,设计并合成含有限制性酶切位点的引物,上游引物为:5’-TAGGATCCATGGCGCTCTCCGAC-3’,下游引物为:5’-CGGTCGACTCAAGCACTGGCTTTCAA-3’(下划线部分分别为BamH I和Sal I酶切位点)。通过酶切连接的方法将纯化后的PCR产物与改造后的表达载体pCAMBIA2300-eGFP连接构建融合表达载体,转化大肠杆菌DH5α并扩大培养阳性菌液提取质粒。分别将测序鉴定正确的重组质粒和空载体质粒(对照试验组)转化农杆菌GV3101,涂板后置于28℃培养48 h挑取单菌落进行菌液PCR检测和测序验证。再将测序验证的阳性单克隆扩大培养12 h,收集菌体并用10 mmol/L的MgCl2悬浮混匀。采用注射渗透法转化本生烟草,正常培养36 h后用于共聚焦显微镜检测。
2 结果 2.1 基因克隆和序列分析通过RT-PCR的方法,根据玉米B73的参考序列设计的特异性引物,以耐低磷玉米自交系Mo17的cDNA为模板,扩增获得ZmPht3;1的CDS序列。测序验证后分析发现该基因开放阅读框全长1 101 bp,编码366个氨基酸(图 1),分子量为38.65 kD,等电点(pI)为9.29,其中亲水氨基酸只有84个,疏水氨基酸为150个。保守结构域预测显示该编码区包含有Mito_carr超家族(图 2),结构特征与线粒体转运家族(mitochondrial carrier family,MCF)高度一致,是行使其特定生物学功能的基础。6个保守的跨膜区与5段亲水序列相间连接,N端和C端序列均位于膜外侧基质面,膜内侧的亲水环为一段极端保守的基序Px[D/E]xx[R/K]。氨基酸序列的功能性修饰位点分析表明,该序列中不存在N连接的糖基化位点,但包含有一个潜在的酪氨酸激酶II的磷酸化位点(图 1)。推导的氨基酸序列疏水性分析结果(图 3),进一步证实了该基因具有6个疏水的跨膜结构。Cello v2.5预测显示该基因的表达蛋白定位于细胞膜周质。

|
| 图 1 ZmPht3; 1的全长CDS序列及其推导的氨基酸序列 划线部分分别是6个跨膜区, 双划线部分为酪氨酸激酶II的磷酸化位点 |

|
| 图 2 ZmPht3; 1的保守结构域预测 |
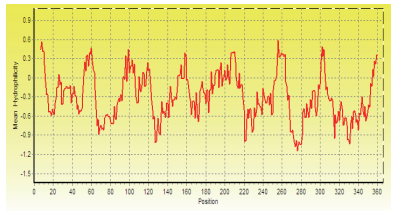
|
| 图 3 ZmPht3; 1的疏水性分析 |
为了探究基因ZmPht3;1的时空表达特征,对耐低磷自交系Mo17和低磷敏感自交系B73分别进行缺磷胁迫处理,并通过荧光定量PCR的方法,检测该基因在根系和叶片中的表达模式差异。实验结果(图 4)表明,ZmPht3;1在根系与叶片中的表达模式在两个极端材料中差异显著。在低磷敏感自交系B73根系与叶片中的表达模式极为相似,表达水平整体上表现为一定程度的动态平衡(图 4-A)。缺磷胁迫早期6 h时,在根系与叶片中均强烈响应缺磷环境,显著的下调抑制表达;胁迫中期,表现为对缺磷环境的适应性,表达量有所增加;在胁迫后期(72 h)的相对表达量又下降至稳态水平。而在耐低磷自交系Mo17的根系与叶片中,ZmPht3;1的表达模式截然相反(图 4-B)。缺磷胁迫早期,在根系与叶片中均诱导上调表达,但随着胁迫时间的延长,胁迫中期在叶片中的相对表达量继续增加,而在根系中表现为下调的抑制表达。在胁迫后期,叶片中ZmPht3;1的相对表达量逐步降低,在72 h时达到最低值;而在根系中显著的上调增强表达,在72 h时达到最高值,表现为对缺磷胁迫环境的诱导表达现象,这可能与该材料对磷环境的敏感性和适应性有关。

|
| 图 4 ZmPht3; 1在B73(A)和Mo17(B)根系与叶片中的表达模式 R和L分别表示根系与叶片 |
为了验证该基因表达蛋白亚细胞定位的在线预测结果,构建了融合表达载体pCAMBIA2300-ZmPht3;1-eGFP,并以空载体pCAMBIA2300-eGFP为对照。通过注射农杆菌渗透法转化本生烟草,经10 μmol/L的FM4-64染色后,置于荧光显微镜上进行镜检观察,再用共聚焦显微镜成像(图 5)。结果显示,空载体pCAMBIA2300-eGFP表达的绿色荧光蛋白分布于整个细胞(图 5-B),而染色剂FM4-64将细胞膜染成红色(图 5-D),重组质粒pCAMBIA2300-ZmPht3;1-eGFP表达的融合蛋白分布于细胞膜和细胞膜周质(图 5-E),与基因序列在线预测(Cello v.2.5)的亚细胞定位结果一致,初步证实ZmPht3;1定位于细胞膜或其周质。

|
| 图 5 融合蛋白ZmPht3; 1-eGFP在烟草中的亚细胞定位 A:明场成像照片; B, E:融合蛋白在绿光激发下产生的荧光; C, F:叠加的照片; D:细胞被FM4-64染色的照片 |
随着测序技术和基因组学的发展,越来越多物种的全基因组序列测序完成,为分子水平上鉴定和克隆特定的功能基因提供了便利。本研究利用同源克隆的方法,在耐低磷玉米自交系Mo17中克隆得到一个磷转运蛋白基因ZmPht3;1,并对该基因的序列进行生物信息学预测分析发现其翻译氨基酸的理化性质和结构特征与MCF高度一致(图 1-图 3),并且膜内侧的亲水环包含一段MCF家族所特有的基序Px[D/E]xx[R/K][19],暗示该家族基因在进化中相对保守。本研究发现ZmPht3;1蛋白定位于细胞膜上,同时基因序列的功能性修饰预测显示在亲水环中存在一个酪氨酸激酶II的磷酸化位点(图 1),可能与其在低磷信号的识别和传导中作出响应有关。
磷转运蛋白基因作为一类具有特定生物学功能的功能基因,在植物响应磷胁迫过程中发挥重要的调节作用[6, 10]。根据磷转运蛋白对土壤中可溶性磷吸附能力的差异,可将其划分为高亲和力磷转运蛋白(Km值为1.8-9.9 μmol/L)和低亲和力磷转运蛋白(Km值为mmol/L数量级)[20]。其中属于高亲和力磷转运蛋白的Pht1家族是H2PO4-/ nH+共运体[21],而归属于低亲和力的线粒体转运家族(MCF)的Pht3主要位于线粒体膜或质膜上,负责将细胞质中无机磷转入质体[22],维持植株体内的磷素循环和再利用。不同磷转运蛋白的基因家族间存在功能分化,而同一家族中部分成员之间的生物学功能存在补偿效应或叠加效应[23]。有研究表明,玉米中属于高亲和力的Pht1家族基因ZmPht1;9响应低磷胁迫诱导,但主要功能是调节根系与叶片中磷素的动态平衡[24]。推测磷转运蛋白五大家族成员之间可能存在功能互补或协同调节作用,以及复杂的反馈抑制调节系统,综合应答低磷胁迫环境。
已有的研究发现,多种植物的磷转运蛋白基因分别有其组织表达特异性和时空表达特征。本研究中ZmPht3;1在两极端材料的根系和叶片中均有表达,而相对表达量的变化模式差异显著(图 4),是MCF蛋白维持植株体内磷素动态平衡的生物学功能的基本体现。在耐低磷玉米自交系Mo17根系和叶片中,缺磷胁迫早期ZmPht3;1均不同程度的上调诱导表达,后期随着胁迫时间的延长表达模式变化差异显著(图 4-B),表明该基因的表达特征属于缺磷胁迫早期的一般性反应和胁迫后期的特异性反应[25]。然而,缺磷胁迫条件下,ZmPht3;1在耐低磷玉米自交系Mo17的根系中胁迫早期被诱导上调表达,相对表达量在胁迫中期有所减少,在24 h时达到最低值,胁迫后期又逐渐增加(图 6),表明该基因对缺磷胁迫的响应存在阶段性差异。正常供磷条件下,ZmPht3;1在前期显著增强表达,后期的相对表达水平逐渐降低(图 6),暗示该基因的表达可能存在不同生长发育时期的差异[26]。由此有理由推测ZmPht3;1可能与少数Pht1家族的高亲和力磷转运蛋白一样,是一个在高磷条件下和低磷条件下均起作用的双亲和转运体[23],在玉米响应磷饥饿胁迫过程中发挥重要的适应性调节作用,为玉米磷高效分子育种研究奠定了基础。
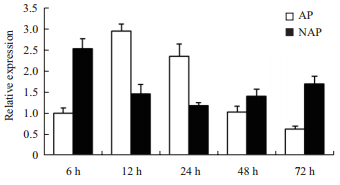
|
| 图 6 ZmPht3; 1在Mo17根系中的表达模式 NAP和AP分别缺磷与正常供磷水平 |
以耐低磷玉米自交系Mo17的总RNA反转录的cDNA为模板,通过同源克隆PCR获得一个磷转运蛋白基因ZmPht3;1。序列分析显示其氨基酸序列中存在6个跨膜结构,包含有Mito_carr超家族保守结构域。基因表达分析表明,ZmPht3;1在两个极端材料中根系与叶片的表达模式差异显著。亚细胞定位显示该基因的表达蛋白主要分布于细胞膜上,结合其在耐低磷玉米自交系Mo17中的表达模式表现为缺磷胁迫早期的一般性反应和胁迫后期的特异性反应,推测ZmPht3;1可能是一个双亲和转运体,在玉米响应磷饥饿胁迫的适应性过程中发挥重要的调节作用。
| [1] | Raghothama KG, Karthikeyan AS. Phosphate acquisition. Plant and Soil, 2005, 274 (1-2): 37–49. DOI:10.1007/s11104-004-2005-6 |
| [2] | Shen J, Yuan L, Zhang J, et al. Phosphorus dynamics: from soil to plant. Plant Physiology, 2011, 156 (3): 997–1005. DOI:10.1104/pp.111.175232 |
| [3] | Calderon-Vazquez C, Sawers RJ, Herrera-Estrella L. Phosphate deprivation in maize: genetics and genomics. Plant Physiology, 2011, 156 (3): 1067–77. DOI:10.1104/pp.111.174987 |
| [4] | Lopez-Arredondo DL, Leyva-Gonzalez MA, Gonzalez-Morales SI, et al. Phosphate nutrition: improving low-phosphate tolerance in crops. Annual Review of Plant Biology, 2014, 65 : 95–123. DOI:10.1146/annurev-arplant-050213-035949 |
| [5] | York LM, Nord EA, Lynch JP. Integration of root phenes for soil res-ource acquisition. Frontiers in Plant Science, 2013, 4 : 355. |
| [6] | Secco D, Jabnoune M, Walker H, et al. Spatio-temporal transcript profiling of rice roots and shoots in response to phosphate starvation and recovery. The Plant Cell, 2013, 25 (11): 4285–4304. DOI:10.1105/tpc.113.117325 |
| [7] | Wu P, Shou H, Xu G, et al. Improvement of phosphorus efficiency in rice on the basis of understanding phosphate signaling and homeostasis. Current Opinion in Plant Biology, 2013, 16 (2): 205–212. DOI:10.1016/j.pbi.2013.03.002 |
| [8] | Li C, Gui S, Yang T, et al. Identification of soybean purple acid phosphatase genes and their expression responses to phosphorus availability and symbiosis. Annals of Botany, 2012, 109 (1): 275–285. DOI:10.1093/aob/mcr246 |
| [9] | Bariola PA, Howard CJ, Taylor CB, et al. The Arabidopsis ribonuclease gene RNS1 is tightly controlled in response to phosphate limitation. The Plant Journal, 1994, 6 (5): 673–685. DOI:10.1046/j.1365-313X.1994.6050673.x |
| [10] | Calderon V, Ibarra L, Caballero P, et al. Transcript profiling of Zea mays roots reveals gene responses to phosphate deficiency at the plant-and species-specific levels. Journal of Experimental Botany, 2008, 59 (9): 2479–2497. DOI:10.1093/jxb/ern115 |
| [11] | Pei L, Wang J, Li K, et al. Overexpression of the llungiella halophila H+-pyrophosphatase gene improves low phosphate tolerance in maize. PLoS One, 2012, 7 (8): e43501. DOI:10.1371/journal.pone.0043501 |
| [12] | Wang X, Bai J, Liu H, et al. Overexpression of a maize transcription factor ZmPHR1 improves shoot inorganic phosphate content and growth of Arabidopsis under low-phosphate conditions. Plant Molecular Biology Reporter, 2012, 31 (3): 665–677. |
| [13] | Wang S, Zhang S, Sun C, et al. Auxin response factor (OsARF12), a novel regulator for phosphate homeostasis in rice (Oryza sativa). The New Phytologist, 2014, 201 (1): 91–103. DOI:10.1111/nph.12499 |
| [14] | 杨存义, 刘灵, 沈宏, 等. 植物Pht1家族磷转运子的分子生物学研究进展. 分子植物育种, 2006, 4(2): 153–159. |
| [15] | Zhang L, Li J, Rong T, et al. Large-scale screening maize germplasm for low-phosphorus tolerance using multiple selection criteria. Euphytica, 2014, 197 (3): 435–446. DOI:10.1007/s10681-014-1079-3 |
| [16] | Kaeppler SM, Parke JL, Mueller SM, et al. Variation among maize inbred lines and detection of quantitative trait loci for growth at low phosphorus and responsiveness to arbuscular mycorrhizal fungi. Crop Science, 2000, 40 : 358–364. DOI:10.2135/cropsci2000.402358x |
| [17] | Letunic I, Doerks T, Bork P. SMART 7: recent updates to the protein domain annotation resource. Nucleic Acids Research, 2012, 40 (D1): D302–D5. DOI:10.1093/nar/gkr931 |
| [18] | Yu CS, Chen YC, Lu CH, et al. Prediction of protein subcellular localization. Proteins: Structure, Function, and Bioinformatics, 2006, 64 (3): 643–651. DOI:10.1002/prot.21018 |
| [19] | Picault N, Hodges M, Palmieri L, et al. The growing family of mitochondrial carriers in Arabidopsis. Trends Plant Sci, 2004, 9 (3): 138–146. DOI:10.1016/j.tplants.2004.01.007 |
| [20] | Muchhal US, Pardo JM, Raghothama K. Phosphate transporters from the higher plant Arabidopsis thaliana. Proceedings of the National Academy of Sciences, 1996, 93 (19): 10519. DOI:10.1073/pnas.93.19.10519 |
| [21] | Raghothama K. Phosphate transport and signaling. Current Opinion in Plant Biology, 2000, 3 (3): 182–187. DOI:10.1016/S1369-5266(00)00062-5 |
| [22] | Poirier Y, Bucher M. Phosphate Transport and Homeostasis in Arabidopsis[M]. The Arabidopsis Book, 2002. |
| [23] | Shin H, Shin HS, Dewbre GR, et al. Phosphate transport in Arabidopsis: Pht1; 1 and Pht1; 4 play a major role in phosphate acquisition from both low-and high-phosphate environments. Plant J, 2004, 39 (4): 629–642. DOI:10.1111/tpj.2004.39.issue-4 |
| [24] | 苏顺宗, 吴锋锴, 刘丹, 等. 一个玉米Pht1家族磷转运蛋白基因克隆和功能分析. 核农学报, 2013, 27(7): 885–894. |
| [25] | Hammond JP, Bennett MJ, Bowen HC, et al. Changes in gene expression in Arabidopsis shoots during phosphate starvation and the potential for developing smart plants. Plant Physiology, 2003, 132 (2): 578–596. DOI:10.1104/pp.103.020941 |
| [26] | Ai P, Sun S, Zhao J, et al. Two rice phosphate transporters, OsPht1; 2 and OsPht1; 6, have different functions and kinetic properties in uptake and translocation. The Plant Journal, 2009, 57 (5): 798–809. DOI:10.1111/tpj.2009.57.issue-5 |



