随着人类基因组计划(Human genome project,HGP)的完成以及高通量测序技术的不断发展完善,众多生物的基因组信息得以确定。在分析基因组序列过程中,大量的重复序列的发现引起了科研工作人员的重视,其多样性和广泛性[1-3]也得到了越来越多的研究。重复序列是指基因组中不同位置出现的相同或对称性片段,其中,反向重复列(Inverted repeat,简称IR)是指同一DNA链内下游存在着与上游某一段序列互补的序列,是重复序列中一类具有生物学意义的序列[4],其特点是可形成具有双螺旋茎部和单链环的发卡结构,即在同一条DNA链内碱基发生互补配对形成DNA双螺旋的茎部,而两个反向重复序列单元之间的序列形成单链的环部。反向重复序列在真核和原核生物基因组中广泛存在[5],并且在生物体中具有多种功能[6],如与真核病毒、哺乳动物细胞和质粒的复制起点有关[7, 8],参与调控基因的表达,能够终止原核生物的转录[9]等;同时也是生物体基因组不稳定的原因之一[10-13]。长的回文序列与基因扩增[14, 15]和减数分裂中基因重组[16, 17]有密切联系。另外,DNA双链中的反向重复序列在体外[18]和体内[19]都可形成十字形结构(Cruciform structure),该在基因的复制[20]以及生物体正常和病理[21, 22]的调节中发挥了重要作用。有研究表明,DNA双链中反向重复序列之间间隔碱基小于5 nt时,负超螺旋带来的拓扑张力可使之形成十字形结构[23]。形成这些高级结构的难易程度除与间隔序列的长短和序列有关外,还与反向重复序列本身的长短和GC含量有关。
在研究生物体基因组中反向重复序列时,需要大量的模板,通常获得大量模板的常规方法是PCR扩增,而以具有发卡结构序列作为模板时,分子内部的杂交会对PCR引物的结合产生影响,使扩增效率降低(图 1),这一问题容易被忽视,且缺乏相关研究进行指导。针对具有反向重复序列的模板,如果能通过设计引物使其顺利扩增,这将会拓展PCR的应用范围。因此,本文深入研究了发卡结构的环部大小、茎部长度、引物相对于茎部的位置等对PCR的影响,旨在探讨通过引物设计及改变退火温度等条件来实现目的模板有效扩增的方法。
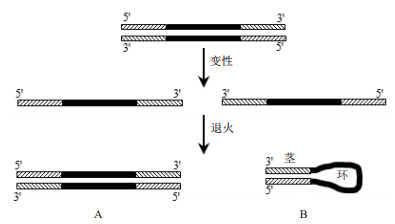
|
| 图 1 反向重复序列形成发卡结构示意图 斜线部分为反向重复序列,退火形成的图B中发卡结构 |
所有寡核苷酸序列均由英潍捷基(上海)贸易有限公司合成;DreamTaq DNA聚合酶购自Thermo Scientific公司;dNTPs购自天根生化科技(北京)有限公司。Life-express热循环仪为杭州博日科技有限公司产品;DYY-6C型电泳仪购自北京市六一仪器厂; Molecular imager Gel Doc XR+imaging system型凝胶成像系统为美国Bio-Rad公司产品,PikoReal荧光定量PCR仪购自Thermo Scientific公司。
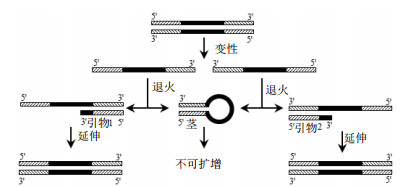
1.2 方法 1.2.1 具有不同环部长度的发卡结构模板的PCR扩增所用模板和引物分别在表 1、表 2中,以发卡结构中茎部长25 bp、环部长度分别为72、62、52、42、32、22和12 nt的DNA发卡序列H25S-72L、H25S-62L、H25S-52L、H25S-42L、H25S-32L、H25S-22L和H25S-12L为模板进行PCR反应,以P1(与发卡结构中茎部完全互补)作为引物,扩增示意图如图 2;同时以长度相同、不具有发卡结构的线性模板N25S-72L、N25S-62L、N25S-52L、N25S-42L和N25S-32L、N25S-22L和N25S-12L作为对照,以P1和P3作为引物对。考察发卡结构中不同环部长度对PCR扩增的影响。

|
| 图 2 具有发卡夹结构模板的PCR示意图 注:斜线部分表示反向重复序列部分(下同) |
PCR反应体系(20 μL)中含有1×的Dream-Taq Buffer(2 mmol/L MgCl2,1 mmol/L DTT,50 mmol/L Tris-HCl(pH8.0)),0.5 U DreamTaq DNA聚合酶,0.6 μmol/L引物,200 μmol/L dNTPs,模板拷贝数为106。
反应条件:94℃ 3 min;94℃ 30 s,61℃ 30 s,72℃ 30 s,30个循环;72℃ 10 min。取2 μL扩增产物,采用2.5%琼脂糖凝胶电泳1 h,EB染色15 min后,置于凝胶成像仪上成像。
1.2.2 具有不同茎长度的发卡结构模板的PCR扩增以发卡结构中环部长度为52 nt,茎部长度分别为20 bp、15 bp、10 bp DNA发卡序列H20S-52L、H15S-52L、H10S-52L(表 2)为模板,分别以P1和P2、P1和P4、P1和P5作为引物(表 3),扩增示意图如图 3;以长度相同、不含发卡结构的线性模板N20S-52L、N15S-52L、N10S-52L作为对照,分别以P2和P3、P3和P4、P3和P5作为引物对,考察发卡结构中不同茎部长度对模板扩增的影响。PCR反应体系和反应条件同1.2.1。
|
| 图 3 不同茎部长度模板扩增示意图 |
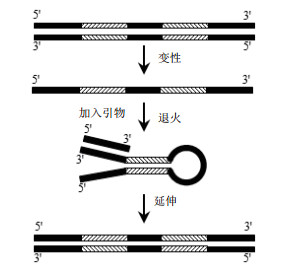
以长度为102 bp,可形成发卡结构的环部长度10 nt,茎部长度为20 bp以及茎部两端各26 nt游离端的序列H20S-10L为模板(表 2),以P3和P6(表 3)作为引物对进行扩增,引物与游离端部分互补,扩增示意图如图 4。以长度相同、不具有发卡结构序列N25S-52L作为对照,以P1和P3作为引物对。PCR反应体系和反应条件同1.2.1。
|
| 图 4 引物处于茎两端游离端时模板扩增过程 |
以发卡结构中茎部长度25 bp,环部长度分别为52、42和32 nt的DNA发卡序列H25S-52L、H25S-42L、H25S-32L为模板,以P1作为引物;同时以长度相同、不具有发卡结构的N25S-52L、N25S-42L和N25S-32L的序列作为对照,以P1和P3作为引物对,进行梯度退火PCR反应,退火温度设置为63℃、60℃和57℃,PCR反应体系和反应条件同1.2.1。
1.2.4.2 不同引物浓度下发卡结构模板的扩增以发卡结构中茎部长度25 bp,环部长度为52 nt的DNA序列H25S-52L为模板,同时以长度相同、不具有发卡结构DNA序列N25S-52L作为对照进行PCR反应。引物浓度设置为0.6 μmol/L、0.1 μmol/L、0.05 μmol/L,其他PCR反应体系和反应条件同1.2.1。
1.2.5 部分发卡结构熔点的测定检测S-25L和S-25R、S-20L和S-20R、S-15L和S-15R合成的双链的熔点;检测发卡结构中茎部长度为20 bp,环部长度分别为20、30和50 nt的DNA序列H20S-20L、H20S-30L、H20S-50L的熔点;检测发卡结构中茎部长度分别为25 bp、20 bp、15 bp序列H25S-32L、H20S-42L、H15S-52L的熔点。
熔点检测体系(10 μL):1×EvaGreen,1×Dr-eamTaq Buffer,200 μmol/L dNTPs,500 nmol/L的DNA。检测条件:首先95℃加热3 min对DNA序列进行充分变性;然后以0.1℃/s缓慢降温至20℃以形成其最适结构,此时荧光染料EvaGreen与DNA充分结合并释放最大荧光;随后缓慢升温至95℃进行DNA序列的熔解,该过程中每升高0.1℃进行荧光信号收集,获得体系随温度变化的荧光熔解曲线。采用Piko Real Software 2.1对荧光熔解曲线进行微分运算处理(-d(RFU)/dT,即荧光强度相对温度的一阶微分运算的负数),获得荧光微分曲线和序列相应的熔点。
1.2.6 扩增产物定量及显著性分析利用Molecular imager Gel Doc XR+imaging system对电泳结果进行分析,以ladder中已知质量的标准条带作为参照,对电泳图中各扩增产物进行定量,并重复3次实验。对扩增产物定量后的数据经Tukey's检验后进行单因素方差分析(ANOVA)。所有数据以x±s表示,当P < 0.05时,定为差异显著。数据统计检验采用SPSS软件进行。
2 结果 2.1 模板发卡结构中环部长度对PCR扩增的影响由图 5可知,不具有发卡结构的模板都得到较好的扩增,而具有发卡结构模板当环的长度为22、32 nt(即72 bp和82 bp模板)时均未检测到产物,当环的长度为42 nt、52 nt、62 nt、72 nt(即92 bp、102 bp、112 bp和122 bp模板)时可检测到强弱不等的目的条带,而且随着环长度的增加,条带亮度逐渐增强。通过对25 bp茎长,环长分别为42 nt、52 nt、62 nt、72 nt的模板扩增产物条带定量后通过显著性分析发现,产物的量分别为13.0±1.7 ng/μL、28.4±2.7 ng/μL、64.5±3.4 ng/μL、102.9±2.4 ng/μL(P < 0.05),可以看出当发卡结构茎部长度为25 bp,环部长度为42 nt、52 nt、62 nt、72 nt时,产物的量随着环长度的增长约呈2倍增多,并且产物的量之间呈现显著性差异。
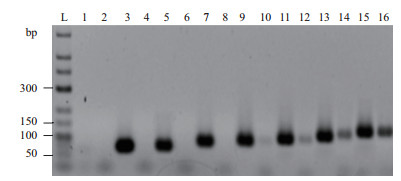
|
| 图 5 具有不同环部长度发卡结构模板的扩增结果 L:low range ladder(Thermo Scientific,#SM1191/2/3);1、2:分别表示不具有发卡结构和具有发卡结构空白;4、6、8、10、12、14、16:发卡结构中茎部长度25 bp,环部长度分别为12 nt、22 nt、32 nt、42 nt、52 nt、62 nt和72 nt的模板;3、5、7、9、11、13、15:分别表示长度相同,不具有发卡结构的对照 |
探究发卡结构中不同茎部长度对模板扩的影响,由图 6可知,相应的对照组,即反应过程中不形成发卡结构的模板均得到较好的扩增条带。反应过程中形成发卡结构环部长度为52 nt,茎部长度分别为10、15和20 bp的模板也都检测到了目的条带,但是当发卡结构中茎部长度为20 bp时,产物的条带亮度明显比发卡结构中茎部长度为15 bp和10 bp的弱。
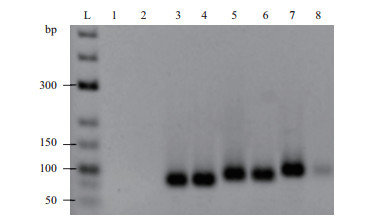
|
| 图 6 发卡结构中具有不同茎长度模板扩增结果 L:low range ladder(Thermo Scientific,#SM1191/2/3);1、2:分别为不具有发卡结构和具有发卡结构的空白;4、6、8:发卡结构中环部长度为52 nt,茎长度分别为10 bp、15 bp、20 bp的模板;3、5、7:分别为长度相同,不具有发卡结构的对照 |
当引物结合位点在发卡结构茎部外侧时的扩增结果见图 7,随着模板浓度的降低,目的片段的条带强度逐渐减弱,此时的最低检出限为1 000拷贝;而使用不形成发卡结构的模板时最低检出限为10拷贝,表明本实验中模板发卡结构的存在使PCR的检测灵敏度降低了约100倍。

|
| 图 7 引物处于两端游离端时模板扩增结果 L:low range ladder(Thermo Scientific,#SM1191/2/3);1、2:分别为不具有发卡结构和具有发卡结构的空白;4、6、8、10、12:具有发卡结构、拷贝数分别为104、103、102、10、1拷贝的模板;3、5、7、9、11:分别为长度相同,不具有发卡结构,拷贝数分别为104、103、102、10、1拷贝的对照 |
选取发卡结构中茎部长度25 bp,总长度为82 bp、92 bp和102 bp的DNA链为模板,同时设置长度相同,不具有发卡结构的模板作为对照。实验共分为3组,退火温度分别为63℃、60℃、57℃,探讨反应过程中不同退火温度对具有发卡结构模板的扩增的影响,实验结果如图 8。不具有发卡结构的模板在不同退火温度下均检测到目的条带。而具有发卡结构的模板在不同的退火温度下扩增结果却不同,当退火温度为63℃时,最短可检测到92 bp的条带;当退火温度为60℃时,仅检测到102 bp的目的条带,并且条带亮度比退火温度为63℃时要弱;当退火温度为57℃时,没有检测到目的条带。

|
| 图 8 不同退火温度下模板的扩增结果 L:low range ladder(Thermo Scientific,#SM1191/2/3);N1、N2:分别为不具有发卡结构和具有发卡结构的空白;2、4、6分别为发卡结构中茎部长度为25 bp,环部长度分别为32 nt、42 nt、52 nt的模板;1、3、5分别为长度相同,不具有发卡结构的对照 |
为探究引物浓度的改变对具有发卡结构模板扩增的影响,以发卡结构中茎部长度25 bp、环部长度52 bp的DNA序列为模板,以长度相同、不具有发卡结构模板作为对照,在不同的引物浓度梯度下进行PCR扩增。由图 9可知,当反应体系中引物浓度为0.6 μmol/L时,具有发卡结构和不具有发卡结构模板均检测到目的条带,并且不具有发卡结构模板条带亮度强于具有发卡结构模板的条带亮度。当反应过程中形成发卡结构的模板在引物浓度降至0.1 μmol/L时便部能检测到产物条带;而反应过程中不形成发卡结构的对照模板在引物浓度降至0.05 μmol/L时才不能检测到目的产物条带。

|
| 图 9 不同引物浓度时模板扩增结果 L:low range ladder(Thermo Scientific,#SM1191/2/3);N1、N2:分别为具有发卡结构和不具有发卡结构模板空白;1、2、3:发卡结构中茎部长度为25 bp、环部长度为52 nt,引物浓度分别为0.6 μmol/L、0.1 μmol/L、0.05 μmol/L的模板;4、5、6:长度相同,不具有发卡结构,引物浓度分别为0.6 μmol/L、0.1 μmol/L、0.05 μmol/L的对照 |
利用Piko Real Software 2.1获得荧光微分曲线和序列相应的熔点(图 10,表 4),图 10-A中25 bp、20 bp、15 bp双链熔点分别为72.5℃、71.1℃、66.7℃,双链熔点随着双链长度的增长而升高;图 10-B中长度为82 nt,发卡结构中茎长分别为25 bp、20 bp、15 bp的序列熔点分别为80.5℃、79.1℃、76.1℃,并且序列的熔点随着发卡结构中茎部长度的增长而升高;图 10-C中发卡结构中茎部长度20 bp,环部长度分别为20 nt、30 nt、50 nt的序列的熔点分别是82.6℃、81.5℃、78.8℃,并且序列的熔点随着发卡结构中环部长度的增长而降低。另外,图 10-B和图 10-C中每个熔点曲线出现了两条峰,推测熔点较低的峰可能是模板发卡结构中环部形成的二级结构所造成的结果。

|
| 图 10 双链DNA熔点测定结果 A:25 bp、20 bp、15 bp双链的熔点;B:长度为82 nt,发卡结构中茎长分别为25 bp、20 bp、15 bp的序列熔点;C:发卡结构中茎部长度20 bp,环部长度分别为20 nt、30 nt、50 nt的序列的熔点 |
本文深入研究了发卡结构的环部大小、茎部长度、引物相对于茎部的位置等对PCR扩增的影响,同时探讨了通过引物的设计和改变退火温度等PCR条件来实现具有发卡结构模板的有效扩增。
通过对发卡结构中不同环部长度对PCR扩增影响的探究发现,发卡结构能够明显抑制模板PCR扩增,并且当发卡结构的茎部长度一定时,环越短,发卡结构对模板扩增的抑制作用越强,当发卡结构中环部长度达到50 nt以上时,发卡结构对模板PCR扩增的抑制作用明显减弱。推测原因为反应过程中引物-模板结合和模板自身的发卡结构形成竞争关系,导致模板扩增效率下降。另外,模板发卡结构中环部越长,PCR反应过程中模板自身形成发卡结构的能力越弱,进而导致引物有更多的机会与模板结合,当发卡结构中环部长度达到50 nt以上时,发卡结构对模板PCR扩增的抑制作用明显减弱。缩短发卡结构中茎的长度,能够减弱发卡结构对模板PCR扩增的抑制作用,当发卡结构中茎部长度小于15 bp时,具有发卡结构模板的检测线与不可形成发卡结构模板的检测线接近。可能是由于发卡结构的茎部长度变短,反应过程中模板自身形成的双螺旋发卡结构不稳定,导致引物有更多机会与模板结合,使得具有卡结构的模板能够有效扩增。为了使具有发卡结构模板能够有效扩增,探究了当引物位于发卡结构中茎部外侧时,模板扩增情况。研究发现,当引物位于模板发卡结构中茎部外侧时,具有发卡结构模板检测线是不具有发卡结构模板的检测线的100倍;另外,将2.1中具有发卡结构茎部长25 bp、环部长52 bp的模板进行10倍梯度稀释,使用与发卡结构中茎部完全互补的引物对其进行PCR扩增,发现其检测限为105拷贝,是引物位于发卡结构茎部外侧时模板检测线的100倍。由此可知,在扩增具有发卡结构模板时,使用位于发卡结构茎部两侧游离端的引物能够明显提高具有发卡结构模板的有效扩增。由于反向重复序列广泛存在于生物体基因组中[5],在实际PCR扩增过程中除本研究针对的情况外(反向重复序列位于扩增片段的上下游引物区),还可能出现重复序列分布于扩增子内部的情况,针对这类待扩增模板,可在引物设计时避开重复序列区,即引物结合区为非重复序列。
另外,探究了退火温度和引物浓度的改变对具有发卡结构模板扩增的影响。研究发现,较高的退火温度,有利于具有发卡结构模板的扩增。原因可能为,在PCR循环迅速降温的过程中分子内的互补链优先于外源引物而进行退火,在降低温度至Tm值以下后分子内退火形成的二级结构相对于温度较高时更稳定,导致外源引物无法与其结合,而用较高的退火温度,虽然在温度下降的过程中分子内部可能优先退火,但由于最终的退火温度较高,分子内的退火处于动态平衡,因此外源引物有更多的机会与模板进行退火进而发生聚合反应,从而提高扩增效率。通过对反应体系中不同引物浓度对具有发卡结构模板扩增的影响发现,提高反应体系中引物浓度,能够在一定程度上提高具有发卡结构模板的扩增量。原因可能是形成发卡结构的模板在退火时,引物竞争性与模板结合,提高引物浓度,相应提高了引物与模板结合的竞争能力。
最后,检测25 bp、20 bp、15 bp双链;长度为82 nt,发卡结构中茎长分别为25 bp、20 bp、15 bp的序列;茎部长度20 bp,环部长度分别为20 nt、30 nt、50 nt序列的熔点。发卡结构中茎部长度越长,形成的发卡结构熔点越高,并且发卡结构的熔点高于与茎部长度相同的双链的熔点;当发卡结构中茎部长度相同时,环部越长,其熔点越低。熔点的测定,从理论上支持了发卡结构中茎部长度的改变、环部长度改变以及退火温度改变时,发卡结构模板扩增规律原因的猜想。
4 结论在对具有反向重复序列的DNA进行PCR扩增时,反向重复序列形成的发卡结构会对DNA的PCR扩增产生抑制作用,并且这种抑制作用随发卡结构中茎部长度的变长而增强,随发卡结构中环部长度的变长而减弱。当引物与DNA中非反向重复序列部分结合时能够明显提高具有反向重复序列DNA的扩增效率。为了使具有反向重复序列的DNA能够有效扩增,在扩增具有反向重复序列的模板时,建议使用的引物尽量与DNA序列中非反向重复序列部分结合。另外发现,当使用引物与模板发卡结构中茎部结合时,适当提高退火温度和引物浓度也可以实现目标DNA的有效扩增。
| [1] | Lander ES, Linton LM, Birren B, et al. Initial sequencing and analysis of the human genome. Nature, 2001, 409 (6822): 860–921. DOI:10.1038/35057062 |
| [2] | 毛源泽, 韦芳萍, 岑华佗, 等. HIV全序列中重复序列的研究. 大众科技, 2012, 14(5): 131–133. |
| [3] | Richard GF, Kerrest A, Dujon B. Comparative genomics and molecular dynamics of DNA repeats in eukaryotes. Microbiology and Molecular Biology Reviews, 2008, 72 (4): 686–727. DOI:10.1128/MMBR.00011-08 |
| [4] | Akberova NI, et al.http://www.netsci.org/Science/Bioinform/feature05.html |
| [5] | Warburton PE, Giordano J, Cheung F, et al. Inverted repeat structure of the human genome :the X-chromosome contains a preponderance of large, highly homologous inverted repeats that contain testes genes. Genome Research, 2004, 14 (10a): 1861–1869. DOI:10.1101/gr.2542904 |
| [6] | Pearson CE, Zorbas H, Price GB, et al. Inverted repeats, stem-loops, and cruciforms :significance for initiation of DNA replication. Journal of Cellular Biochemistry, 1996, 63 (1): 1–22. DOI:10.1002/(ISSN)1097-4644 |
| [7] | Chew DSH, Choi KP, Leung MY. Scoring schemes of palindrome clusters for more sensitive prediction of replication origins in herpesviruses. Nucleic Acids Research, 2005, 33 (15): e134–e134. DOI:10.1093/nar/gni135 |
| [8] | Leung MY, Choi KP, Xia A, et al. Nonrandom clusters of palindromes in herpesvirus genomes. Journal of Computational Biology, 2005, 12 (3): 331–354. DOI:10.1089/cmb.2005.12.331 |
| [9] | Stauffer GV, Stauffer LT. Cloning and nucleotide sequence of the Salmonella typhimurium LT2 metF gene and its homology with the corresponding sequence of Escherichia coli. Molecular and General Genetics, 1988, 212 (2): 246–251. DOI:10.1007/BF00334692 |
| [10] | Gordenin DA, Lobachev KS, Degtyareva NP, et al. Inverted DNA repeats :a source of eukaryotic genomic instability. Molecular and Cellular Biology, 1993, 13 (9): 5315–5322. DOI:10.1128/MCB.13.9.5315 |
| [11] | Bi X, Liu LF. DNA rearrangement mediated by inverted repeats. Proceedings of the National Academy of Sciences of USA, 1996, 93 (2): 819–823. DOI:10.1073/pnas.93.2.819 |
| [12] | Eykelenboom JK, Blackwood JK, Okely E, et al. SbcCD causes a double-strand break at a DNA palindrome in the Escherichia coli chromosome. Molecular Cell, 2008, 29 (5): 644–651. DOI:10.1016/j.molcel.2007.12.020 |
| [13] | Lee GS, Neiditch MB, Sinden RR, et al. Targeted transposition by the V (D) J recombinase. Molecular and Cellular Biology, 2002, 22 (7): 2068–2077. DOI:10.1128/MCB.22.7.2068-2077.2002 |
| [14] | Butler DK, Yasuda LE, Yao MC. Induction of large DNA palindrome formation in yeast :implications for gene amplification and genome stability in eukaryotes. Cell, 1996, 87 (6): 1115–1122. DOI:10.1016/S0092-8674(00)81805-X |
| [15] | Inagaki K, Lewis SM, Wu X, et al. DNA Palindromes with a modest arm length of 20 base pairs are a significant target for recombinant adeno-associated virus vector integration in the liver, muscles, and heart in mice. Journal of Virology, 2007, 81 (20): 11290–11303. DOI:10.1128/JVI.00963-07 |
| [16] | Moore H, Greenwell PW, Liu CP, et al. Triplet repeats form secondary structures that escape DNA repair in yeast. Proceedings of the National Academy of Sciences, 1999, 96 (4): 1504–1509. DOI:10.1073/pnas.96.4.1504 |
| [17] | Butler DK, Gillespie D, Steele B. Formation of large palindromic DNA by homologous recombination of short inverted repeat sequences in Saccharomyces cerevisiae. Genetics, 2002, 161 (3): 1065–1075. |
| [18] | Kouzine F, Levens D. Supercoil-driven DNA structures regulate genetic transactions. Front Bioscience A Journal and Virtual Library, 2007, 12 (8-12): 4409–4423. DOI:10.2741/2398 |
| [19] | Dayn A, Malkhosyan S, Mirkin SM. Transcriptionally driven cruciform formation in vivo. Nucleic Acids Research, 1992, 20 (22): 5991–5997. DOI:10.1093/nar/20.22.5991 |
| [20] | Zannis-Hadjopoulos M, Yahyaoui W, Callejo M. 14-3-3 cruciformbinding proteins as regulators of eukaryotic DNA replication. Trends in Biochemical Sciences, 2008, 33 (1): 44–50. DOI:10.1016/j.tibs.2007.09.012 |
| [21] | Kim EL, Peng H, Esparza FM, et al. Cruciform-extruding regulatory element controls cell-specific activity of the tyrosine hydroxylase gene promoter. Nucleic Acids Research, 1998, 26 (7): 1793–1800. DOI:10.1093/nar/26.7.1793 |
| [22] | Shlyakhtenko LS, Hsieh P, Grigoriev M, et al. A cruciform structural transition provides a molecular switch for chromosome structure and dynamics. Journal of Molecular Biology, 2000, 296 (5): 1169–1173. DOI:10.1006/jmbi.2000.3542 |
| [23] | Singleton CK. Effects of salts, temperature, and stem length on supercoil-induced formation of cruciforms. Journal of Biological Chemistry, 1983, 258 (12): 7661–7668. |








