线粒体是细胞进行呼吸的主要场所,在细胞代谢旺盛的需能部位比较集中。线粒体主要功能是通过氧化磷酸化作用合成ATP,为细胞各种生理活动提供能量,固有“细胞发电厂”之称;同时线粒体电子传递链的本质作用是促进天冬氨酸的合成[1, 2]。线粒体是半自主细胞器,含有线粒体DNA,可以编码构成呼吸链复合物的若干亚基;具有独立的遗传体系,能够进行DNA复制、转录和蛋白质翻译。粟酒裂殖酵母的线粒体DNA编码25种tRNA、8种mRNA、2种rRNA和一个rnpB[3]。所以,线粒体DNA的正常转录翻译对于线粒体正常的执行自身的功能具有十分重要的意义。
线粒体功能障碍会引起线粒体代谢酶的缺陷,导致ATP合成障碍、能量来源不足。许多研究表明,线粒体功能异常与帕金森氏症、阿尔兹海默症、糖尿病、肿瘤的发生发展过程密切相关[4-8]。线粒体的表达障碍直接导致各种各样的疾病,如何治疗线粒体疾病受到较大关注。
线粒体蛋白质表达异常是线粒体功能紊乱的主要原因,而线粒体蛋白质组学正是从整体角度,分析线粒体的蛋白质组成、表达水平与修饰状态等的动态变化,旨在阐明线粒体内全部蛋白质的表达模式和功能模式,从而在蛋白质水平探索线粒体生理功能和相关疾病的联系。通过线粒体蛋白质组学可系统研究正常和病变组织中线粒体蛋白分布与表达的差异,从而为研究线粒体相关疾病的分子机制和以线粒体为靶标的药物研发奠定理论基础。近年来,人类基因组测序的完成、串级质谱和蛋白质数据库的发展加速了线粒体蛋白组学的研究。线粒体蛋白质组学的发展对线粒体相关疾病的研究起到了积极的推动作用[9]。
Shy1是SURF家族的蛋白。酵母基因组数据库预测shy1的缺失可能会导致线粒体表达障碍和Leigh综合征。Leigh综合征是由SURF1突变引起在婴儿期严重的脑肌病。shy1在芽殖酵母里的同源基因SHY1,在芽殖酵母中已经有人报道[10-12]。在芽殖酵母中,SHY1是哺乳动物SURF1基因的酵母同源物,编码呼吸所需要的线粒体蛋白质。还有文献报道,芽殖酵母Shy1是线粒体内膜的不可或缺的组成部分,是酵母细胞色素c氧化酶的高效组装所必须的。本研究旨在揭示粟酒裂殖酵母中同源蛋白Shy1的定位及相关功能。首先,通过生物信息学对粟酒裂殖酵母的线粒体信号肽进行分析和观察Shy1的GFP绿色荧光位置,确定Shy1的定位;为了研究粟酒裂殖酵母蛋白Shy1的功能,以单倍体酵母菌株yHL6381为出发菌株构建shy1基因缺失突变株,研究shy1缺失菌株在非发酵性培养基上的生长状况;最后,通过提取线粒体,利用Western blotting技术进一步分析shy1的缺失对线粒体相关蛋白表达量的影响。
1 材料与方法 1.1 材料 1.1.1 菌株与质粒E.coli Top10、粟酒裂殖酵母单倍体菌株yHL6381(h+,his3-D1,leu1-32,ura4-D18,ade6-M210)、pFA6a-KanMX6、pYJ19又称(pJK148-nmt1new-trz1-gfp)为本实验室保存。
1.1.2 培养基LB培养基:蛋白胨10 g/L,酵母膏5 g/L,氯化钠10 g/L,固体培养基添加1.5%的琼脂,固体和液体培养基在需要时添加氨苄霉素至50 mg/L。YES培养基:葡萄糖30 g/L,酵母膏5 g/L,4种添加营养物(如ade,leu,ura,his)至终浓度为225 mg/L,固体培养基添加2%的琼脂。YES+3%甘油:yeast extract 5 g/L,glycerol 3%,4种添加营养物(ade,leu,ura,his)至终浓度为225 mg/ L,固体培养基添加2%的琼脂。YES+6%甘油:yeast extract 5 g/L,glycerol 6%,4种添加营养物(如ade,leu,ura,his)至终浓度为225 mg/L,固体培养基添加2%的琼脂。YES+KanMX抗性(KanMX是一种氨基苷类抗菌素衍生物,对真核细胞均有毒性作用,能阻断细胞内蛋白质合成。KanMX用在酵母体系中,可扩大筛选使用菌株的范围,便于提高外源基因的表达产量):每100 mL YES固体培养基+100 μL KanMX,KanMX的浓度为100 mg/mL;EMM(Edinburgh minimal medium)-leu培养基(g/L):3 g邻苯二甲酸氢钾,5.55 g十二水合磷酸氢二钠,5 g氯化铵,10 mL 100×salt stock,1 mL的1 000×vitamin,0.1 mL 10 000×mineral stock,20 g葡萄糖,3种添加营养物(如ade,ura,his)至终浓度为225 mg/L,葡萄糖分开灭菌,固体培养基加2.0%的琼脂粉。
1.1.3 仪器与试剂各种限制性内切酶、solution I[kite code.6022]、rTaq DNA聚合酶、PrimeSTAR DNA Polymerase、蛋白marker、均购自TaKaRa公司;DNA割胶回收试剂盒、PCR过柱纯化试剂盒购自博尔迪公司;DNA marker购自北京全式金生物技术有限公司;PCR引物由上海生工生物工程技术服务有限公司合成;氨苄霉素购自Sigma公司。
1.2 方法 1.2.1 生物信息学分析粟酒裂殖酵母shy1基因序列来源于酵母基因组数据(S.pombe_GeneDB,http://www.pombase.org/);粟酒裂殖酵母Shy1的查找使用NCBI数据库(http://www.ncbi.nlm.nih.gov/),芽殖酵母同源蛋白SHY1的查找使用SGD数据库(http://www.yeastgenome.org/);用AlignX对所查到蛋白序列进行Clustal W序列比对分析;线粒体信号肽预测分析采用MitoProt Ⅱ(http://ihg.gsf.de/ihg/mitoprot.html)。
1.2.2 PCR扩增Shy1-GFP融合蛋白的构建:根据S.pombe_GeneDB数据库中shy1(SPBC1215.01)的基因序列设计上下游引物:shy1-Pst I-up:5'-AACTGCAGATGTTTTGGTGGAAAAGTGCTACT-3',shy1-Sal I-down:5'-ACGCGTCGACTAATCGTTTGTTGTTTAGAATCTTGC-3'(下划线代表酶切位点)。以粟酒裂殖酵母yHL6381基因组为模板扩增目的片段,扩增条件为98℃ 2 min,98℃ 10 s,60℃ 15 s,72℃ 1 min,30个循环;72℃ 5 min,4℃保存,扩增产物用1.0%琼脂糖凝胶电泳检测。
构建重组质粒shy1-del-KanMX:根据shy1的基因序列设计上游同源臂和下游同源臂的引物:shy1-5-Sma I-up:5'-TCCCCCGGGGCATAAGTAGTGTGGGAGCAAG-3',shy1-5-Bgl II-dw:5'-GAAGATCTGATAGACAGGAATCCAAGATTCAATCG-3',shy1-3-Sac I-up:5'-CGAGCTCAAAATTTTCATATTTATAAGTTTCTAAATATTATCTACC-3',shy1-3-Spe I-dw:5'-GACTAGTCGATTATGAGAACTCCACGCCT-3'。扩增条件和模板同上。
1.2.3 重组质粒的构建和鉴定纯化的Shy1-GFP的扩增片段和pYJ19空载质粒,用Pst Ⅰ和SalⅠ双酶切、纯化、酶连;将酶连产物导入感受态细胞中,涂布到含氨苄抗性的LB培养基。随机挑取转化子用碱裂解法提取质粒,进行Pst Ⅰ和Sal Ⅰ双酶切验证。酶切正确的重组质粒委托南京思普金生物科技有限公司进行DNA测序。
重组质粒shy1-del-KanMX构建时,先构建用上游同源臂,再构建下游同源臂。上游同源臂所用酶切位点为Sma Ⅰ和Bgl Ⅱ,下游同源臂所用酶切位点Sac Ⅰ和Spe Ⅰ。构建及验证过程同上。
1.2.4 将目的片段转入酵母菌株及菌种验证将构建好的Shy1-GFP重组质粒用Nru Ⅰ单切以获得目的片段;以shy1-del-KanMX重组质粒为模板,shy1-5-Sma Ⅰ-up和shy1-3-Spe Ⅰ-dw为引物PCR,扩增出目的片段;经过醋酸锂转化进入酵母菌株中,通过同源重组的方式整合到酵母的基因组上。
荧光显微镜观察验证shy1-gfp菌株。PCR验证Δshy1菌株:以粟酒裂殖酵母yHL6381(作为对照)和醋酸锂转化子的基因组为模板,shy1-5-yz:5'-ATCACTGCAGTTAAGCGTCCTGAG-3'和shy1-3-yz:5'-CAAGACAGCATA-GTAAACGGCG-3'为引物PCR,比较野生型菌株与Δshy扩增片段的大小。
1.2.5 ∆shy1在非发酵培养基上的生长实验将活化好的yHL6381、∆shy1的单菌落分别接种于10 mL的YES液体中,于30℃,220 r/min恒温摇床中培养过夜;将种子液转接至起始OD600为0.2左右,于30℃,220 r/min恒温摇床中继续培养至OD600为1-2之间;收菌,水洗;调整初始OD600为3左右,10倍差异梯度稀释,每个浓度各取3 μL点圈于YES、YES+3%甘油、YES+6%甘油培养基;30℃恒温培养箱培养5 d,拍照。
1.2.6 荧光显微镜观察Shy1的定位挑取LiAc转化子单菌落划线于相应的选择性平板,挑取少量菌于EMM-leu液体中,转接使起始OD600约0.2,培养6-8 h后,OD600约0.8到1之间,取1 mL菌液PBS洗细胞一次,最终悬浮于200 μL PBS(含有终浓度为2 nmol/L的染料)中,30℃,染色1-2 min,加入1 mL PBS稀释染料,离心弃上清,加入100 µL PBS,取3 µL用于显微镜观察使用。制片后,利用Zeiss Axio imager A1 microscope(Zeiss,Jena,Germany)进行荧光观察。先调节粗准焦螺旋再调节细准焦螺旋,在显微镜中找到细胞,并在DIC通道拍照,然后转换显微镜至GFP通道(GFP滤光片为Catalog No.41017),激发波长为488 nm,GFP信号通过Chroma(Brattleboro,VT)获得,观察荧光信号,并拍照,最后转换显微镜至DsRed通道观察红色荧光信号,并拍照[13, 14]。相机系统为Sensicam QE cooled digital camera system(Cooke Corp);图像整合系统为MetaMorph/MetaFluor combination package analysis software(Uniwersal Imaging,West Chester,PA);图像后期处理使用Adobe Photoshop7.0(Adobe,San Jose,CA)。
1.2.7 ∆shy1菌株中线粒体蛋白表达量的检测提取∆shy1、yHL6381菌株的线粒体[15, 16]用于Western blotting检测。处理线粒体的粗提液:线粒体蛋白Cox2、Cox4、内参Hsp60,在100℃处理10 min,Cox1、Cob1、Cox3在45℃处理3 min。Western blot-ting检测用ODDESSY进行扫描显色。本研究选择线粒体基质的蛋白Hsp60作为内参,检测线粒体相关蛋白Cox1、Cox2、Cox3、Cob1、Cox4的表达量,抗体及稀释比例分别为anti-HSP60(1:1 000),anti-Cox1(1:1 000),anti-Cox2(1:500),anti-Cox3(1:400),anti-Cox4(1:1 000)and anti-Cob1(1:500)。
2 结果 2.1 重组质粒的构建及酶切验证重组质粒Shy1-GFP的目的片段的PCR扩增结果(图 1-A)显示,在750-1 000 bp之间且有单一、清晰的条带,与目的基因873 bp相吻合;重组质粒Shy1-GFP双酶切,在750-1 000 bp之间有释放片段,大小与PCR扩增片段一致图(1-B),酶切正确的重组质粒进行DNA测序;重组质粒shy1-del-KanMX上游同源臂和下游同源臂扩增产物结果(图 1-C),与目的片段大小634 bp和545 bp相吻合;重组质粒shy1-del-KanMX上游同源臂酶切后释放片段(黑色粗箭头处)与PCR大小相符(图 1-D),进行DNA测序;重组质粒shy1-del-KanMX酶切后释放片段(黑色粗箭头处)大小相符(图 1-E),进行DNA测序;即本实验成功构建了重组质粒Shy1-GFP和shy1-del-KanMX。

|
| 图 1 重组质粒的构建及酶切验证 M:DNA分子量标准;(100 bp-1 kb);A:1-6号泳道为重组质粒Shy1-GFP的目的片段的PCR扩增;B:1-3号泳道是Pst Ⅰ和Sal Ⅰ对重组质粒Shy1-GFP双酶切验证;C:1-4号泳道为重组质粒shy1-del-KanMX上游同源臂PCR扩增,5-8号泳道为重组质粒shy1-del-KanMX下游同源臂PCR扩增;D:1-4泳道号为Sma Ⅰ和Bgl Ⅱ对含有上游同源臂的重组质粒shy1-del-KanMX的双酶切验证;E:1-2号泳道为Sac Ⅰ和Spe Ⅰ对含有下游同源臂的重组质粒shy1-del-KanMX的双酶切验证 |
由图 2-A可以看到绿色荧光,即菌株shy1-gfp构建成功;PCR扩增结果(图 2-B)显示,敲除菌株扩增出目的基因比野生型菌株扩增出目的基因大,说明shy1基因缺失突变株构建成功。

|
| 图 2 菌株shy1-gfp和Δshy1的验证 A:通过荧光显微镜观察鉴定菌株shy1-gfp;B:野生型菌株扩增出目的基因大小应为2 723 bp,敲除菌株扩增出目的基因大小应为3 124 bp(M:DNA分子量标准(100-1 000 kb) |
通过在GeneDB和NCBI数据库的搜索,得到裂殖酵母shy1基因序列,其系统名为SPBC1215.01。Shy1蛋白由290个氨基酸残基组成,预测的分子量约为33.38 kD(图 3)。

|
| 图 3 芽殖酵母SHY1和裂殖酵母Shy1的蛋白序列比对 “*”表示参与比对的序列在该位点是完全一致的;“:”表示在该列序列可以观察到保守的替换;“.”表示在该列序列可以观察到半保守的替换同源性分析结果显示,裂殖酵母shy1基因编码的蛋白和芽殖酵母中同源蛋白SHY1有大约36%的一致性 |
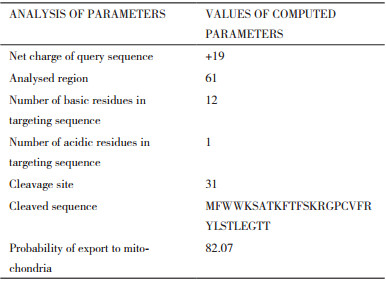
借助在线工具MitoProt Ⅱ对Shy1含有定位于线粒体的信号肽的可能性进行分析。通过MitoProt Ⅱ的预测,SpShy1p存在定位于线粒体概率为82.07%。实验已证明,对已知定位于线粒体的信号肽预测结果大于76%[17],可以确定SpShy1p中含有定位于线粒体的信号肽。根据预测结果(表 1),SpShy1p定位于线粒体的信号肽为其N端的30 aa残基,具体序列为:MFWWKSATKFTFSKRGPCVFRYLSTLEGTT。
根据预测结果(表 1),可以得知靶向序列中含有12个碱性残基和1个酸性残基,Shy1在细胞质合成后,运往线粒体的过程中,在第31为氨基酸处切割,线粒体信号肽不进入线粒体;线粒体的信号肽为其N端的30 aa残基,具体序列为:MFWWKSATKFTFSKRGPCVFRYLSTLEGTT,Shy1输入线粒体的概率为82.07%。
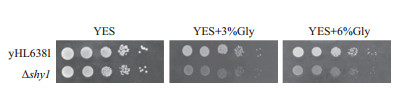
2.4 shy1的缺失导致在以甘油为唯一碳源的培养基上生长缺陷为了确定Shy1在粟酒裂殖酵母中的功能,本实验研究了shy1基因缺失突变株在以甘油为唯一碳源的非发酵培养基上的生长状况。实验结果(图 4)显示,与yHL6381的生长情况相比,在以葡萄糖为碳源的YES培养基上,shy1基因缺失突变株和yHL6381生长状况基本一致;而在以甘油为唯一碳源的非发酵性培养基上,shy1基因缺失突变株生长受损,表现出明显的呼吸缺陷。说明Δshy1是呼吸缺陷菌株,即shy1的缺失影响粟酒裂殖酵母线粒体功能的正常发挥。
|
| 图 4 Δshy1在非发酵培养基上的生长情况 |
生物信息学分析可知,Shy1含有线粒体的信号肽的概率高达82.07%。为了进一步确定Shy1的定位,用线粒体示踪染料来标记线粒体的位置,作为线粒体的Marker。荧光显微镜观察可以看到红色荧光;含有绿色荧光标签的shy1-gfp菌株在荧光显微镜观察下呈现绿色荧光。如图 5所示,同一视野下的绿色荧光和红色荧光可以重叠,Shy1与线粒体共定位,即Shy1定位于线粒体。

|
| 图 5 shy1-gfp的荧光显微镜观察 RFP:线粒体红色荧光探针染色后的观察结果;GFP:绿色荧光蛋白示踪结果;Merge:将MitoTracker Red染料和GFP观察结果融合 |
由已取得的实验结果可知,shy1缺失菌株在非发酵型培养基上生长明显受损,影响粟酒裂殖酵母线粒体功能的正常发挥。为了探究这种受损的原因,在实验室已有实验条件的前提下,通过Western blotting分析线粒体复合体中部分亚基的含量。Cox1、Cox2、Cox3参与组成呼吸链复合体Ⅳ,Cob1则参与组成呼吸链复合体Ⅲ,这些都属于线粒体自身编码的蛋白,Cox4是由核编码定位于线粒体的蛋白。
与野生型菌株yHL6381相比,Δshy1菌株中Cox1、Cox2、Cox3、Cox4、Cob1蛋白在线粒体中的含量明显降低(图 6),即shy1的缺失导致线粒体呼吸链相关蛋白的表达量显著降低。这些蛋白表达量的下降使得线粒体呼吸链功能严重受损,这也就解释了Δshy1缺失突变体在非发酵碳源培养基上不能正常生长的原因。总之,shy1的缺失导致线粒体呼吸链复合物的合成缺陷,使得线粒体呼吸链功能严重受损。

|
| 图 6 Western Blot检测Δshy1线粒体蛋白质的表达量 |
根据人类的基因图谱,估计大约有1 000-2 000种线粒体蛋白,大约有600多种已经被鉴定出来。线粒体蛋白质只有2%是线粒体自己合成的,98%的线粒体蛋白质是由细胞核编码、细胞质核糖体合成后运往线粒体的;粟酒裂殖酵母线粒体中含有300多个蛋白,其中有8个蛋白由线粒体基因组编码的,其他的也均由细胞核基因组编码。线粒体的蛋白质参与机体许多生理、病理过程,线粒体蛋白质的研究日益受到关注。
利用突变体进行基因功能研究是经典遗传学的重要方法,在粟酒裂殖酵母中经典的基因敲除方法是通过同源重组将外源基因定点整合到靶细胞基因组上某一确定的位点,以达到定点修饰改造染色体上某一基因的目的。它克服了随机整合的盲目性和偶然性,是一种理想的修饰、改造生物遗传,并进一步推测被替代基因功能的方法。为了研究Shy1的结构与功能,本研究首先获得了shy1基因缺失菌株。
特定基因的突变往往导致酵母菌株表型的变化,根据表型变化确定基因功能是检测基因功能最直接的方法之一。对于酵母而言,存在以葡萄糖为碳源的发酵型培养基和以甘油为唯一碳源的非发酵型培养基,这两种培养基涉及线粒体的功能差异,发酵型不需要呼吸作用,非发酵的需要呼吸作用,即发酵型不依赖线粒体,而非发酵的需要线粒体功能的正常发挥[18]。首先构建shy1基因缺失菌株,在以甘油为唯一碳源的非发酵型培养基上表现出明显的生长缺陷。在以甘油为唯一碳源的情况下,细胞不能进行糖酵解过程只能通过线粒体进行呼吸作用,但是敲除菌由于线粒体受损不能正常利用甘油进行三羧酸循环为细胞提供能量,最终导致在甘油培养基上不能正常生长。
线粒体呼吸链主要由线粒体呼吸链酶,包括NADH脱氢酶(也称为复合物Ⅰ)、琥珀酸氧化还原酶(也称为复合物Ⅱ)、细胞色素c还原酶(复合物Ⅲ)、细胞色素c氧化酶(也称为复合物Ⅳ)和ATP合成酶(也称为复合物Ⅴ)组成。Cob1由复合物Ⅲ编码,Cox1、Cox2、Cox3由复合物Ⅳ编码,而Atp6、Atp8、Atp9则由复合物Ⅴ编码。线粒体呼吸链酶的作用为通过一系列的氧化还原过程最终形成ATP,为肌体组织提供能量。线粒体呼吸链酶复合物缺陷是导致线粒体病的重要原因。细胞色素氧化酶基因上的缺陷,包括突变,能够导致严重的常常甚至是致命的代谢紊乱[19-21]。这种紊乱通常出现在幼儿时代的早期,并且主要影响需要高能量的组织器官(脑、心脏、肌肉)。在许多分级的线粒体疾病中,与细胞色素氧化酶组装缺陷的疾病被认为是最严重的。在shy1基因缺失菌株中,线粒体呼吸链相关蛋白Cox1、Cox2、Cox3、Cob1、Cox4的表达量下降,即shy1的缺失破坏了线粒体呼吸链的功能。
根据序列比对,裂殖酵母Shy1和芽殖酵母中同源蛋白SHY1有一定的同源性,且具有芽殖酵母的功能保守位点,即第137位的G和344位的Y。在芽殖酵母里,这两个功能位点影响复合物的组装;这两个位点突变后,线粒体呼吸链复合体不能正常组装,导致呼吸受损。在裂殖酵母中,初步推测Shy1具有类似芽殖酵母Shy1的功能,即参与线粒体呼吸链复合体的正常组装。因而,shy1的缺失导致在以甘油为唯一碳源的非发酵培养基上生长缺陷和线粒体蛋白表达的下降。
4 结论粟酒裂殖酵母shy1基因大小为873 bp,共编码290个氨基酸。本文主要研究的为芽殖酵母同源蛋白SHY1的同源Shy1,经序列比对发现两者的同源性为36%。线粒体信号肽分析和GFP荧光观察,最终确定Shy1是一个定位于线粒体的蛋白。同时,在以甘油为唯一碳源的非发酵型培养基上shy1缺陷菌株表现出明显的生长缺陷,后续研究发现这种呼吸呼吸受损影响的生长缺陷是由于Shy1缺失导致线粒体呼吸复合体不能正常组装,目前证明复合物Ⅵ的部分亚基包括Cox1、Cox2、Cox3、复合物Ⅲ的Cob1、细胞核编码的蛋白Cox4的含量都发生明显的减少,最终导致线粒体呼吸不能正常进行而引发在甘油培养基上生长缺陷。
| [1] | Sullivan LB, Gui DY, Hosios AM, et al. Supporting aspartate biosynthesis is an essential function of respiration in proliferating cells. Cell, 2015, 162 (3): 552–563. DOI:10.1016/j.cell.2015.07.017 |
| [2] | Birsoy K, Wang T, Chen WW. An essential role of the mitochondrial electron transport chain in cell proliferation is to enable aspartate synthesis. Cell, 2015, 162 (3): 540–551. DOI:10.1016/j.cell.2015.07.016 |
| [3] | Kühl I, Dujeancourt L, Gaisne M, et al. A genome wide study in fission yeast reveals nine PPR proteins that regulate mitochondrial gene expression. Nucleic Acids Research, 2011, 39 (18): 8029–8041. DOI:10.1093/nar/gkr511 |
| [4] | Cleland MM, Youle RJ. Mitochondrial dynamics and apoptosis[M]. Springer Netherlands, 2011: 109-138. |
| [5] | 程书珍, 苏安英, 高丽萍, 等. 阿尔兹海默病中细胞凋亡的机制研究进展. 邯郸医学高等专科学校学报, 2003, 16(5): 487–489. |
| [6] | 马丽晶, 徐勉. 线粒体基因突变所致糖尿病发病机制及治疗进展. 医学综述, 2010, 16(2): 275–277. |
| [7] | 王勉. 线粒体与肿瘤关系的研究进展. 承德医学院学报, 2009, 26(3): 320–324. |
| [8] | 张颖, 胡国华. 线粒体呼吸链功能异常在帕金森病中作用的研究进展. 中风与神经疾病杂志, 2010, 27(1): 89–91. |
| [9] | Lemaire C, Dujardin G. Preparation of respiratory chain complexes from Saccharomyces cerevisiae wild-type and mutant mitochondria : Activity measurement and subunit composition analysis. Methods in Molecular Biology, 2008, 432 : 65–81. DOI:10.1007/978-1-59745-028-7 |
| [10] | Reinhold R, Bareth B, Balleininger M, et al. Mimicking a SURF1 allele reveals uncoupling of cytochrome c oxidase assembly from translational regulation in yeast. Human Molecular Genetics, 2011, 20 (12): 2379–2393. DOI:10.1093/hmg/ddr145 |
| [11] | Nijtmans LG, Sanz MA, Bucko M, et al. Shy1p occurs in a high molecular weight complex and is required for efficient assembly of cytochrome c oxidase in yeast. Febs Letters, 2001, 498 (1): 46–51. DOI:10.1016/S0014-5793(01)02447-4 |
| [12] | Mashkevich G, Repetto B, Glerum DM, et al. Shy1, the yeast homolog of the mammalian SURF-1 gene, encodes a mitochondrial protein required for respiration. Journal of Biological Chemistry, 1997, 272 (22): 14356–14364. DOI:10.1074/jbc.272.22.14356 |
| [13] | Greenberg RA, Sobhian B, Pathania S, et al. Multifactorial contributions to an acute DNA damage response by BRCA1/BARD1-containing complexes. Genes & Development, 2006, 20 (1): 34–46. |
| [14] | Chalfie M. Green fluorescent protein as a marker for gene expression. Science, 1994, 263 (5148): 151. |
| [15] | Meisinger C, Pfanner N, Truscott KN. Isolation of yeast mitochondria. Methods in Molecular Biology, 2006, 313 (313): 33–9. |
| [16] | Boldogh IR, Pon LA. Purification and subfractionation of mitochondria from the yeast Saccharomyces cerevisiae. Methods in Cell Biology, 2007, 80 (9): 45–64. |
| [17] | Claros MG, Vincens P. Computational method to predict mitochondrially imported proteins and their targeting sequences. European Journal of Biochemistry, 1996, 241 (3): 779–786. DOI:10.1111/ejb.1996.241.issue-3 |
| [18] | De JP, van Dijken JP, Pronk JT. Metabolic fluxes in chemostat cultures of Schizosaccharomyces pombe grown on mixtures of glucose and ethanol. Microbiology, 1996, 142(Pt 6) (1): 1399–1407. |
| [19] | Herbert CJ, Golik P, Bonnefoy N. Yeast PPR proteins, watchdogs of mitochondrial gene expression. Rna Biology, 2013, 10 (9): 1477–1494. DOI:10.4161/rna.25392 |
| [20] | Roloff GA, Henry MF. Mam33 promotes cytochrome c oxidase subunit itranslation in Saccharomyces cerevisiae mitochondria. Molecular Biology of the Cell, 2015, 26 (16): 2885–2894. DOI:10.1091/mbc.E15-04-0222 |
| [21] | Mootha VK, Lepage P, Miller K, et al. Identification of a gene causing human cytochrome c oxidase deficiency by integrative genomics. Proceedings of the National Academy of Sciences, 2003, 100 (2): 605–610. DOI:10.1073/pnas.242716699 |