2. 中南大学教育部生物冶金重点实验室,长沙 410083
2. Key Laboratory of Biometallurgy of Ministry of Education, Central South University, Changsha 410083
硫化矿生物浸出技术在复杂低品位原生硫化矿生物提取、金矿表面硫化矿的氧化预处理、以及硫化矿尾矿的治理和金属回收等方面可以发挥重大作用[1]。常见的硫化矿生物浸出微生物可根据其最适生长温度划分为常温微生物(30-40℃)、中度嗜热微生物(40-60℃)和极端嗜热微生物(80℃以上)[2]。由于嗜热菌与嗜中温菌相比具有耐高温、提取速率快和浸出率高等特点,在工业应用上有着较大的潜力。然而目前关于嗜酸热古菌-硫化矿作用下的硫氧化过程研究极少,人们对嗜酸热古菌硫氧化过程与高温生物浸出的关系缺乏清晰的认识和了解[3]。
单质硫(S0)的生物氧化过程十分复杂,基于对典型中温菌Acidithiobacillus ferrooxidans ATCC 23270的研究表明,S0必须经过嗜酸硫氧化菌胞外蛋白对其有效的活化作用,并经膜蛋白的转运进入细胞周质空间后,才能被细菌有效地氧化[4]。在这几个步骤中胞外蛋白对S0的活化作用起着重要的作用,决定着整个S0氧化过程效率的高低,并且已有研究表明S0的活化需要胞外和外膜上富含巯基(-SH)蛋白的参与反应形成-SnH(n≥2)的复合体才能进一步被氧化[5, 6]。我们课题组之前基于比较蛋白质组学和同步辐射的原位巯基表征,验证了-SH在中温菌A. ferrooxidans硫活化中的重要作用[7, 8]。由于嗜酸热古菌具有独特的生理生化特性,其S0的活化机制及其与A. ferrooxidans的区别与联系还不清楚,因此,进一步基于比较蛋白质组学的方法开展嗜酸热古菌胞外S0活化相关蛋白质的筛选和作用机制的研究,有助于揭示嗜酸热古菌对S0的活化途径和氧化利用机制[9, 10]。
本研究基于比较蛋白质组学的方法,对典型极端嗜酸热古菌Acidianus manzaensis进行研究,以期获得A. manzaensis菌胞外与S0活化相关的蛋白质基因,为以后阐明嗜酸热古菌硫活化氧化机制的相关研究奠定基础。
1 材料与方法 1.1 材料主要试剂有丙烯酰胺,甲叉双丙烯酰胺,苯甲基磺酰氟(PMSF),十二烷基硫酸钠(SDS),过硫酸铵,甘氨酸,考马斯亮蓝G-250,考马斯亮蓝R-250,二硫苏糖醇(DTT),碘乙酰胺,Tris-碱,溴酚蓝,尿素,硫脲,丙三醇,丙酮,8-10 kD透析袋等均购自于北京鼎国昌盛生物技术有限责任公司;固相pH梯度干胶条(IPG干胶条,pH3-10,24 cm)IPG buffer(pH3-10)Ettan IP Gphor 3等电聚焦系统和电泳系统均购自于GE公司;质谱鉴定在串联飞行时间质谱仪(5800 MALDI-TOF/TOF,AB SCIEX)上完成;TIANamp细菌基因组DNA提取试剂盒和TIANamp总RNA提取试剂盒均购自于天根生化科技有限公司;ReverTra Ace-α-第一链cDNA合成试剂盒,HUNDERBIRDTM SYBR® qPCR Mix均购自于日本东洋纺株式会社生命科学事业部(Toyobo co.,LTD.,Osaka,Japan);RT-qPCR在iCycler iQTM Real-time PCR仪(Bio-Rad Laboratories,Inc.,Hercules,USA)上完成。
1.2 方法 1.2.1 菌株及培养基菌株A. manzaensis YN-25由中南大学生物冶金重点实验室提供[11],A. manzaensis培养于9K基础培养基(0.5 g/L,MgSO4·7H2O;0.5 g/L,K2HPO4;3.0 g/L,(NH4)2SO4;0.1 g/L,KCl;0.01 g/L,Ca(NO3)2),添加0.02%(W/V)酵母浸出液,并分别加入10 g/L的S0、20 g/L的FeSO4·7H2O作为能源底物。A. manzaensis菌在S0为能源底物时初始pH用硫酸调为2.0,在Fe2+为能源底物培养时的初始pH调为1.5;取200 mL上述培养基于500 mL的三角瓶中在121℃灭菌锅中灭菌20 min。A. manzaensis的初始接种量均为107个/mL,置于65℃、170 r/min的空气浴摇床中培养至对数中期,4℃下10 000×g离心15 min收集细胞,菌体先用pH2.0的稀硫酸轻轻震荡洗涤除去杂质后离心收集菌体,然后悬浮于pH7.3的磷酸盐缓冲液(PBS)中。
1.2.2 不同能源底物下生长的A. manzaensis胞外蛋白质的提取胞外硫活化蛋白的提取采取80℃热水浴加热的方法[12],并以Fe2+为能源底物下培养的A. manzaensis提取的胞外蛋白质作为对照筛选出与硫活化相关的胞外蛋白质。具体步骤如下:(1)分别将在Fe2+和S0培养至对数期下收集的细胞悬液于80℃水浴条件下缓慢摇动孵育;(2)4℃下10 000×g离心15 min收集上清液即为胞外蛋白质;(3)向胞外蛋白质溶液中加入4倍体积的冷丙酮,并于-20℃下过夜静置沉淀蛋白质;(4)将第(3)步中用丙酮沉淀产生的蛋白质于4℃下10 000×g离心30 min,弃上清,加入适量超纯水溶解沉淀所得蛋白质,置于8-10 kD的透析袋中用灭菌的超纯水透析96 h(每12 h换一次水);(5)透析后的蛋白质经冷冻干燥后溶解于裂解液(7 mol/L尿素,2 mol/L硫脲,4% CHAPS,1% NP-40,2% IPG缓冲液,65 mmol/L DTT),Bradford法[13]测定蛋白质浓度后于-80℃保存备用。为确定最佳的水浴时间在步骤(1)中选取不同的水浴处理时间,对离心收集到的上清液采用考马斯亮蓝法测定其中的蛋白浓度,并绘制水浴时间和胞外蛋白质含量图。
1.2.3 双向电泳分离蛋白质双向电泳采用固相pH梯度干胶条(pH3-10,24 cm),上样量为500 mg。分别将上述A. manzaensis在S0/Fe2+为能源底物时提取得到的含500 mg蛋白质裂解液与600 μL水化液(7 mol/L尿素,2 mol/L硫脲,2% CHAPS,1% NP-40,0.5% IPG缓冲液,18 mmol/L DTT以及0.002%的溴酚蓝)充分混匀后采用胶内水化上样方式上样[14]。等电聚焦结束后取出胶条,将IPG胶条于10 mL平衡缓冲液(6 mol/L尿素,2% SDS,50 mmol/L、pH8.8 Tris-HCl,30%甘油,0.002%溴酚蓝)中水平摇床振动平衡两次,每次15 min。第一次在10 mL平衡缓冲液中加入1% DTT;第二次在10 mL平衡缓冲液中加入2.5%碘乙酰胺。平衡好的胶条移至12.5%聚丙烯酰胺凝胶上端,上部用0.5 %低熔点琼脂糖封胶后进行二向垂直平板SDS-PAGE。取出电泳结束后的第二向SDS-PAGE用考马斯亮兰R-250染色。
1.2.4 A. manzaensis胞外硫活化相关蛋白质胶内水解与质谱鉴定采用ImageMaster 2D Platinum 6.0软件对双向电泳分离得到的图谱进行分析,以Fe2+为能源底物的电泳图谱作为对照,筛选出在以S0为能源底物时差异表达的斑点,选择挖取蛋白质点,转入1.5 mL的离心管中,用去离子水反复清洗后进行蛋白质斑点胶块脱色、胶内蛋白质胰蛋白酶水解[15]。质谱鉴定采用串联飞行时间质谱仪(5800 MALDI-TOF/TOF,AB SCIEX)进行测试分析,参数设定:Nd:YAG激光器发射激光源波长355 nm,加速电压2.0 kV;采集数据时用正离子模式以及自动获取数据模式;其中一级质谱(MS)范围为800-4 000 Da,根据MS结果选择信噪比大于50的母离子进行二级质谱(MS/MS)分析,每组实验样品选择8个母离子,MS/MS累计叠加2 500次,碰撞能量2.0 kV,CID闭。
1.2.5 数据库检索鉴定蛋白质质谱测试后获得的原始文件用Mascot 2.2软件检索NCBI和Swissprot数据库,对蛋白质进行鉴定,检索鉴定的蛋白质参考NCIB(http://www.ncbi.nlm.nih.gov/)数据库公布的信息进行功能分类和生物信息学分析[16]。
1.2.6 RT-qPCR验证筛选蛋白质在硫活化中的作用以数据库检索鉴定的A. manzaensis在S0为能源底物时表达量明显上调的胞外蛋白基因,采用Primer 3(v.0.4.0)(http://bioinfo.ut.ee/primer3-0.4.0/primer3/)在线设计引物并由上海生工公司合成,用于RT-qPCR的细胞收集于对数中期。总RNA的提取和纯化采用总RNA提取试剂盒(天根生化科技有限公司),然后立刻以总RNA作为模板用ReverTra Ace-α-第一链cDNA合成试剂盒(Toyobo co.,LTD.,Osaka,Japan)反转录合成cDNA,均按试剂盒说明操作。以上述反转录所得cDNA为模板,使用高效率SYBR荧光定量PCR mix QPK-201(Toyobo co.,LTD.,Osaka,Japan)配制反应体系后在iCycler iQTM Real-time PCR仪(Bio-Rad Laboratories,Inc.,Hercules,USA)上进行,每个反应设3个重复。对RT-qPCR采集的数据,用16S rDNA基因作为内部参照校正,用(1+E)-ΔΔCq法进行计算(其中E为扩增效率,Cq值为荧光信号达到域值时的循环数),分析各个基因在不同能源底物培养时的相对表达量[17]。
2 结果 2.1 热水浴法提取胞外蛋白时间的确定通过测定水浴时间和上清液中胞外蛋白质含量所得的关系图(图 1)可知,A. manzaensis分别在S0和Fe2+生长时收集的相同体积菌悬液(在600 nm波长下吸光值均为1),在80℃水浴中缓慢摇动孵育时均随着时间的延长胞外蛋白的提取量增多,而且由图 1还可知A. manzaensis在S0中生长时的胞外蛋白质含量明显高于在以Fe2+为能源底物时的胞外蛋白质含量,说明在在S0能源底物下A. manzaensis分泌更多的胞外蛋白质。水浴30 min后蛋白质含量均不再增加,因此为保证在既能充分提取胞外蛋白又不对细胞进行破坏的前提下,选择80℃水浴时间30 min提取胞外蛋白质。
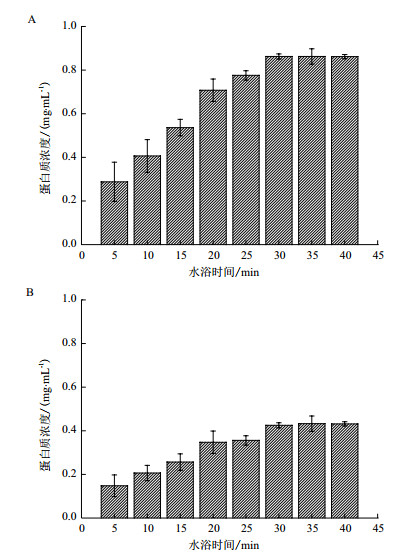
|
| 图 1 80℃水浴时间和A.manzaensis分别在S0(A)和Fe2+(B)能源底物下生长时细胞胞外蛋白质提取量关系图 |
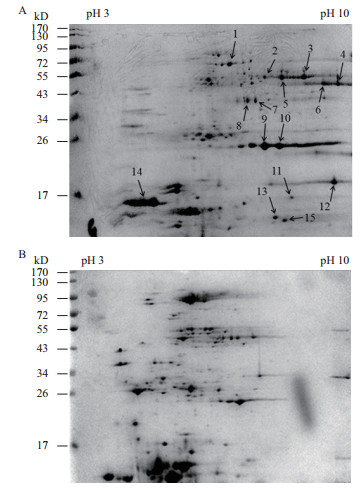
A. manzaensis在S0/Fe2+能源底物下胞外蛋白质的2-DE差异图谱如图 2所示。分析2-DE图谱可知当蛋白质上样量为500 mg时在8 000 V下等点聚焦10 h可以得到清晰的蛋白质斑点,无明显的拖尾情况,高丰度的蛋白质能有效的分离,而且低丰度的蛋白质斑点也能完全展示,说明该2-DE差异图谱可以进一步用于后续比较蛋白质组学的差异蛋白质斑点的筛选和质谱鉴定,从而获得出与S0活化相关的胞外蛋白质信息。
|
| 图 2 A.manzaensis分别在S0(A)和Fe2+(B)能源底物下生长时细胞胞外蛋白质2-DE图谱 A中箭头所指为用于质谱鉴定的胞外蛋白质斑点,数字表示蛋白质斑点序号 |
通过采用ImageMaster 2D Platinum 6.0版本软件对双向电泳分离得到的图谱进行分析可知,A. manzaensis分别在S0和Fe2+能源底物下生长的胞外蛋白质2-DE图谱存在显著的差异,这表明细胞在这两种能源底物中生长时,有各自的代谢机制从而诱导表达不同的胞外蛋白质。分析结果表明S0能源底物下的胞外蛋白质斑点(图 2-A)在55-72 kD、34-43 kD和17 kD范围附近展现出很多,在Fe2+(图 2-B)底物下没有的斑点,说明这部分蛋白质可能与细胞对S0的活化相关。例如,图 2-A中序号14箭头所指的斑点虽然在Fe2+底物中(图 2-B)也有表达,但是在S0为能源底物下表达量明显上调,说明也与S0的活化紧密相关。通过比较分析2-DE差异图谱后,选择性挖取15个只有在S0能源底物中表达和表达显著上调的蛋白质斑点(图 2-A中用箭头指示并标注斑点序号)进行胶内水解与MALDI-TOF/TOF质谱鉴定。
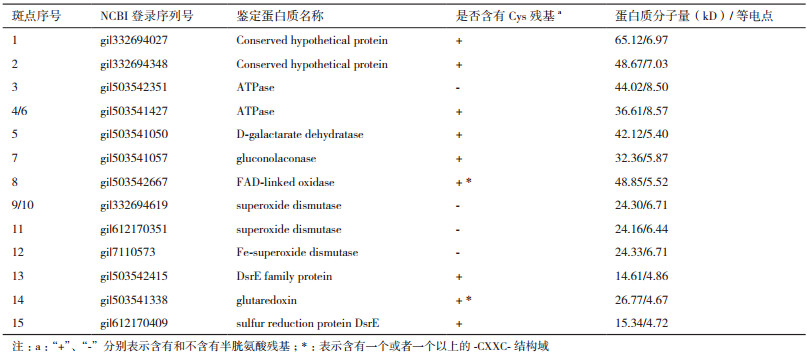
2.3 A. manzaensis胞外S0活化蛋白的质谱鉴定和生物信息学分析基于比较蛋白质组学筛选的A. manzaensis菌胞外S0活化相关蛋白质斑点经过胶内酶切,然后进行MALDI-TOF/TOF分析和指纹鉴定,得到结果如表 1所示。值得注意的是,虽然一共挖取了15个蛋白斑点,但是其中4号和6号斑点均为ATP酶(ATPase),9号和10号斑点鉴定均为个超氧化物歧化酶(superoxide dismutase),表明该蛋白质有不同的同分异构体存在,构象异构的差异导致了等电点的轻微不同;其次鉴定的蛋白质斑点中有2个ATP酶(ATPase)同功酶(3号和4/6号斑点),3个超氧化物歧化酶(superoxide dismutase)同功酶(9/10,11和12号斑点),同功酶是由不同基因编码的蛋白质,它们能催化相同的化学反应,但分子结构和生理功能有所不同的一类酶。对所鉴定的蛋白质功能进行汇总分析可知,所筛选的13个S0活化相关蛋白质中有2个保守的未知功能的蛋白质斑点(Conserved hypothetical protein),2个ATP酶(ATPase)同功酶,1个D-粘酸脱水酶(D-galactarate dehydratase),1个葡萄糖内酯酶(gluconolaconase),1个FAD氧化酶(FAD-linked oxidase),3个超氧化物歧化酶(superoxide dismutase)同功酶,1个谷氧还蛋白(glutaredoxin),一个DsrE家族蛋白质和1个硫氧还蛋白(sulfur reduction protein DsrE)。通过鉴定所得到的13个蛋白质中经分析可知共有9个蛋白质富含Cys残基,并且其中glutaredoxin和FAD-linked oxidase均含有一个或者一个以上的-CXXC-结构域,说明胞外富含巯基(-SH)的蛋白质及含有-CXXC-结构域的蛋白质可能参与了S0活化过程。
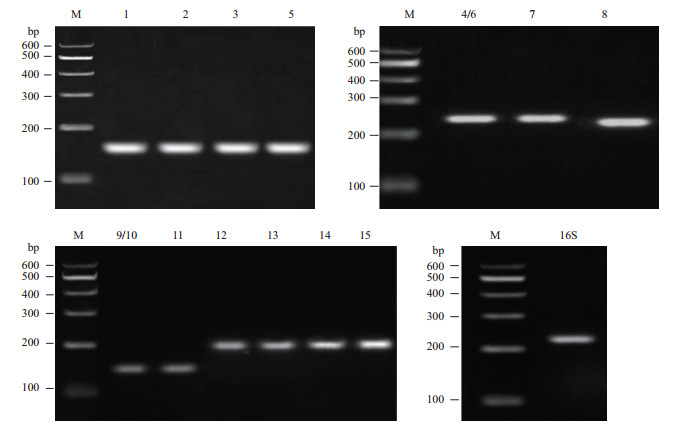
根据质谱鉴定和Mascot检索筛选得到S0活化相关蛋白质后查找到对应的基因设计相应的引物,引物信息如表 2所示,以提取的A. manzaensis菌基因组DNA为模板进行PCR,对产物进行琼脂糖凝胶电泳,电泳结果如图 3所示。琼脂糖凝胶电泳结果显示所有设计的基因引物的PCR扩增产物均为单一明亮的条带,说明所设计的引物特异性较好,而且产物分子量也和理论扩增的产物大小一致,经测序确定为目的基因,可用于后续的RT-qPCR实验。
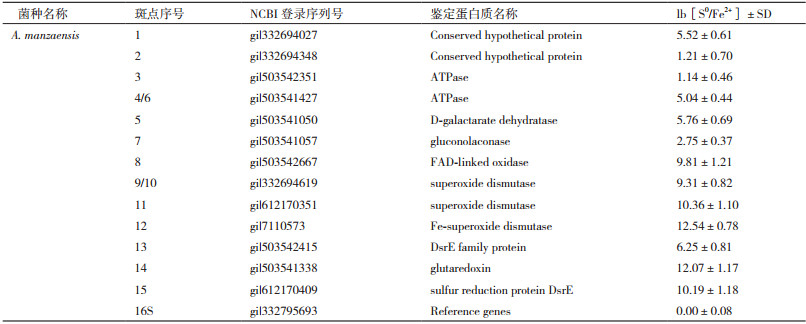
A. manzaensis菌分别在S0硫和Fe2+中生长至对数中期,提取RNA进行反转录及cDNA第二链的合成后,以cDNA为模板,利用表 2中的引物序列进行RT-qPCR。细胞在S0/Fe2+不同能源底物下相关基因的表达差异用各自16S rDNA基因作为内参校正RT-qPCR操作中的随机误差和系统误差,结果用lb[S0/Fe2+]±SD表示。根据统计学的分析当lb[S0/Fe]±SD的值大于1时,表明该基因的表达量在以S0能源底物时明显上调,筛选得到的胞外蛋白质基因RT-qPCR表达结果见表 3。RT-qPCR结果表明通过比较蛋白质组学的方法筛选得到的A. manzaensis菌胞外蛋白质基因的表达量在S0能源底物中均显著高于在Fe2+能源底物中,说明筛选到的蛋白质对应的基因在转录水平上均显著表达上调,从而验证了双向电泳结果,进一步证明所筛选的蛋白质在S0活化中起重要作用。
细胞对S0吸附和活化是整个S0生物氧化过程的关键环节,被认为是整个生物S0氧化的限速步骤。胞外物质很可能介导了细菌对疏水性S0的吸附和活化过程[18]。以S0为能源生长的A. ferrooxidans细胞表面会释放出大量由脂类、多糖和蛋白质等物质组成的外膜泡可能是细菌与元素硫吸附的“桥梁”[19]。半胱氨酸(Cys)残基上的巯基(-SH)在生化反应中具有特殊性质,使得半胱氨酸存在的结构域往往能够为生物体的一系列生理生化反应提供氧化还原对,成为功能蛋白质的氧化还原活性中心[20]。Rohwerder和Sand[21]提出S0活化的模式可能是经过细菌EPS蛋白质的活性巯基(P-SH)与环状硫作用,形成类似的P-SnH结构化合物(n≥2)(公式1),后者再经外膜蛋白转运到细胞周质后被氧化。Peng等[22]通过比较蛋白质组学和基因组学的实验方法比较了S0和Fe2+基质中生长的A. ferrooxidans胞外蛋白质表达差异,筛选出了与硫活化相关的蛋白质,这些蛋白质绝大多数含有-SH,并且发现-CXXC-基序在硫活化中起到非常重要的作用,但是关于极端嗜酸热古菌的胞外S0活化蛋白质的筛选鉴定没有相关文献报道。
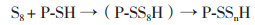 (1)
(1)
本实验基于比较蛋白质组学的研究方法找到的13个与极端嗜酸热古菌A. manzaensis活化S0相关的胞外蛋白质中,D-粘酸脱水酶是催化产生环烃类碳水化合物的重要酶类,其表达量(lb[S0/Fe2+]±SD)为5.76±0.69,说明其可能通过催化合成能吸附到疏水性S0表面的环烃类碳水化合物(如脂质,多糖),在A. manzaensis适应和活化S0中起到“桥梁”的作用。谷氧还蛋白质是巯基-二硫键氧化还原酶家族的重要组分,在调节氧化还原反应中起到重要作用;生物信息分析发现谷氧还蛋白质和FAD键合氧化酶均含有-CXXC-结构域,并且两种蛋白质的基因表达量都非常高,说明含有-CXXC-结构域的两种蛋白质在极端嗜酸热古菌A. manzaensis活化S0的过程中也起到重要的作用。硫还原蛋白质DsrE在许多还原反应中作为氢供体,是一类广泛存在的作为氢载体的热稳定蛋白质[23]。通过生物信息学分析表 1中的蛋白质序列发现约一半以上的蛋白质具有较多的半胱氨酸残基,结合表 3中硫活化相关蛋白质基因的RT-qPCR表达结果,表明巯基在A. manzaensis菌活化氧化S0的过程中起到了非常重要的作用。筛选出来的蛋白质中有2个属于保守未知功能蛋白且也含有较多的半胱氨酸残基,它们在端嗜酸热古菌A. manzaensis活化S0的作用有待进一步研究。值得注意的是,在所筛选得到的蛋白质中有1个ATP酶(3号斑点)和3个超氧化物歧化酶同功酶(9/10,11和12号斑点)不含有半胱氨酸残基,其蛋白质基因的表达量在S0能源底物中也显著高于在Fe2+能源底物中,说明这部分蛋白质也参与了极端嗜酸热古菌A. manzaensis对S0的活化过程,但这几种蛋白质在目前已有的中温菌S0活化过程中的作用均没有报道,说明对于极端嗜酸热古菌A. manzaensis而言,其活化S0的过程与典型的中温菌A. ferrooxidans相比存在差异。推测其中ATP酶可能在整个S0的活化过程中为其他的反应提供能量;另外3个不含巯基的超氧化物歧化酶同功酶(9/10,11和12号斑点)可能通过维持胞外的还原性环境从而保证巯基(P-SH)的还原性,最终保证胞外EPS蛋白质的活性巯基与S0的反应(公式1),而且根据表 3可知这几个同功酶的表达上调倍数不一样,说明这几种酶可能在胞外存在的位置不同,可能分别定位于胞外的荚膜层和粘液层。
4 结论本研究主要通过基于比较蛋白质组学的方法筛选和鉴定了13个A. manzaensis胞外与硫活化相关的蛋白质基因,并从转录水平对其进行了验证。筛选得到的蛋白质中一半以上含有较多的Cys残基,说明胞外富含-SH的蛋白参与了S0活化;其中谷氧还蛋白质和FAD键合氧化酶均含有-CXXC-结构域,且两种蛋白质的基因表达量都非常高,说明含有-CXXC-结构域的蛋白质在极端嗜酸热古菌A. manzaensis活化S0的过程中也起到重要的作用。
| [1] | 周吉奎, 钮因健. 硫化矿生物冶金研究进展. 金属矿山, 2015(04): 24–30. |
| [2] | 周洪波, 毛峰, 王玉光. 嗜酸微生物与生物冶金技术. 矿物岩石地球化学通报, 2015(2): 269–276. |
| [3] | Li Y, Kawashima N, Li J, et al. A review of the structure, and fundamental mechanisms and kinetics of the leaching of chalcopyrite. Advances in Colloid & Interface Science, 2013, 197-198 (9): 1–32. |
| [4] | Peng AA, Xia JL, Liu HC, et al. Differential utilization of cyclic, orthorhombic α-and chain-like polymeric μ-sulfur by Acidithiobacillus ferrooxidans. Transactions of Nonferrous Metals Society of China, 2014, 24 (5): 1562–1570. DOI:10.1016/S1003-6326(14)63226-X |
| [5] | Rohwerder T, Sand W. Properties of thiols required for sulfur dioxygenase activity at acidic pH. Journal of Sulfur Chemistry, 2008, 29 : 293–302. DOI:10.1080/17415990802146972 |
| [6] | Zeng J, Davies M. Evidence for the formation of adducts and s-(carboxymethyl) cysteine on reaction of α-dicarbonyl compounds with thiol groups on amino acids, peptides, and proteins. Chemical Research in Toxicology, 2005, 18 : 1232–1241. DOI:10.1021/tx050074u |
| [7] | Xia JL, Liu HC, Nie ZY, et al. Synchrotron radiation based STXM analysis and micro-XRF mapping of differential expression of extracellular thiol groups by Acidithiobacillus ferrooxidans grown on Fe (2+) and S (0). Journal of Microbiological Methods, 2013, 94 (3): 257–261. DOI:10.1016/j.mimet.2013.06.030 |
| [8] | Liu HC, Xia JL, Nie ZY, et al. Differential expression of extracellular thiol groups of moderately thermophilic Sulfobacillus thermosulfido-oxidans, and extremely thermophilic Acidianus manzaensis, grown on S0, and Fe2+. Archives of Microbiology, 2015, 197 (6): 1–9. |
| [9] | Friedrich CG, Bardischewsky F, Rother D, et al. Prokaryotic sulfur oxidation. Current Opinion in Microbiology, 2005, 8 (3): 253–259. DOI:10.1016/j.mib.2005.04.005 |
| [10] | Quatrini R, Appia-Ayme C, Denis Y, et al. Extending the models for iron and sulfur oxidation in the extreme Acidophile Acidithiobacillus ferrooxidans. Bmc Genomics, 2009, 10 (9): 507–521. |
| [11] | Liu HC, Xia JL, Nie ZY, et al. Iron L-edge and sulfur K-edge XANES spectroscopy analysis of pyrite leached by Acidianus manzaensis. Transactions of Nonferrous Metals Society of China, 2015, 25 (7): 2407–2414. DOI:10.1016/S1003-6326(15)63856-0 |
| [12] | 彭安安.嗜酸硫氧化细菌元素硫活化氧化机制研究[D].长沙:中南大学, 2012. http://cdmd.cnki.com.cn/Article/CDMD-10533-1013358403.htm |
| [13] | Hammond JBW, Kruger NJ. The bradford method for protein quantitation. Methods in Molecular Biology, 1994, 32 (32): 9–15. |
| [14] | 孔令琼, 管政兵, 陆健, 等. 绍兴黄酒成品麦曲中微生物胞外酶的双向电泳技术的建立. 食品与生物技术学报, 2011, 30(3): 453–457. |
| [15] | Ramírez P, Guiliani N, Valenzuela L, et al. Differential Protein Expression during Growth of Acidithiobacillus ferrooxidans on ferrous iron, sulfur compounds, or metal sulfides. Applied & Environmental Microbiology, 2004, 70 (8): 4491–4498. |
| [16] | Bonnefoy V.Bioinformatics and Genomics of Iron-and SulfurOxidizing Acidophiles[M]//Geomicrobiology :Molecular and Environmental Perspective.Springer Netherlands, 2010 :169-192. |
| [17] | Pfaffl MW, Tichopad A, Prgomet C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity :BestKeeper -Excel-based tool using pair-wise correlations. Biotechnology Letters, 2004, 26 (6): 509–515. DOI:10.1023/B:BILE.0000019559.84305.47 |
| [18] | Wang H, Liu S, Liu X, et al. Identification and characterization of an ETHE1-like sulfur dioxygenase in extremely acidophilic Acidithiobacillus spp. Applied Microbiology & Biotechnology, 2014, 98 (17): 7511–22. |
| [19] | Boretska M, Bellenberg S, Moshynets O, et al. Change of extracellular polymeric substances composition of Thiobacillus thioparus in presence of sulfur and steel. Microbial & Biochemical Technology, 2013, 5 (3): 68–73. |
| [20] | Quatrini R, Appia-Ayme C, Denis Y, et al. Extending the models for iron and sulfur oxidation in the extreme Acidophile Acidithiobacillus ferrooxidans. Bmc Genomics, 2009, 10 (1): 507–521. DOI:10.1186/1471-2164-10-507 |
| [21] | Rohwerder T, Sand W. The sulfane sulfur of persulfides is the actual substrate of the sulfur-oxidizing enzymes from Acidithiobacillus and Acidiphilium spp. Microbiology, 2003, 149 (Pt 7): 1699–710. |
| [22] | Peng AA, Xia JL, Liu HC, et al. Thiol-rich proteins play important role in adhesion and sulfur oxidation process of Acidithiobacillus ferroxidans. Advanced Materials Research, 2013, 825 : 137–140. DOI:10.4028/www.scientific.net/AMR.825 |
| [23] | Weissgerber T, Watanabe M, Hoefgen R, et al. Metabolomic profiling of the purple sulfur bacterium Allochromatium vinosum during growth on different reduced sulfur compounds and malate. Metabolomics, 2014, 10 (6): 1094–1112. |