端粒是真核染色体末端一段特殊的结构,由端粒重复序列和端粒结合蛋白组成。端粒能够防止染色体末端的降解、融合和重排,从而在维持染色体的完整性和稳定性上起重要作用[1-3]。人类二倍体细胞有46条染色体92个端粒,序列是由富含G的短双链重复序列TTAGGG串联组成,双链区长度一般在0.5-20 kb。其中富含G碱基的一条链在3′末端比其互补链多出100-200 bp核苷酸,形成一条突出的3′单链DNA。这条突出的端粒单链DNA被反折插入到端粒DNA的双链区,从而使染色体DNA终止于一种T型环(T-Loop)结构,将末端端粒DNA与细胞内环境隔绝,端粒作为染色体的保护性帽来防止DNA损伤信号对染色体末端的识别[4, 5]。邻近端粒重复序列的区域被称作亚端粒(sub-telomere)区,目前亚端粒的功能并不完全清楚[6]。
由于DNA聚合酶不能完整的复制染色体的末端(DNA复制问题),几乎所有的正常人体细胞会随着细胞分裂出现端粒逐渐缩短并最终引发细胞复制性衰老,端粒是细胞衰老的分子钟[7]。一部分丧失关键细胞周期检测点功能的细胞会跃过细胞复制性衰老继续分裂最终进入危机期。这个时期的极少数细胞获得了端粒酶活性,并且继续分裂转化为肿瘤细胞。端粒在人类的衰老与肿瘤的发生进程中扮演了极其重要的角色,端粒重复序列的长度的变化决定着细胞的命运。因此,端粒长度检测是端粒生物学研究中的重要实验方法。此外,端粒长度检测在衰老疾病和肿瘤中也具有重要诊断价值[8-11]。就目前而言,最常用的端粒长度检测方法包括: 端粒末端限制性片段分析(terminal restriction fragment,TRF),定量PCR(qPCR),荧光原位杂交(fluores-cence in situ hybridization,FISH)和单链端粒长度分析(single telomere length analysis,STELA)。但每一种检测方法都有其独有的特点,我们有必要对端粒长度的检测方法进行比较分析,了解每种方法的优势和局限及适用范围,以便于在研究时选择合适的端粒长度检测方法。
1 端粒末端限制性片段分析(terminal restr-iction fragment,TRF)端粒末端限制性片段分析是最早最经典的端粒长度检测的方法,此方法也被认为是端粒长度检测的金标准。此方法是评估群体细胞的平均端粒长度。根据端粒序列特异且重复的特性,用一组缺乏端粒识别位点的频繁切割限制性内切酶(如Hinf I和Rsa I)消化基因组DNA,基因组DNA被消化成短片段,端粒DNA不被切割以较长的片段保留。不同长度的DNA消化产物在琼脂糖凝胶上分离,用端粒DNA特异的探针通过Southern blotting方法检测端粒长度[12, 13]。不同长度的端粒产生弥散的信号(图 1),通过软件与已知分子量DNA 梯度条带比较来评估平均端粒长度,通过标准化参照样本来修正实验之间的“胶效应”。
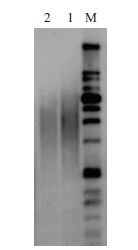
|
| 图 1 肿瘤细胞端粒末端限制性片段分析 M: DNA Marker;1,2: 肿瘤细胞端粒信号 |
用此方法来检测端粒长度时,提取的基因组DNA的完整性对定量端粒长度十分重要,DNA降解会导致对端粒长度评估的不准确,因此提取过程要注意不要污染核酸酶,存储时要分装避免反复冻融。此方法的优势是不需要特殊的设备,经济,易于操作,错误相对较小,是评价其他新的端粒长度检测技术的标准方法。TRF目前仍是端粒生物学研究最常用的端粒长度检测方法。TRF 的局限在于需要的DNA量多,另外由于短端粒与探针结合的效率不高,TRF对检测较短的端粒是不敏感的,而最短的端粒很可能在诱发DNA损伤中具有重要作用[14]。此外,这种方法检测的端粒包含了亚端粒DNA,因此有可能过多评估端粒长度。
2 定量PCR(quantitative PCR,qPCR)为了克服分析端粒长度需要DNA量多的局限,PCR为基础的端粒长度检测方法得以研发,应用较广泛的是2002年Cawthon[15]报道的qPCR和之后改进的MMqPCR方法[16]。用与端粒C链和G链都能退火但与其他碱基不匹配的引物通过PCR扩增端粒,前两个循环用低温退火使引物与端粒DNA模板配对产生端粒产物,其余循环用高温退火确保仅扩增前两个循环得到的端粒产物(抑制引物和端粒DNA模板退火和引物二聚体产生)。端粒扩增产物(T)的量与另一管中扩增的单拷贝基因的量(S)进行比值来对端粒长度定量。然而在配制T反应管和S反应管溶液时,由于吸取体积产生的误差可能降低检测的精确性。2009年Cawthon[16]改进了这种方法使端粒和单拷贝基因在同一管中完成扩增,此方法称为MMqPCR方法。改进的MMqPCR方法使得来源于不同质量的DNA样本可以比较,而且端粒和单拷贝基因本身扩增效率的差异可以用比值来控制。2011年O′Callaghan 和 Fenech[17]也对qPCR方法进行了改进称为aTL qPCR,这种方法使用标准曲线,标准曲线是根据一系列14拷贝TTAGGG序列(84 bp长)稀释值绘制的,可以评估样本端粒碱基对的长度。
qPCR同样需要高质量的DNA,但需要的DNA量很少(ng)。另外,这种方法比较经济,需要的设备容易获得(定量PCR仪),并且可以用于高通量检测,适用于临床诊断和流行病学等大规模样本研究。基于PCR的端粒长度检测方法已经对GERA(genetic epidemiology research on adult health and aging)队列研究中的约100 000例唾液DNA样本进行了端粒长度检测,并分析了端粒长度与年龄的相关性[18]。这项研究是迄今为止最大样本量的相对端粒长度分析。qPCR的局限是需要扩增对照基因,而扩增的对照基因在基因组中是独特的,其拷贝数的变异和染色体的复制会改变基因拷贝数,从而显著改变T/S值。因此,qPCR法只适合用于二倍体和核型稳定的细胞和样本,转化细胞系和肿瘤组织样本是不适合的。此外,与TRF一样只能评估平均端粒长度,而且样本内和样本间的差异还是比较高的,不同实验室得出的结果相差很大。TRF方法变异系数是1.74%,qPCR方法的变异系数是6.45%,说明qPCR法分析端粒长度的精确性和稳定性不高。
3 定量荧光原位杂交(quantitative fluoresc-ence in situ hybridization,Q-FISH)Q-FISH是用荧光标记的(CCCTAA)3肽核酸探针与分裂中期细胞的变性端粒DNA重复序列杂交,荧光信号可以被检测,通过软件与已知端粒长度的标准品比对来分析端粒长度[19, 20]。Q-FISH能检测每条染色体上的端粒长度,Q-FISH还能检测极短端粒重复序列(<0.5 kb)的末端和端粒融合事件。分裂中期Q-FISH分析不仅能评估不同染色体端粒的差异也能分析无端粒染色体不稳定的频率。这种方法可以研究多种背景下的端粒生物学,对于检测细胞量较少的细胞端粒长度也是比较好的方法。局限是不能检测衰老细胞、不分裂细胞和高度畸变细胞的端粒,对检测低有丝分裂指数的细胞型也是很困难的。随后又研究了改进的方法分裂间期(Interphase)Q-FISH,这种方法能够分析来源于分裂间期血细胞和组织切片细胞的端粒长度,通过端粒探针荧光信号和中心体探针信号的比值分析端粒长度[21]。这种方法的优势在于对组织切片进行Q-FISH分析能同时获得端粒长度和病理学特征的信息。但是,组织切片可能不含有完整的胞核,此外分裂间期Q-FISH不能分析每个染色体端粒长度和无端粒染色体,得出的也是平均端粒长度。
自动化高通量Q-FISH(HT Q-FISH)适用于大样本的研究。如Canela等[22]用HT Q-FISH的方法检测了198例健康人外周血淋巴细胞的端粒,发现了年龄、性别和地理区域对端粒长度的影响。Tengumnuay等[23]用HT Q-FISH的方法检测了间充质干细胞的端粒长度,通过检测间充质干细胞的端粒长度能够评估细胞复制能力从而筛选适合用于治疗的间充质干细胞。但HT Q-FISH检测精确性和内在的重复性是有限的。
4 流式荧光原位杂交(flow cytometry and flow fluorescence in situ hybridization,Flow FISH)Flow FISH是用荧光标记的(CCCTAA)3肽核酸探针与悬浮细胞杂交,端粒荧光信号用流式细胞仪分析[24]。Flow FISH能够精确检测每个细胞的平均端粒长度,能够用于检测经流式分离的细胞亚群(根据大小、抗体标记等)的端粒长度。Flow FISH因此广泛用于检测不同亚型造血细胞的端粒长度,后来通过使用半自动96孔板形式,Flow FISH适用于更高通量的检测,并且具有较好的重复性。
Flow FISH是第一个应用于临床诊断的端粒长度检测方法,如辅助诊断患有先天角化不良的病人[25, 26],这种方法也为推断细胞中端粒信号的3维分布提供了一种手段。Flow FISH也比较适合血液中不同类型亚群细胞端粒长度分析,自动化的多色Flow FISH是目前最快最敏感的检测人血液中粒细胞、T细胞、B细胞和天然杀伤细胞(NK)平均端粒长度的方法。目前已有商品化的Flow FISH试剂盒用于检测体外扩增的NK细胞端粒长度,评估NK细胞的活性,用于肿瘤免疫治疗。但是Flow-FISH要求完整的细胞核,粒细胞在体外制备不稳定,而在体内半衰期也极短,如果检测粒细胞的端粒长度,就要求迅速处理样本。这也限制了其在流行病学研究的应用。Flow FISH的局限是检测的也是平均端粒长度,对技术要求比较高,耗时且不易操作。
5 单链端粒长度分析(single telomere length analysis,STELA)所有端粒末端都有富含G的3′单链突出,以突出的3′单链为模板退火并连接一个接头到端粒的5′末端,以接头引物和一条染色体上特异亚端粒引物扩增单个染色体端粒单链区。但不是所有端粒末端都有合适的用于设计亚端粒引物的序列,因此这种方法仅适合于几个特征性染色体末端如XpYp、2p、11q、12q 和 17p[27]。STELA的优势是能通过少量样本精确分析端粒长度,pg级的DNA就足够用于分析,亚端粒引物的完整碱基是已知的,并且在细胞、样本和个体间是比较稳定的,因此分析的端粒长度比较精确。此法不需要特殊设备,能够分析单个端粒,可以清楚分析自然缩短和异常缩短的端粒动态,也能够分析不分裂的衰老细胞。非常适合于检测异常短的端粒,而短端粒往往诱发衰老和产生融合。但STELA方法需要具备单分子PCR技术经验,对技术操作要求较高,操作过程比较耗时。尽管能检测单个染色体末端端粒长度的微小改变,但不能代表细胞整体端粒长度状态,也不适合分析较长的端粒(>20 kb,如小鼠)。STELA是一种低通量的端粒长度检测方法,不适合临床应用。
6 基于全基因组测序(WGS)的端粒长度分析随着二代测序技术的发展和测序成本的下降,WGS在研究和临床中的应用越来越广泛。最近,WGS数据已经被用于分析端粒长度[28, 29]。端粒是基因组上的特殊生物学元件,WGS能够捕获端粒序列信息,通过生物信息学软件可以分析WGS数据中端粒含量和长度。Parker等[30]用235个儿科肿瘤的WGS数据分析了肿瘤中端粒DNA含量的改变,提示在肿瘤进展过程中检测端粒DNA的改变可能用于临床诊断。基于WGS的端粒长度分析方法与qPCR法有较好的一致性,然而与TRF法一致性较差。由于WGS数据本身分析起来就十分复杂,WGS数据分析方法对端粒长度的预测会有较大影响。此外,WGS法依赖于细胞单倍体的已知染色体数,正常健康细胞单倍体染色体数是23,但肿瘤细胞通常是非整倍体。因此如果细胞倍数未知,基于WGS的端粒长度分析方法并不精确。
7 结语对比分析这些端粒长度检测方法(表 1),我们可以看出并没有任何一种单一的方法可以精确、简单、快速的检测端粒长度,因此端粒长度检测方法的选择需要根据具体的科学问题。TRF检测的是平均端粒长度,检测不到端粒的微小改变,从而不适合用于高精确度端粒长度分析,但仍是端粒长度分析方法的金标准及最常用方法之一。这种方法已经广泛应用于基础研究、临床研究和流行病学研究,基础研究通常检测培养的细胞或组织端粒长度,临床研究和流行病学研究通常检测外周血淋巴细胞的端粒长度。如果有大规模的样本需要进行高通量检测,例如流行病研究及临床大样本的端粒长度筛查,qPCR是比较好的选择,qPCR用于流行病学研究有简便、经济、高通量,DNA用量少等优势,但不适用于总体样本数量少和精确度要求高的检测。Flow FISH比qPCR更精确,更适用于临床检验[31]。Q-FISH 和STELA适合于小量样本的实验研究,它们需要的DNA很少(<100 pg),样本量较少较珍贵时可选择这两种方法。此外,这两种方法高度精确,能检测单个染色体异常短的端粒,从而适于基本端粒生物学问题的精细研究,但目前还不适用于流行病学研究。基于WGS的端粒分析方法是在二代测序技术快速发展和大量样本WGS数据已知情况下新出现的方法,目前还存在很多问题,其应用还需进一步的探索。
端粒在衰老和肿瘤研究中是极为引人注目的,但就目前而言,没有任何一种端粒检测方法是通用的。在特定的实际应用或科学研究中,没有选择合适的端粒长度分析方法很可能导致错误的发现或阻碍其应用前景,因此必须根据实际需要而选择最合适的端粒长度检测方法。当然,由于目前每种方法都有一定的局限,同一样本不同实验室间检测数据存在较大的差异[32],这就要求我们对端粒长度检测方法进行不断的完善和创新,相信端粒检测方法学的创新将会推动端粒基础和流行病学研究及临床应用的极大进展。
| [1] | Blackburn EH. Structure and function of telomeres. Nature , 1991, 350 : 569–73. DOI:10.1038/350569a0 |
| [2] | Blackburn EH. Switching and signaling at the telomere. Cell , 2001, 106 : 661–73. DOI:10.1016/S0092-8674(01)00492-5 |
| [3] | Lu W, Zhang Y, Liu D, et al. Telomeres-structure, function, and regulation. Experimental Cell Research , 2013, 319 : 133–41. DOI:10.1016/j.yexcr.2012.09.005 |
| [4] | Griffith JD, Comeau L, Rosenfield S, et al. Mammalian telomeres end in a large duplex loop. Cell , 1999, 97 : 503–514. DOI:10.1016/S0092-8674(00)80760-6 |
| [5] | Sfeir A, de Lange T. Removal of shelterin reveals the telomere end-protection problem. Science , 2012, 336 : 593–597. DOI:10.1126/science.1218498 |
| [6] | Mefford HC, Trask BJ. The complex structure and dynamic evolution of human subtelomeres. Nature Reviews Genetics , 2002, 3 : 91–102. |
| [7] | Blackburn EH. Telomere states and cell fates. Nature , 2000, 408 : 53–56. DOI:10.1038/35040500 |
| [8] | Cawthon RM, Smith KR, O'Brien E, et al. Association between telomere length in blood and mortality in people aged 60 years or older. Lancet , 2003, 361 : 393–395. DOI:10.1016/S0140-6736(03)12384-7 |
| [9] | Heaphy CM, Meeker AK. The potential utility of telomere-related markers for cancer diagnosis. Journal of Cellular and Molecular Medicine , 2011, 15 : 1227–1238. DOI:10.1111/jcmm.2011.15.issue-6 |
| [10] | Weischer M, Nordestgaard BG, Cawthon RM, et al. Short telomere length, cancer survival, and cancer risk in 47102 individuals. Journal of the National Cancer Institute , 2013, 105 : 459–468. DOI:10.1093/jnci/djt016 |
| [11] | Seow WJ, Cawthon RM, Purdue MP, et al. Telomere length in white blood cell DNA and lung cancer: a pooled analysis of three prospective cohorts. Cancer Research , 2014, 74 : 4090–4098. DOI:10.1158/0008-5472.CAN-14-0459 |
| [12] | Kimura M, Stone RC, Hunt SC, et al. Measurement of telomere length by the Southern blot analysis of terminal restriction fragment lengths. Nature Protocols , 2010, 5 : 1596–1607. DOI:10.1038/nprot.2010.124 |
| [13] | Mender I, Shay JW. Telomere Restriction Fragment(TRF)analysis. Bio-Protocol , 2015, 5 (22) : e1658. |
| [14] | Hemann M, Strong MA, Hao LY, et al. The shortest telomere, not average telomere length, is critical for cell viability and chromosome stability. Cell , 2001, 107 : 67–77. DOI:10.1016/S0092-8674(01)00504-9 |
| [15] | Cawthon RM. Telomere measurement by quantitative PCR. Nucleic Acids Research , 2002, 30 : e47. DOI:10.1093/nar/30.10.e47 |
| [16] | Cawthon RM. Telomere length measurement by a novel monochrome multiplex quantitative PCR method. Nucleic Acids Research , 2009, 37 : e21. DOI:10.1093/nar/gkn1027 |
| [17] | O'Callaghan NJ, Fenech M. A quantitative PCR method for measuring absolute telomere length. Biological Procedures Online , 2011, 13 : 3. DOI:10.1186/1480-9222-13-3 |
| [18] | Lapham K, Kvale MN, Lin J, et al. Automated assay of telomere length measurement and informatics for 100, 000 subjects in the genetic epidemiology research on adult health and aging(GERA)cohort. Genetics , 2015, 200 : 1061–1072. DOI:10.1534/genetics.115.178624 |
| [19] | Slijepcevic P. Telomere length measurement by Q-FISH. Methods in Cell science: an Official Journal of the Society for in Vitro Biology , 2001, 23 : 17–22. |
| [20] | Ferlicot S, Youssef N, Feneux D, et al. Measurement of telomere length on tissue sections using quantitative fluorescence in situ hybridization(Q-FISH). The Journal of Pathology , 2003, 200 : 661–666. DOI:10.1002/(ISSN)1096-9896 |
| [21] | Narath R, Lorch T, Greulich-Bode KM, et al. Automatic telomere length measurements in interphase nuclei by IQ-FISH. Cytometry Part A: the Journal of the International Society for Analytical Cytology , 2005, 68 : 113–120. |
| [22] | Canela A, Vera E, Klatt P, et al. High-throughput telomere length quantification by FISH and its application to human population studies. Proceedings of the National Academy of Sciences of the United States of America , 2007, 104 : 5300–5305. DOI:10.1073/pnas.0609367104 |
| [23] | Toutain J, Prochazkova-Carlotti M, Horovitz J, et al. Evaluation of Quantitative Fluorescence in situ hybridization for relative measurement of telomere length in placental mesenchymal core cells. Gynecologic and Obstetric Investigation , 2016, 81 : 54–60. |
| [24] | Baerlocher GM, Vulto I, de Jong G, et al. Flow cytometry and FISH to measure the average length of telomeres(flow FISH). Nature Protocols , 2006, 1 : 2365–2376. DOI:10.1038/nprot.2006.263 |
| [25] | Alter BP, Baerlocher GM, Savage SA, et al. Very short telomere length by flow fluorescence in situ hybridization identifies patients with dyskeratosis congenita. Blood , 2007, 110 : 1439–47. DOI:10.1182/blood-2007-02-075598 |
| [26] | Alter BP, Rosenberg PS, Giri N, et al. Telomere length is associated with disease severity and declines with age in dyskeratosis congenita. Haematologica , 2012, 97 : 353–359. DOI:10.3324/haematol.2011.055269 |
| [27] | Britt-Compton B, Rowson J, Locke M, et al. Structural stability and chromosome-specific telomere length is governed by cis-acting determinants in humans. Human Molecular Genetics , 2006, 15 : 725–733. DOI:10.1093/hmg/ddi486 |
| [28] | Ding Z, Mangino M, Aviv A, et al. Estimating telomere length from whole genome sequence data. Nucleic Acids Research , 2014, 42 : e75. DOI:10.1093/nar/gku181 |
| [29] | Lee M, Napier CE, Yang SF, et al. Comparative analysis of whole genome sequencing-based telomere length measurement techniques. Methods , 2016, S1046-2023 (16) : 30263–8. |
| [30] | Parker M, Chen X, Bahrami A, et al. Assessing telomeric DNA content in pediatric cancers using whole-genome sequencing data. Genome Biology , 2012, 13 : R113. DOI:10.1186/gb-2012-13-12-r113 |
| [31] | Gutierrez-Rodrigues F, Santana-Lemos BA, Scheucher PS, et al. Direct comparison of flow-FISH and qPCR as diagnostic tests for telomere length measurement in humans. PLoS One , 2014, 9 : e113747. DOI:10.1371/journal.pone.0113747 |
| [32] | Martin-Ruiz CM, Baird D, Roger L, et al. Reproducibility of telomere length assessment: an international collaborative study. International Journal of Epidemiology , 2015, 44 : 1673–1683. DOI:10.1093/ije/dyu191 |