2. 农业部转基因生物食用安全监督检验测试中心,北京 100083
2. The Supervision,Inspection and Testing Center of Genetically Modified Organisms,Ministry of Agriculture of P. R. China,Beijing 100083
大肠埃希氏菌(Escherichia coli)通常被称为大肠杆菌,为埃希氏菌属的代表菌种。有鞭毛及动力,为无芽孢的革兰氏阴性短杆菌。大肠杆菌为兼性厌氧菌,生长温度范围是8-46℃,最适生长温度为37℃。其菌体抗原为“O”型抗原,鞭毛抗原为“H”型抗原,表面抗原为“K”型抗原,根据其抗原结构的差异被分为180多种血清型别。致病性大肠杆菌是指能产生和携带肠毒素(LT、ST和SLT)、定居因子(CFA-Ⅰ和CFA-Ⅱ)、K抗原或类似物质,以及带有性菌毛、能分泌内毒素的大肠杆菌。根据其不同的生物学特性可将致病性大肠杆菌分为6种类型[1]: 产肠毒素性大肠杆菌(enterotoxigenic Escherichia coli,ETEC)、 肠侵袭性大肠埃希杆菌(enteroinvasive E. coli,EIEC)、肠致病性大肠杆菌(enteropathogenic Escherichia coli,EPEC)、肠出血性大肠杆菌(enterohemorrhage E. coli,EHEC)、肠聚集性大肠杆菌(enteroaggregative Escherichia coli,EAEC)及弥散黏附性大肠杆菌(DAEC)。
自1885年Esherich发现大肠埃希氏菌以来,人们通常将其认作肠道菌群中的正常组成部分。直到1982年,Riley和Remis等[2]通过对当时美国密歇根州和俄勒冈州爆发的伴有腹泻和肠出血的肠道疾病进行调查研究,首次发现这种不同寻常的肠道疾病是由肠出血性大肠杆菌O157: H7感染所引起的,人们才意识到大肠杆菌具有致病性。此后30多年,致病性大肠杆菌引发的食物中毒事件不胜枚数[3, 4],事件爆发后需要对污染的菌体进行分析鉴定,明确其致病机制、耐药情况等,才能对诊断做出有效的判定。因此探究致病性大肠杆菌的污染情况,能够更加明确其容易侵染的基质,为食物中毒事件的分析提供参考;总结致病性大肠杆菌的感染机制及症状能够协助对病发状态做出更加准确的分析;分析耐药性、致病因子及现有检测技术能够进一步研究新型快检技术以及核酸诊断药物等,对治疗具有重要意义。基于此,本文结合国内外研究现状从污染情况、感染途径及症状、耐药性、风险毒力因子及检测方法等方面综述了致病性大肠杆菌的现状分析及检测技术研究进展。
1 污染情况及限量情况 1.1 生物体污染大肠杆菌O157所引起的疾病属于人畜共患病,其中动物为重要的传染源,可于宰前感染[5],较常见的有牛、鸡、羊、狗、猪等,动物的粪便中也常常能够分离出O157: H7大肠杆菌。其中以牛的带菌率最高,可高达16%。
大肠杆菌常寄生于家畜及其奶中,或是被家畜粪便污染了的水中,以及用被污染了的水灌溉的作物中。人们食用了这些被污染过的食品,就可能引发腹泻等症状。世界范围内爆发的严重大肠杆菌感染事件中,几乎都是由食物传播引起的。屠宰与加工中的污染,肉类带大肠杆菌,在加工烹调过程中加热不彻底或生吃都会导致进食者感染[6]。
1.2 环境污染大肠杆菌O157引起的环境污染主要在于土壤及水源。目前我国有关污水和禽畜粪便等随意排放导致病原微生物污染环境的报道屡见不鲜[7]。肠道致病性微生物也是土壤中最为常见的病原菌,主要是通过粪肥的施用进入土壤,Gagliardi等[8]通过研究发现土壤中的大肠杆菌O157: H7还具有向地表和地下迁移的特性。
在我国,类似的污染也不时被报道。周淑玉等[9]报道称,用污水灌溉的土壤中粪大肠杆菌的检出率高达93.9%。朱奇等[10]调查发现,使用被大肠杆菌及肠道致病菌污染的水源水灌溉导致2000年聊城上市蔬菜中,高达47%都受到不同程度的污染。由此可见,土壤与水源都是大肠杆菌污染传播的重要媒介。叶小梅等[11]对江苏省10家畜禽养殖场的排放物及周边水、土样进行了分析,并以粪大肠菌群和沙门菌为指标,结果发现10家养殖场中有9家废物排放不合格,其中粪大肠菌群全部严重超标,施于土壤的粪肥中粪大肠菌群数在105/g以上,此外,从中分离出的大肠杆菌能够表现出多重耐药性。
大肠杆菌极易通过土壤作物进入人类食物链,引发大规模的传染,因此保护灌溉水源及使用经过无害化处理的粪肥也是生态环境保护的重要工作。
1.3 国内外对于大肠杆菌的限量情况针对致病菌的限量,不同的国家和地区有着相似标准,但食品微生物限量指示菌在不同的食品中标准限量值的差异很大[5]。欧盟对市场销售的27类食品进行了微生物限量规定。以总菌落数、大肠埃希氏菌、肠杆菌科指示菌限量,对水果、蔬菜及其制品、乳与乳制品、肉及肉制品,以及水产品、蛋制品的加工进行了规范。欧盟对大肠埃希氏菌的限量采用三级采样手段,在市场销售的产品中仅限定了水产品,限量为n=1,c=0,m=230 MPN/100 g;在生产加工中限定的种类较多,规定在加工过程中果蔬制品及乳制品的限量为n=5,c=2,m=100 MPN/g,M=1 000 MPN/g;生产过程结束时加工生鲜肉的限量为n=5,c=2,m=50 MPN/g,M=500 MPN/g;乳制品的限量为n=5,c=2,m=10 MPN/g,M=100 MPN/g;水产品限量为n=5,c=2,m=1 MPN/g,M=10 MPN/g。澳大利亚、新西兰微生物通用标准中,共26类食品涉及到微生物限量,其中14类食品具有指示菌限量,其中对大肠埃希氏菌做出限量的食品共有9种,在这9种食品中,矿泉水、瓶装水、定型包装的冰及豆腐中均不得检出大肠埃希氏菌。此外,干酪中要求n=5,c=1,m=10 MPN/g,M=100 MPN/g,未经巴氏消毒牛乳中要求n=5,c=1,m=3 MPN/g,M=9 MPN/g,肉粉中要求n=5,c=1,m=3.6 MPN/g,M=9.2 MPN/g,双贝壳类软体动物要求n=5,c=1,m=2.3 MPN/g,M=7 MPN/g。加拿大主要对果蔬、乳制品、即食食品中菌落总数、大肠菌群作出限量以进行规范,22种具有指示菌限量的食品中,有15种对大肠杆菌做出限量。其中巴氏消毒奶制备的奶酪限定标准为n=5,c=2,m=100 MPN/g,M=2 000 MPN/g,非巴氏消毒奶制备的奶酪限定标准为n=5,c=2,m=500 MPN/g,M=2 000 MPN/g,婴儿配方食品的限定标准为n=5,c=1,m<1.8 MPN/g,M=10 MPN/g,面制品及焙烤食品的限定标准为n=5,c=2,m<1.8 MPN/g,M=1 000 MPN/g,经热处理的发酵香肠的限定标准为n=5,c=1,m=10 MPN/g,M=1 000 MPN/g,生的发酵香肠的限定标准为n=5,c=1,m=100 MPN/g,M=1 000 MPN/g,即食类香肠、豆制品、调味品以及鲜果蔬等的限定标准为n=5,c=2,m=100 MPN/g,M=1 000 MPN/g。
在我国于2010年新发布的15项乳制品产品标准中,针对11种产品规定了大肠菌群限量标准,分别是巴氏杀菌乳、调制乳、发酵乳、炼乳、乳粉、稀奶油/奶油/无水奶油、干酪、再制干酪、婴儿配方食品、较大婴儿和幼儿配方食品以及婴幼儿谷类辅助食品。在《GB29921-2013 食品安全国家标准 食品中致病菌限量》中,要求牛肉制品和生食果蔬制品中不得检出大肠埃希氏菌O157: H7,其他无说明。香港只是在即食食品中规定了菌落总数、大肠埃希氏菌的限量,要求大肠埃希氏菌的检出量为20-100 CFU/g,方可认定为满意级别。
2 大肠杆菌感染机制途径与症状 2.1 感染途径根据众多致病性大肠杆菌引发的食物中毒事件的分析可知,大肠杆菌的感染途径有3种方式: 食物传播、水传播、亲密接触传播,其中食物传播为最主要的感染途径。在过去的40年内,较为严重的大肠杆菌爆发事件均是通过食物传播引起的,例如美国爆发的O157食物中毒基本都与汉堡包肉馅有关,1996年日本爆发的疫情与进食牛肉、牛肝有关。另外,牛奶及其制品、生蔬菜、饮料等也常作为传播介质,这些都是通过食品进行传播的。通过水进行传播主要是由粪便污染导致的。此外,通过亲密接触感染者,并可产生二代污染,二代病人出现的症状通常较轻,出血性肠炎的发病率很低。
2.2 感染机制与症状肠产毒性大肠杆菌(enterotoxigenic Escherichia coli,ETEC)是一类能侵染小肠上皮细胞的病原菌,能够在细胞上定植和增殖,并产生肠毒素。肠毒素是由质粒或染色体编码的,具有热敏感或热不稳定的特点[12]。肠道感染患者常伴有轻微或严重腹泻,这类病原菌的感染对肠道的营养吸收、儿童的生长发育甚至于全球的死亡率都有着深远的影响[13]。临床上ETEC感染的症状通常为急性水样腹泻,有从温和到严重多种程度。感染者也曾出现头痛、发烧、恶心、呕吐等症状。
肠侵袭性大肠杆菌(enteroinvasive Escherichia coli,EIEC)是一类具有强致病毒力的病原菌,能够感染较大儿童及成年人。常见的临床症状主要是水样腹泻,少数人出现痢疾症状,通常是以食物或水源为媒介进行感染的,也可经过亲密接触传播产生散发病例[14]。
肠致病性大肠杆菌(enteropathic Escherichia co-li,EPEC)是在小肠上段寄居繁殖的一类病原微生物,EPEC在1945年就被认定为致泻大肠杆菌,主要引起婴幼儿急性腹泻或慢性腹泻以及成人散发腹泻。
肠出血性大肠杆菌(enterohemorrhagic Escheri-chia coli,EHEC)是一类毒力强、致病性高的病原菌,肠出血性大肠杆菌感染是一种人畜共患病,传染源通常是带菌者、感染病人及畜禽。其中牛的带菌率最高,因此,牛肉、奶制品等动物来源食品通常是主要的污染源。畜禽的粪便同样会对环境中的土壤和水源产生污染使其污染范围更加广泛。其主要临床症状为右下腹剧烈疼痛、腹泻、便血,并伴有低热或不发热,病程7-10 d,少数病人特别是幼儿及老人可并发溶血尿毒综合征,病情较为严重[15]。
肠聚集性大肠杆菌(enteroaggregative Escheric-hia coli,EAEC)是Nataro等于1987年首次从智利腹泻儿童的粪便中分离出的一种大肠杆菌,其特点是在组织培养过程中能以特有的“聚集样”或“砖堆样”的方式黏附于组织培养的上皮细胞株HEp-2,后被命名为肠聚集性大肠杆菌(EAEC或EAggEC)。其在侵染时不侵入肠上皮细胞,不产生LT、ST或是VT毒素,临床表现为急性、持续性腹泻和腹泻,含粘液的水样分泌性腹泻是其典型表现,部分病人伴有发热、呕吐、腹痛,1/3病人有便血症状[16]。
弥散黏附性大肠杆菌(dispersion adhesion Escherichia coli,DAEC)的感染病例较为少见,该菌曾引起墨西哥儿童腹泻从而被分离发现。
2.3 防范措施针对致病性大肠杆菌的污染情况以及感染途径,国家应加强对其监测力度,掌握大肠杆菌的分布特征及疾病流行趋势并建立早期预警机制,一旦发生疫情立即进行诊断并及时预警,而后进行严密监测,及时防控,防止病情蔓延。中央还应对地方进行统一应急管理,疫情发生后及时向民众通报实施状况。有关部门还需加强对食品和水源的管理,并确保禽畜养殖场对污染物进行处理后排放。
个人也应加强卫生习惯以避免感染病原菌,应保持双手清洁,饮用煮沸的自来水,避免进食未经彻底杀菌的带菌率高的食物。体质较弱者及外出旅游者应补食乳酸菌,以细菌间的拮抗作用防止大肠杆菌的侵染。
3 大肠杆菌耐药性的相关研究 3.1 耐药性与菌株血清型近年来的研究表明,血清型大肠杆菌已产生较高的耐药性,且相对于其他血清型,O141含有更多的独立因子。此外近些年研究结果表明O6、O8、O18三种血清型的菌株普遍对氨苄西林产生较高抗性[17],抗生素的滥用是细菌产生耐药性主要原因。
3.2 耐药性与毒力基因相较于国内,国外对于毒力基因与耐药性有更多研究。Ojo等[18]针对O157、O26、O91、O103、O111、O128、O145这7种产志贺毒素的大肠杆菌(shiga toxin Escherichia coli,STEC)进行了一系列研究,结果表明STEC菌株携带的主要毒力基因为Stx1、Stx2、EaeA及HlyA。STEC菌株对氨苄西林、四环素、链霉素及氯霉素等耐药率超过40%。Soufi等[19]以从突尼斯家禽肉中分离出来的166株大肠杆菌为研究对象,对其抗药性表现和基因型及其耐磺胺基因的侧翼区及整合子进行了研究分析发现,大肠杆菌对氨苄青霉素、链霉素、萘啶酸、磺胺类及四环素都具有较高抗性,而对庆大霉素、阿莫西林-克拉维酸及头孢西丁的抗性则不够显著,分析可知大肠杆菌携带的耐药基因主要为Bla(TEM)、Tet(A)/Tet(B)、Aph(3’)-Ia,Aac(6’)-Ib-cr、Aac(3)-II及CmlA。因此,大肠杆菌针对氨苄西林、四环素及链霉素表现出明显的耐药性。
3.3 耐药性与动物来源根据近年来报道,禽畜肉及奶制品成为主要污染源。Ewers等[20]对禽致病性大肠杆菌进行研究发现,从已爆发疾病的病鸡体内分离出的121株菌株、从健康鸡粪便中分离出的211株以及从它们所生存的环境中分离的35株菌株中,有高达79.2%-89.3%的菌株存在耐药性。de Jong和Bywater等[21]研究了从鸡、猪、牛中分离的大肠杆菌对几种新型抗生素(头孢吡肟、头孢噻肟和环丙沙星)的耐药力,根据研究资料显示,虽然大肠杆菌对这些新型抗生素表现出了较低的抗性,但从鸡体内分离出的大肠杆菌对这些新型抗生素已经出现敏感性降低的现象。研究进一步表明,除欧洲国家外,相较于从牛源分离出的大肠杆菌,猪源、鸡源分离出的通常具有更高的耐药性及毒力基因。以上研究均说明不同动物来源的大肠杆菌有着不同的耐药性。
4 风险毒力因子的研究致病性大肠杆菌具有多种毒力因子,主要分为黏附素和毒素两种类别。
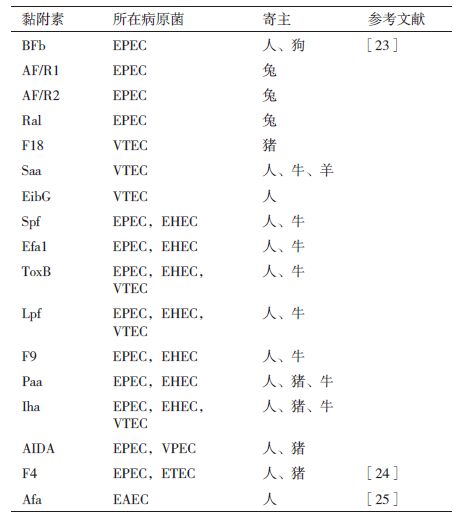
4.1 黏附素和定植因子黏附素是存在于细菌表面的某种特定的蛋白质结构或糖脂成分,存在于细菌的细胞壁、外膜蛋白、鞭毛、菌毛等结构中,能够使细菌黏附到宿主靶细胞上,对细菌的定植起着重要的作用。黏附素又分为菌毛和非菌毛黏附物质[22]。大肠杆菌的17种黏附素主要存在于肠致病性大肠杆菌、产Vero毒素大肠杆菌(verocytotoxin-producing Escherichia coli,VTEC)和肠出血性大肠杆菌(EHEC)中,少数存在于肠聚集性大肠杆菌(EAEC)和肠产毒性大肠杆菌(ETEC)中,主要的寄主是人、牛、猪、兔等(表 1)。对黏附素的研究在大肠杆菌的致病机制及诊断疫苗研制等方面具有重要意义。
首次在猪和牛中分离出来的产肠毒素大肠杆菌中发现了菌毛和菌毛黏附素,后来又在人源大肠杆菌中发现[23]。最典型的是由肠致病性大肠杆菌(EPEC)产生的F4菌毛[26]。Devriendt等[27]通过研究发现F4菌毛是影响细菌黏附肠上皮细胞及影响肠上皮细胞分泌细胞因子(cytokine)的主要因素。已有研究表明肠上皮细胞通过分泌一些细胞因子调节肠道免疫系统,F4菌毛通过抑制IL-6和IL-8这两种细胞因子的分泌使感染了产肠毒素大肠杆菌的仔猪产生严重的腹泻现象。
4.1.2 非菌毛黏附素Duguid等于1995年发现无菌毛的大肠杆菌也能造成红细胞的聚集,非菌毛黏附素(Afa)家族由这些导致红细胞凝集的血凝素组成,编码基因位于质粒或染色体上[25]。Afa家族中的大肠杆菌可在Hep-2细胞、HeLa细胞或MDCK细胞周围表现扩散粘连,这类大肠杆菌成为弥散黏附性大肠杆菌(DAEC),在这个家族中,还有一类大肠杆菌虽然不表现扩散粘连,但也可以聚集黏附到Hep-2细胞、HeLa细胞上,具有这些特点的大肠杆菌都叫做肠黏附性大肠杆菌(EAEC)[28, 29]。
4.1.3 外膜蛋白外膜蛋白(outer membrane protei-ns,OMPs)是一种非常重要的蛋白质,广泛存在原核生物的细胞外膜和真核生物的细胞器外膜中。大肠杆菌的黏附素也能存在于外膜蛋白中,这种外膜蛋白能够引发细菌与真核细胞之间的黏附。EPEC和VTEC两种病原菌的质粒能够编码不同特异性的外膜蛋白从而使人和猪受到弥散黏附感染[26]。
4.1.4 定植因子定植因子(colonization factors,CFs)是一类异质性蛋白质样的表面结构,目前已发现至少25种定植因子,多数由质粒编码。最常见的定植因子是CFA/A,包括其在内的已鉴别的定植因子中大多数被认为是糖蛋白偶联物,除此之外仍有部分定植因子还未被鉴别[30]。
4.2 LEE毒力岛及其主要编码的致病基因毒力岛(pathogenicity islands)是编码毒素、黏附素等一些毒力因子的毒力基因位于病原菌染色体上的特定区域[31]。EPEC的肠细胞脱落位点(LEE)毒力岛包含了与黏附和脱落损伤(AE损伤)及一些其他毒力因子相关的所有编码基因,主要编码Ⅲ型分泌系统(ESC或SEP)、分泌型蛋白质(ESP)及其分子伴侣、外膜蛋白紧密素(EAE)和紧密素易位受体(TIR)等[32]。
Ⅲ型分泌系统是病菌分泌毒力蛋白或向宿主细胞注入毒力蛋白的通道,由多组分蛋白复合体形成,其一系列基因位于LEE的左侧。位于LEE右侧的Esps则编码Ⅲ型分泌系统所要分泌的蛋白质。外膜蛋白紧密素(EAE)所有能引起AE损伤的病原菌都可检测到的基因,或可检测到其同源序列,是引起AE损伤的主要基因。Tir基因位于Eae基因前方,编码一种成为宿主与致病菌结合桥梁的蛋白,是重要的体内致病因子。
4.3 毒素病原微生物产生的毒素具有多种类型。有些病原菌可以产生寡肽毒素,寡肽素可黏附于胞质膜受体上并激活胞内联级反应,从而干扰真核细胞新陈代谢,大肠杆菌可产生的寡肽毒素主要是耐热肠毒素、热稳定性肠毒素。有些病原菌产生AB毒素,这种毒素以A亚基为毒力中心,B亚基则使该毒素与细胞膜上受体结合,两个亚基共同组成AB毒素的编码区域。有的产生一种细胞溶解素-RTX毒素,这种毒素可以使细胞质膜产生缝隙,从而使得细胞溶解、死亡[33]。
5 国内外大肠杆菌检测方法的研究进展 5.1 大肠杆菌传统检测方法根据食品安全国家标准(GB 4789.3-2010)规定,现行的大肠杆检测方法主要有两种,大肠菌群MPN计数法和大肠菌群平板计数法,也是最为传统的检测方法。在MPN检测法中,首先根据待检菌在LST肉汤管中是否产气来进行初步判断,不产气则判断为大肠杆菌阴性,产气的进行BGLB肉汤培养,若在BGLB肉汤中培养仍产气则报告为大肠杆菌阳性,否则报告为大肠杆菌阴性,最后通过查MPN表计数。平板计数法是将10倍系列稀释的菌液接种VRBA平板,计数典型可疑菌落,并接种可疑菌落于BGLB肉汤中,报告实验结果。具体实验材料及步骤可参见《食品安全国家标准 食品微生物学检验 大肠菌群计数》。
5.2 传统检测方法的劣势及发展趋势传统的大肠杆菌检测方法耗时较长且操作复杂,不适合进行食品检疫等大批量样品的检测。医疗中,如果检测时间过长也可能会导致诊治的延迟。因此,多年来相关研究人员一直致力于研究快速、灵敏、特异的大肠杆菌检测方法。
5.3 新型检测方法 5.3.1 免疫学检测技术主要包括酶联免疫吸附技术、免疫层析技术、量子点荧光探针技术。
酶联免疫吸附技术(ELISA)是指利用抗原抗体能够特异结合的特性,使之产生颜色反应从而定性定量的进行分析。Ge等[34]建立了一种高灵敏度、高特异性的PCR-ELISA技术用于检测大肠埃希氏菌O157: H7以及其他产志贺毒素的食源性大肠杆菌。Galikowska等[35]利用噬菌体代替抗体,进行ELISA实验用来检测大肠杆菌O157: H7,这种方法具有简单、快速、经济等特点。
免疫层析技术是通过毛细管作用使抗原或抗体与已知抗原抗体的微孔滤膜载体相连接,通过标记物进行定性检测,具有快速,适用范围广、稳定易判断等优点,但灵敏度稍显逊色。Park等[36]应用免疫层析技术对大肠杆菌O157: H7、鼠伤寒沙门氏菌、金黄色葡萄球菌等病原菌进行了检测,其中大肠杆菌O157: H7的检测范围是9.2×103-9.2×105 CFU/mL。Yonekita等[37]开发了一款免疫层析试纸从115种肉制品中成功检测出5株E.coli O26菌株,检测范围是2.2×103 到1.0×105 CFU/mL。Cui等[38]应用胶体金免疫层析法结合免疫磁珠分离技术对大肠杆菌O157: H7进行了快速检测,该方法特异性强、灵敏度低,检测限达10 CFU/g。
量子点荧光探针技术通过激发使量子点使其具有荧光特性,后与相关抗体结合形成荧光免疫复合物,从而通过复合物的荧光强度测定目标菌浓度。这种方法的优势在于效率高、稳定性好且具有较高的灵敏度。Mitchell等[39]在免疫磁珠和DNA上修饰量子点,形成免疫磁珠-DNA-量子点复合物,对大肠杆菌O157: H7的EaeA基因进行了特异性检测,通过荧光检测系统检测反应后体系的荧光强度的大小来判定样品中含大肠杆菌量的多少,该方法最低检测限为49×10-15 mol/L。Sanvicens等[40]建立了一种量子点荧光抗体阵列检测大肠杆菌O157: H7,最低检测限为10 CFU/mL,相较于传统的ELISA方法,这种方法将检测限提高了3个数量级。
5.3.2 分子生物学检测技术分子生物学检测技术主要是建立在PCR基础上衍生出来的检测方法。包括常规PCR、实时荧光定量PCR、多重PCR、基因芯片及新型核酸扩增技术等。
5.3.2.1 PCR常规PCR技术是在短时间内对特定DNA序列进行大量体外扩增,后通过电泳技术完成特定序列的检测。优点是能够快速进行微量的检测,且具有较高的灵敏度,但是容易产生假阳性。Sangdee等[41]针对肠杆菌基因重复序列(ERIC)设计引物用于大肠杆菌的检测。该方法具有良好的特异性,检测限100 CFU/ mL。
5.3.2.2 定量PCR实时荧光定量PCR是在传统PCR的基础上发展起来的一种定量方法,通过在反应体系中加入特定荧光物质(荧光探针或核酸染料),对扩增产物进行跟踪标记,也可进行实时监控,最后根据标准曲线进行监测分析。该方法优势在于灵敏度较高,操作简单,实现靶物质的定量分析。Wang等[42]针对大肠杆菌和志贺氏菌的Tuf基因设计特异性引物和探针实现了定量检测,该方法操作简便,短时间内能从生牛奶中同时检测出两种目标致病菌,其中大肠杆菌在102 CFU/mL-106 CFU/mL范围内具有良好的线性关系。Kim等[43]针对大肠杆菌O157: H7的EaeA基因设计了新引物和探针,实现了对鲜切卷心菜中的大肠杆菌O157: H7的定量PCR检测,检测灵敏度提高两个数量级至1.53 CFU/mL。Khatami等[44]开发了一种基于荧光PCR的特异性杂交方法(FLASH-PCR)检测大肠杆菌O157: H7中由STX编码的Stx-1基因,实验结果表明该方法与传统方法检测结果相符,与后者相比检测过程更为快速、灵敏、简单。
5.3.2.3 高通量PCR高通量检测技术是近几年基于PCR技术发展起来的新技术,相比传统的病原微生物检测方法,该方法解决了每次只能检测一种或一类样品的局限性,其具有反应体系简单、自动化操作程度较高、快速省时、无污染及诊断结果精确等特点。目前用于病原微生物鉴定的高通量检测技术主要有: 多重PCR技术、焦磷酸测序技术、二代测序、变性高效液相色谱分析技术和芯片技术等。
多重PCR是一种可以同时进行多个目标菌或基因检测的技术,在反应体系中同时加入多种特异性引物即可扩增出多种DNA片段。这种方法的优点在于检测速度快、特异性高及简便高效。缺点在于反应体系中各种引物容易相互干扰,产生竞争性抑制。Son等[45]针对产志贺毒素大肠杆菌的Stx1、Stx2、Eae、EhxA及UidA五种毒力基因建立了多重PCR检测方法,该方法有效提高了检测的准确性,对其毒力基因的分析更加便捷,同时设计探针实现定量检测。Gordillo等[46]针对大肠杆菌O157: H7的FliCh7和 RfbE基因构建了引物和探针,建立了双重定量PCR技术用于肉制品的检测,检测结果显示本方法的准确度要优于传统方法,两个基因的结果均具有良好的线性关系。
随着大规模基因组学的兴起,二代测序技术(新一代测序技术)也随之产生。二代测序技术大多采用大型矩阵结构的微阵列分析技术,使用引物和DNA聚合酶或连接酶扩展延伸核酸链。这种方法通过连续记录光信号周期来获取序列信息,可用于大规模的测序。市场上已有多种测序仪,如Illumina公司的Genome Analyzer及HiSeq 2000 系统,ABI公司的AB SoLid sequencer等。随着2008年Helicos Biosciences公司的第一台DNA分子测序仪的问世,基因组学迎来了新的发展时期,同时也为微生物检测提供给了新的途径。焦磷酸测序(Pyrosequencing)技术是在20世纪90年代年发明的DNA序列分析技术,这种技术是由酶级联化学发光反应发展出的,该反应由4种酶(DNA聚合酶、三磷酸腺硫酸化酶、荧光素酶和双磷酸酶)催化的同一反应体系产生。该技术是在每一轮测序反应中,只加入一种dNTP,当加入的这种dNTP能够与模板进行配对时,聚合酶就会使其连接到引物链上,形成磷酸二酯键并释放出等量的焦磷酸基团(PPi),通过检测转化为可见光信号的PPi量来确定参与反应中的核苷酸数目[47]。
虽然第二代测序技术在全球测序市场上占有绝对优势,但新一代的测序技术又快速发展起来。以PacBio公司的SMRT和Oxford Nanopore Technologies纳米孔单分子测序技术,被称之为第三代测序技术。与前两代相比最大的特点就是单分子测序,并且在测序过程不需要PCR扩增。SMRT技术以SMRT芯片为测序载体边合成边测序,通过碱基配对发出光的波长与峰值来判断插入的碱基类型。SMRT技术的测序速度达到每秒约10个dNTP,最大的劣势就是测序错误率比较高。纳米单分子测序技术基于电信号的测序技术,DNA穿过特殊的纳米孔时,通过监测电荷变化从而鉴定所通过的碱基。第三代测序技术是为了解决第二代所存在的缺点而开发的,它最大的特点是单分子测序,且能避免由于PCR扩增造成的错误,极大的提高了测序长度,为微生物的测序分析提供了新的解决方法[48]。
变性高效液相色谱分析技术(denaturing high-performance liquid chromatography,DHPLC)是利用离子配对逆相层析的原理来分析核酸的检测方法,这种方法利用离子配对试剂TEAA(triethylammonium acetate)在核酸非变性条件下与DNA的磷酸基团离子配对,带正电的TEAA会将带负电的核酸包裹,并以其非极性的烷基端吸附在非极性的固定相上,DNA因此滞留于分离管柱中。DHPLC系统对DNA片段的分离是基于其长度,而非序列,DNA链越长,所带磷酸基团就越多,与之结合的TAEE就越多,其在柱内保留的时间也就相应增长。William等[49]通过不同谱系39种细菌进行DHPLC检测,成功地把DHPLC技术应用于细菌菌种的鉴定。
基因芯片技术是指将与荧光标记探针结合后的样品PCR扩增产物与芯片上的寡核苷酸进行杂交,通过荧光强度及分布进行分析鉴定。Suo等[50]对基因芯片进行了优化用于检测食源性致病菌,并对26种鲜肉样品进行了检测。Kim等[51]对基因芯片技术进行了改进,使得检出假阴性概率几乎为零。
5.3.2.4 等温PCR等温扩增技术的出现不仅使得反应时间大大缩短,同时也简化了操作过程中所使用的仪器,大大满足了快速简便的要求。目前,多种基于PCR的等温扩增技术逐渐发展起来,如核酸序列依赖的扩增(nucleic acid sequence based amplification,NASBA),高温解旋酶依赖性扩增(thermophilic helicase-dependent amplification,tHDA),链置换扩增(strand-displacement amplification,SDA),环介导等温扩增(loop-mediated isothermal amplification,LAMP),滚环扩增(rolling circle amplification,RCA),重组聚合酶扩增(recombinase polymerase reaction,RPA)恒温和嵌合引物引发的核酸扩增(chimeric primer-initiated amplification of nucleic acids,ICAN)以及切刻内切酶信号放大反应(nicking endonuclease signal amplification,NESA)。这些技术在反应速度、反应温度、工艺简化、对DNA或RNA的特异性、对酶的依赖、检测范围、引物设计及成本等方面都有着自身的优缺点[52]。
环介导等温扩增(LAMP)是一种发展最为成熟的等温核酸扩增技术,其反应温度为60-65℃,是DNA双链复性和延伸的中间温度,DNA在此温度中处于一种单双链的动态平衡。依据6个区域设计4条引物,实现循环扩增,具有高效的特异性和灵敏度[53]。Wang等[54]利用这种技术对生乳中的大肠杆菌O157进行了检测,同时结合核酸染料实现活细胞的检测,检测灵敏度是 PCR 方法的 1 000 倍。Wang等[55]分别针对产志贺毒素大肠杆菌的Stx1、Stx2和Eae基因设计引物,利用LAMP 技术对莴苣、菠菜样品中的产志贺毒素大肠杆菌进行了检测,该方法的准确性和灵敏度与定量PCR方法几乎一致,其显示了在食品中大肠杆菌现场快速检测方面良好的应用前景。
5.3.2.5 ERIC-PCR肠杆菌属间的共有重复序列(enterobacterial repetitive intergenic consensus,ERIC)是Sharples等[56]于1990年在大肠杆菌发现的一组序列,它具有一段保守性很高的反向重复序列,序列长约44 bp。虽然功能尚未研究清楚,但了解到其在基因组的分配比较均匀,且存在的位置和拷贝随物种的不同而有差异。Versalovic[57]根据这一核心序列设计了反向引物,用于扩增ERIC两端之间的片段,建立了ERIC-PCR技术。由于在不同种属或同种不同菌株之间的拷贝数和定位都不同,通常被用于细菌基因组图谱的研究,用于细菌的分类与鉴定。在应用该方法的过程中发现,ERIC结果的带型和条带的强弱易受PCR的反应参数和电泳条件的影响,因此有人建议将所有的样本在相对集中的时间用同一条件进行分析,以保证其结果的可比性[58]。
5.3.3 死活菌检测技术传统的死活菌检测技术是根据每个活菌可以在平板上长出一个菌落来进行活菌计数的,但该方法局限于检测能形成菌落的细菌,并且需要将菌液浓度稀释到一定程度,因此,于PCR的新型死活菌检测技术应运而生。目前发展出的主要有RT-PCR、EMA-PCR和PMA-PCR。
RT-PCR是以细菌中适当的mRNA作为靶基因,由于细菌死亡过后mRNA会快速降解,不会像DNA一样降解缓慢,所以运用 RT-PCR 技术只能检测到具有活性的细菌,从而避免了假阳性的出现[59]。EMA-PCR是根据叠氮溴乙锭(ethidiummonoazide bromide,EMA)能够选择性地进入死细胞的细胞膜内并与DNA发生不可逆的共价结合从而抑制DNA的PCR扩增,因此只有活菌能够进行PCR扩增[60]。Shi等[61]对EMA的反应条件进行了优化,包括体系的温度、pH、渗透压,并记录了EMA-qPCR 的检测结果。当EMA染料进入死菌并与之核酸结合时,扩增反应会被抑制。研究还发现光的存在可以推动EMA与DNA的结合。优化后EMA的使用浓度为10 μg/mL,光照时间为20 min,加热可以使不同状态的细胞都能够与EMA充分结合,对细胞进行85℃灭活35 min后,EMA-qPCR 即无法检出活菌,说明了这种检测方式的有效性。高渗透压(≥4%)会对EMA-qPCR产生抑制,且渗透压越高,抑制效果越明显。当渗透压达到8%时,EMA-qPCR扩增出的菌数量急剧减少。将细胞在不同的pH溶液中处理后再用EMA处理发现,pH从1升高到5时,其Ct值显著增加,pH大于5以后无明显变化,试验中还发现pH为3时,细胞数量已经开始下降。用LB培养基对已经损伤酸化了的细胞进行40 min孵育,损伤的细胞即可复原且EMA不能进入到其内部。PMA-PCR是对于EMA-PCR的一种改进方法,由于叠氮溴化乙锭(EMA)具有细胞毒性,使得EMA-PCR应用受限,因此,选用PMA来代替EMA进行死活菌检测可以有效扩大其应用范围,如临床医学、食品安全等领域[62]。
5.3.4 其他新型检测技术 5.3.4.1 红外光谱技术红外光谱技术主要用于测定微生物的傅里叶变换红外光谱来获得微生物及其生物大分子结构的信息,由此鉴定微生物种类及状态。慈云祥等[63]利用红外光谱技术对大肠杆菌、金黄色葡萄球菌、酵母菌及谷氨酸菌进行了研究,大肠杆菌的实验结果表明,培养时间不同、微生物含量不同对FTIR图谱没有明显影响,并且测定出大肠杆菌的吸收带波数为1 080和1 238 cm-1。Siripatrawan等[64]利用近红外光谱技术结合化学统计学方法定性、定量地对大肠杆菌ATCC25922及大肠杆菌K12两种菌株进行了分析检测。
5.3.4.2 生物传感器技术传感器分为3种类型: 物理传感器、化学传感器及生物传感器。目前用于微生物快速检测的传感器技术主要有电化学传感器、光学传感器等。电化学传感器是微生物利用低电导率的大分子物质进行代谢,使其分解物带有一定电荷,因此电导率发生变化,将电信号放大后记录其变化即可对微生物量进行分析。此方法具有快速廉价等优点。光学传感器是利用微生物产生的代谢产物使预先加入的指示剂变色,由光学检测器检测其颜色变化,找到突变点时间从而确定微生物的数量。
在各种类型的传感器中,生物传感器是最具优势的一类,它具有敏感性、专一性,可以进行实时测定,且具有时效性。生物传感器可与纳米材料结合,用于测定核酸、酶、抗体、噬菌体甚至整个细胞。一种具有高特异性和敏感性的RNA生物传感器被构建用于检测具有活性的大肠杆菌,用mRNA(ClpB)基因构造的基于NASBA的生物传感器可用于对活性大肠杆菌进行定性和定量[65]。Varshney等[66]构建了一种电阻生物传感器,用于鉴定具有活性的大肠杆菌O157: H7,如果细菌生长,就会监测到有电阻产生。Chang 等[67]利用EMA构造了一种微流控平台,用于检测区分有无活性的大肠杆菌,采用纳米金探针对活性细胞进行探测,并使得具有生物活性的细胞得以扩增,这是第一次在同一微流控平台上对活性细菌进行这两项连续的操作。
侧向层析检测(lateral flow testsL,FT)及其试纸条已经成为实验室的诊断工具,是一种基于纸张的生物传感器,这种技术具有简单、准确、快速、廉价的优点,不需要专业的技术或复杂的仪器。这种试纸条通常被胶体金、乳胶、碳、磷、单链核酸等标记,最常见的是胶体金粒子试纸条。目前,LFT被认为具有高效检测细菌的巨大潜力,大肠杆菌、金黄色葡萄球菌、李斯特菌等都可以用这种试纸条进行快速检测。
除了传统的生物传感器,一些新型的传感器也被构建出来用于检测一些特定的细菌。Li等[68]构建出了一种用于检测大肠杆菌O157: H7 的新型传感器,这种传感器采用了一种由二茂铁与抗菌肽相组成的薄膜,其上的马加宁抗菌肽为生物识别元件。Chan等[69]基于免疫传感器研究出一种用于检测大肠杆菌O157: H7的生物功能磁珠,可用于在纳米多孔氧化铝膜上进行细菌细胞浓度的测定,这种传感器具有高度的敏感性。
5.3.4.3 适配体适配体是通过指数富集配体进化技术从体外筛选得到的一类单链寡核苷酸,能够特异性的与靶分子结合并具有较高的亲和性,是由Ellington和 Tuerk于1990年首次提出的[70-72]。适配体与靶分子的特异性结合基于其单链结构和空间结构的多样性,高亲和性基于核酸分子间的碱基互补配对、静电作用、氢键以及其自身的适应性折叠。人们利用基于细胞层面的SELEX技术发现了一种仅结合于E. coli O157: H7血清型菌株细胞表面的一种RNA适配体,该适配体还具有核酸酶抗性,这种在适配体可用于大肠杆菌O157: H7菌株的特异性鉴别。化脓链球菌(Streptococcus pyogenes)是一类曾引起过许多严重临床疾病的致病菌,是A型链球菌(group A Streptococcus,GAS)的一种,目前发现一种能够选择性识别10种主要A型链球菌的DNA适配体序列,可用于A型链球菌的特异性检测。
6 展望目前,国内外大肠杆菌的疫情爆发依然时有发生,究其根本主要还是通过食物传播。因此,大肠杆菌在分析、诊断、治疗等各个方面均有待进一步的研究。随着外界环境的影响,可能会引发大肠杆菌基因的变化,如基因突变、单核苷酸的多态性等,因此在今后的研究中,需要将研究重点向基因组、尤其是毒力基因的分析方面进行拓展,为大肠杆菌的风险评估提供更加可靠的理论基础。随着生物技术的大力发展,对于大肠杆菌的新型检测方法不断被开发应用,但快速、特异、灵敏、高通量仍然是新型检测方法开发需要继续改进方向,迎合企业及政府等现场筛查检测的需求。目前研究重点均是围绕检测技术展开,因此在今后的研究中,需要将重点向寻找新的抑菌方法进行拓展,保证大肠杆菌的有效治疗。
目前大量抗生素的使用使病原菌变得更加耐药,为了防止动物感染病原菌,饲料中添加的大量抗生素最终使食用这些动物的人类产生耐药性,针对此应加强对病原菌的监控以及抗生素类用药的管理。此外,对于大肠杆菌新毒力因子的挖掘以及风险评估研究也应继续拓宽、深入,掌握致病机理能够方便人们针对不同的致病原因寻求更加有效治疗方案。
| [1] | Rodríguez-Angeles G. Principal characteristics and diagnosis of the pathogenic groups of Escherichia coli. Salud Publica de Mexico , 2001, 44 (5) : 464–475. |
| [2] | Riley LW, Remis RS, Helgerson SD, et al. Hemorrhagic colitis associated with a rare Escherichia coli serotype. New England Journal of Medicine , 1983, 308 (12) : 681–685. DOI:10.1056/NEJM198303243081203 |
| [3] | 王燕, 谢贵林, 杜琳. 大肠杆菌O157: H7感染流行概况. 微生物学免疫学进展 , 2008, 1 : 51–58. |
| [4] | 汪华, 景怀琦, 李红卫, 等. 江苏省淮北地区肠出血性大肠埃希菌O157: H7感染性腹泻并发急性肾衰的研究. 中华流行病学杂志 , 2004, 11 : 24–26. |
| [5] | 徐进, 庞璐. 食品安全微生物学指示菌国内外标准应用的比较分析. 中国食品卫生杂志 , 2011, 5 : 472–477. |
| [6] | 黄介枚. 国外大肠杆菌O157感染的爆发流行近况与防治策略. 广东卫生防疫 , 1997, 1 : 89–92. |
| [7] | 张桃香. 中国东部地带性土壤中大肠杆菌O157: H7存活和吸附机制的研究[D]. 杭州: 浙江大学, 2014. http://cdmd.cnki.com.cn/article/cdmd-10335-1014361610.htm |
| [8] | Gagliardi JV, Karns JS. Leaching of Escherichia coli O157: H7 in diverse soils under various agricultural management practices. Applied and Environmental Microbiology , 2000, 66 (3) : 877–883. DOI:10.1128/AEM.66.3.877-883.2000 |
| [9] | 周淑玉, 王俊起, 王京京. 北京污灌区土壤的细菌污染及其存活时间的实验研究. 卫生研究 , 1987, 2 : 17–20. |
| [10] | 朱奇, 陈彦, 许伟. 聊城市春季蔬菓生物污染的初步调查. 山东师大学报: 自然科学版 , 2000, 15 (2) : 186–189. |
| [11] | 叶小梅, 常志州, 陈欣, 等. 畜禽养殖场排放物病原微生物危险性调查. 生态与农村环境学报 , 2007, 2 : 66–70. |
| [12] | 蒋磊, 周明旭, 夏芃芃, 等. 产肠毒素大肠杆菌感染的分子致病机制. 中国兽医学报 , 2014, 9 : 1551–1560. |
| [13] | Petri WA, Miller M, Binder HJ, et al. Enteric infections, diarrhea, and their impact on function and development. The Journal of Clinical Investigation , 2008, 118 (4) : 1277–1290. DOI:10.1172/JCI34005 |
| [14] | Kaper JB, Nataro JP, Mobley HL, et al. Pathogenic Escherichia coli. Nature Reviews Microbiology , 2004, 22 : 123–140. |
| [15] | 朱蓓. 肠出血性大肠杆菌感染的流行病学及临床医学资料概述. 解放军预防医学杂志 , 2011, 4 : 309–311. |
| [16] | 李惠民, 骆金芝. 肠聚集性大肠杆菌腹泻的研究进展. 国外医学(儿科学分册) , 2000, 4 : 207–209. |
| [17] | 安微, 张秀英, 李蕊, 等. 致病性大肠杆菌毒力因子和耐药性研究进展. 畜牧与兽医 , 2013, 8 : 106–110. |
| [18] | Ojo OE, Ajuwape ATP, Otesile EB, et al. Potentially zoonotic shiga toxin-producing Escherichia coli serogroups in the faeces and meat of food-producing animals in Ibadan, Nigeria. International Journal of Food Microbiology , 2010, 142 (1) : 214–221. |
| [19] | Soufi L, Sáenz Y, Vinué L, et al. Escherichia coli of poultry food origin as reservoir of sulphonamide resistance genes and integrons. International Journal of Food Microbiology , 2010, 144 (3) : 497–502. |
| [20] | Ewers C, Ant?o EM, Diehl I, et al. Intestine and environment of the chicken as reservoirs for extraintestinal pathogenic Escherichia coli strains with zoonotic potential. Applied and Environmental Microbiology , 2009, 75 (1) : 184–192. DOI:10.1128/AEM.01324-08 |
| [21] | de Jong A, Bywater R, Butty P, et al. A pan-European survey of antimicrobial susceptibility towards human-use antimicrobial drugs among zoonotic and commensal enteric bacteria isolated from healthy food-producing animals. Journal of Antimicrobial Chemotherapy , 2009, 63 (4) : 733–744. DOI:10.1093/jac/dkp012 |
| [22] | 严亚贤, 华修国. 大肠杆菌O157和其它产志贺毒素大肠杆菌的毒力因子-黏附素. 中国预防兽医学报 , 2002, 6 : 81–84. |
| [23] | Gyles CL, Fairbrother JM. Pathogenesis of bacterial infections in animals[M]. USA: Wiley-Blackwell, 2010. |
| [24] | Bianchi ATJ, Scholten JW, Van ZAM, et al. Parenteral vaccination of mice and piglets with F4+ Escherichia coli suppresses the enteric anti-F4 response upon oral infection. Vaccine , 1996, 14 (3) : 199–206. DOI:10.1016/0264-410X(95)00192-4 |
| [25] | Harrington SM, Dudley EG, Nataro JP. Pathogenesis of enteroaggr-egative Escherichia coli infection. FEMS Microbiology Lett-ers , 2006, 254 (1) : 12–18. DOI:10.1111/fml.2006.254.issue-1 |
| [26] | Bardiau M, Szalo M, Mainil JG. Initial adherence of EPEC, EHEC and VTEC to host cells. Veterinary Research , 2010, 41 (5) : 57. DOI:10.1051/vetres/2010029 |
| [27] | Devriendt B, Stuyven E, Verdonck F, et al. Enterotoxigenic Escherichia coli(K88)induce prionflammatory responses in porcine intestinal epithelial cells. Developmental and Comparative Immunology , 2010, 34 (11) : 1175–1182. DOI:10.1016/j.dci.2010.06.009 |
| [28] | Piérard D, De GH, Haesebrouck F, et al. O157: H7 and O104: H4 Vero/Shiga toxin-producing Escherichia coli outbreaks: respective role of cattle and humans. Veterinary Research , 2012, 43 (1) : 1. DOI:10.1186/1297-9716-43-1 |
| [29] | Scheutz F, Nielsen EM, Frimodt MJ, et al. Characteristics of the enteroaggregative Shiga toxin/verotoxin-producing Escherichia coli O104: H4 strain causing the outbreak of haemolytic uraemic syndrome in Germany, May to June 2011. Euro Surveill , 2011, 16 (24) : 19889. |
| [30] | Jansson L, Tobias J, Lebens M, et al. The major subunit, CfaB, of colonization factor antigen i from enterotoxigenic Escherichia coli is a glycosphingolipid binding protein. Infection and Immunity , 2006, 74 (6) : 3488–3497. DOI:10.1128/IAI.02006-05 |
| [31] | HackerJ, Blum-OehlerG, MühldorferI, 等. Pathogenicity islands of virulent bacteria: structure, function and impact on microbial evolution. Molecular Microbiology , 1997, 23 (6) : 1089–1097. |
| [32] | McDaniel TK, Jarvis KG, Donnenberg MS, et al. A genetic locus of enterocyte effacement conserved among diverse enterobacterial pathogens. Proceedings of the National Academy of Sciences , 1995, 92 (5) : 1664–1668. DOI:10.1073/pnas.92.5.1664 |
| [33] | Marchès O, Nougayrède JP, Boullier S, et al. Role of tir and intimin in the virulence of rabbit enteropathogenic Escherichia coli serotype O103: H2. Infection and Immunity , 2000, 68 (4) : 2171–2182. DOI:10.1128/IAI.68.4.2171-2182.2000 |
| [34] | Ge B, Zhao S, Hall R, et al. A PCR-ELISA for detecting Shiga toxin-producing Escherichia coli. Microbes and Infection , 2002, 4 (3) : 285–290. DOI:10.1016/S1286-4579(02)01540-X |
| [35] | Galikowska E, Kunikowska D, Tokarska-Pietrzak E, et al. Specific detection of Salmonella enterica and Escherichia coli strains by using ELISA with bacteriophages as recognition agents. European Journal of Clinical Microbiology & Infectious Diseases , 2011, 30 (9) : 1067–1073. |
| [36] | Park J, Park S, Kim YK. Multiplex detection of pathogens using an immunochromatographic assay strip. BioChip Journal , 2010, 4 (4) : 305–312. DOI:10.1007/s13206-010-4407-2 |
| [37] | Yonekita T, Fujimura T, Morishita N, et al. Simple, rapid, and reli-able detection of Escherichia coli O26 using immunochromatogr-aphy. Journal of Food Protection , 2013, 76 (5) : 748–754. DOI:10.4315/0362-028X.JFP-12-423 |
| [38] | Xi C, Xiong QR, Xiong YH, et al. Establishing of a method combined immunomagnetic separation with colloidal gold lateral flow assay and its application in rapid detection of Escherichia coli O157: H7. Chinese Journal of Analytical Chemistry , 2013, 41 (12) : 1812–1816. DOI:10.1016/S1872-2040(13)60696-9 |
| [39] | Mitchell KA, Chua B, Son A. Development of first generation in-situ pathogen detection system(Gen1-IPDS)based on NanoGene assay for near real time E. coli O157: H7 detection. Biosensors and Bioelectronics , 2014, 54 : 229–236. DOI:10.1016/j.bios.2013.10.056 |
| [40] | Sanvicens N, Pascual N, Fernández-Argüelles MT, et al. Quantum dot-based array for sensitive detection of Escherichia coli. Analytical and Bioanalytical Chemistry , 2011, 399 (8) : 2755–2762. DOI:10.1007/s00216-010-4624-5 |
| [41] | Sangdee A, Natphosuk S, Srisathan A, et al. Development of SCAR primers based on a repetitive DNA fingerprint for Escherichia coli detection. Journal of Microbiology , 2013, 51 (1) : 31–35. DOI:10.1007/s12275-013-2244-4 |
| [42] | Wang Y, Zhao P, Zhang H, et al. A simple and rapid real-time PCR assay for the detection of Shigella and Escherichia coli species in raw milk. Journal für Verbraucherschutz und Lebensmittelsicherheit , 2013, 8 (4) : 313–319. DOI:10.1007/s00003-013-0837-9 |
| [43] | Kim YJ, Kim JG, Oh SW. Rapid detection of Escherichia coli O157: H7 in fresh-cut cabbage by real-time polymerase chain reaction. Journal of the Korean Society for Applied Biological Chemistry , 2011, 54 (2) : 264–268. |
| [44] | Khatami F, Heidari M, Khatami M. Rapid Detection of Escherichia coli O157: H7 by Fluorescent Amplification-Based Specific Hybridization(FLASH)PCR. Iranian Red Crescent Medical Journal , 2012, 14 (9) : 594. |
| [45] | Son I, Binet R, Maounounen LA, et al. Detection of five Shiga toxin-producing Escherichia coli genes with multiplex PCR. Food Microbiology , 2014, 40 : 31–40. DOI:10.1016/j.fm.2013.11.016 |
| [46] | Gordillo R, Rodríguez A, Werning ML, et al. Quantification of viable Escherichia coli O157: H7 in meat products by duplex real-time PCR assays. Meat Science , 2014, 96 (2) : 964–970. DOI:10.1016/j.meatsci.2013.10.018 |
| [47] | Ronaghi M, Uhlen M, Nyren P. A sequencing method based on real-timepyrophosphate. Science , 1998, 281 (5375) : 363–365. DOI:10.1126/science.281.5375.363 |
| [48] | 张得芳, 马秋月, 尹佟明, 等. 第三代测序技术及其应用. 中国生物工程杂志 , 2013, 33 (5) : 125–131. |
| [49] | William H, Dadid S, Eerik H, et al. Denaturing HPLC for identifyingbacteria. Bio Techniques , 2002, 33 (8) : 386–391. |
| [50] | Suo B, He Y, Irwin P, et al. Optimization and application of a custom microarray for the detection and genotyping of E. coli O157: H7 in fresh meat samples. Food Analytical Methods , 2013, 6 (5) : 1477–1484. DOI:10.1007/s12161-013-9639-z |
| [51] | Kim H, Kane MD, Kim S, et al. A molecular beacon DNA microarray system for rapid detection of E. coli O157: H7 that eliminates the risk of a false negative signal. Biosensors and Bioelectronics , 2007, 22 (6) : 1041–1047. DOI:10.1016/j.bios.2006.04.032 |
| [52] | Huang S, Xu Y, Yan X, et al. Development and application of a quantitative loop-mediated isothermal amplification method for detecting genetically modified maize MON863. Journal of the Science of Food and Agriculture , 2015, 95 (2) : 253–259. DOI:10.1002/jsfa.2015.95.issue-2 |
| [53] | 冯瑜菲. 猪水肿病大肠杆菌毒力因子鸡卵黄抗体制备及 Stx2基因 LAMP方法建立[D]. 哈尔滨: 东北农业大学, 2011. |
| [54] | Wang DG, Huo GC. Rapid detection viable Escherichia Coli O157 in raw milk using Loop-Mediated Isothermal Amplification with aid of ethidiu monoazide. Advanced Materials Research , 2012, 343 : 1217–1221. |
| [55] | Wang F, Yang Q, Qu Y, et al. Evaluation of a loop-mediated isothermal amplification suite for the rapid, reliable, and robust detection of Shiga toxin-producing Escherichia coli in produce. Applied and Environmental Microbiology , 2014, 80 (8) : 2516–2525. DOI:10.1128/AEM.04203-13 |
| [56] | Sharples G J, Lloyd RG. A novel repeated sequence located in the intergenic regions of bacterial chromosomes. Nucleic Acids Reserch , 1990, 18 (22) : 6503–6508. DOI:10.1093/nar/18.22.6503 |
| [57] | Versalovic J, Koeuth T, Lupski JR. Distribution of repetitive DNA sequences in eubacteria and application to fingerprinting of bacterial genomes. Nucleic Acids Reserch , 1991, 19 (24) : 6823–6831. DOI:10.1093/nar/19.24.6823 |
| [58] | 陈玉婷, 程楠, 许文涛. 食源性致病微生物的检测新技术. 食品安全质量检测学报 , 2015, 9 : 3405–3413. |
| [59] | Takayama K, Kjelleberg SA. The role of RNA stability during bacterial stress responses and starvation. Environ Microbiol , 2000, 2 (4) : 355–365. DOI:10.1046/j.1462-2920.2000.00119.x |
| [60] | Nogva HK, Dr?mtorp SM, Nissen H, et al. Ethidium monoazide for DNA-based differentiation of viable and dead bacteria by 5-nuclease PCR. Biotechniques , 2003, 34 (4) : 804–808. |
| [61] | Shi H, Xu W, Luo Y, et al. The effect of various environmental factors on the ethidium monazite and quantitative PCR method to detect viable bacteria. Journal of Applied Microbiology , 2011, 111 (5) : 1194–1204. DOI:10.1111/jam.2011.111.issue-5 |
| [62] | 于小龙, 徐进, 张昊, 等. PMA-PCR方法快速检测VBNC状态青枯菌. 植物保护 , 2016, 1 : 144–149. |
| [63] | 慈云祥, 臧凯赛, 高体玉. 几种微生物的红外光谱研究. 高等学校化学学报 , 2002, 23 (6) : 1047–1049. |
| [64] | Siripatrawan U, Makino Y, Kawagoe Y, et al. Near infrared spectroscopy integrated with chemometrics for rapid detection of E. coli ATCC 25922 and E. coli K12. Sensors and Actuators B: Chemical , 2010, 148 (2) : 366–370. DOI:10.1016/j.snb.2010.05.024 |
| [65] | Baeumner AJ, Cohen RN, Miksic V, et al. RNA biosensor for the rapid detection of viable Escherichia coli in drinking water. Biosensors and Bioelectronics , 2003, 18 (4) : 405–413. DOI:10.1016/S0956-5663(02)00162-8 |
| [66] | Varshney M, Li Y. Double interdigitated array microelectrode-based impedance biosensor for detection of viable Escherichia coli O157: H7 in growth medium. Talanta , 2008, 74 (4) : 518–525. DOI:10.1016/j.talanta.2007.06.027 |
| [67] | Chang WH, Wang CH, Lin CL, et al. Rapid detection and typing of live bacteria from human joint fluid samples by utilizing an integrated microfluidic system. Biosensors and Bioelectronics , 2015, 66 : 148–154. DOI:10.1016/j.bios.2014.11.006 |
| [68] | Li Y, Afrasiabi R, Fathi F, et al. Impedance based detection of pathogenic E. coli O157: H7 using a ferrocene-antimicrobial peptide modified biosensor. Biosensors and Bioelectronics , 2014, 58 : 193–199. DOI:10.1016/j.bios.2014.02.045 |
| [69] | Chan KY, Ye WW, Zhang Y, et al. Ultrasensitive detection of E. coli O157: H7 with biofunctional magnetic bead concentration via nanoporous membrane based electrochemical immunosensor. Biosensors and Bioelectronic , 2013, 41 : 532–537. DOI:10.1016/j.bios.2012.09.016 |
| [70] | Ellington AD, Szostak JW. In vitro selection of RNA molecules that bind specific ligands. Nature , 1990, 346 (6287) : 818–822. DOI:10.1038/346818a0 |
| [71] | Tuerk C, Gold L. Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase. Science , 1990, 249 (4968) : 505–510. DOI:10.1126/science.2200121 |
| [72] | Jayasena SD. Aptamers: an emerging class of molecules that rival antibodies in diagnostics. Clinical Chemistry , 1999, 45 (9) : 1628–1650. |