2. 中国农业科学院生物技术研究所,北京 100081
2. Biotechnology Research Institute,Chinese Academy of Agricultural Sciences,Beijing 100081
侧翼序列是指染色体中特定位点两侧的DNA序列。整合进入转基因作物染色体的外源目的基因插入位点的侧翼序列,对目的基因的正常转录、表达及调控有重要影响,且对研究转基因作物的稳定遗传性以及转基因作物的安全生产具有重要意义,因此,侧翼序列的分析成为植物转基因育种安全评估的重要研究手段,受到广大科学研究者的重视[1, 2]。此外,侧翼序列也包括基因的启动子和调控位点,因此对侧翼序列的分析对基因的表达调控研究也有重要作用。目前已经建立了许多关于侧翼序列的分析方法,根据其技术原理,可将这些方法归为3类: 反向PCR、外源接头介导PCR、半随机引物PCR。
1 反向PCR(inverse or inverted PCR,I-P-CR)反向PCR方法的重要功能是扩增目标序列的侧翼序列。在进行PCR扩增前,先用合适的限制性内切酶消化基因组,然后让酶切后的片段环化,最后通过扩增引物扩增获得上、下游片段。现已制备了酵母人工染色体(YAC)大的线状DNA片段的杂交探针,这对于转座子插入序列的确定和基因库染色体上DNA片段序列的识别十分重要。该方法的关键之处在于选择一种合适的酶,且这种选择不能在目的位点切断靶DNA[4],其原理见图 1。
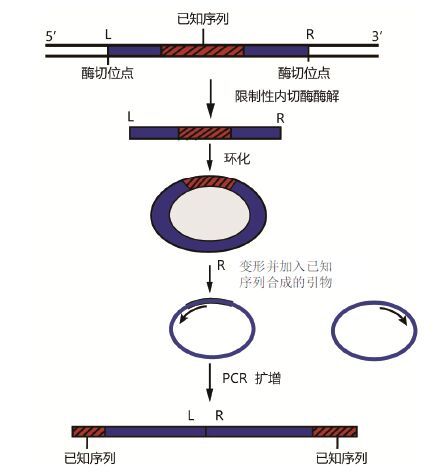
|
| 图 1 反向PCR原理示意图 |
I-PCR在分子生物学研究中应用广泛,可以用于鉴定外源基因或转座子在基因组中的整合位点,或者用于克隆基因的邻接序列[5]。下面对I-PCR的应用实例进行简单介绍;首先,I-PCR在基因启动子克隆的应用,Digeon等[6]用该方法成功克隆了小麦puroindoline基因启动子约1 000 bp片段;Van和Chen等[7, 8]基于I-PCR技术克隆了小麦花粉特异性基因TaPSG719启动子序列;2006年,冯丽等[9]在I-PCR的基础上通过技术优化,建立互补末端连接反向PCR(CELI-PCR)技术,该技术能够用于快速分离目的基因全长cDNA和启动子序列;Forester等[10]用I-PCR技术分离出豌豆种子脂肪加氧酶基因的启动子区,约800 bp片段;Heel等[7]扩增了人类ITGB7基因启动子;Tsaftaris等[11]成功克隆出了番红花CsatAP1/FUL启动子。
除了克隆启动子区外,I-PCR在分析基因旁侧序列上也得到广泛应用。研究表明,甲酰胺、二甲基亚砜(DMSO)等能够在不影响Taq酶活力的情况下降低非特异性扩增。1998年,李竹红等[12]依据以上原理改进反向PCR方法,通过优化的I-PCR技术克隆获得Tc1转座子左侧反转重复序列的旁侧序列。2001年,Yu[13]在疱疹病毒中通过IPCR技术获得GTFP基因旁侧序列。同年,韩志勇[14]通过反向PCR建立了分离水稻外源基因旁侧序列技术体系,该技术在套式PCR中结合了热启动PCR和降落PCR技术,成功克隆了35个转基因水稻品系的外源基因侧翼序列,得到的侧翼序列长度在300-750 bp,并且通过用Southern杂交对实验结果进行了后期验证。洪宇植[5]在0.5 μg DNA/mL的反应体系中,用DNA酶消化基因组并使酶解片段充分环化连接,最后用特异性引物进行长距离PCR使得酶解片段充分自身环化连接,其产物用25-30 nt的序列特异引物进行长距离PCR,从而获得漆酶LacA基因上下游9 kb的序列信息。2010年,俄广鑫等[15]利用优化的I-PCR技术,参考限制性内切酶EaeI Kit的具体实验方法,对cDNA进行酶切消化,通过T4连接酶连接环化,获得环状DNA分子,最后通过PCR扩增得到猪部分猪精氨酸-丝氨酸蛋白激酶1(SRPK1)的5′ UTR序列。2012年,张晓等[16]为获取atpA基因的侧翼序列,对I-PCR技术在2个方面进行了优化,分别是将DNA片段自连时间延长到24 h、将PCR的起始模板量增加到500 ng,以保证目标产物高的扩增效率。Zimmermann[17]获得了Bt11玉米的左右旁侧序列。但是,该方法仍然存在许多不足之处: (1)需要从多种限制性内切酶中选择合适的限制酶,只有这样才能获得大小合适的DNA片段,因此,由于酶切位点的限制,导致部分靶标DNA的扩增不能使用该方法。(2)大多数有核基因组含有大量中度和高度重复序列,而在YAC或Cosmid中的未知功能序列中有时也会有这些序列,因而通过反向PCR得到的探针就有可能与多个基因序列杂交。
2 外源接头介导PCR该技术是将已知的序列通过连接酶的作用与待分离侧翼区域连接起来,且已知序列位于限制性片段外侧,然后根据未知序列两旁的序列设计引物,从而进行PCR扩增获得未知序列。根据连接到未知序列的接头不同,将外源接头介导PCR分为单链接头、双链接头、载体接头3类[4]。
2.1 单链接头PCR单链接头PCR,又称panhandle PCR(P-PCR)。1997年,Jones[18]发明了该方法,其基本原理: 首先根据物种选择合适的限制性内切酶消化基因组DNA,使酶切下的片段产生5′端黏性末端;在5′端黏性末端上接上一条单链接头,单链接头有以下两个特点: 一是接头的5′端和黏性末端互补,二是单链接头上,除了酶切序列以外,其余序列必须和已知序列中的一部分序列反向互补;由于单链接头的末端是一个反向重复序列,故用该方法分离未知序列只需要一条引物。由于该方法的复杂性以及较低的特异性,导致该方法的应用较少。
Chen[19]通过对P-PCR改良,成功从线虫中分离得到FOG-3基因启动子序列;1997年,Jones对该方法进行了优化,在PCR复性前,在3′末端加上ddCTP,利用改进的 P-PCR方法从人类基因组DNA 扩增得到4-9 kb的片段。P-PCR在分离基因旁侧序列研究中具有广泛的应用价值。1999年,黄君健等[20]应用该技术扩增出端粒催化亚基hTERT基因5′端上游2 090 bp的旁侧序列,Fan等[21]利用该技术克隆出漆酶基因lac48424-1启动子序列;Shang等[22]成功克隆出灵芝羊毛固醇合酶(LS)基因启动子序列。2014年,采用单接头连接PCR 法顺利地分离到了甘蔗SPSⅢ基因5′侧翼序列。序列提交GenBank,查询登录号KC422670,为公共数据库中首次提交的甘蔗SPSⅢ基因5′侧翼序列[23]。
2.2 双链接头PCR双链接头PCR,基本原理是先用限制性内切酶消化基因组DNA,让基因组片段能产生黏性末端,将末端配对的接头和限制性片段连接,以其作模板,用接头引物和已知序列特异引物进行PCR扩增,即可获得包含侧翼序列的目标片段[24]。双链接头PCR 在克隆基因转录起始位点或启动子方面得到广泛应用。
1994年,张国裕[25]用Sma I和EcoR V酶切的总DNA及双链接头介导PCR的染色体步行扩增出大小分别约为4.5 kb和900 bp的条带,选择较长的片段克隆测序,测序结果表明该技术成功克隆了青花菜脱氢抗坏血酸还原酶DHAR(dehydroascorbate reductase)上游的启动子序列。2002年,北京大学蛋白质工程及植物基因工程国家重点实验室选用Bcl I消化油菜基因组,用双链接头PCR成功克隆出一条专一条带条,长约1.6 kb,经测序表明该条带与油菜质膜水孔蛋白BnPIPI基因启动子区完全相同(54 bp)[26]。2006年,张国裕等通过该技术克隆了青花菜雄性不育相关基因BoDHAR上游序列并获得了DHAR基因cDNA及基因全长序列,且研究者表明,该技术的关键在于接头的防自身扩增的设计以及通过两步高退火温度的PCR扩增来减少非特异性扩增[25]。2009年,钟秋月[27]利用改良的双链介导接头PCR技术,通过两轮极高退火温度的PCR扩增,获得番茄GGPS基因上游侧翼序列。
2.3 载体接头PCR载体接头PCR,是以基因组DNA的特异序列为引物和载体的通用引物扩增目的片段[28]。王新国等[29]通过载体接头的技术克隆了胡萝卜II型转化酶基因启动子。小鼠生长释放激素基因5′端侧翼序列和矮牵P450基因的5′端侧翼序列也通过该方法获得[30, 31]。
3种外源接头介导PCR虽然在分离基因启动子与侧翼序列等研究上得到广泛应用,但该方法仍然存在一些明显的缺点: (1)由于DNA缺口是被直接检测,故需要合适酶或化学方法对基因组进行消化处理;(2)因为必须对PCR扩增后的产物进行连接,因此需要酸化DNA分子的5′端,使其具有可连接性;(3)参与平端连接只能在引物延伸进行到模板链的末端之后,因此控制好PCR的延伸时间十分重要。
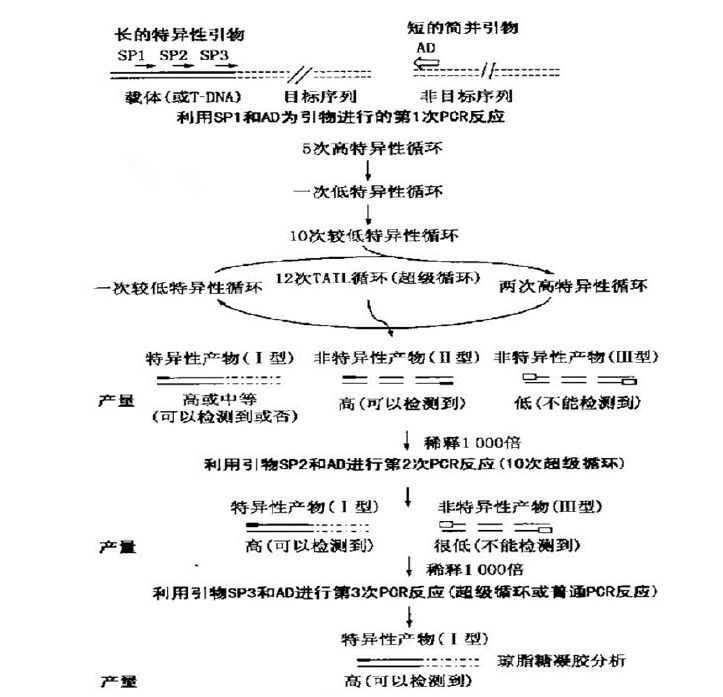
3 半随机引物PCR 3.1 热不对称交错PCR法(TAIL-PCR)热不对称交错PCR法是一种依据已知基因序列来得到未知序列的PCR扩增技术,其原理为: 通过高简并性引物结合目标DNA;利用巢式PCR增强特异性、高、低温超级PCR提高特异性产物的扩增效率,减少非特异性扩增;PCR产物作为下一轮模板,需进行一定程度的稀释,以降低非特异性模板的比例;将第2轮和第3轮PCR产物通过电泳分析来判断获得的条带是否为目标带。该技术由Liu和Whitter[32]于1995年首先研究并报道,TAIL-PCR利用已知序列设计嵌套式特异性,且特异性引物的退火温度均大于65℃,而兼并引物含有15-16个碱基,退火温度在45℃左右,其兼并性在65-256倍之间,因此特异与非特异扩增产物的相对效率能通过温度很好的控制。该技术的具体原理见图 2,具体的扩增方法见Liu等[32]的方法。
目前,应用该方法克隆了许多启动子片段,通常能克隆约0.2-2 kb的旁侧片段。因此,该方法在克隆基因启动子的应用中表现出简单、快速、高效等特点。如陈军营[34]利用改良的TAIL-PCR通过4轮PCR反应,成功克隆到小麦PMH+-ATPase上游363 bp的旁侧序列,通过生物信息学分析发现,该序列包含该基因的启动子区域;财音青格乐[35]通过3轮PCR扩增,分离得到300 bp的大豆β伴球蛋白α-亚基基因5′侧翼序列,其中含有其启动子片段;韩兆雪等[36]成功克隆了青稞2个B组醇溶蛋白基因Hordein基因的上游分别为395 bp、396 bp的调控序列、嗜碱芽孢杆菌PB92碱性蛋白酶基因aprE的619 bp的启动子序列被成功克隆等[37];邵彦春等[38]采用TAIL-PCR法,以红曲霉色素突变株基因组为模板,获得7个显著片段,测序分析表明有一个片段为T-DNA插入位点侧翼序列,番薯的pal和 pgi基因的5′侧翼序列和银杏查尔酮合成酶基因全序列也是通过该方法克隆得到的[39, 40]。
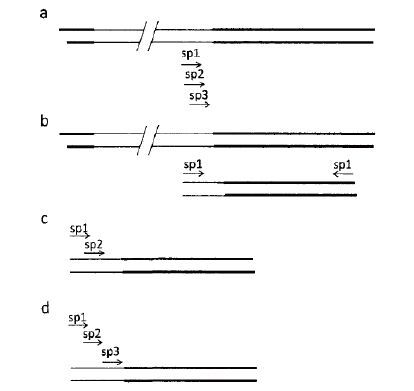
3.2 高效热不对称PCR(hi-TAIL-PCR)Liu等[41]结合TAIL-PCR以及抑制PCR的原理,提出的新的侧翼序列分析方法,该方法在兼并引物的结构,长短,兼并性上做了优化,且运用了抑制PCR的原理,使得该方法比TAIL-PCR扩增得到的片段更长,更特异。与TAIL-PCR 相比,hi-TAIL-PCR用的具有更高简并性(2304)的简并引物(图 3),hi-TAIL-PCR由3轮PCR组成,第1轮由11次高特异性(Tm=65℃)扩增、一次极低退火温度(25℃)的低特异性扩增、12次较高特异性(Tm=58℃)扩增;第2轮由2次较高特异性(Tm=65℃)扩增、2次高特异性(Tm=68℃)扩增和1次较低特异性(Tm=68℃)扩增构成的14个超级循环;第3轮由2次高特异性(Tm=68℃)扩增和1次较低特异性(Tm=68℃)扩增构成的6-7个超级循环。如Terauchi和Kahl[42]从甘薯中克隆获得苯丙氨酸氨裂解酶基因(Pal)启动子;芸苔 BcMF21 基因ATG上游约779 bp的启动子都通过该技术克隆获得[43-45]。侯娜等[46]采用hi-TAIL-PCR的方法,获得了抗虫棉06N-119的外源DNA插入位点的侧翼序列。2013年,梁丽霞等[47]通过Hi-TAIL-PCR分离了转SCK基因抗虫水稻科丰2号的插入载体插入位点的旁侧序列,并建立事件特异性检测方法。

|
| 图 3 Hi-TAIL-PCR简并引物示意图 |
由于基因组的复杂性,很多侧翼序列扩增技术的应用受到很大程度的限制。1993年Sarkar[48]发明了RS-PCR,该方法中运用到的酶切位点分别是Taq I、EcoR I、Sau3A和BamH I。这些酶切位点具有以下特点: 在生物基因组中分布具有随机性;出现的频率较高;不带重复序列。Upcroft等[49]利用RS-PCR技术,通过SacⅠ酶切消化鞭毛虫基因组,并用PCR方法成功扩增出抗药性相关基因5′侧翼序列片段。在这一成功案例的启示下,Ji等[50]设计了一种无需酶切连接反应的染色体步移法,称为“Restriction Site Extension PCR(RSE-PCR)”。
3.4 单寡核苷酸巢式PCR(single oligonucleotide nested PCR,SON-PCR)SON-PCR由TAIL-PCR改进而来,用嵌套式特异引物代替其兼并引物,且用特殊的PCR循环进行扩增。具体实验设计方法,见图 4。
Antal等[51]利用SON-PCR分离了两基因(palH和pacC)的旁侧序列。2006年,Guo等[52]研究者利用SON-PCR扩增了2个光合多样性基因位点。
4 其他方法 4.1 质粒拯救法(plasmid rescue)Plasmid rescue技术的原理如下: 首先选择合适的限制性内切酶消化基因组DNA,用连接酶连接环化消化产物,通过转化方法将环化产物转入大肠杆菌,通过菌落PCR等方法鉴定阳性菌株,经测序分析获得侧翼序列。该方法应用范围窄,筛选繁琐,工作量大,只能作为染色体步移的补充工具。质粒拯救法是一种经典的分子生物学方法,目前该方法主要用于转座子或自杀质粒插入位点周围序列的鉴定。细菌基因组大片段的克隆是细菌功能研究的一个难点,王玉飞[54]通过质粒拯救法,将待克隆的基因一侧序列连接到自杀载体上,构建出打靶质粒并将其整合到原细菌基因组上,通过酶切、自连及转化后将自杀载体与需要克隆的目标大片段一起拯救出来。质粒拯救法除了在克隆细菌大片段上用得较为广泛外,其在克隆已知基因的插入位点及其相关基因上大量应用。鲍晓明[55]将转座标签导入酵母,建立插入突变株库,通过质粒拯救法,在非生物胁迫的下筛选出抗逆相关基因。袁亮[56]通过TAIL-PCR与质粒拯救PCR法再8个水稻突变体中快速分离水稻条斑病菌(Xanthomonas oryzae pv. oryzicola)Tn5致病相关基因,谭晓红等[57]通过该技术获得了来自诱捕载体整合位点附近的新基因。
4.2 PCR-walking法PCR-walking法是利用DNA探针作为“诱饵”,依次钓出与探针有重叠区域的DNA片段,从而获得目的基因的旁侧序列,只将PCR与探针结合就提高了该方法的特异性,且免去建库的繁琐步骤,操作更加简明[58]。Sibert等[59]利用该技术克隆了tPA基因上游分别为1.8、4.5和0.9 kb的片段。
4.3 Sequence-base分离法Sequence-base分离法通常通过PCR扩增获得T-DNA与被研究基因之间的连接区域。该方法的关键在于引物,一端引物根据插入T-DNA的末端序列设计,该引物3′端向外,另一条扩增引物根据目标基因设计。拟蓝芥actin基因的研究就用到了此方法。
4.4 连接介导PCR(Ligation-mediated PCR,LMPCR)连接介导PCR法首先使用小的寡核苷酸接头连接通过限制性内切酶消化的靶DNA片段,然后选择与接头结合的PCR引物来扩增未知的靶片段。该方法在扩增单侧序列上应用较多,此外,该方法在分离启动子和基因侧翼序列也有应用。
5 小结综上所述,基因侧翼序列的分析方法很多,但均有其优势与不足(表 1)。它们在分子生物学的研究中起重要的作用,并且随着人们对生物体乃至基因的了解不断深入,对基因的调控序列、侧翼序列、启动子序列的研究将会越来越多,这些用于克隆侧翼未知序列的技术将会有更广阔的应用空间。目前对克隆侧翼序列方法的研究从未停止过,因此,随着分子生物物技术的不断进步,更多简单、高效的旁侧序列克隆方法一定会不断涌现。
| [1] | 祁洋, 李燕, 王永智, 等. 扩增 T-DNA插入位点侧翼序列的方法及其应用进展. 安徽农业科学 , 2009, 37 (17) : 7907–7908. |
| [2] | 颜静宛, 林琳, 王锋. 获得基因侧翼序列位点信息的几种扩增方法. 福建农业学报 , 2005, 20 (增刊) : 125–129. |
| [3] | 洪登峰, 万丽丽, 杨光圣. 侧翼序列克隆方法评价. 分子植物育种 , 2006, 4 (2) : 280–288. |
| [4] | 康丹, 等. 染色体步移技术克隆已知序列侧翼启动子的研究进展. 农 业生物技术学报 , 2013, 21 (3) : 355–366. |
| [5] | 洪宇植, 肖亚中, 房伟, 等. 长距离反向 PCR技术高效扩增已知 DNA片断的侧翼序列. 激光生物学报 , 2006, 1 : 46–49. |
| [6] | Digeon JF, Guiderdoni E, Alary R, et al. Cloning of a wheat puroindoline gene promoter by IPCR and analysis of promoter regions required for tissue-specific expression in transgenic rice seedsR. Plant Molecular Biology , 1999, 39 (6) : 1101–1112. DOI:10.1023/A:1006194326804 |
| [7] | van Heel DA, Allen M, Jewell DP, Carey AH. A revised sequence of the human β7 integrin gene(ITGB7)promoter region obtained by inverse PCR. Immunogenetics , 2000, 51 (10) : 863–865. DOI:10.1007/s002510000218 |
| [8] | Chen L, Tu Z, Hussain J, et al. Isolation and heterologous transformation analysis of a pollen-specific promoter from wheat(Triticum aestivum L. )[. Mol Biol Rep , 2010, 2 : 737–744. |
| [9] | 冯丽, 任茂智, 罗洪发, 等. 分离植物目的基因全长cDNA 和启动子的新方法-快速定位转录起始位点(RITIS). 分子植物育种 , 2006, 1 (4) : 23–28. |
| [10] | Forester C, Arthur E, Crespi S, et al. Isolation of a pea(Pisum sativum)seed lipoxygenase promoter by inverse polymerase chain reaction and characterization of its expression in transgenic tobacco. Plant Mol Biol , 1994, 26 (1) : 235–248. DOI:10.1007/BF00039535 |
| [11] | Tsaftaris A, Pasentzis K, Argiriou A. Rolling circle amplification of genomic templates for inverse PCR(RCA-GIP): A method for 5’- and 3’-genome walking without anchoring. Biotechnology Letters , 2010, 32 (1) : 157–161. DOI:10.1007/s10529-009-0128-9 |
| [12] | 李竹红, 等. 改进的反向 PCR 技术克隆转移基因的旁侧序列. 生物化学与生物物理进展 , 1996, 26 (6) : 600–603. |
| [13] | Yu QG, Hu NJ, Lu YN, et al. Yanagihara. rapid acquisition of entire DNA polymerase gene of a novel herpes virus from green turtle fibropap illoma by a genomic walking technique. J Virol Methods , 2001, 91 (2) : 183–195. DOI:10.1016/S0166-0934(00)00267-6 |
| [14] | 韩志勇, 王新其, 沈革志, 等. 反向 PCR 克隆转基因水稻的外源基因旁侧序列. 上海农业学报 , 2001, 17 (2) : 27–32. |
| [15] | 俄广鑫, 刘娣, 张冬杰, 等. 猪精氨酸-丝氨酸蛋白激酶1(SRPK1)克隆表达及功能初步分析. 畜牧兽医学报 , 2010, 41 (11) : 1379–1386. |
| [16] | 张晓, 张锐, 孙国清, 等. 优化的反向PCR结合TAIL-PCR法克隆棉花线粒体atpA双拷贝基因及其侧翼序列. 生物工程学报 , 2012, 28 (1) : 104–115. |
| [17] | Zimmermann A, Liithy J, Pauli U. Event specific transgene detection in Btl1 corn byquantitative PCR at the integration site. LWT-Food Science and Technology , 2000, 33 (3) : 210–216. DOI:10.1006/fstl.2000.0637 |
| [18] | Jones DH, Winistorfer SC. Sequence specific generation of a DNA panhandle permits PCR amplification of unknown flanking DNA. Nucl Acid Res , 1997, 20 (3) : 595–600. |
| [19] | Chen PJ, Cho S, Jin SW, Ellis RE. Specification of germ cell fates by FOG-3 has been conserved during nematode evolution. Genetics , 2001, 158 (4) : 1513–1525. |
| [20] | 黄君健, 李杰之, 林坚, 等. 人端粒酶催化亚基 hTERT 基因启动子的克隆. 生物技术通讯 , 1999, 10 (3) : 36–39. |
| [21] | Fan F, Zhuo R, Sun S, et al. Cloning and functional analysis of a new laccase gene from Trametes sp. 48424 which had the high yield of laccase and strong ability for decolorizing different dyes. Bioresource Technology , 2011, 102 (3) : 3126–3137. DOI:10.1016/j.biortech.2010.10.079 |
| [22] | Shang CH, Shi L, Ren A, et al. Molecular cloning, characterization, and differential expression of a lanosterol synthase gene from Ganoderma lucidum. Bioscience, Biotechnology, and Biochemistry , 2010, 74 (5) : 974–978. DOI:10.1271/bbb.90833 |
| [23] | 周平, 何炜, 金光, 等. 甘蔗SPSⅢ基因组DNA及5'侧翼序列的克隆与分析. 农业生物技术学报 , 2014, 22 (1) : 1–9. |
| [24] | Rosenthal A, Jones DSC. Genomic walking and sequencing by oligo-cassette mediated polymerase chain reaction. Nucleic Acids Res , 1990, 18 (10) : 3095–3096. DOI:10.1093/nar/18.10.3095 |
| [25] | 张国裕, 等. 青花菜雄性不育相关基因BoDHAR的克隆与表达分析. 生物工程学报 , 2006, 22 (5) : 752–756. |
| [26] | 于秋菊, 杜丽, 胡鸯雷, 等. 油菜质膜水孔蛋白基因BnPIPI启动子区的克隆及初步的功能分析. 中国科学(C辑) , 2002, 6 (32) : 521–525. |
| [27] | 钟秋月, 国艳梅, 梁燕. 两个不同来源的番茄GGPS基因克隆和序列分析. 华北农学报 , 2009, 24 (3) : 15–22. |
| [28] | Shyamala V, Ames GFL. Genome walking by single-specific-primer polymerase chain reaction: SSP-PCR. Gene , 1989, 84 (1) : 1–8. DOI:10.1016/0378-1119(89)90132-7 |
| [29] | 王新国, 张国华. 用衔接头 PCR 克隆新的胡萝卜Ⅱ型转化酶基因启动子. 中国生物学与分子生物学报 , 2001, 1 : 61–65. |
| [30] | Mizobuchi M, Frohman L. Rapid amplification of genomic DNA ends. Biotechniques , 1993, 15 (2) : 214–221. |
| [31] | Shimada Y, Ohbayashi M, Nakano-Shimada R, et al. A novel metho clone P450s with modified single-specific-primer PCR. Plant Molecular Biology Reporter , 1999, 17 (4) : 355–361. DOI:10.1023/A:1007681726174 |
| [32] | Liu YG, Whittier RF. Thermal asymmetric interlaced PCR: Automatable amplification and sequencing of insert end fragments from P1 and YAC clones for chromosome walking. Genomics , 1995, 25 (3) : 674–681. DOI:10.1016/0888-7543(95)80010-J |
| [33] | 罗丽娟, 施季森. 一种DNA侧翼序列分离技术—TAIL-PCR. 南京林业大学学报 , 2003, 27 (4) : 87–90. |
| [34] | 陈军营, 沈向磊, 辛玉茹, 等. 改良TAIL-PCR技术在小麦PM H+-ATP-Pase基因启动子克隆中的应用. 河南农业大学学报 , 2008, 42 (1) : 1–5. |
| [35] | 财音青格乐, 李明春, 蔡易, 等. 大豆种子特异性启动子的克隆及序列分析. 作物学报 , 2005, 31 (1) : 11–17. |
| [36] | 韩兆雪, 吴芳, 赵桃, 等. 青稞B组醇溶蛋白基因5'上游调控区的TAIL-PCR克隆及序列分析. 麦类作物学报 , 2007, 27 (4) : 613–618. |
| [37] | 陈坤, 黎明, 成堃, 等. 嗜碱芽孢杆菌PB92碱性蛋白酶基因启动子的克隆及应用. 遗传 , 2008, 30 (11) : 1513–1520. |
| [38] | 邵彦春, 丁月娣, 陈福生, 等. TAIL-PCR法快速分离红曲霉色素突变株T-DNA插入位点侧翼序列. 微生物学通报 , 2007, 34 (2) : 323–326. |
| [39] | Teruchi R, Kahl G. Rapid isolation of promoter sequences by TAIL-PCR: the 5’-flanking regions of pal and pgi genes from yams. Mol Gen Genet , 2000, 263 (3) : 554–560. DOI:10.1007/s004380051201 |
| [40] | 许锋, 程水源, 王燕, 等. TAIL-PCR方法快速克隆银杏查尔酮合成酶基因及序列分析. 果树学报 , 2007, 2 : 237–243. |
| [41] | Liu YG, Chen YL. High-efficiency thermal asym metric interlaced PCR for amplification of unknown flanking sequences. Biotechniques , 2007, 43 (5) : 649–654. DOI:10.2144/000112601 |
| [42] | Terauchi R, Kahl G. Rapid isolation of promoter sequences by TAIL-PCR: The 5’-flanking regions of Pal and Pgi genes from yams(Dioscorea). Mol Gen Genet MGG , 2000, 63 (3) : 554–560. |
| [43] | Wang P, Sun Y, Li X, et al. Rapid isolation and functional analysis of promoter sequences of the nitrate reductase gene from Chlorella ellipsoidea. J Appl Phycol , 2004, 16 (1) : 11–16. DOI:10.1023/B:JAPH.0000019048.56489.3c |
| [44] | Du HW, Zhang ZX, Li JS. Isolation and functional characterization of a waterlogging-induced promoter from maize. Plant Cell Reports , 2010, 29 (11) : 1269–1275. DOI:10.1007/s00299-010-0913-x |
| [45] | Jiang JJ, Yu XL, Miao Y, et al. Sequence characterization and expression pattern of BcMF21, a novel gene related to pollen development in Brassica campestris ssp. chinensis. Molecular Biology Reports , 2012, 39 (7) : 7319–7326. DOI:10.1007/s11033-012-1563-6 |
| [46] | 侯娜, 贺辉群, 董美, 等. 转基因抗虫棉外源DNA插入整合结构分析和转化体特异性检测方法的建立. 分子植物育种 , 2012, 10 (3) : 317–323. |
| [47] | 梁利霞. 转基因水稻检测方法研究[D]. 北京: 中国农业科学院, 2013. |
| [48] | Sarkar G, Turner RT, Bolander ME. Restriction-site PCR: A direct method of unknown sequence retrieval adjacent to a known locus by using universal primers. Genome Res , 1993, 4 : 318–322. |
| [49] | Upcroft P, Healey A. PCR priming from the restriction endonuclease site 3’extension. Nucleic Acids Research , 1993, 21 (20) : 4854. DOI:10.1093/nar/21.20.4854 |
| [50] | Ji J, Braam J. Restriction sSite extension PCR: A novel method for high-throughput characterization of tagged DNA fragments and genome walking. PLoS ONE , 2010, 5 (5) : e10577. DOI:10.1371/journal.pone.0010577 |
| [51] | Antal Z, Rascle C, Fevre M, Bruel C. Single oligonucleotide nested PCR: A rapid method for the isolation of genes and their flanking regions from expressed sequence tags. Current Genetics , 2004, 46 (4) : 240–246. DOI:10.1007/s00294-004-0524-6 |
| [52] | Guo H, Xiong J. A specific and versatile genome walking technique. Gene , 2006, 381 : 18–23. DOI:10.1016/j.gene.2006.06.002 |
| [53] | 杨琳. T-DNA在农杆菌介导转基因玉米中的整合模式[M]. 雅安: 四川农业大学, 2012. |
| [54] | 王玉飞, 陈泽良, 乔凤, 等. 质粒拯救法克隆细菌基因组大片段. 微生物学报 , 2008, 48 (4) : 532–538. |
| [55] | 鲍晓明, 等. 转座子标签及其在酿酒酵母基因功能研究中的应用. 中国生物工程杂志 , 2003, 11 : 53–55. |
| [56] | 袁亮, 李玉蓉, 张希福, 等. 热不对称PCR(TAIL-PCR)和Tn5转座子质粒拯救法快速分离水稻条斑病菌致病相关基因. 农业生物技术学报 , 2009, 17 (6) : 1089–1095. |
| [57] | 谭晓红, 程置, 毛春明, 等. 利用基因诱捕技术进行小鼠基因剔除的初步研究. 生物化学与生物物理进展 , 2004, 31 (7) : 606–610. |
| [58] | Rosenthal A, Jones DS. Gene walking and sequencing by oligo-cassette mediated polymerase chain reaction. Nucleic Acids Res , 1990, 18 (10) : 3095–3096. DOI:10.1093/nar/18.10.3095 |
| [59] | Siebert PD, Chenchik A, Kellogg DE, et al. An improved PCR method for walking in uncloned genomic DNA. Nucleic Acids Research , 1995, 23 (6) : 1087–1088. DOI:10.1093/nar/23.6.1087 |