木聚糖是一类由β-1,4-糖苷键连接木糖单元形成主链,而主链又被不同基团修饰的异质多糖[1]。木聚糖是半纤维素的主要组成成分,在植物细胞壁中的含量仅次于纤维素。由于其结构的复杂性,木聚糖的完全降解需要多种糖苷水解酶的协同作用,其中木聚糖酶首先作用于木聚糖主链将其降解为低聚木糖,随后β-木糖苷酶进一步将低聚木糖降解为木糖,α-L-阿拉伯呋喃糖苷酶负责水解其侧链的取代基[2]。β-木糖苷酶和α-L-阿拉伯糖苷酶是木聚糖降解酶系中的关键酶,近年来人们发现了一类双功能木糖苷酶/阿拉伯呋喃糖苷酶,它们可以同时降解多种底物,从而减少酶的用量,降低生产成本,而且能够与木聚糖酶协同降解木聚糖,提高还原糖的生成量,解除低聚木糖对木聚糖酶的反馈抑制作用[3]。
β-木糖苷酶和α-L-阿拉伯糖苷酶被广泛应用于食品、生物转化和造纸行业等工业[4-6],但是在实际应用中面临着一个比较突出的问题:为了减少食品中活性成分、风味物质和营养成分的损失,一些如葡萄酒发酵和果汁澄清等食品加工过程通常需要在低温条件下进行(<30℃)[7],但当前工业应用的木糖苷酶都属于中高温酶(40-60℃),它们在低温条件下(0-20℃)的活力很低甚至完全丧失[8, 9],如果不解决木糖苷酶在低温条件下活力低的问题,将严重限制其在食品加工及其他需要低温处理工业中的应用。
具有嗜冷特性的低温酶为解决这一问题提供了一个新思路,低温酶的最适温度一般在30℃以下,在低温条件下仍具有较高的催化活性;而且低温酶在中高温条件下热稳定性差,只用温和的热处理就可终止催化反应,从而减少加热和冷却过程,降低生产成本[10]。但是目前有基因序列和酶学性质研究报道的低温木糖苷酶极少,所以亟需发掘具有低温特性的木糖苷酶和阿拉伯呋喃糖苷酶。本研究从大兴安岭多年冻土中的枝顶孢属菌Acremonium sp. WCQ6A中克隆得到一个新的GH43家族低温木糖苷酶基因AX543,对该基因在毕赤酵母中进行表达,并对其酶学性质进行系统的研究,以期开发具有低温应用食品工业及其他需要低温处理的工业潜力的酶。
1 材料与方法 1.1 材料 1.1.1 菌株和质粒枝顶孢菌Acremonium sp. WCQ-6A是本实验室前期从大兴安岭多年冻土分离得到,已保藏于中国普通微生物菌种保藏中心(CGMCC),菌种保藏号为CGMCC11804。大肠杆菌(Escherichia coli)DH5α、Pichiapastoris GS115和pPIC9质粒由本实验室保存;pMD19-T购自宝生物工程(大连)股份有限公司。
1.1.2 试剂LA Taq购自TaKaRa公司;限制性内切酶EcoRⅠ、NotⅠ和PmeⅠ购于Fermentas公司;异丙基硫代-β-D-半乳糖苷(IPTG)、质粒小提试剂盒和DNA纯化试剂盒购自北京索莱宝公司;BCA蛋白定量试剂盒购自Thermo Scientific公司。底物对硝基苯基-α-L-阿拉伯呋喃糖苷(pNPA)、对硝基苯基-β-D-吡喃木糖苷(pNPX)、榉木木聚糖、桦木木聚糖和小麦阿拉伯木聚糖均购自Sigma公司;其他化学试剂购于天津市江天化工技术有限公司,均为分析纯。
1.1.3 培养基YPD培养基:1%酵母提取物、2%蛋白胨、2%葡萄糖;BMGY培养基:1%酵母浸出物、2%蛋白胨、1%甘油;BMMY培养基:1%酵母浸出物、2%蛋白胨;NB培养基:0.3%酵母浸出物、0.5%蛋白胨、0.6%葡萄糖、1% NaCl。
1.2 方法 1.2.1 基因组DNA的提取将枝顶孢菌WCQ6A在NB培养基中30℃条件下培养48 h后收集菌体,立即加入液氮,反复研磨破碎菌体,并用CTAB-SDS法提取基因组DNA[11]。
1.2.2 双功能酶AX543基因序列的克隆与分析以枝顶孢菌WCQ6A基因组DNA为模板,根据GH43家族木糖苷酶基因的保守区序列设计简并引物XylDF和XylDR(表 1),利用Touchdown PCR克隆AX543保守区序列[12],扩增程序为:94℃ 5 min;94℃ 30 s,65-55℃ 45 s,72℃ 1 min(每个循环降0.5℃);94℃ 30 s,55℃ 45 s,72℃ 1 min,共30个循环;72℃ 10 min。将扩增得到的大小约600 bp的基因片段纯化后,连接pMD19-T载体,进行测序。根据测序正确的保守区序列分别设计上游和下游的特异性嵌套引物,通过Tail-PCR获取侧翼基因序列[11],通过Vector NTI 7.0(Invitrogen)拼接获取全长,通过BlastX和BlastP(http://blast.ncbi.nlm.nih.gov/Blast.cgi)比对分析基因序列。
利用SignalP 4.0软件分析其信号肽切割位点(http://www.cbs.dtu.dk/services/SignalP/);利用NetNGlyc 1.0 sever和NetOGlyc 4.0 sever软件预测蛋白的N-糖基化位点和O-糖基化位点(http://www.cbs.dtu.dk/services/NetNGlyc/;http://www.cbs.dtu.dk/services/NetOGlyc/);利用FGENESH 软件预测内含子信息(http://linux1.softberry.com/);利用ClustalW软件进行多序列比对分析(http://www.ebi.ac.uk/clustalW);利用Vector NTI7.0软件预测蛋白分子量和等电点。
表达载体的构建:以Acremonium sp. WCQ6A基因组DNA为模板(基因无内含子),根据AX543基因序列设计带有EcoRⅠ和NotⅠ酶切位点的扩增引物AX543F和AX543R(表 1中下划线部分),为了方便后续纯化,在蛋白C-端引入His标签,PCR反应条件:94℃预变性5 min;94℃ 30 s,52℃ 30 s,72℃ 1 min 10 s,30个循环;72℃ 10 min。将双酶切后的PCR产物与pPIC9载体连接,构建重组表达质粒pPIC9-ax543,转化大肠杆菌,测序正确后备用。
重组菌株pPIC9-ax543-GS115的获得和高酶活菌株的筛选:提取表达质粒用限制性内切酶PmeⅠ线性化后电转入毕赤酵母GS115感受态细胞中,涂布于MD平板,30℃培养2-3 d;待长出His+菌落后,挑取单克隆到3 mL BMGY培养基中,30℃培养48 h后5 000 r/min离心5 min去上清,加入1 mL BMMY培养基(0.5% V/V甲醇)进行诱导;诱导结束后,离心获得上清,测定木糖苷酶酶活。将筛选获得的酶活最高的转化子进行摇瓶发酵培养,先在400 mL BMGY中培养36 h后,12 000 r/min离心5 min去上清,然后用200 mL BMMY培养基进行甲醇诱导(甲醇初始浓度0.5 mL/100 mL培养基),每24 h补加0.5 mL/100 mL培养基的甲醇进行持续诱导,96 h后离心收集上清,用于后续蛋白纯化和酶学性质测定。
重组蛋白的纯化:重组蛋白AX543用镍亲和层析进行纯化,用含不同浓度梯度咪唑(10-300 mmol/L)的缓冲液(20 mmol/L Tris,50 mmol/L NaCl,pH8.0)洗脱目的蛋白,收集不同浓度梯度的洗脱液进行SDS-PAGE蛋白电泳检测纯度,并通过BCA蛋白定量试剂盒进行蛋白浓度的测定。
1.2.4 重组双功能酶酶学性质分析 1.2.4.1 酶的最适温度、热稳定性、最适反应pH值和pH稳定性测定(1)最适温度测定:在pH6.0的条件下,测定纯化后的酶在不同温度下(0℃-50℃)的酶活。(2)热稳定性测定:将重组酶分别在40℃、50℃和60℃处理60 min,并在不同时间点取样(0、1、2、5、10、20、30和60 min),在25℃和pH6.0条件下测定剩余酶活。(3)最适pH测定:测定纯化后的酶在pH3.0-11.0条件下的酶活,缓冲液如下:0.2 mol/L磷酸氢二钠-柠檬酸pH3.0-8.0;0.2 mol/L甘氨酸-氢氧化钠缓冲液,pH8.0-11.0,25℃反应10 min。(4)pH稳定性测定:将纯化后的酶先用不同pH的缓冲液稀释,37℃处理1 h,再用最适pH的缓冲液再进行一定稀释,在25℃和pH6.0条件下测定剩余酶活。每个实验重复测定3次。
1.2.4.2 金属离子及化学试剂对酶活性的影响在反应体系中加入终浓度为5 mmol/L的不同金属离子及化学试剂,在25℃和pH6.0的条件下测定酶活性[13]。以只加入对应的金属离子、化学试剂而不加入酶液作为对照。每个实验重复测定3次。
1.2.4.3 底物特异性分别以1%榉木木聚糖、桦木木聚糖、小麦阿拉伯木聚糖为底物,在25℃,pH6.0的条件下测定酶活性。
1.2.4.4 比活力和动力学参数的测定1个酶活性单位(U)定义为在最适反应条件下,每分钟分解底物pNPX或者pNPA生成1 μmoL对硝基酚(pNP)所需的酶量。在pH6.0,25℃条件下测得重组酶AX543的木糖苷酶/阿拉伯呋喃糖苷酶活性,同时采用BCA法测定纯化后AX543的蛋白浓度,每个实验重复测定3次,最终计算出每毫克蛋白所含的酶活力单位,即酶的比活力。配制不同浓度的pNPX和pNPA(0.4-2 mmol/L),在25℃和pH6.0条件下测定酶活性,利用米氏方程双倒数法(Lineweaver-Burk)作图并绘制曲线,求出Km值和Vmax。
1.2.4.5 木糖耐受性和阿拉伯糖耐受性的测定配制2 mmol/L和3 mmol/L的底物pNPX和pNPA,向反应体系中加入0-400 mmol/L不同浓度的木糖/阿拉伯糖,在最适反应条件25℃和pH6.0测定酶活并分别计算其Ki值。
1.2.5 薄层色谱(TLC)水解产物分析在100 μL的1%(W/V)榉木木聚糖中加入6 U纯化后的酶液,25℃、pH6.0条件下分别反应0.5、1、2、4、8和12 h;在50 μL木二糖和木三糖(5 mg/mL)底物中加入2 U纯化后酶液在25℃、pH6.0条件下反应1 h;将上述反应体系于沸水中煮沸5 min后,12 000 r/min离心5 min,取上清点样于硅胶板G-254,在展开剂[正丁醇∶水∶乙酸(V∶V∶V)=2∶1∶1]中展开两次,展开后置于显色剂(丙酮∶二苯胺∶苯胺∶磷酸=50 mL∶1 g∶1 mL∶5 mL)中浸泡数秒后立即于90℃烘箱烘干显色。
1.2.6 双功能酶AX543与木聚糖酶Xyn11-1的协同作用在900 μL的0.5%(W/V)榉木木聚糖中加入100 μL酶液(0.5 U AX543/0.5 U Xyn11-1)。第一步反应在pH6.0、30℃条件下进行,6 h后置于100℃沸水中煮沸10 min终止反应;接着进行第二步反应(6 h),反应结束后通过DNS法测定酶活并计算协同效率[11]。协同效率的定义为协同反应产生的总还原糖与各单酶反应产生总还原糖总和的比值[13]。每个实验重复测定3次。
2 结果 2.1 双功能酶AX543基因序列的克隆与分析通过Touch-down PCR及Tail-PCR分别扩增出一条长622 bp的保守区序列,以保守区序列为模板,通过Tail-PCR扩增得到了AX543的编码基因,为975 bp,编码324个氨基酸和一个终止密码子,无糖基化位点、信号肽和内含子。预测其成熟蛋白分子量为41.8 kD,等电点为4.99。BlastP比对结果显示AX543属于GH43家族,与来源于Acremenium chrysogenum的木糖苷酶/阿拉伯呋喃糖苷酶的一致性为86%;来源菌环带状枝顶孢菌WCQ6A的ITS和基因ax543序列已提交至NCBI数据库,其检索号分别为(KU376503)和(KU353562)。结合多序列比对分析和已报道的GH43家族木糖苷酶的晶体结构分析[14],找到了AX543存在有3个保守的催化位点(图 1),Asp12和Glu225为酸碱催化残基,Asp131参与调节催化氨基酸的pKa和底物与催化氨基酸的定位。

|
| 图 1 AX543多序列比对 其他来源木糖苷酶氨基酸序列包括Penicillium herquei(AB093564),Aspergillus oryzae(BAE55732),Humicola insolens(KC962400),Paecilomyces thermophila(GU937001),深灰色阴影部分代表完全保守的氨基酸位点,浅灰色阴影部分代表相对保守的氨基酸位点,*代表木糖苷酶保守催化氨基酸位点(AX543中Asp12,Asp131,Glu225) |
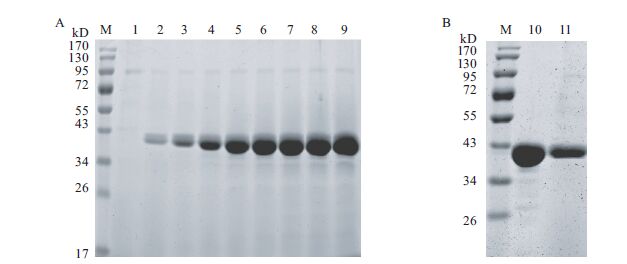
将筛选得到酶活力最高的7#转化子于1 L摇瓶发酵培养以确定最佳诱导时间,结果(图 2-A)显示在诱导96 h时AX543的酶活力达到最高;将发酵液上清用镍亲和柱纯化,其中200 mmol/L洗脱液中的酶活最高,纯化后的蛋白大小约为42 kD,与理论值相符(图 2-B)。
|
| 图 2 重组蛋白AX543的诱导表达条件优化(A)和纯化(B) M:蛋白Marker;1-9:分别为诱导0、6、12、24、48、72、96、120和144 h后的发酵液上清粗蛋白;10,11:分别为纯化后的AX543和未纯化AX543 |
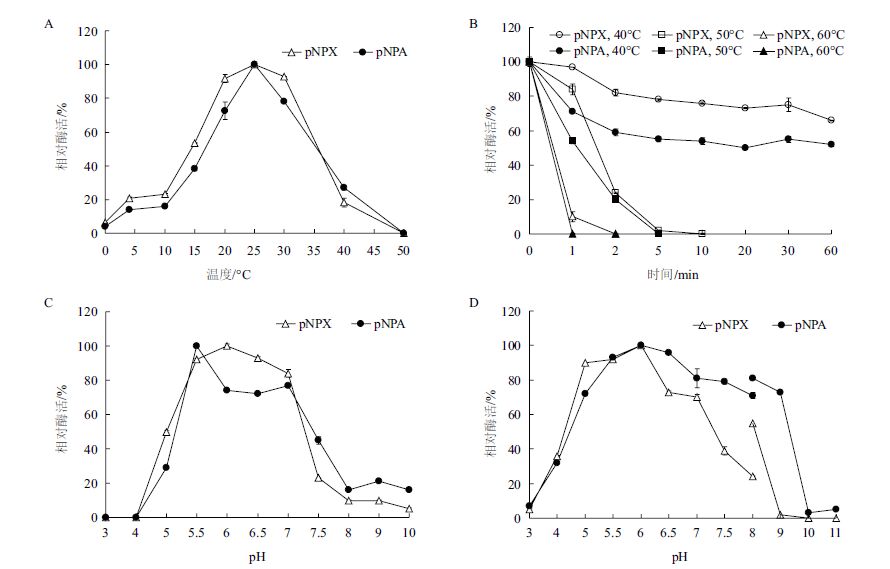
AX543的最适温度为25℃,在15℃、4℃和0℃时,以pNPX为底物时的相对酶活分别为53.61%、20.57%和6.28%,以pNPA为底物时的相对酶活较低,分别为38.44%、14.04%和4.15%(图 3-A);该酶的热稳定较差,其木糖苷酶活性/阿拉伯呋喃糖苷酶活性在40℃处理1 h后的剩余酶活为50%,但在50℃处理5 min后酶活全部丧失(图 3-B);AX543的最适pH为6.0(图 3-C),在pH5.5-7.0范围内,其木糖苷酶活性高于阿拉伯呋喃糖苷酶活性,pH稳定性结果表明,以pNPA为底物时在pH5.0-9.0范围内较稳定(图 3-D)。
|
| 图 3 温度(A和B)及pH(C和D)对AX543的影响 |
金属离子及化学试剂试验表明,Mn2+、Ca2+和β-巯基乙醇对该酶的激活作用十分显著,为114.9%-344.0%;此外,乙醇能激活其木糖苷酶活性至206.5%,但是却抑制其阿拉伯呋喃糖苷酶活性至96.1%(表 2)。木糖苷酶/阿拉伯呋喃糖苷酶AX543的比活分别为4.99 U/mg和1.16 U/mg,动力学常数Km、Vmax和Kcat分别为5.94 mmol/L、3.53 μmol/(min·mg)、2.46 s-1和14.29 mmol/L、3.23 μmol/(min·mg)、2.25 s-1;AX543对榉木木聚糖、桦木木聚糖和小麦阿拉伯木聚糖均有降解作用,在以上3种聚糖为底物时的比活分别为17.95 U/mg、10.35 U/mg和1.19 U/mg。
以不同浓度梯度的木糖和阿拉伯糖作为抑制剂,以2 mmol/L和3 mmol/L的pNPX和pNPA为底物测定AX543对木糖和阿拉伯糖的耐受性,计算得AX543的Ki值分别为84.78 mmoL/L和45.01 mmoL/L。
2.5 TLC水解产物分析TLC水解产物结果(图 4)表明,AX543处理榉木木聚糖0.5、1、2、4、8和12 h后的降解产物都是木糖,同时AX543可以将木二糖和木三糖水解生成木糖。

|
| 图 4 TLC水解产物分析 1-6:分别为AX543作用于榉木木聚糖0.5、1、2、4、8和12 h;7:木糖标准品;8:阿拉伯糖标准品;9:AX543降解木二糖产物;10:AX543降解木三糖产物;11:木二糖标准品;12:木三糖标准品 |
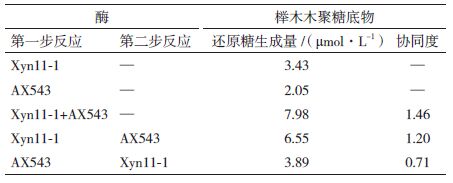
协同作用实验中,当先加入木聚糖酶Xyn11-1[15]后再加入双功能酶AX543时,还原糖的释放量比单独加入这两种酶时提高了1.2倍;同时加入Xyn11-1和AX543时的协同率最高,为1.46倍;但先加入AX543后再加入Xyn11-1则会抑制榉木木聚糖的降解(表 3)。
低温酶可以在低温下具有较高的催化活性,可以克服中高温酶在低温条件下活性低下的缺点,在食品及其它需要低温处理的工业中具有巨大的应用潜力。目前发现的大多数木糖苷酶的最适温度在40-60℃之间,具有低温特性木糖苷酶和阿拉伯呋喃糖苷酶非常少,只有P. herquei IFO4674来源的S2a、A. oryzae来源的XylB和Fusarium graminearum[16]来源的XyloA 属于低温木糖苷酶,其最适温度都是30℃。本研究从大兴安岭多年冻土来源的枝顶孢菌WCQ6A中获得了一个低温的GH43家族木糖苷酶AX543,与其他低温木糖苷酶相比,AX543的最适温度更低(25℃),更加接近于室温条件,是目前报道的最适温度最低的木糖苷酶,在低温工业应用中具有很好的潜力。
一种酶具有多种底物活性可以扩展其应用范围,但是目前发现的一些木糖苷酶只能降解一种底物,如来源于P. herquei和A. oryzae中的两个低温木糖苷酶只能水解糖苷酶pNPX;一些木糖苷酶可以降解少数几种底物,如来源于Enterobacter sp.的木糖苷酶Xyl43只可以降解pNPX和木二糖[17];来源于Phanerochaete chrysosporium的PcXyl可作用于pNPX和pNPA但不能降解木聚糖[18]。本研究的AX543具有广泛的底物特异性,能够作用于pNPX、pNPA、桦木木聚糖、榉木木聚糖、小麦阿拉伯木聚糖、木二糖和木三糖,而且降解物终产物都为木糖。
研究发现一些木糖苷酶可以与木聚糖酶一起协同作用水解木聚糖,这种特性可以大大提高木糖产量,从而降低生产成本[13]。本研究的木糖苷酶AX543可以与木聚糖酶Xyn11-1协同催化榉木木聚糖,其作用协同率高于Humicola insolens来源的Xyl43A(1.29倍),但是要低于Alicyclobacillus sp. A4来源的双功能酶Ac-Abf51A(2.92倍)。同时我们还发现木糖苷酶AX543和木聚糖酶同时加入时的协同率要高于先加木聚糖酶Xyn11-1后再加木糖苷酶AX543的协同率,这种现象与来源于Alicyclobacillus sp. A4的双功能酶Ac-Abf51A相似[19],其潜在原因可能是两种酶同时作用于木聚糖底物时,木糖苷酶可以与低聚木糖结合,减少低聚木糖对木聚糖酶的抑制作用,从而使木聚糖酶更好地发挥催化作用[20]。此外,我们还发现先加木糖苷酶AX543再加木聚糖酶Xyn11-1时会抑制木聚糖酶本身的降解能力,木糖苷酶Xyl43A与木聚糖酶协同作用时也发现了这种现象[13],其原因可能是先加入的木糖苷酶占据了木聚糖底物中的酶结合位点,从而导致后续加入的木聚糖酶不能与木聚糖底物结合,不能进一步作用于木聚糖的主链产生木糖。
木糖是木糖苷酶降解木聚糖和低聚木糖的产物,同时也是木糖苷酶的抑制剂,在实际生产过程中提高酶的木糖耐受性将会在很大程度上解除酶的抑制作用,提高酶的催化能力,节约成本。目前发现的大多数木糖苷酶的木糖耐受性能力都较差,如来源于Trichoderma reesei、Humicola grisea var. thermoidea和Aureobasidium pullulans的木糖苷酶的Ki值都在2-10 mmol/L之间[21]。AX543具有较好的木糖耐受性(Ki =84.78 mmol/L),这与来源于Humicola insolens的Xyl43A(Ki=79 mmol/L)[13]相似,低于T. thermarum来源的xynB3(Ki=1 000 mmol/L)[22];此外AX543对阿拉伯糖耐受性也很高,高于Penicillium purpurogenum来源的阿拉伯呋喃糖苷酶/木糖苷酶XYL1(Ki=2.5 mmol/L)[14]以及双功能酶deAX(Ki=18.5 mmol/L)[23]。
4 结论本研究从枝顶孢菌中得到一个新的双功能木糖苷酶/阿拉伯呋喃糖苷酶基因ax543,并在毕赤酵母中进行了异源表达和高效制备。AX543是目前报道的最适反应温度最低的双功能木糖苷/阿拉伯呋喃糖苷酶,并具有较为广泛的底物特异性,较高的木糖耐受性和阿拉伯糖耐受性,还可以与木聚糖酶协同作用高效降解木聚糖,这些催化特性使双功能木糖苷酶/阿拉伯呋喃糖苷酶AX543具有应用于食品工业及其他需要低温处理工业的潜力。
| [1] | Bastawde KB. Xylan structure, microbial xylanases, and their mode of action. World Journal of Microbiol and Biotechnol , 1992, 8 (4) : 353–368. DOI:10.1007/BF01198746 |
| [2] | Lagaert SA, Pollet A, Courtin CM, et al. β-Xylosidases and α-l-arabinofuranosidases:accessory enzymes for arabinoxylan degradation. Biotechnol Advances , 2014, 32 (2) : 316–332. DOI:10.1016/j.biotechadv.2013.11.005 |
| [3] | Till M, Goldstone D, Card G, et al. Structural analysis of the GH43 enzyme Xsa43E from Butyrivibrio proteoclasticus. Acta Crystallogr F Struct Biol Commun , 2014, 70 (9) : 1193–1198. DOI:10.1107/S2053230X14014745 |
| [4] | Juturu V, Wu JC. Microbial exo-xylanases:a mini review. Applied Biochemistry and Biotechnology , 2014, 174 (1) : 81–92. DOI:10.1007/s12010-014-1042-8 |
| [5] | Camargo D, Sene L, Variz DI, et al. Xylitol bioproduction in hemicellulosic hydrolysate obtained from sorghum forage biomass. Applied Biochemistry and Biotechnology , 2015, 175 (8) : 3628–3642. DOI:10.1007/s12010-015-1531-4 |
| [6] | Bosetto A, Justo PI, Zanardi B, et al. Research progress concerning fungal and bacterial β-Xylosidases. Applied Biochemistry and Biotechnology , 2016, 178 (4) : 766–795. DOI:10.1007/s12010-015-1908-4 |
| [7] | Sarmiento F, Peralta R, Blamey JM. Cold and hot extremozymes:industrial relevance and current trends. Frontiers in Bioengineering and Biotechnology , 2015, 3 : 148. |
| [8] | Ito T, Yokoyama E, Sato H, et al. Xylosidases associated with the cell surface of Penicillium herquei IFO 4674. Journal of Bioscience and Bioengineering , 2003, 96 (4) : 354–359. DOI:10.1016/S1389-1723(03)90136-8 |
| [9] | Suzuki S, Fukuoka M, Ookuchi H, et al. Characterization of Aspergillus oryzae glycoside hydrolase family 43 β-xylosidase expressed in Escherichia coli. Journal of Bioscience and Bioengineering , 2010, 109 (2) : 115–117. DOI:10.1016/j.jbiosc.2009.07.018 |
| [10] | Siddiqui KS. Some like it hot, some like it cold:temperature dependent biotechnological applications and improvements in extremophilic enzymes. Biotechnology Advances , 2015, 33 (8) : 1912–1922. DOI:10.1016/j.biotechadv.2015.11.001 |
| [11] | Li Z, Xue X, Zhao H, et al. A C-terminal proline-rich sequence simultaneously broadens the optimal temperature and pH ranges and improves the catalytic efficiency of glycosyl hydrolase family 10 ruminal xylanases. Applied and Environmental Microbiology , 2014, 80 (11) : 3426–3432. DOI:10.1128/AEM.00016-14 |
| [12] | Teng C, Jia H, Yan Q, et al. High-level expression of extracellular secretion of a β-xylosidase gene from Paecilomyces thermophila in Escherichia coli. Bioresource Technology , 2011, 102 (2) : 1822–1830. DOI:10.1016/j.biortech.2010.09.055 |
| [13] | Yang X, Shi P, Huang H, et al. Two xylose-tolerant GH43 bifunctional β-xylosidase/α-arabinosidases and one GH11 xylanase from Humicola insolens and their synergy in the degradation of xylan. Food Chemistry , 2014, 148 (148) : 381–387. |
| [14] | Ravanal MC, Alegria-Arcos M, Gonzalez-Nilo FD, et al. Penicillium purpurogenum produces two GH family 43 enzymes with β-xylosid-ase activity, one monofunctional and the other bifunctional:bioc-hemical and structural analyses explain the difference. Archi-ves of Biochemistry and Biophysics , 2013, 540 (1-2) : 117–124. |
| [15] | Li K, Li Z, Luo X, et al. Cloning and expression of a novel xylanase Xyn11-1 from alkaline soil[C]. Springer Berlin Heidelberg, 2015, 333:75-81. |
| [16] | Carapito R, Carapito C, Jeltsch JM, et al. Efficient hydrolysis of hemicellulose by a Fusarium graminearum xylanase blend produced at high levels in Escherichia coli. Bioresource Technology , 2009, 100 (2) : 845–850. DOI:10.1016/j.biortech.2008.07.006 |
| [17] | Campos E, Negro Alvarez MJ, Sabaris di Lorenzo G, et al. Sabaris di Lorenzo G, et al. Purification and characterization of a GH43 β-xylosidase from Enterobacter sp. identified and cloned from forest soil bacteria. Microbiological Research , 2014, 169 (2-3) : 213–220. DOI:10.1016/j.micres.2013.06.004 |
| [18] | Huy ND, Thayumanavan P, Kwon TH, et al. Characterization of a recombinant bifunctional xylosidase/arabinofuranosidase from Phanerochaete chrysosporium. Journal of Bioscience and Bioengineering , 2013, 116 (2) : 152–159. DOI:10.1016/j.jbiosc.2013.02.004 |
| [19] | Yang W, Bai Y, Yang P, et al. A novel bifunctional GH51 exo-a-L-arabinofuranosidase/endo-xylanase from Alicyclobacillus sp. A4 with significant biomass-degrading capacity. Biotechnology for Biofuels , 2015, 8 –197. |
| [20] | Teleman A, Tenkanen M, Jacobs A, et al. Characterization of O-acetyl-(4-O-methylglucurono)xylan isolated from birch and beech. Carbohydrate Research , 2002, 337 (4) : 373–377. DOI:10.1016/S0008-6215(01)00327-5 |
| [21] | Zanoelo FF, Polizeli Md Mde L, Terenzi HF, et al. Purification and biochemical properties of a thermostable xylose-tolerant β- D-xylosidase from Scytalidium thermophilum. Journal of Industrial Microbiology and Biotechnology , 2004, 31 (4) : 170–176. DOI:10.1007/s10295-004-0129-6 |
| [22] | Shi H, Li X, Gu H, et al. Biochemical properties of a novel thermostable and highly xylose-tolerant β-xylosidase/alpha-arabinosidase from Thermotoga thermarum. Biotechnology for Biofuels , 2013, 6 (1) : 1–10. DOI:10.1186/1754-6834-6-1 |
| [23] | Wagschal K, Heng C, Lee CC, et al. Biochemical characterization of a novel dual-function arabinofuranosidase/xylosidase isolated from a compost starter mixture. Applied Microbiology and Biotechnology , 2008, 81 (5) : 855–863. |