植物表皮毛是一种特化的表皮细胞,是地上部组织表皮向外突出形成的一种单细胞或多细胞结构[1, 2]。作为与外界环境接触的最外层结构,具有重要的生物学功能。表皮毛的存在能减少植株热量的散失,提高植物对冷害的抗性[3, 4];减少植株对太阳辐射的吸收,保护组织免受紫外线的伤害[5];防止水分的散失,提高植物的抗旱性等[6];吸收重金属元素,提高植物对土壤重金属污染的防御[7, 8]。另外,表皮毛还能通过建立物理屏障来抵御食植性昆虫对植物体的危害[9]。同时,表皮毛细胞发育分化的独特性使其成为研究细胞命运决定和细胞发育调控的模式系统,因此研究植物表皮毛的发育调控机制具有重要的理论意义和潜在的应用价值。
模式植物拟南芥表皮毛是一种典型的单细胞结构,无腺体,一般有3个分支,广泛分布于叶片、茎、莲座叶和萼片上[10]。目前,拟南芥中已报道的控制表皮毛发育的基因有70多个,HD-Zip转录因子GLABRA2(GL2)是最早鉴定的控制植物表皮毛发育的关键调控因子之一[11]。GL2正调控表皮毛的形成,GL2功能缺失突变体表现为表皮毛严重缺失,发育受阻,只在叶片的边缘有少量的毛状突起或者1分支、2分支的表皮毛。GL2基因编码一个HD-Zip转录因子,位于连接表皮毛形成及随后发育的咽喉位置,其表达受到上游的GL1-GL3/EGL3-TTG1转录复合体的调控,但是其下游的调控路径还不明确[11]。
本研究通过筛选以gl2-3突变体为背景建立的甲基磺酸乙酯(ethyl methane sulphonate,EMS)二次诱变库,得到了一个gl2-3的增强突变体M12-01,其表型为叶表面完全没有表皮毛。与Col-0回交获得了M12-01单突变体(M12-01 single)。遗传学分析表明,M12-01 single是单基因控制的隐性性状。通过图位克隆发现该突变基因是TTG1基因的一个新等位基因[12]。本研究旨在为研究GL2调控表皮毛发育的过程提供新的遗传材料,为进一步解释GL2调控植物表皮毛发育的分子机制奠定基础。
1 材料与方法 1.1 材料拟南芥(Arabidopsis thaliana)野生型 Columbia(Col-0),Landsberg erecta(Ler)、SALK_039825(gl2-3)纯合体、ttg1-1突变体由本实验室保存。gl2-3背景的EMS突变体库由本实验室创制保存。
1.2 方法 1.2.1 植物的种植和培养将gl2-3背景的EMS诱变库按pool种植在充分吸水后的商业营养上(pindstrup substrate peat moss)上,4℃处理3 d保证种子萌发的均一性,然后移入培养温度为22℃左右,相对湿度为70%的24 h持续光照培养间中生长。待植物长出第3、4片真叶(15 d左右)时观察叶片表皮毛发育状况,筛选能回复或者增强gl2-3表皮毛突变表型的植株作为潜在的目标突变体。
1.2.2 突变体背景的纯化和遗传分析以拟南芥Col-0植株为母本,挑选出的表皮毛明显异常的突变体为父本杂交得到F1代植株,F1代植株自交得到F2代种子。种植并观察F2代植株表型,统计F2代群体植株分离的表型和比例,挑选出Col-0背景的单突变体。Col-0与单突变体连续回交3次,纯化单突变体的遗传背景并获得BC3。在Col-0与单突变体回交的过程中,通过观察杂交F1代植株的表型判断突变基因的显隐性;通过统计F2群体中的分离比判断该突变是否为单基因突变。同时,通过正反交来确定突变体的遗传方式。
1.2.3 图位克隆将单突变体与Ler生态型拟南芥进行杂交,在杂交F2代植株中挑选具有单突变体表型的植株作为图位克隆的作图群体。通过CTAB法提取95棵突变体植物DNA,每个样品中吸取5 μL等量混合得到一个DNA池(bulk),利用已经公布的均匀覆盖拟南芥5条染色体的27对分子标记为引物,对突变基因进行初步的粗定位。确定突变位点在染色体上的连锁区间和侧翼分子标记后,继续扩大作图群体,增加分子标记密度进行精细定位,进一步缩小突变位点所在区间范围。
1.2.4 候选基因的确定根据精细定位的区间,从拟南芥基因组数据库网站(www.arabidopsis.org)上获得区间内所包含的全部基因,查看基因注释信息及相关参考文献,筛选出候选突变基因,然后针对这些候选突变基因设计引物,进行PCR扩增并测序,最终确定出突变位点。
1.2.5 ttg1-1与gl2-3突变体之间的遗传互作分析将gl2-3与ttg1-1杂交,观察F2代植株表型,判断ttg1-1是否能增强gl2-3突变体的表型。
1.2.6 实时荧光定量RT-PCR分析将不同基因型的植物种子分别种植在充分吸水的商业营养土上,置于光照培养间中培养。待植物生长10 d后取除子叶外的全部地上部组织,TRIzol法提取总RNA并进行DNAase处理,将1.5 µg 处理后的总RNA用Roche反转录试剂盒进行反转录合成cDNA。以合成的cDNA为模板,利用Bio-Rad实时荧光定量PCR试剂盒和TTG1和GL2基因特异性引物进行PCR扩增,以UBQ10基因为内参照。相对基因表达量的为2-ΔΔCt,所用的引物序列为:GL2qF1 5'-ATGAAGCTCGTCGGCATGAGTGGG-3';GL2qR1 5'-TGGATTGCCACTGAGTTGCCTCTG-3';TTG1qF1 5'-GCGATTTCCTCCGTCTTTGG-3';TTG1qR1 5'-CGCTCGTTTTGCTGTTGTTG-3';UBQ10qF1 5'-GGTTCGTACCTTTGTCCAAGCA-5';UBQ10qR1 5'-CCTTCGTTAAACCAAGCTCAGTATC-3'。
2 结果 2.1 M12-01突变体的获得和表型分析为了深入研究GL2参与拟南芥表皮毛发育的调控机制,开展了大规模的遗传筛选工作,希望通过筛选gl2-3的修饰突变体从而发掘GL2的新互作因子。在对以gl2-3背景进行的EMS诱变库的筛选过程中,获得了一个表皮毛突变体,将其命名为M12-01(代表第12个诱变pool中筛选到的第1个突变体)。与gl2-3相比,M12-01 突变体叶片上完全没有表皮毛,其他发育表型与野生型相比没有明显差异(图 1),表明M12-01的突变基因可能是GL2基因的增强子。由于M12-01突变体中还包含有gl2突变位点,为了获得M12-01 single,将M12-01突变体与Col-0进行回交,在F2代植株中挑选出Col-0背景的M12-01 single,并继续将M12-01 single与Col-0连续回交3次,一方面清除遗传背景,一方面观察M12-01 single的突变表型是否能稳定遗传。结果(图 1)发现,M12-01 single与Col-0相比,其叶片表面几乎没有表皮毛,只在边缘有少量的单分支的表皮毛,并且M12-01 single的突变表型能稳定遗传。
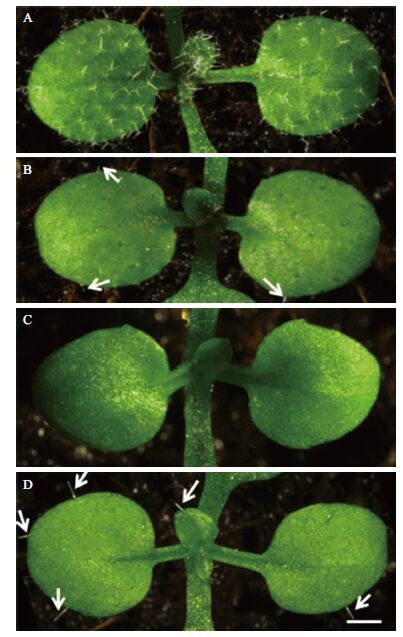
|
| 图 1 M12-01及M12-01 single突变体的表皮毛发育表型 A:Col-0;B:gl2-3突变体;C:M12-01突变体;D:M12-01 single突变体。图中白色箭头分别指示gl2-3及M12-01 single 突变体中叶边缘的表皮毛。Bar=1 mm |
由于M12-01 single突变体表型明显,并且可能与GL2存在一定的互作关系,所以利用图位克隆的方法对突变基因进行了克隆,以期能发现GL2的新遗传因子。首先我们进行了正反交试验,结果显示无论是正交还是反交,所获得的F1代植物均表现与野生型一致的表型,表明该突变体是由核基因控制的隐性突变体。在F1自交后代F2中野生型表型与突变体表型个体的分离比为3∶1,符合孟德尔隐性单基因遗传规律,这说明M12-01 single表型是受单个隐性的核基因控制的。
2.2 M12-01 single突变基因的定位 2.2.1 M12-01single突变位点的粗定位首先将Ler生态型拟南芥与M12-01 single BC3突变体植株进行杂交,在F2代中挑选M12-01 single表型植株作为图位克隆的作图群体,利用实验室已有的覆盖且等分拟南芥5条染色体的27个分子标记(图 2)和M12-01 single突变体DNA混合池bulk进行了染色体的粗定位。根据与突变位点连锁的分子标记所扩增出的条带的遗传背景偏向突变体来源的Col-0,而用于杂交的另一亲本Ler条带不能扩增或扩增量弱于对照的原理,我们发现突变位点与第五条染色体上分子标记F13G24(2、501和825),MOJ16(7、464和555),F14I23(9、935和849)及K18L3(15、085和156)连锁较为紧密(图 2)。
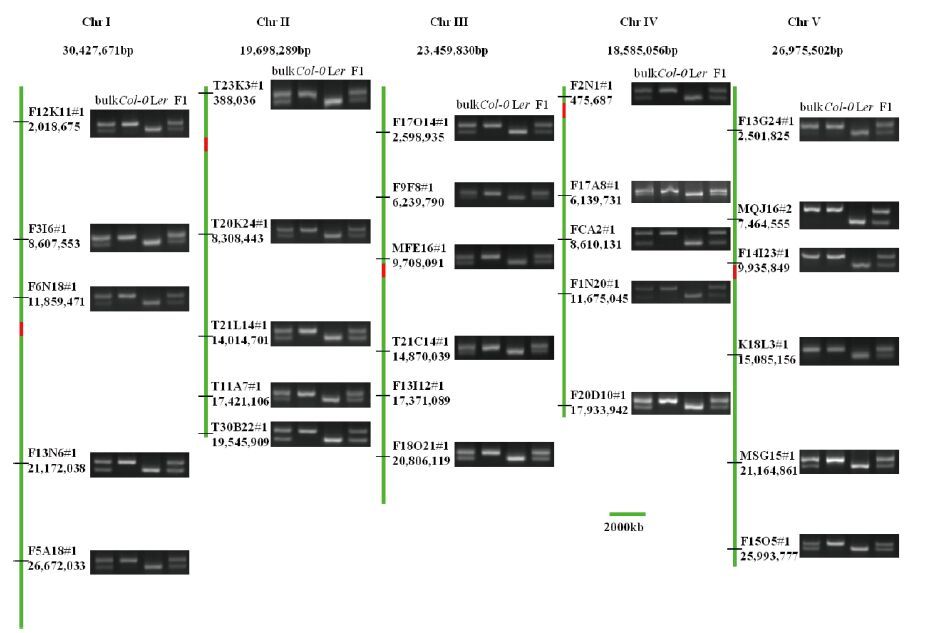
|
| 图 2 M12-01 single突变位点的粗定位 |
确定突变位点与染色体的连锁关系后,为了进一步确定突变位点在染色体上所在的区间,以 95个突变体DNA为模板,以F13G24#1(2、501和825)和K18L3(15、085和156)为引物进行PCR(表 1),结果显示突变位点位于分子标记F13G24#1和K18L3#1之间,所以我们将分子标记F13G24#1和K18L3#1作为后续定位的侧翼分子标记(flanking marker)(图 3)。接着我们通过在flanking marker区间内增加分子标记密度对M12-01 single 突变位点进行精细定位。利用2个新的插入/删除(In/Del)分子标记MQJ16#1(7、464和555)和F14I23#1(9、935和849)(表 1)对F2作图群体中95株突变体表型植物的基因型分析,最终将突变基因定位在分子标记MQJ16#1和F14I23#1的区间内,该区间的物理距离为2、471和294 bp(图 3)。
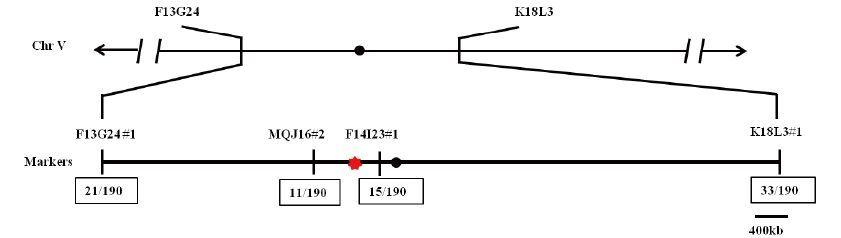
|
| 图 3 M12-01 single突变基因的精细定位 分子标记下面的方框内数字表示染色体交换个数;红色的星号表示M12-01 single突变基因At5g24520所在的位置 |
确定了M12-01 single突变位点所在的区间后,对该区间内基因注释进行分析,重点关注与表皮毛发育相关的基因。在这个区间里,基因At5g24520被报道与表皮毛发育有关,At5g24520基因也被称为TRANSPARENT TESTA GLABRA 1(TTG1),它编码一个含有4-5个WD重复的小蛋白[12]。并且,TTG1基因的功能缺失突变体ttg1-1与野生型相比,叶表面光滑,只在叶片边缘有少量表皮毛,种皮颜色为黄色[13],与M12-01 single突变体表型相似(图 4)。据此推测M12-01 single突变体的表型可能为TTG1基因功能缺陷导致。

|
| 图 4 M12-01 single与ttg1-1突变体的表型 A和A':野生型的表皮毛(A)及种皮颜色(A')表型;B和B':M12-01的表皮毛(B)及种皮颜色(B')表型;C和C':ttg-1突变体的表皮毛(C)及种皮颜色(C')表型。图中白色箭头分别指示M12-01 single 及ttg1-1突变体中叶边缘的表皮毛。A、B、C:Bar=1 mm;A'、B'、C':Bar=500 µm |
为了验证我们的推测,设计TTG1基因的特异性引物,以M12-01 single BC3突变体DNA为模板对TTG1编码区的DNA片段进行PCR扩增并对扩增产物进行测序。引物序列如下:At5g24520F:5'-GATACTGATTAATCCAGAGG-3';At5g24520R:5'-GACTATCTGTAATGCAACAC-3'。测序结果(图 5)显示,TTG1基因的+445 bp处发生了单碱基突变,由鸟嘌呤(G)突变为腺嘌呤(A),导致中性氨基酸Gly149突变为碱性氨基酸Arg149。这说明TTG1基因的单碱基突变很可能是造成M12-01 single突变表型产生的原因,TTG1可能就是M12-01 single的突变基因。
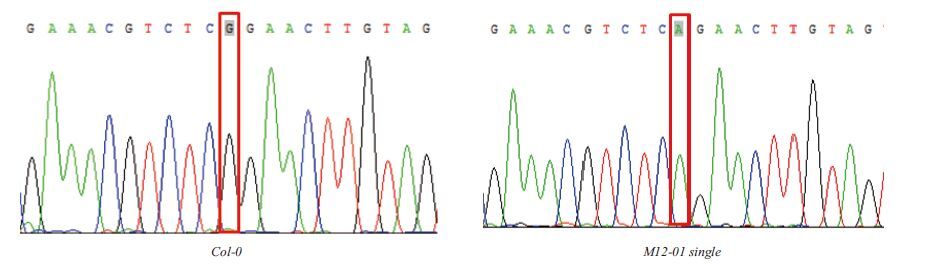
|
| 图 5 M12-01single 突变位点的确定 红色方框表示突变碱基所在位置 |
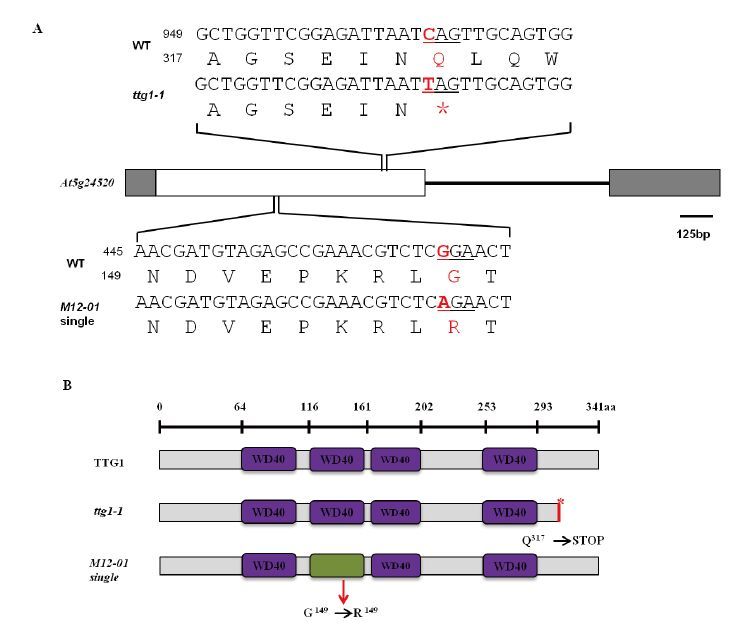
为了进一步确定TTG1基因的突变是造成M12-01突变表型产生的原因,并且TTG1突变能增强gl2-3突变体的表型,对M12-01 single和ttg1-1突变体进行等位性分析。ttg1-1的突变表型是由于TTG1基因的+949 bp处发生了单碱基突变,由胞嘧啶(C)突变为胸腺嘧啶(T),导致该位点的氨基酸由Gln变为终止密码子产生无义突变,引起了编码蛋白的提前终止[14](图 6)。
|
| 图 6 M12-01 single和ttg1-1突变体分析 A:ttg1-1和M12-01 single的突变位点;B:TTG1以及ttg1-1和M12-01 single背景中TTG1蛋白结构域;紫色方框为WD40结构域,红色星号为ttg1-1突变体中TTG1蛋白氨基酸残基突变位置,绿色方框为M12-01 single突变体中TTG1蛋白氨基酸残基发生突变所在区域 |
首先我们将M12-01 single与ttg1-1进行了杂交,观察F1植物的表型,发现F1植物的表型与M12-01 single相似(图 7-a),表明M12-01 single与ttg1-1不能互补彼此的突变表型,M12-01 single与TTG1是等位基因。其次我们将ttg1-1 突变体与gl2-3进行了杂交,构建ttg1-1 gl2-3 双突变体,观察ttg1-1是否也能增强gl2-3突变体的表型,结果(图 7-b)表明ttg1-1 gl2-3双突变的表型与M12-01相似,叶片上完全没有表皮毛,这表明ttg1-1像M12-01 single一样也能增强gl2-3突变体的表型。这些结果说明M12-01 single 的突变表型就是由TTG1基因的单碱基突变造成的,TTG1与GL2基因之间存在遗传互作,TTG1突变可以增强GL2功能缺失突变体的表型。

|
| 图 7 M12-01single和ttg1-1突变体的等位性分析 A-G:分别为M12-01 single,ttg1-1,M12-01 single ttg1-1 F1,gl2-3,ttg1-1,ttg1-1gl2-3及M12-01突变体表皮毛表型;A'-G':分别为对应的A-G图的放大。Bar=1mm |
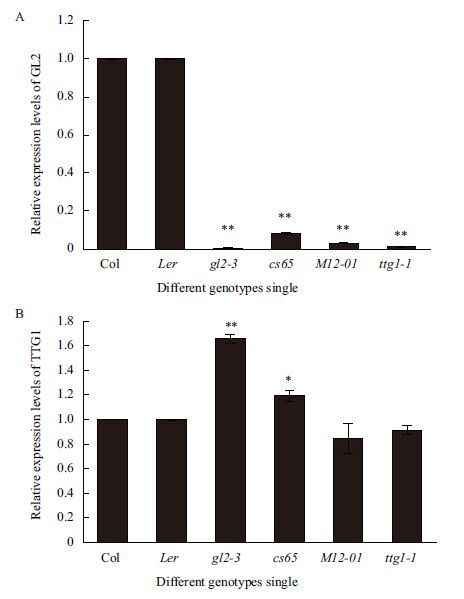
确定GL2与TTG1在遗传互作中的上下游关系。通过实时荧光定量RT-PCR检测TTG1突变对GL2基因表达水平的影响以及GL2基因突变对TTG1基因表达水平的影响。结果(图 8)显示在M12-01 single突变体中,GL2的表达水平显著受到抑制,在ttg1-1突变体中,也看到了相似的结果。这表明TTG1作用于GL2的上游。在gl2-3突变体中,TTG1基因的表达水平显著增加,在GL2功能缺失突变体cs65中,TTG1的表达水平也有所上升(图 8),这表明GL2可能对TTG1的转录有潜在的调节作用。
|
| 图 8 GL2(A)及TTG1(B)基因的表达水平分析 *表示P值在0.05水平上显著,**表示在0.01水平上显著 |
遗传突变体的获得是研究基因功能的强有力手段。为了进一步发掘与GL2互作的新因子,揭示GL2参与调控表皮毛发育的作用机制,本实验利用EMS高效诱变拟南芥gl2-3突变体所建立的二次突变体库,筛选gl2-3的修饰突变体,以期能获得GL2的新的互作因子。通过对拟南芥表皮毛发育状态的观察,筛选出了一个gl2-3的增强突变体M12-01并获得了M12-01 single。基因克隆表明M12-01 single的突变表型是由TTG1基因的+445 bp处发生由鸟嘌呤到腺嘌呤的单碱基突变,从而使中性氨基酸Gly149突变为碱性氨基酸Arg149所导致的。进一步的等位性分析证实TTG1基因突变是造成M12-01 single表型产生的原因,M12-01 single是TTG1的一个新的等位突变体。
早期对GL2基因的研究发现其编码一个HD-Zip转录因子,是调控表皮毛形成和发育的关键因子,它的功能丧失导致表皮严重缺失和发育受阻[15]。GL2和TTG1均参与早期表皮毛发育过程,TTG1编码一个含有4-5个WD重复的小蛋白,它能与GL3相互作用形成bHLH/WD40转录复合体,通过调控GL2的表达从而调控表皮毛细胞的形成及发育。本研究发现TTG1突变能增强GL2突变体的表型,这表明TTG1和GL2在调控表皮毛发育的过程中可能处于不同的途径上,GL2在表皮毛发育过程中对TTG1部分依赖又相互独立。
本实验所获得的M12-01突变体为进一步研究TTG1与GL2基因之间的互作关系提供了新的遗传材料,为深入研究GL2参与植物表皮毛发育的调控关系奠定了基础。
4 结论通过对gl2-3突变体背景的EMS诱变体库的筛选获得一株完全没有表皮毛的突变体M12-01,图位克隆发现该突变基因是TTG1的等位基因。遗传分析证实TTG1突变可以增强GL2突变体的表型。
| [1] | Johnson HB. Plant pubescence:an ecological perspective. Bot Rev , 1975, 41 (3) : 233–258. DOI:10.1007/BF02860838 |
| [2] | Yang C, Li H, Zhang J, et al. A regulatory gene induces trichome formation and embryo lethality intomato. Proc Natl Acad Sci USA , 2011, 108 (29) : 11836–11841. DOI:10.1073/pnas.1100532108 |
| [3] | Levin DA. The role of trichomes in plant defence. Rev Biol , 1973, 48 (1) : 3–15. DOI:10.1086/407484 |
| [4] | Saltveit ME, Hepler PK. Effect of heat shock on the chilling sensiti-vity of trichome and petiole of African violet(Saintpaulia ionantha). Physiol Plant , 2004, 121 (1) : 35–43. DOI:10.1111/ppl.2004.121.issue-1 |
| [5] | Karabourniotis G, Papadopoulos K, Papamarkou M, et al. Ultraviolet-B radiation absorbing capacity of leaf hairs. Physiol Plant , 1992, 86 (3) : 414–418. DOI:10.1111/ppl.1992.86.issue-3 |
| [6] | Huttunen P, K?rkk?inen K, L?e G, et al. Leaf trichome production and response to defoliation and drought in Arabidopsis lyrata(Brassicaceae). Ann Bot Fennici , 2010, 47 (3) : 199–207. DOI:10.5735/085.047.0304 |
| [7] | Davidian JC, Grill D, Kok LJD, et al. Leiden:Backhuys Publishers[M]. 2003 : 1933-1949. |
| [8] | Li W, Chen T, Chen Y, et al. Role of trichome of Pterisvittata L. in arsenichyper accumulation. Sci China Ser CLife Sci , 2005, 48 (2) : 148–154. |
| [9] | Loughner R, Goldman K, Loeb G, et al. Influence of leaf trichomes on predatory mite(Typhlodromus pyri)abundance in grape varieties. Exp Appl Acarol , 2008, 45 (3) : 111–122. |
| [10] | Hülskamp M, Misra S, Jurgens G. Genetic dissection of trichome cell development in Arabidopsis. Cell , 1994, 76 (3) : 555–566. DOI:10.1016/0092-8674(94)90118-X |
| [11] | Szymanski DB, Jilk RA, Pollock SM, et al. Control of GL2 expression in Arabidopsis leaves and trichome. Development , 1990, 125 (7) : 1161–1171. |
| [12] | Walker AR, Davison PA, Bolognesi-Winfield AC, et al. The TRANSPARENT TESTA GLABRA1 locus, which regulates trichome differentiation and anthocyanin biosynthesis in Arabidopsis, encodes a WD40 repeat protein. Plant Cell , 1999, 11 (7) : 1337–1350. DOI:10.1105/tpc.11.7.1337 |
| [13] | Koornneef M. The complex syndrome of ttg mutants. Arabidopsis Inf Serv , 1981, 18 : 45–51. |
| [14] | Chopra D, Wolff H, Span J, et al. Analysis of TTG1 function in Arabisalpina. Bmc Plant Biol , 2014, 14 (1) : 1–14. DOI:10.1186/1471-2229-14-1 |
| [15] | 曹敏, 张璐, 高新梅, 等. 植物表皮毛发育分子调控机制的研究进展. 安徽农业科学 , 2013, 41 (10) : 4231–4235. |





