2. 中国农业大学园艺学院, 北京100193
2. College of Horticulture, China Agricultural University, Beijing 100193
黑果枸杞(Lycium ruthenicum Murr.)是在我国西北干旱盐碱地生境广泛分布的多年生灌木,是一种集盐碱地绿化价值、防护林价值、药食用价值等于一体的野生优良水土保持植物。黑果枸杞果实中富含枸杞多糖[1, 2]、色素[3, 4]、类黄酮[5]、甜菜碱[6]、微量元素、脂肪酸、挥发油、维生素等多种成分,其果实提取物在抗氧化[7]、调节免疫力[8]、抗疲劳[9]、降血脂[10]、治疗心血管系统疾病、抗动脉硬化[11]、抗衰老等方面均有明确作用。
除对黑果枸杞植物生理、产物成分等研究外,近年来研究者开始进行黑果枸杞遗传基因的研究[12],包括针对黑果枸杞灌木种群遗传学[13]、序列扩增多态性(SRAP)[14]和微卫星标记(SSR)[15]等,探究黑果枸杞在紫外线胁迫下转录组的变化[16],以及对宁夏枸杞和黑枸杞花青素合成的转录组对比[17]等,以期在分子生物层面揭示其生理发育等过程的机制。
果实成熟发育过程是果实功能与品质形成的基础,这个过程受到遗传、基因表达和环境因素的影响[18-20]。随着基因组测序技术和生物信息学的迅速发展,基因组、转录组、蛋白组和代谢组学等技术手段被广泛应用于果实发育与品质形成研究中,为理解果实成熟变化和利用基因资源控制品质形成提供了重要研究数据[21-24]。果实发育过程中,伴随果实细胞生理和生化代谢的变化,糖分、氨基酸、色素、脂肪酸和芳香酯含量发生变化,以及其它次生代谢物、特色营养物质积累,从而产生不同的风味和功能特性[25]。由于我国目前黑果枸杞主要以野生和半野生分布生长于不同地区,果实品质存在一定差异。利用果实转录组学相关手段,展开黑果枸杞果实生长发育基因表达变化特性研究,对理解和指导我国不同生态条件下黑果枸杞发育特征和果实品质提高具有重要意义。
本研究利用RNA-seq技术在RNA水平对黑果枸杞果实座果后青果期、变色期、黑熟期3个不同生长和成熟时期进行基因表达组学分析,探讨果实发育的相关基因功能表达差异,旨在为黑果枸杞品质形成和的基因调控及优良株系选育提供基础数据。
1 材料与方法 1.1 材料黑果枸杞(Lycium ruthenicum Murr.)从花期座果后到成熟期果实,采自宁夏回族自治区银川市永宁县南郊半干旱地域(年平均气温8.7℃,夏季各月平均气温在20℃以上,无霜期平均167 d,多年年平均降水量为201 mm),分级后选取早期结实成型的青色果实(早期,果实颜色为青绿色,简称青果期,S1),开始变色时期的果实(中期,果身大部分为青绿色、局部为淡紫色,简称变色期,S2)和完全成熟的果实(晚期,果实迅速膨大、通体呈黑紫色,简称黑熟期,S3)各3份,每份10 g,液氮速冻,于-80℃冰箱保存用于后续总RNA提取、RNA-seq样品制备和测序分析。
1.2 方法 1.2.1 黑果枸杞果实发育各时期总RNA的提取和测序总RNA的提取采用改良的CTAB法[26],对3个时期的黑果枸杞果实及其重复样本提取总RNA。选取RNA浓度≥200 ng/mL,OD260/280比值为1.8-2.2的黑果枸杞果实总RNA样品,送深圳华大基因研究院进行后续测序。
去除rRNA后质控合格的mRNA样品按参考样品(S1+S2+S3)和各果实时期S1-3样品分别进行RNA-seq样品制备、合并和测序。
1.2.2 测序数据处理对测序得到的原始序列去除接头(adaptor)序列、低质量标签(Tag)及长度过小和过大的Tag等数据,得到高质量读长(clean reads);进行数据产出统计及测序数据的成分和质量评估;用短reads组装软件Trinity组装;对测序结果进行饱和度和质控分析。筛选获得的clean reads用于Unigene后续分析和GO、NR、NT Swiss-Prot和KO等数据库注释,并分别对注释到每个库以及所有注释上的Unigene数目进行统计;去除其中的杂质数据,用SOAP2[27](http://soap.genomics.org.cn/soapaligner.html)对黑果枸杞转录组数据进行数据库对比分析。基因表达量的计算使用 RPKM 法(Reads per kb per million reads)[28],基因表达量符合P<0.005,错配率(FDR)≤0.001,且|log2(差异表达倍数)|>1(即|log2ratio|>1)的基因被定义为显著差异表达的基因。差异基因检测方法参考“The significance of digital gene expression profiles”[29],并对差异表达基因进行COG分析和KEGG功能与代谢途径分析。
2 结果总转录组数据达到5.14 G,总clean read数51 393 116,以黑果枸杞果实发育总表达基因数据作为果实参考转录组,对黑果枸杞果实不同发育期的青果期(S1)、变色期(S2)和黑熟期(S3)的表达谱及相互间的差异进行分析。
2.1 测序质量评价对黑果枸杞参考转录组和各时期果实测序饱和度的数据分析,显示黑果枸杞发育的3个不同时期的样品都呈现出在标签数量较少时,检测到的基因数目较少,基因数目随着测序量的增加也急剧增加的模式。当S1的read数达到80×100 k时,read数接近不再上升,这表明此时样本的测序量已经基本覆盖到细胞中基因组可表达的全部基因。图 1为S1样品的测序随机性统计分析的结果,表明黑果枸杞mRNA样本在RNA-Seq实验过程中,通过超声方法片段化后的测序的read分布较均匀,为随后的各项转录组差异分析提供了很好的保障。

|
| 图 1 黑果枸杞S1期果实转录组基因测序频数与基因位置分布 |
通过Gene Ontology(GO)基因功能分类体系注释分析黑果枸杞果实各发育期间差异表达基因在分子功能(molecular function)、细胞组分(cellular component)、参与的生物过程(biological process)中的分布(图 2),可以反映果实基因表达所表征的代谢富集差异和相应生物学功能的变化。本研究中黑果枸杞果实大部分差异基因的功能集中在分子功能的结合和催化活性方面,细胞组件功能的细胞、细胞组件和细胞器上,在生物过程功能中主要参与细胞过程和代谢过程中。在生物过程中,基因差异数最多;在细胞组件和分子功能中,差异基因为前者的1/3-1/2;而在分子功能组中,差异表达基因数量更少。其中,与亮氨酸合成途径有关的Unigene有14个,其中在S3/S1对比中差异表达基因为9个,S3/S2中为8个,S2/S1中无差异表达基因。

|
| 图 2 黑果枸杞果实不同时期基因的功能分类 |
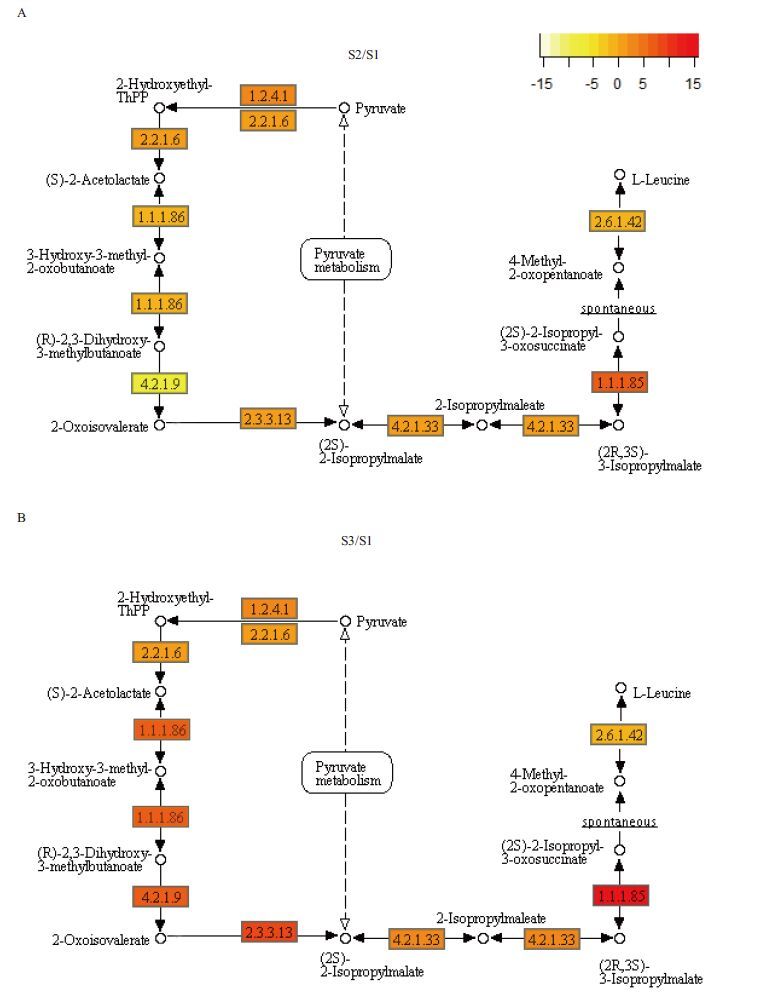
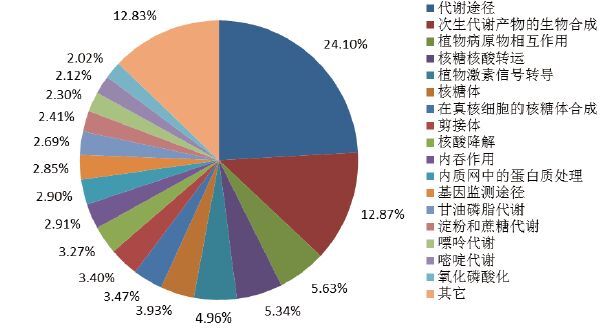
通过KEGG代谢途径富集分析(metabolic pathway enrichment analysis),在黑果枸杞果实中鉴定出次生代谢物合成代谢途径(biosynthesis of secondary metabolites)基因为3 380个,占到注释参与代谢途径基因总数的12.9%(图 3)。在S2/S1、S3/S2和S3/S1中分别注释到3 917、5 133和7 259个差异表达基因,说明黑果枸杞随果实发育进程,代谢途径所需基因数量与模式不断变化。亮氨酸生物合成属于基础代谢物中重要的中间代谢物或前体,黑果枸杞果实成熟发育过程的亮氨酸生物合成代谢途径基因表达变化,见图 4。S2/S1中亮氨酸生物合成代谢途径仅有7个差异表达基因,其中3个上调基因,4个下调基因;S3/S2有35个差异表达基因,全部为上调基因,总体上(S3/S1)有39个差异表达基因,其中36个上调基因,3个下调基因,说明此代谢对黑果枸杞果实成熟与功能性物质转化特别是花青素苷元类物质代谢,具有重要意义。
|
| 图 3 黑果枸杞不同代谢通路中基因占总注释基因的百分比 |
S2/S1中,二羟基酸脱水酶(ilvD,EC:4.2.1.9)和酮醇酸还原异构酶(ilvC,EC:1.1.1.86)基因有显著的下调;在S3/S1中,除支链氨基酸氨基转移酶(ilvE,EC:26.6.1.42)外,所有基因有显著的上调,其中3-异丙基苹果酸脱氢酶(leuB,EC:1.1.1.85)出现非常显著的上调,log2(差异表达倍数)达到15.6,乙酰羟酸合酶(ilvI,EC:2.2.1.6)和2-异丙基苹果酸合酶(leuA,EC:2.3.3.13)的log2(差异表达倍数)分别为9.04和9.19。
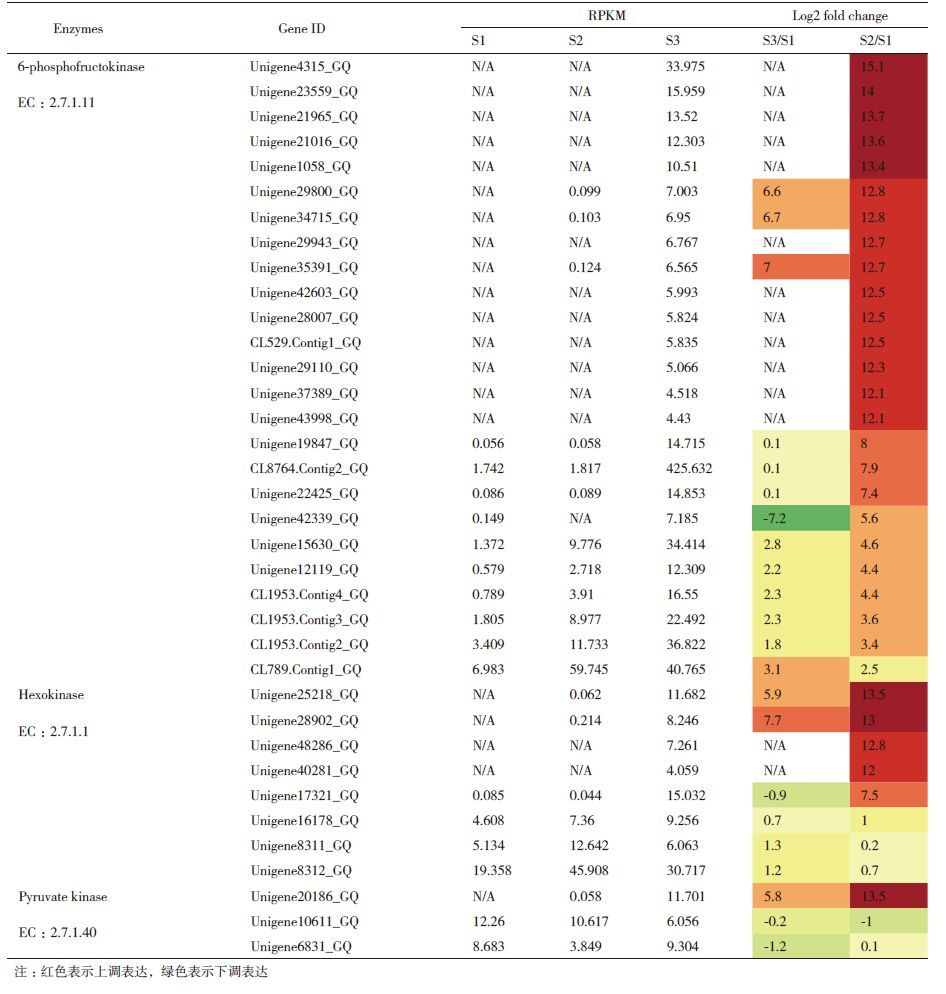
糖酵解是为细胞生命活动提供部分能量及为其它代谢途径提供中间产物的基础代谢途径。表 1列出了黑果枸杞果实不同发育时期的糖酵解途径关键酶基因转录的差异情况。在糖酵解途径中,S2/S1有55个差异表达基因,其中上调表达22个,下调表达33个;S3/S1中,有207个差异表达基因,其中上调表达182个,下调表达25个,表明黑果枸杞果实成熟时,糖酵解的代谢重要性在明显增强,尤其是糖酵解过程中3个不可逆的反应步骤限速酶己糖激酶(hexokinase,EC:2.7.1.1)、6-磷酸果糖激酶(6-phosphofructokinas,EC:2.7.1.11)和丙酮酸激酶(pyruvate kinase,EC:2.7.1.40)的表达。在不同的果实发育阶段,编码上述3个酶的家族基因表达量都呈现出逐渐增高的规律,其中6-磷酸果糖激酶的Unigenes在青果期基本不表达或者表达量较少,而在果实完全成熟阶段表达量急剧增加,其中Unigene4315_GQ在青果期和变色期未见表达,而完全成熟阶段RPKM值为33.975;CL8764.Contig2_GQ在3个时期的RPKM值分别为1.742、1.817和425.632,青果期与变色期无明显差异,而黑熟期其表达量急剧增加;仅Unigene42339_GQ在变色期未见表达,相对青果期表现为下调表达,在黑熟期重新表现为上调表达。
由于黑果枸杞基因组测序尚未完成,本研究中黑果枸杞转录组的注释采用参考转录组以及对已有植物基因和蛋白资源的相对注释,可注释已知功能基因达59 537个,未知基因为25 431个,能够总体反映出黑果枸杞果实发育成熟过程的基因转录谱特征。黑果枸杞果实中含有丰富的氨基酸,黑果枸杞果实中除色氨酸外,其他 7 种必需氨基酸含量较丰富,其中质量分数最高的是亮氨酸(Leu),为9.366 g/kg(干重)[31]。探索黑果枸杞成熟过程中亮氨酸生物合成变化,有助于了解其氨基酸合成与积累情况,对选育更富有营养的品种具有重要作用。
L-亮氨酸、L-缬氨酸和L-异亮氨酸属于支链氨基酸,因其疏水脂质链都具有分支的甲基基团,又称之为分支链氨基酸[32],3种支链氨基酸的生物合成途径是紧密相连的[33]。在丙酮酸合成L-亮氨酸的代谢途径中,乙酰羟酸合酶(acetohydroxyacid synthase,AHAS)由ilvBN编码,是合成途径上的第一个关键酶[34]。在 L-亮氨酸生物合成中,第二个关键酶为异丙基苹果酸合成酶(Isopropylmalatesynthetase,IPMS),IPMS 由leuA基因编码,催化2-酮异戊酸生成异丙基苹果酸[35],IPMS受到 L-亮氨酸的反馈抑制和反馈阻遏。ilvC基因与ilvBN基因序列在基因组上相邻共同形成一个操纵子,表达ilvBNC基因有利于L-亮氨酸生物合成前体物α-酮基异戊酸的生成[36]。这2个关键酶的基因,在S2/S1中仅有2-3倍的增加,而在S3/S1 中急剧增加了527倍和584倍,说明在果实初步成型期和果实开始变色期的果实,其亮氨酸的合成并不丰富,而在果实完全成熟阶段,编码亮氨酸合成的基因表达急剧增加,有利于亮氨酸的生物合成和积累。
在黑果枸杞转录组分析中,分析出萜类化合物骨架生物合成Unigene 216个,二萜类化合物的生物合成Unigene 147个,说明黑果枸杞的萜类合成能力较强。糖酵解产物3-磷酸甘油醛、丙酮酸及乙酰CoA是萜类物质合成的前体[37],己糖激酶、6-磷酸果糖激酶和丙酮酸激酶基因表达的上调将为成熟果实中的萜类合成提供更多的碳源。6-磷酸果糖激酶的Unigene在果实发育前期,表达量较少或者不表达,而在果实成熟过程中表达量逐渐增加。糖酵解在果实发育后期的增加,除了代谢中间产物生成的增加同时也体现了果实成熟后期物质积累所需要能量的增加,果实通过加大糖酵解的反应获得部分所需能量,糖分代谢对果实干物质形成具有重要意义。萜类化合物骨架生物合成后可以为N-聚糖生物合成途径提供二磷酸多萜醇(dolichol diphosphate),在该途径中糖基转移酶催化单糖从糖供体转移到糖受体分子,参与天然产物黄酮类生物合成路径,为花青素花色苷合成提供糖苷配体。
高通量测序技术具有海量数据、可靠性高、实验操作简单、节约时间等优点,现已经广泛应用于生命科学、农业等领域[38]。此外,如何对测序获得的海量数据进行有效分析,也对研究人员在专业性和生物信息学分析能力提出了更高的要求。
4 结论在黑果枸杞果实中鉴定出次生代谢物合成代谢途径基因3 380个。黑果枸杞果实成熟发育过程中亮氨酸生物合成代谢途径,总体上有39个差异表达基因,其中36个上调基因,3个下调基因。编码糖酵解过程中3个不可逆的反应步骤限速酶:己糖激酶、6-磷酸果糖激酶和丙酮酸激酶的家族基因,其表达量在黑果枸杞果实发育阶段呈现出逐渐增高的规律。
| [1] | Peng Q, Lv X, Xu Q, et al. Isolation and structural characterization of the polysaccharide LRGP1 from Lycium ruthenicum. Carbohydrate Polymers , 2012, 90 : 95–101. DOI:10.1016/j.carbpol.2012.04.067 |
| [2] | 李艳, 孙萍, 鲁建疆, 等. 新疆黑枸杞多糖的提取及含量测定. 数理医药学杂志 , 2001, 14 : 164–165. |
| [3] | 李进, 瞿伟菁, 吕海英, 等. 黑果枸杞色素的提取和精制工艺研究. 天然产物研究与开发 , 2006, 18 : 650–654. |
| [4] | 孙奎. 柴达木盆地黑果枸杞色素最佳提取工艺研究. 湖北农业科学 , 2011, 50 : 2318–2320. |
| [5] | 李淑珍, 李进, 杨志江, 等. 大孔树脂分离纯化黑果枸杞总黄酮的研究. 食品科学 , 2009, 30 : 19. |
| [6] | 刘增根, 陶燕铎, 邵赟, 等. 柴达木枸杞和黑果枸杞中甜菜碱的测定. 光谱实验室 , 2012, 29 : 694. |
| [7] | 陈晨, 赵晓辉, 文怀秀, 等. 黑果枸杞的抗氧化成分分析及抗氧化能力测定. 中国医院药学杂志 , 2011, 31 : 1305–1306. |
| [8] | 贾琦珍, 陶大勇, 陈瑛, 等. 黑果枸杞色素对巨噬细胞的激活作用研究. 中兽医医药杂志 , 2008, 27 : 29–30. |
| [9] | 汪建红, 陈晓琴, 张蔚佼. 黑果枸杞果实多糖抗疲劳生物功效及其机制研究. 食品科技 , 2009 : 203–207. |
| [10] | 吕海英, 林丽, 等. 黑果枸杞叶总黄酮抗氧化和降血脂成分测定. 新疆师范大学学报:自然科学版 , 2012, 31 : 43–48. |
| [11] | 林丽, 李进, 吕海英, 等. 黑果枸杞花色苷对小鼠动脉粥样硬化的影响. 中国中药杂志 , 2012, 37 : 1460–1466. |
| [12] | 孙晓东, 李军, 施京红. 枸杞基因组 DNA 的提取与分析. 陕西中医 , 2004, 24 : 1129–1130. |
| [13] | Chen H, Zeng L, Yonezawa T, et al. Genetic population structure of the desert shrub species Lycium ruthenicum inferred from chloroplast dna. Pakistan Journal of Botany , 2014, 46 : 2121–2130. |
| [14] | Liu Z, Shu Q, Wang L, et al. Genetic diversity of the endangered and medically important Lycium ruthenicum Murr. revealed by sequence-related amplified polymorphism(SRAP)markers. Biochemical Systematics and Ecology , 2012, 45 : 86–97. DOI:10.1016/j.bse.2012.07.017 |
| [15] | Chen H, Zhong Y. Microsatellite markers for Lycium ruthenicum(Solananeae). Molecular Biology Reports , 2014, 41 : 5545–5548. DOI:10.1007/s11033-014-3442-9 |
| [16] | Chen H, Feng Y, Wang L, et al. Transcriptome profiling of the UV-B stress response in the desert shrub Lycium ruthenicum. Molecular Biology Reports , 2015, 42 : 639–649. DOI:10.1007/s11033-014-3809-y |
| [17] | Zeng S, Wu M, Zou C, et al. Comparative analysis of anthocyanin biosynthesis during fruit development in two Lycium species. Physiologia Plantarum , 2014, 150 : 505–516. DOI:10.1111/ppl.12131 |
| [18] | Klee HJ, Giovannoni JJ. Genetics and control of tomato fruit ripening and quality attributes. Annual Review of Genetics , 2011, 45 : 41–59. DOI:10.1146/annurev-genet-110410-132507 |
| [19] | Giovannoni JJ. Genetic regulation of fruit development and ripening. The Plant Cell , 2004, 16 : S170–S180. DOI:10.1105/tpc.019158 |
| [20] | Grierson D, Kader AA. Fruit ripening and quality. // The Tomato Crop[M]. Atherton JG, et al. Chapman and Hall Ltd, USA, 1986:241-280. |
| [21] | Lombardo VA, Osorio S, Borsani J, et al. Metabolic profiling during peach fruit development and ripening reveals the metabolic networks that underpin each developmental stage. Plant Physiology , 2011, 157 : 1696–1710. DOI:10.1104/pp.111.186064 |
| [22] | Zhang J, Ma H, Feng J, et al. Grape berry plasma membrane proteome analysis and its differential expression during ripening. Journal of Experimental Botany , 2008, 59 : 2979–2990. DOI:10.1093/jxb/ern156 |
| [23] | Wang Z, Zhao F, Zhao X, et al. Proteomic analysis of berry-sizing effect of GA3 on seedless Vitis viniferaL. Proteomics , 2012, 12 : 86–94. DOI:10.1002/pmic.v12.1 |
| [24] | Chai L, Li Y, Chen S, et al. RNA sequencing reveals high resolution expression change of major plant hormone pathway genes after young seedless grape berries treated with gibberellin. Plant Science , 2014, 229 : 215–224. DOI:10.1016/j.plantsci.2014.09.010 |
| [25] | 吕英民, 张大鹏. 果实发育过程中糖的积累. 植物生理学通讯 , 2000, 36 : 258–265. |
| [26] | Reid KE, Olsson N, Schlosser J, et al. An optimized grapevine RNA isolation procedure and statistical determination of reference genes for real-time RT-PCR during berry development. BMC Plant Biology , 2006, 6 : 27. DOI:10.1186/1471-2229-6-27 |
| [27] | Li R, Yu C, Li Y, et al. SOAP2:an improved ultrafast tool for short read alignment. Bioinformatics , 2009, 25 : 1966–1967. DOI:10.1093/bioinformatics/btp336 |
| [28] | Mortazavi A, Williams BA, McCue K, et al. Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nature Methods , 2008, 5 : 621–628. DOI:10.1038/nmeth.1226 |
| [29] | Audic S, Claverie JM. The significance of digital gene expression profiles. Genome Research , 1997, 7 : 986–995. |
| [30] | Xu H, Zhang Y, Guo X, et al. Isoleucine biosynthesis in Leptospira interrogans serotype lai strain 56601 proceeds via a threonine-independent pathway. Journal of Bacteriology , 2004, 186 (16) : 5400–5409. DOI:10.1128/JB.186.16.5400-5409.2004 |
| [31] | 矫晓丽, 迟晓峰, 董琦, 等. 柴达木野生黑果枸杞营养成分分析. 氨基酸和生物资源 , 2011, 33 : 60–62. |
| [32] | Bonnefoy M, Laville M, et al. Effects of branched amino acids sup-plementation in malnourished elderly with catabolic status. The Journal of Nutrition, Health & Aging , 2010, 14 : 579–584. |
| [33] | 张伟国, 郭燕风. 支链氨基酸生物合成及其代谢工程育种研究进展. 食品与生物技术学报 , 2014, 33 : 120–126. |
| [34] | Park JH, Lee KH, et al. Metabolic engineering of Escherichia coli for the production of L-valine based on transcriptome analysis and in silico gene knockout simulation. Proceedings of the National Academy of Sciences , 2007, 104 : 7797–7802. DOI:10.1073/pnas.0702609104 |
| [35] | Ruklisha M, Paegle L, Denina I. L-Valine biosynthesis during batch and fed-batch cultivations of Corynebacterium glutamicum:relationship between changes in bacterial growth rate and intracellular metabolism. Process Biochemistry , 2007, 42 : 634–640. DOI:10.1016/j.procbio.2006.11.008 |
| [36] | Kalinowski J, Bathe B, Bartels D, et al. The complete Corynebacterium glutamicum ATCC 13032 genome sequence and its impact on the production of L-aspartate-derived amino acids and vitamins. Journal of Biotechnology , 2003, 104 : 5–25. DOI:10.1016/S0168-1656(03)00154-8 |
| [37] | 郝宏蕾, 朱旭芬. 类异戊二烯的生物合成及调控. 浙江大学学报:农业与生命科学版 , 2002, 28 : 224–230. |
| [38] | 闫绍鹏, 杨瑞华, 冷淑娇, 等. 高通量测序技术及其在农业科学研究中的应用. 中国农学通报 , 2012, 28 : 171–176. |