2. 海南大学农学院, 海口 570228
2. College of Agriculture, Hainan University, Haikou 570228
巴西橡胶树(Hevea brasiliensis)是热带山地主要的经济作物,种植面积大,林产品产量高,其林产品是由胶工通过周期性切割橡胶树树干树皮(即割胶)刺激乳管细胞释放出的天然胶乳,是一种丰富的天然橡胶资源。因其胶乳量极为丰富,并且干胶含量较高,已广泛应用于商业、防御和运输等行业[1]。从分子生物学的角度分析胶乳的形成机制将有助于对天然橡胶生产过程的全面了解。高质量胶乳总RNA的提取是研究橡胶树胶乳形成分子机制的基础。目前已报道的橡胶树RNA提取方法主要有十六烷基三甲基溴化铵(CTAB)法、十二烷基硫磺酸钠(SDS)法、异硫氰酸胍法、Trizol试剂盒法等[2-4],尽管不同的橡胶树组织及在不同生理状态下的供试材料,其细胞与组织的成分各不相同,但这些方法对于不同生理状态橡胶树的根、树皮、花、叶和胶乳都有其一定的适用性,其中SDS法及试剂盒法是近年来常用的提取胶乳RNA方法,且沉淀胶乳RNA的常用方法有异丙醇沉淀法、乙醇沉淀法和LiCl沉淀法[3-7]。已有研究表明,橡胶树胶乳中富含的蛋白、多糖、橡胶粒子等成分,在一定程度上使提取高质量胶乳RNA成为困难[8, 9]。集中表现为胶乳中高含量的蛋白质难以有效去除,导致提取的RNA纯度不高;胶乳的橡胶粒子含量达到了30%-50%,严重地影响RNA提取。本研究针对橡胶树胶乳的特点,在本实验室改良的SDS法[4]的基础上,分别用4种单方或复方沉淀剂对RNA进行沉淀,并将不同沉淀方法获得的橡胶树胶乳总RNA和以核糖体18S rRNA基因和胶乳优势表达基因橡胶延伸因子REF(rubber elongation factor)mRNA作为研究对象的扩增效果进行比较,找出受蛋白和橡胶粒子干扰最小的沉淀剂,改进RNA沉淀方法,旨在为提高改良SDS法提取胶乳总RNA的产量和质量提供理论支持。
1 材料与方法 1.1 材料实验材料为大规模推广级橡胶树优良品种热研7-33-97,林段位于中国热带农业科学院(儋州)试验场十二队,种植于2005年,至2012年开割,实验当年为3 a割龄的成龄幼树。
1.2 方法 1.2.1 胶乳的采集橡胶树割胶后,弃去前30 s流出的胶乳,然后引流胶乳滴入保温瓶里的液氮中,采集好的胶乳分装入40 mL的保鲜管内,并将保鲜管放入装有液氮的保温瓶中带回实验室,可直接用于RNA提取,或-80℃冰箱中保存待用。
1.2.2 总RNA的提取根据所处理样品的特性,对本实验室改进的SDS法略作了改良:在沉淀RNA时设计了4个处理。因此,本实验使用改良SDS法提取橡胶树胶乳总RNA的具体操作如下:
(1)量取2 mL新鲜胶乳或-80℃保存的样品迅速转移至预冷(4℃)的含有2 mL 2×SDS提取液(0.1 mol/L Tris-HCl,0.3 mol/L LiCl,10 mmol/L EDTA-Na2,100 g/L SDS,pH9.5)的EP管中,加入等体积的酚∶氯仿∶异戊醇(25∶24∶1),涡旋振荡5 min至充分混匀,4℃、12 000 r/min 离心 10 min。
(2)将上清液转移到另一新的EP管中,加入等体积的酚∶氯仿∶异戊醇(25∶24∶1),涡旋振荡5 min至充分混匀,4℃、12 000 r/min 离心10 min。
(3)将上清转移到另一新的EP管中,加入等体积的氯仿∶异戊醇(24∶1),涡旋振荡5 min至充分混匀,4℃、12 000 r/min离心10 min。
(4)将上清液转移到另一新的EP管中,随后从上清液中取样并等量分装于4个新的EP管中,每管作不同处理。处理A:上清液中加入1/3体积的8 mol/L LiCl,使其终浓度为2 mol/L,-20℃沉淀RNA 2 h以上,4℃、15 000 r/min离心25 min。处理B:加入1/4体积的5 mol/L NaCl,再加入总体积1倍的-20℃预冷的异丙醇,-20℃放置2 h以上,4℃、15 000 r/min离心25 min。处理C(乙醇沉淀法I):加入1/4体积的5 mol/L NaCl和1/10体积的3 mol/L NaAc(pH5.2),再加入总体积2.5倍-20℃预冷的无水乙醇,-20℃放置2 h 以上,4℃、15 000 r/min离心25 min。处理D(乙醇沉淀法Ⅱ):加入1/10 体积 3 mol/L NaAc(pH5.2),再加入总体积2.5倍-20℃预冷的无水乙醇,-20℃放置2 h,4℃、15 000 r/min离心25 min。
(5)4℃、15 000 r/min离心15 min,弃上清。
(6)用70%乙醇洗涤RNA沉淀3次。
(7)4℃、15 000 r/min离心15 min,弃乙醇。
(8)将RNA沉淀置于超净台内晾干,加入20 μL无菌DEPC·H2O溶解沉淀,-80℃保存备用。
1.2.3 总RNA的质量检测 1.2.3.1 RNA含量与纯度的紫外光吸收法检测取1 μL总RNA溶液,加入DEPC-H2O稀释至200 μL,用紫外分光光度计测定其在230、260、280 nm波长下的紫外吸光值(A),每个样品重复3次,取平均值。计算A260nm/A280nm、A260nm/A230nm值,判定其纯度。并按照下式计算总RNA的得率:

用1×TBE电泳缓冲液配制含0.5 μg/mL溴化乙锭(EB)的12 g/L琼脂糖凝胶,取1-1.5 μg RNA(由原液换算成)与适量的loading buffer混匀后上样,90 V电泳30 min,采用凝胶成像分析系统观察电泳结果并拍照记录。并利用Quantity One软件分析电泳条带的光密度。
1.2.3.3 RNA质量及完整性的RT-PCR检测为了进一步验证RNA的质量,以等摩尔的上述总RNA样品作为反转录的模板,采用RevertAidTM First Strand cDNA Synthesis Kit(#K1621;Fermentas,Lithuania)并按照其操作程序合成第一链cDNA,并以稀释10倍的此1st cDNA为模板。PCR扩增反应采用20 μL反应体系,在该体系中,加入了cDNA 2 μL,2×Taq PCR MasterMix(TaKaRa)10 μL,上游和下游引物各 0.5 μL,无核酸酶的双蒸水7 μL。PCR程序为:94℃预变性5 min;94℃变性30 s,60℃退火30 s,72℃延伸30 s,35个循环;72℃终延伸7 min,4℃保存。检测RNA质量引物根据橡胶树18S rRNA和延伸因子(REF)序列设计,Hb18SF:5'-GGTCGCAAGGCTGAAACT-3',Hb18SR:5'-ACGGGCGGTGTGTACAAA-3';RefF1:5'-GGCTACAGTTTATGCCAGGGCT-3',RefR1:5'-GACGGGTCTAACCACGCTCTTA-3',所有引物由上海英骏生物公司合成。
2 结果 2.1 不同方法沉淀的RNA纯度比较用紫外分光光度法测定不同沉淀方法得到的胶乳总RNA纯度,结果如表 1所示,LiCl沉淀法所得RNA的A260nm/A280nm为2.04-2.16,A260nm/A230nm均大于2.0;异丙醇沉淀法所得RNA的A260nm/A280nm为2.04-2.16,A260nm/A230nm均大于1.9;乙醇沉淀法I所得RNA的A260nm/A280nm为2.05-2.12,A260nm/A230nm均大于1.8;乙醇沉淀法Ⅱ所得RNA的A260nm/A280nm为2.03-2.11,A260nm/A230nm均大于1.8。这表明4种方法所沉淀的终产物含多糖、多酚、DNA和蛋白质等杂质较少,RNA纯度较高。
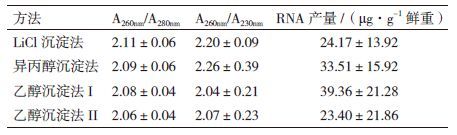
2.2 不同方法沉淀的RNA完整性比较对不同方法沉淀的橡胶树胶乳RNA进行琼脂糖凝胶电泳,4种方法的电泳结果见图 1。前3泳道的28S、18S两个条带均完整清晰,且前者的亮度约是后者的1.26-1.33倍,表明前3种方法提取的RNA降解少,完整性好,基本没有DNA、多糖和蛋白污染;而泳道4出现一条清晰5S条带,且28S的亮度仅为18S的1.02倍,说明泳道中RNA出现降解,其原因可能是乙醇沉淀法Ⅱ提取的RNA中存在RNA水解酶的污染。

|
| 图 1 橡胶树胶乳总RNA的电泳检测结果 A:胶乳总RNA的琼脂糖电泳图(1:LiCl沉淀法;2:异丙醇沉淀法;3:乙醇沉淀法I;4:乙醇沉淀法Ⅱ);B:胶乳总RNA的光密度统计结果 |
由表 1可知,LiCl沉淀法、异丙醇沉淀法、乙醇沉淀法I和乙醇沉淀法Ⅱ沉淀的RNA的平均产量分别为24.17、33.51、39.36、和23.40 μg/g鲜重,但差异显著性检验结果表明4种方法间均无明显差异。结合2.1和2.2分析结果说明,用这4种方法得到的RNA可用于cDNA合成或RT-PCR扩增等分子实验。而且异丙醇沉淀法、乙醇沉淀法I的变异系数分别比乙醇沉淀法Ⅱ的93.40%小45.91%、39.33%,前二者产量高且稳定,还能满足RNA用量大的Northern分析等实验的需要。
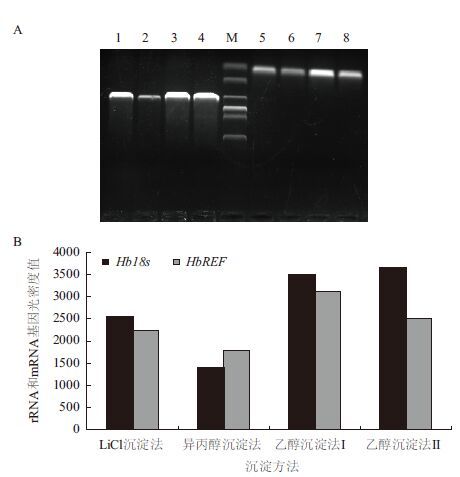
实验利用4种沉淀法成功地从橡胶树新鲜胶乳中提取了RNA,并对不同沉淀方法提取的总RNA分别进行反转录,再以两对基因特异性引物(18S、REF1)分别对4种反转录产物cDNA进行RT-PCR扩增,PCR反应体系中其余成分及其浓度完全一致,PCR循环参数均相等。RT-PCR检测结果(图 2)显示,各方法分别在500、100 bp左右得到一条清晰的RT-PCR扩增条带,特异性强,无杂带出现;扩增产物大小与设计的长度529、124 bp相符,即为所要扩增的目的基因片段。可见,这4种方法提取的RNA均能满足RT-PCR技术对扩增带长度的要求。
|
| 图 2 不同质量的cDNA对PCR效率的影响 A:cDNA质量对PCR效率影响的琼脂糖电泳检测(1、5:LiCl沉淀法;2、6:异丙醇沉淀法;3、7:乙醇沉淀法I;4、8:乙醇沉淀法Ⅱ;M:分子量标准(DL2000)。1、2、3、4:HbREF;5、6、7、8:Hb18S);B:cDNA 质量对PCR效率影响的光密度统计结果 |
图 2还显示,cDNA模板来源对18S、REF扩增结果的影响主要表现在条带丰度随其来源的不同而不同。当扩增18S的模板cDNA为乙醇沉淀法I时呈现出较为清晰条带,并且当模板为乙醇沉淀法Ⅱ时最为清晰,当模板cDNA为LiCl沉淀法、异丙醇沉淀法时,扩增条带逐渐变淡;其中,乙醇沉淀法I的18S表达量分别是LiCl沉淀法、异丙醇沉淀法18S的1.38、2.50倍,乙醇沉淀法Ⅱ的18S的表达量分别是LiCl沉淀法、异丙醇沉淀法18S的1.44、2.61倍。当扩增REF的模板为异丙醇沉淀法时扩增条带很浅,LiCl沉淀法为模板时与异丙醇沉淀法时相比扩增条带略微清晰,乙醇沉淀法I和乙醇沉淀法Ⅱ同LiCl沉淀法相比扩增条带表现出逐渐清晰的趋势;其中,乙醇沉淀法I的REF表达量分别是LiCl沉淀法、异丙醇沉淀法REF的1.40、1.75倍,乙醇沉淀法Ⅱ中REF的表达量分别是LiCl沉淀法、异丙醇沉淀法REF的1.12、1.40倍。这说明乙醇沉淀法I、乙醇沉淀法Ⅱ提取的RNA均能满足RT-PCR技术对扩增带丰度的要求。
3 讨论橡胶树胶乳组织中含有大量的多糖、蛋白类物质,严重干扰完整RNA的提取,进而影响下游的实验操作。去除多糖的方法很多,除通常的低浓度乙醇沉淀法[10]和醋酸钾沉淀法[11]外,还可通过提高缓冲液中NaCl浓度也有助于去除多糖[12]。本实验主要使用了含有Tris-HCI、LiCl、EDTA-Na2的SDS提取液,并通过在RNA沉淀时结合使用乙醇+醋酸钠、乙醇+氯化钠+醋酸钠,对SDS法进行改良,最后去除多糖的效果均较好。
在沉淀方法上,本研究采用了4种方法沉淀橡胶树胶乳RNA,这几种方法都能获得18S、28S带型清晰、完整性好、纯度和产率高的总RNA,完全适用于后续的分子生物学研究。但是在沉淀质量、产量、成本等方面,不同方法是有差别的。例如,从提取RNA的电泳图、吸光度比值以及逆转录后内参基因18S和目标基因REF的qRT-PCR检测值,可以明显地看出乙醇结合NaCl和NaAc沉淀法与LiCl沉淀法、NaCl +异丙醇沉淀法以及NaAc +乙醇沉淀法沉淀的结果相比,其RNA的完整性、得率和基因扩增效果的优势均明显。尽管用LiCl沉淀的RNA完整性比NaCl和异丙醇,NaAc和乙醇沉淀的RNA完整性更好,但用NaCl和异丙醇沉淀的RNA产量比LiCl,NaAc和乙醇沉淀的RNA产量高,其中LiCl沉淀时导致RNA产率低的可能原因是LiCl专门沉淀溶液中的高分子量RNA。而且,使用NaCl +异丙醇、NaCl + NaAc +乙醇、NaAc +乙醇沉淀RNA比用LiCl沉淀RNA所需要的成本低。综上所述,在4种RNA沉淀方法中,以NaCl + NaAc +乙醇沉淀法最优。笔者应用该方法还成功地提取了杜仲、橡胶草的胶乳RNA,说明该方法可以适用于大多数产胶植物胶乳的提取,这对胶乳RNA为基础的分子生物学研究具有重要的现实意义。此外,用乙醇结合两种高浓度单价盐沉淀的方法富集RNA,既提高了总RNA的浓度,同时也相应地提高了miRNA的浓度,进而为miRNA后续实验奠定了基础。
据文献报道,用于SDS法提取橡胶树胶乳总RNA的沉淀方法主要有LiCl沉淀法和乙醇沉淀法[13]。也有研究者将这两种沉淀法同步或分步结合使用,如黄贵修等[14]先使用终浓度为0.3 mol/L的LiCl和2.5倍体积冰乙醇/等体积异丙醇-20℃下沉淀RNA 30 min,随后进行离心和去上清,并对沉淀作溶解处理;当沉淀完全溶解时,再使用终浓度为2 mol/L的LiCl冰浴下沉淀1 h,所获橡胶树胶乳总RNA的纯度、完整性和产量均较高,可以满足Northern杂交实验的要求。曾日中等[4]先使用终浓度为2 mol/L的LiCl -20℃条件下沉淀RNA过夜,然后进行离心和去上清,并对沉淀作溶解处理,当沉淀溶解充分时,再使用终浓度为2 mol/L的LiCl -20℃下沉淀2 h,随后离心,弃上清,并充分溶解沉淀,最后使用终浓度为0.3 mol/L的NaAc和2.5倍体积的无水乙醇-20℃沉淀l h,实验获得质量较好的胶乳总RNA,可以满足cDNA消减文库构建的要求。同时也发现,上述RNA沉淀剂LiCl和乙醇的两种结合方法,其环节多,耗时长,成本高。张治礼等[15]采用2倍体积无水乙醇沉淀RNA 10 min,所得RNA完整性较好,纯度和得率较高,可以满足RT-PCR和RACE实验的要求,但泳道中出现一条十分明显的5S条带,其亮度显著超过28S和18S的值。由此可见,该法更有利于小分子量RNA沉淀。而本研究的NaCl+NaAc+乙醇沉淀法则弥补了传统沉淀法步骤较繁琐和小分子量RNA含量高等不足。
4 结论通过对RNA提取的沉淀步骤研究,发现沉淀剂的选择对RNA提取量与品质的影响较大,采用NaCl、NaAc和无水乙醇配合沉淀RNA,能明显提高RNA纯度和产率,完全满足相应的分子实验要求,还节省成本和时间。由此,改良的SDS酸酚法更适合橡胶树胶乳总RNA提取。
| [1] | Ko JH, Chow KS, Han KH. Transcriptome analysis reveals novel features of the molecular events occurring in the laticifers of Hevea brasiliensis(para rubber tree). Plant Mol Biol , 2003, 53 (4) : 479–492. DOI:10.1023/B:PLAN.0000019119.66643.5d |
| [2] | Deng LH, Luo MW, Zhang CF, et al. Extraction of high-quality RNA from rubber tree leaves. Biosci Biotechnol Biochem , 2012, 76 (7) : 1394–1396. DOI:10.1271/bbb.120014 |
| [3] | Bokma E, Spiering M, Chow KS, et al. Determination of cDNA and genomic DNA sequences of hevamine, a chitinase from the rubber tree Hevea brasiliensis. Plant Physiol Bioch , 2001, 39 (5) : 367–376. DOI:10.1016/S0981-9428(01)01247-5 |
| [4] | 曾日中, 段翠芳, 黎瑜, 等. 茉莉酸刺激的橡胶树胶乳cDNA消减文库的构建及其序列分析. 热带作物学报 , 2003, 24 (3) : 1–6. |
| [5] | Tang C, Qi J, Li H, et al. A convenient and efficient protocol for isolating high-quality RNA from latex of Hevea brasiliensis(para rubber tree). J Biochem Bioph Meth , 2007, 70 (5) : 749–754. DOI:10.1016/j.jbbm.2007.04.002 |
| [6] | Xu J, Aileni M, Abbagani S, et al. A reliable and efficient method for total RNA isolation from various members of spurge family(Euphorbiaceae). Phytochem Anal , 2010, 21 (5) : 395–398. DOI:10.1002/pca.1205 |
| [7] | Spanò D, Pintus F, Mascia C, et al. Extraction and characterization of a natural rubber from Euphorbia characias latex. Biopolymers , 2012, 97 (8) : 589–594. DOI:10.1002/bip.22044 |
| [8] | 代龙军, 项秋兰, 黎瑜, 等. 巴西橡胶树橡胶粒子蛋白质的16-BAC/SDS-PAGE双向电泳及质谱分析. 中国农业科学 , 2012, 45 (11) : 2328–2338. |
| [9] | Dai L, Kang G, Li Y, et al. In-depth proteome analysis of the rubber particle of Hevea brasiliensis(para rubber tree). Plant Mol Biol , 2013, 82 (1) : 155–168. |
| [10] | Lewinsohn E, Steele CL, Croteau R. Simple isolation of functional RNA from woody stems of gymnosperms. Plant Mol Biol Rep , 1994, 12 (1) : 20–25. DOI:10.1007/BF02668660 |
| [11] | Ainsworth C. Isolation of RNA from floral tissue of Rumex acetosa(Sorre1). Plant Mol Biol Rep , 1994, 12 (3) : 198–203. DOI:10.1007/BF02668741 |
| [12] | Fang G, Hammar S, Grumet R. A quick and inexpensive method f or removing polysaccharides from plant genomic DNA. Biotechniques , 1992, 13 : 52–56. |
| [13] | 庄盈婷, 仝征, 王力敏, 等. 橡胶树胶乳 RNA 简易提取和保存方法的比较探索. 热带作物学报 , 2013, 34 (10) : 1907–1913. |
| [14] | 黄贵修, 吴坤鑫, 陈守才. 一种改良的橡胶树胶乳总RNA提取方法. 华南热带农业大学学报 , 2002, 8 (4) : 1–4. |
| [15] | 张治礼, 杨云, 刘宽灿, 等. 一种快速?高效的橡胶树胶乳总RNA提取方法. 植物学通报 , 2007, 24 (4) : 516–520. |