水稻(Oryza sativa)作为重要粮食作物,养活全球一半左右人口,特别是在东亚国家,有超过2/3人口以稻米为主食,但水稻稳定生产与增产一直受到多种病害威胁。在水稻各个生育期,大量病原微生物,包括真菌、细菌,病毒和线虫对水稻不同器官的侵染,是造成水稻严重减产甚至绝收的主要原因。农药的广泛与大量使用在一定程度上减缓了各种水稻病害的发生与危害程度,但同时也严重影响到人类身体健康与生态环境安全。因此,培育与种植水稻病害高抗品种是目前最为经济有效、安全健康与环境友好的水稻病害育种策略。在过去20年间,全世界范围内的科学家们在水稻抗病分子机制研究方面取得了重要进展,包括水稻抗病及其抗病相关蛋白对稻瘟病菌、水稻白叶枯病菌等病原物效应分子识别、病原物效应蛋白对水稻抗病防卫反应抑制、植物激素在水稻免疫反应中的功能以及表观遗传修饰在水稻免疫反应中的作用。基于对上述基础理论知识的认识,育种学家利用目前先进生物技术与常规育种技术相结合的方法开发全新育种策略。
1 水稻主要病害根据科学意义与经济效益层面进行分析,对水稻可以产生重要影响的病害包括:由子囊菌(Magnaporthe oryzae)引起的稻瘟病;由黄单胞杆菌水稻变种(Xanthomonas oryzae pv. oryzae Xoo)引起的水稻白叶枯病;由拟禾本科根结线虫(Meloidogyne graminicola)引起的水稻根结线虫病;由叶芽线虫(Aphelenchoides besseyi)引起的水稻干尖线虫病;由茎线虫(Ditylenchus angustus)引起的水稻茎秆线虫病[4]。近些年来,由于气候与水稻种植方式的改变以及高产但抗病性欠佳水稻品种大面积推广,水稻纹枯病、水稻稻曲病、水稻条纹叶枯病等水稻病害发生频率逐年增加,发生区域与面积也有扩大趋势。在过去的二十年间,由于研究基础与方法便利,水稻-稻瘟病菌与水稻-白叶枯病菌互作关系已被作为植物-微生物互作模式系统进行深入阐释,特别是在水稻抗稻瘟病、白叶枯病的分子机制及稻瘟病菌致病机理等方面取得了重要进展。
2 植物免疫反应分子机制研究表明植物主要通过两种不同方式抵抗病原物入侵[1]:第一种,由位于植物细胞膜上的模式识别受体(pattern-recognition receptors,PRRs)识别病原物分泌的病原相关分子模式(pathogen-associated molecular patterns,PAMPs)而诱导的植物抗性反应,被称为PAMP诱导免疫反应(PAMP-triggered immunity,PTI)。为了克服植物PTI反应,真菌、细菌、病毒、线虫等病原物进化出能够抑制植物PTI的效应蛋白(effector)进而导致效应蛋白诱导的感病反应(effector-triggered susceptibility,ETS)。为了克服病原物ETS,植物进化出第二种防御机制,即通过编码抗性蛋白识别病原物效应蛋白,从而诱导快速而激烈的抗性反应,称为效应蛋白诱导的免疫反应(effector-triggered immunity,ETI)(图 1)。然而,目前研究证明上述两种植物抗病模式并不能完全解释植物对病毒的抗性机制[2]。虽然植物对病毒的抗性机制与对细菌/真菌的抗性机制存在很大不同,但近些年一些研究证明,植物RNA干扰(RNAi silencing,RNAi)机制对病毒双链RNA的识别功能可能与PTI存在类似机制,进而抑制病毒入侵[2, 3]。病毒为了克服植物的这一抗性机制,已进化出具有抑制植物RNAi的抑制子(viral suppressors of RNAi,VSRs)克服植物RNAi反应,这一过程类似于ETS;当VSRs被植物体内相应抗性蛋白识别后,诱导激烈的抗性反应则类似于ETI。
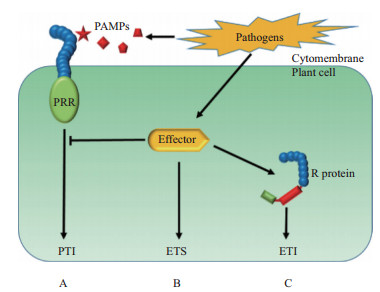
|
| 图 1 植物免疫反应原理图 A :由位于植物细胞膜上的PRRs识别病原物分泌的PAMPs而诱导的植物抗性反应,被称为PTI;B:为了克服植物PTI反应,病原物进化出能够抑制植物PTI的效应蛋白(effector)进而导致效应蛋白诱导的感病反应,被称为ETS;C :为了克服病原物诱导的ETS,植物进化出第二种防御机制,即通过编码抗性蛋白(R protein)识别病原物效应蛋白,从而诱导快速而激烈的抗性反应,称为ETI |
植物PRR蛋白结构包括:含有典型胞外富含亮氨酸重复序列与胞内激酶结构域的类跨膜受体激酶(receptor-like kinases,RLKs)和缺少激酶结构域的类受体蛋白(receptor-like proteins,RLPs)[5]。由于RLP缺少胞内激酶结构域,因此该类蛋白可与其他含有激酶结构域蛋白互作诱导下游抗病信号传导。水稻基因组编码1 131个RLK基因与90个RLP基因,其中,水稻RLK基因是拟南芥中RLK基因数目的2倍。通过进化分析发现水稻中RLK基因如此之多,可能是通过重组复制而来[6, 7]。PRR蛋白可识别并响应外来病原物以及植物内源信号刺激,如脂类、蛋白、核酸和糖类等。目前,大量科学研究证明PRR蛋白可以特异识别病原物PAMPs,如细菌鞭毛蛋白、肽聚糖类、脂多糖类、真菌几丁质等,进而诱导植物的抗病免疫反应[8, 9]。
目前研究较为清楚的PRR蛋白为来自拟南芥的鞭毛蛋白受体激酶(receptor kinase flagellin sensing 2,FLS2)。该蛋白通过特异识别细菌鞭毛蛋白N端一段含22个氨基酸残基的保守肽段(flagellin pep-tide,flg22),激活植物抗病性信号传导复合体[10, 11]。在水稻中,FLS2同源蛋白OsFLS2可通过识别flg22诱导水稻抗病性反应。OsFLS2可与flg22直接识别,并能互补fls2突变体缺陷表型[12, 13]。另外OsFLS2可识别不能被FLS2识别的flg22衍生物[13],表明该类蛋白特异性相互识别在不同物种之间存在很大区别。另外,OsFLS2可通过胞质结构域与OsRac1GEF相互作用,OsRac1GEF对PTI重要参与因子OsRac1有调控作用[14]。另外,OsRac1GEF与识别几丁质的OsCERK1有互作关系,说明鞭毛蛋白与几丁质诱导的植物抗病信号传导通路有一定重合。
水稻RLK基因Xa21是第一个被鉴定到的天然免疫受体基因,并对白叶枯病菌具有广谱抗性[15]。目前,已经利用遗传学与生物化学等方法对XA21介导的抗病信号网络进行了较为深入的研究,鉴定到包括腺苷三磷酸酶(XB24)、E3链接酶(XB3)、PP2C磷酸酶(XB15)、转录因子WRKY62(XB10)、锚定蛋白(XB25)在内的5个XA21结合蛋白(XA21-binding proteins,XBs),这些蛋白在植物天然免疫过程中起重要调控作用[16-20]。XB24通过结合与催化XA21蛋白的丝氨酸与苏氨酸残基磷酸化,保持XA21处于非激活状态。但当XB24/XA21复合体感知到病原物入侵时,XB24与XA21会彼此脱离,XA21进而被激活,进一步诱导下游大量抗病性信号传导,如XA21激酶结构域切割与转运致细胞核[17, 21]。XB15可以通过对XA21的磷酸化,而减弱XA21介导的免疫反应[18]。研究证明,XB25的N端跨膜与正电结构域可与XA21跨膜重复结构域相互作用[17]。下调表达XB25蛋白后,XA21表达量也受到下调,并抑制XA21介导抗性反应。XBs类蛋白结构与功能多样性预示XA21介导水稻抗病信号通路是一个极其复杂的过程。最近有研究证明,定位于内质网的多种分子伴侣蛋白对XA21介导的抗性反应是必须的,初步猜想可能是这些分子伴侣对XA21的折叠与加工有帮助[22]。综上所述,水稻编码大量与多样性蛋白参与到XA21蛋白激活与XA21蛋白介导的水稻免疫反应过程中。
4 水稻抗病蛋白(R蛋白)与ETI研究表明,核酸结合与富亮氨酸重复结构域(NLR)蛋白在植物与动物中均参与调控免疫反应过程[23]。植物基因组含有的NLR基因数量大大多于动物NLR基因,说明NLR类基因介导免疫反应在不同物种中可能存在差别。如,水稻基因组含有480个编码NLR蛋白的基因,而人类基因组中则只含有10个[24]。迄今为止,在水稻中共鉴定到超过100个R基因,并已成功分离了26个稻瘟病菌R基因与9个白叶枯病菌R基因(表 1)。在这些被克隆的R基因中,绝大多数R基因含有NLR结构域[25]。其中不含有NLR结构域的基因有:编码RLK结构域的Pi-d2蛋白[26]、隐性R基因Pi21编码富含脯氨酸的蛋白[27]。有意思的是,NLR基因在植物基因组中往往成簇分布于染色体上,如水稻中的480个NLR基因中的263个NLR基因集中分布于44个基因簇[24]。水稻抗稻瘟病基因Pi2、Pi9和Piz-t是位于6号染色体同一基因簇的3个NLR基因,序列比对分析发现,在野生稻与栽培稻中这一基因簇至少含有8个NLR基因[28]。最近被克隆并对稻瘟病菌具有广谱抗性的R基因Pi50也位于Pi2/Pi9基因簇[29]。同源分析发现Pi50蛋白与Pi2、Pi9、Piz-t蛋白具有超过96%的序列相似性,说明Pi50可能是起源于基因旁系同源复制而导致差异性功能的R基因[30]。该位点的另一等位基因Pi64编码的蛋白由1 288个氨基酸残基组成,该蛋白同时定位于胞质与细胞核。基因表达分析表明,该蛋白在水稻所有组织器官中为组成型表达,并对水稻叶瘟与穗颈瘟有较高抗性[31]。
研究表明,Xa10与Xa23基因编码两个TALE(transcription activator-like effector nucleases)依赖型的水稻白叶枯R蛋白[60, 61]。XA10蛋白以六聚体形式定位于内质网,并以诱导蛋白通过调控内质网与胞内钙离子平衡等保守机制诱导细胞程序性死亡。通过蛋白结构预测软件分析发现XA23与XA10蛋白同源性高达50%,二者具有相似跨膜结构域。研究证明,TALE类病原菌分泌蛋白AvrXa23可以特激活Xa23表达,在水稻、烟草与番茄中,高表达的Xa23可以诱导激烈的免疫反应。另外,XA23与XA10启动子区域含有的TALE结合原件对TALE诱导的植物免疫反应是必须的。上述研究结果证明,水稻基因组进化出的R基因家族成员是通过识别白叶枯病菌分泌的TALE类蛋白来调控水稻抗病反应。
植物R基因与病原物无毒基因(Avr基因)相互作用符合经典基因对基因假说:病原物分泌的无毒蛋白被相应植物抗性蛋白识别后,进而诱导与激活下游植物抗性反应。目前通过对植物与病原物互作的深入研究,已经大大丰富了基因对基因假说。研究表明,植物抗性蛋白与病原物无毒蛋白相互作用可分为两类,第一类为直接互作,第二类为间接互作。其中,水稻抗稻瘟病菌R基因编码蛋白Pita与AvrPita的互作关系为直接互作,其他决大多数R基因与Avr基因均属于间接互作关系。Pita与AvrPita为第一对被报道的存在直接互作关系的蛋白。当把Pita蛋白LRR结构域中一个氨基酸突变后,二者互作关系消失,说明LRR结构域对Pita与AvrPita的互作识别是必须的。到目前为止,虽然还没有数据证明AvrPiz-t与Piz-t存在直接的互作关系,但是,当把AvrPiz-t异源表达于含有Piz-t的水稻中时,会诱发强烈的HR反应,说明AvrPiz-t与Piz-t在水稻内可能存在某种特异识别方式[62]。
5 病原菌效应蛋白与寄主识别因子病原物效应子在广义上可定义为能够改变寄主细胞结构与功能的病原物分泌蛋白或小分子物质[63]。病原菌分泌的无毒效应蛋白可被寄主相应R蛋白直接或间接识别,进而激发下游快速而剧烈的过敏性反应。迄今为止,在水稻病原菌中,共克隆与分离到21个无毒效应子,其中,有13个效应子来自于稻瘟病菌,7个来自于白叶枯病菌,另外一个来自于水稻细菌条斑病菌[4]。无毒效应子的鉴定为更进一步深入研究无毒效应子与R蛋白的互作关系与分子基础提供了有利条件。AvrPib与AvrPi9为最近从稻瘟病菌中分离克隆到的两个无毒效应子,其中AvrPib基因编码含有77个氨基酸蛋白,目前尚未发现其同源蛋白[30]。通过对来自全球5个地理区域的60个稻瘟病菌表型与基因型进行分析发现,AvrPib的进化没有受到寄主选择的影响。对108个生理小种进行AvrPib等位重测序分析证明,转座元件的非特异性插入是导致该基因毒性丧失的主要因素。水稻广谱主效抗稻瘟病基因Pi9对应的无毒基因AvrPi9最近已经通过基因组比较方法被成功克隆。研究证明,AvrPi9在稻瘟病菌侵染早期阶段具有较高水平表达,并随着侵染的进行,AvrPi9蛋白转移至水稻细胞中。与AvrPib同样,转坐元件对AvrPi9毒性的获得与丧失具有重要作用[64]。
当稻瘟病菌在适宜条件下接触水稻后,会分泌大量效应子进入水稻细胞,并可进一步转移至相邻细胞中与寄主蛋白相互作用,为稻瘟病菌进一步侵染与扩散做准备。最近研究发现大量病原菌无毒效应子的寄主内靶标蛋白对调控寄主抗性具有重要作用。如AvrPiz-t与E3泛素连接酶蛋白APIP6相互作用,并抑制水稻PTI。有趣的是,AvrPiz-t与APIP6互作后,会导致后者降解。APIP6的RNAi转基因水稻的PTI受到明显抑制,且稻瘟病菌抗性也同样受到抑制,说明APIP6对水稻免疫反应起正调控作用[62]。最近研究发现,AvrPiz-t与另一个E3类蛋白APIP10相互作用,并促进后者的降解,有趣的是,APIP10可促进AvrPiz-t泛素化水平,进而引起AvrPiz-t降解。在Piz-t背景下下调APIP10表达后,可引起水稻细胞死亡表型,同时,Piz-t表达量明显上调,进而引起稻瘟病菌抗性增强,说明APIP10对水稻免疫反应起负调控作用[65]。
6 激素在水稻抗病反应中的作用在植物免疫反应过程中,水杨酸(salicylic acid,SA)、茉莉酸(jasmonic acid,JA)与乙烯(ethylene,ET)等植物激素作为信号传递因子对植物免疫反应具有重要调控作用[66]。在过去数十年间,植物生长相关激素如植物生长素、赤霉素、油菜素内酯与脱落酸等均参与调控植物免疫反应,对维持植物生长与免疫反应动态平衡起到重要作用[67]。大量研究证明,提高植物抗病性突变体材料往往具有生长受到抑制的表型。同样,对植物生长与发育具有重要调控作用的基因突变后,往往会改变植物对病原物的抗性。因此,植物抗病性的激活与植物正常生长发育可能存在一定拮抗关系。水稻通常会在叶与嫩芽中含有较高含量的SA,而在根与悬浮培养的细胞中含量较低[68]。有趣的是,假单胞菌、稻瘟病菌与水稻纹枯病菌侵染过的水稻体内并不积累过高水平SA[69]。但是,水稻可以响应外源SA处理,包括诱导水杨酸葡糖基转移酶表达以及过氧化氢积累,说明SA对氧化还原反应具有重要调控作用[70]。在过去十几年中,科学实验证明大量SA信号通路因子参与植物免疫反应,说明SA信号通路对水稻免疫反应具有重要调控作用。通过反向遗传学筛选,发现SA信号通路因子编码类肉桂酰辅酶A还原酶的基因SNL6对水稻病原相关蛋白表达与依赖于OsNPR1的白叶枯抗性是必须的[71]。水稻SNL6突变体内的木质素含量会大量积累,但木质素积累与OsNPR1介导植物抗病性之间的关系还需要进一步深入研究。WRKY类转录因子OsWRKY45可以被SA及其衍生物苯并噻二唑(benzothiadiazole,BTH)所诱导。研究证明,超表达OsWRKY45转基因水稻可以提高对稻瘟病抗性,而该基因RNAi转基因水稻则减弱了BTH诱导的稻瘟病抗性。有意思的是OsWRKY45在植物抗病性功能并不依赖于OsNPR1[72]。
茉莉酸(JA)及其衍生物茉莉酸甲酯(methyl jasmonate,MeJA)是一类脂肪酸衍生物,研究结果表明,JA及其衍生物对植物生长发育具有重要意义。另外,当植物处于机械伤害及死体营养病原物侵害等非生物与生物胁迫过程中,JA及其衍生物起重要调控作用[71]。研究表明,水稻OsPR1a、OsPR1b、OsPR2等PR基因受JA诱导表达,另外,外源JA可以提高水稻对稻瘟病抗性[66]。研究证明,在水稻中超表达JA合成相关基因OsAOS2可以上调PR基因表达与对稻瘟病菌抗性[73]。最近研究表明稻瘟病菌编码的一种抗生素合成单氧酶(Abm)可促进水稻内源JA转化为12OH-JA,进而减弱稻瘟病菌侵染过程中水稻免疫反应[74]。上述结果证明,JA在水稻对真菌病害免疫反应过程中起重要作用。
乙烯(ET)作为重要植物激素,参与调控种子萌发、幼苗生长、器官发育、果实成熟、器官衰老及脱离等植物生长发育过程。研究证明ET也参与植物逆境调控反应,如盐胁迫、冷胁迫、水淹及病原微生物侵染等[75]。研究表明,由于病原物与环境条件不同,ET可以作为正、负调控因子对植物免疫反应进行调控[76, 77]。研究表明,水淹与无氧条件下所诱导的ET合成对水稻田间稻瘟病菌抗性非常重要,另外,外源ET合成抑制剂与ET合成促进剂可以下调与上调水稻对稻瘟病菌的抗性[78]。研究表明,ET含量的上升是伴随着HR反应发生,并可以诱导抗病相关基因(PR)表达。当用ET合成抑制剂处理后,ET的释放受到抑制,同时感病反应更加强烈[79]。超表达ET合成相关基因ACS2的转基因水稻对稻病菌与白叶枯病菌表现出明显抗性增强反应,令人兴奋的是,超表达ACS2的转基因水稻生长表型与产量并末受到影响[80]。上述结果证明,ET在植物对真菌病害的基础抗性过程中起重要作用。
赤霉素(gibberellin,GAs)是一种广泛存在的植物激素,最初来源于赤霉菌,并可以引起水稻恶苗病。然而该激素的重要性及其调控植物免疫反应分子机制近些年才得到深入研究。水稻黑条矮缩病毒侵染后,水稻表现明显矮化与深绿叶表型,这种表型与GAs缺陷突变体表型极为相似。研究证明,外源GAs可以减缓由水稻黑条矮缩病毒引起的水稻矮化与深绿叶表型[81]。最近研究表明,GAs合成相关基因GA20ox超表达株系内GAs含量明显提高,并显著降低对稻瘟病抗性。GA20ox RNAi转基因水稻可增强对稻瘟病菌与白叶枯病菌抗性[82]。上述结果证明GAs不仅在调控水稻株高方面起重要作用,在水稻免疫过程中同样起重要作用。
油菜素内酯(brassinosteroids,BR)在调节植物生长发育与多种生理过程中起重要作用,如细胞伸长、维管组织分化、根伸长、光反应、抗逆反应及早衰等[83, 84]。BR受体BRI1是一个定位质膜,编码富含亮氨酸重复类激酶(leucine-rich repeat receptor-like kinase,RLK)的蛋白[85]。研究证明BR与BRI1结合后,可以促进磷酸基团在BRI1与BAK1间转移[86]。有趣的是,BR与BRI1结合,可激活BRI1-XA21嵌合体受体激酶诱导的XA21介导抗病反应[87]。最近研究表明,外源BR合成抑制剂可增强植物对白叶枯病菌抗性。而BR缺陷型突变体同样可以增强植物对白叶枯病菌抗性,并且BR介导的抗病过程是通过SA与GA通路实现的[88]。
7 表观遗传修饰调控水稻抗病反应组蛋白修饰与染色质重塑等表观遗传调控过程在动物与植物抵抗病原物侵染过程中起重要作用[89, 90]。组蛋白乙酰化与去乙酰化是一种可调控相关基因差异表达的组蛋白修饰方式之一。组蛋白乙酰转移酶(histone acetyltransferases,HAT)与组蛋白脱乙酰酶(histone deacetylases,HDAC)分别控制组蛋白乙酰化与去乙酰化。目前,已有报道证明HDAC类蛋白可通过Toll-like receptor(TLR)通路与Interferon(FIN)信号通路调控动物免疫反应。HDT701是水稻中HDAC类蛋白家族成员之一,并为植物所特有,作为负调控因子调节水稻免疫反应。当用亲合性稻瘟病菌生理小种接种后,HDT701表达量明显上调,然而不亲合小种接种水稻后,其表达量受到抑制。HDT701超表达转基因水稻表现出对稻瘟病菌与白叶枯病菌更严重感病表型与组蛋白H4乙酰化水平下降现象,同时,HDT701 RNAi转基因水稻增强对上述两种病原菌抗性与上调组蛋白H4乙酰化水平[91]。
8 生物技术在水稻抗病育种中的应用据统计,由于人口快速增长,到2030年全球粮食增产要达到150%才能满足人类需求[92]。由于各种条件限制,如栽培技术,高产品种培育遇到更大瓶颈及大量病虫害发生与流行严重等限制了全球粮食作物稳产与增产。在过去的20年间,在中国与非洲国家,作物抗病性育种取得了巨大进步。例如,大量作物病害主效抗性基因克隆与分子标记辅助育种(marker-assisted selection,MAS)应用,大大加快了优良抗性品种培育效率[92-94]。随着新技术的开发与应用,大量新技术与方法已经广泛被应用于水稻抗病育种工作中,如全基因组关联分析(genome wide association studies,GWAS)技术,TALEN(tra-nscription activator-like effector nucleases),CRISPR(clustered regularly interspaced short palindromic repe-ats /CRISPR-associated 9,CRISPR/Cas9)技术以及宿主诱导的基因沉默(host induced gene silence,HI-GS)技术等。最近GWAS技术在水稻、玉米等多种作物中已成功应用于控制数量性状基因的定位[95, 96]。最近研究发现,利用来自5个国家的5个稻瘟病菌生理小种对414份来自世界多地区水稻材料进行GWAS分析,共鉴定到66个稻瘟病候选抗性位点,其中53个位点为初次报道[97]。这表明,GWAS分析与传统定位方法有一定的重合性,但基于其大规模性与大数据量的优势,GWAS可为水稻抗病育种提供更多候选资源。最近几年发展起来的基因编辑技术在水稻抗病育种方面具有巨大应用潜能。TALEN是一种利用病原物分泌的TAL效应子识别目标基因特异序列,通过与核酸内切酶结合并对目标序列进行切割的一种基因编辑技术。目前利用TALEN技术,已经得到对Xanthomonas spp.具有良好抗性转基因水稻[98]。新技术CRISPR/Cas9基因编辑技术的应用,使基因定点编辑变得更加方便与快捷[99]。到目前为止,CRISPR基因编辑技术已在小鼠、斑马鱼、果蝇、线虫、拟南芥、玉米及水稻等多个物种中成功应用。利用CRISPR技术对六倍体小麦3个Mlo基因进行定点编辑,得到了抗性持久,稳定遗传的六倍体小麦株系[100]。最新研究报告称来自于Argonaute蛋白(NgAgo)家族的核酸内切酶可以利用DNA作为向导降解入侵DNA。NgAgo蛋白在向导DNA的引导下,能够在人细胞中进行特定基因定点编辑[101]。由于Cas9必须依赖于目标位点位于PAM上游,才可能发挥切割作用,而NgAgo编辑技术则没有这一限制,因此,NgAgo编辑技术比CRISPR编辑技术在作物抗病性方面的遗传改良与培育可能拥有更为广泛应用前景,但这一技术的重复性及效率目前还存在争议,需要进一步的研究证实。HIGS技术是最近几年发展起来的一种利用RNAi原理抑制病原菌侵染的技术。第一个成功应用HIGS技术进行抗性研究结果证明,当在植物内表达PRSV(papaya ringspot virus,PRSV)外壳蛋白可有效抑制PRSV侵染[102]。目前有研究证明,在植物体内表达特异识别病原物重要基因的双链RNA时,可以有效提高植物自身对病原物抗性,并且在大麦与小麦抗白粉病育种中成功应用[103, 104]。
9 结论与展望在过去20年间,科学界在水稻先天免疫分子机制方面取得了巨大进步。迄今为止,有超过40个PRR与R基因被成功克隆,这些基因在抵抗真菌、细菌及病毒入侵过程起重要作用[59]。这些基因也为培育水稻高抗品种提供了重要理论基础与遗传材料,如邹德堂等[105]利用显性标记pB8对黑龙江省96份水稻主栽品种进行Pi9基因筛选,发现早熟青森、垦粳1号等44个品种含有Pi9基因,苗期和分蘖期平均发病级别均为2.4级;青森5号、长白9号等52个品种不含Pi9基因,苗期和分蘖期平均发病级别分别为5.1和5.4级,研究结果可为水稻品种改良以及抗病资源合理利用奠定基础;Pi35是最近被克隆的一个稻瘟病抗性基因,是Pish的等位变异基因[48]。携带Pi35基因的日本粳稻品种北海188(Hokkai 188)和藤系138(Fukei 138)自育成30多年来一直保持稳定的高水平叶瘟抗性;其中,藤系138在20世纪80年代中期被引入黑龙江、吉林等省,并以之作为抗性亲本培育了一批推广品种[106]。另外,对水稻先天免疫受体因子的鉴定及其识别真菌、细菌及病毒分子机制研究、受体复合体的结构分析为水稻抗病性研究与高抗品种的培育提供了新思路与理论基础。以下几个方面还需进一步深入研究:(1)PRR与NLR相互识别所介导的植物抗病性机制;(2)信号传递因子如何识别PRR与NLR互作信号及调控下游PR基因表达;(3)表观遗传修饰参与调控水稻先天免疫机制;(4)如何将目前水稻先天免疫基础理论与抗病育种实际更加有机结合,以提高抗病品种培育效率,缩短新品种选育时间。
| [1] | Boller T, He C. Innate immunity in plants:An arms race between pattern recognition receptors in plants and effectors in microbial pathogens. Science , 2009, 324 (5928) : 742–744. DOI:10.1126/science.1171647 |
| [2] | Nakahara K, Masuta C. Interaction between viral RNA silencing suppressors and host factors in plant immunity. Current Opinion in Plant Biology , 2014, 20 : 88–95. DOI:10.1016/j.pbi.2014.05.004 |
| [3] | Zvereva A, Pooggin M. Silencing and innate immunity in plant defense against viral and non-viral pathogens. Viruses , 2012, 4 (11) : 2578–2597. |
| [4] | Liu W, Wang G. Plant innate immunity in rice:a defense against pathogen infection. National Science Review , 2016 . |
| [5] | Zipfel C. Pattern-recognition receptors in plant innate immunity. Current Opinion in Immunology , 2008, 20 (1) : 10–16. DOI:10.1016/j.coi.2007.11.003 |
| [6] | Shiu S, Karlowski W, Pan R, et al. Comparative analysis of the receptor-like kinase family in Arabidopsis and rice. Plant Cell , 2004, 16 (5) : 1220–1234. DOI:10.1105/tpc.020834 |
| [7] | Fritz-Laylin L, Jones J. Phylogenomic analysis of the receptor-like proteins of rice and Arabidopsis 1. Plant Physiology , 2005, 138 (2) : 611–623. DOI:10.1104/pp.104.054452 |
| [8] | Chen X, Ronald P. Innate immunity in rice. Trends in Plant Science , 2011, 16 (8) : 451–459. DOI:10.1016/j.tplants.2011.04.003 |
| [9] | Liu B, Li J, Ao Y, et al. Lysin motif-containing proteins LYP4 and LYP6 play dual roles in peptidoglycan and chitin perception in rice innate immunity. Plant Cell , 2012, 24 (8) : 3406–3419. DOI:10.1105/tpc.112.102475 |
| [10] | Delphine C, Martin R, Thomas B, et al. The Arabidopsis receptor kinase fls2 binds flg22 and determines the specificity of flagellin perception. Plant Cell , 2006, 18 (2) : 465–476. DOI:10.1105/tpc.105.036574 |
| [11] | Zipfel C, Robatzek S, Navarro L, et al. Bacterial disease resistance in Arabidopsis through flagellin perception. Nature , 2004, 428 (6984) : 764–767. DOI:10.1038/nature02485 |
| [12] | Shinya T, Osada T, Desaki Y, et al. Characterization of receptor proteins using affinity cross-linking with biotinylated ligands. Plant & Cell Physiology , 2010, 51 (2) : 262–270. |
| [13] | Takai R, Isogai A, Takayama S, et al. Analysis of flagellin perception mediated by flg22 receptor OsFLS2 in rice. Molecular Plant-Microbe Interactions , 2009, 21 (12) : 1635–1642. |
| [14] | Akimoto K, Katakami H, Kim H, et al. Epigenetic inheritance in rice plants. Annals of Botany , 2007, 100 (2) : 205–217. DOI:10.1093/aob/mcm110 |
| [15] | Song W, Wang G, Chen L, et al. A receptor kinase-like protein encoded by the rice disease resistance gene, Xa21. Science , 1995, 270 (5243) : 1804–1806. DOI:10.1126/science.270.5243.1804 |
| [16] | Chen X, Chern M, Canlas P, et al. An ATPase promotes autophosphorylation of the pattern recognition receptor XA21 and inhibits XA21-mediated immunity. Proceedings of the National Academy of Sciences of the United States of America , 2010, 107 (17) : 8029–8034. DOI:10.1073/pnas.0912311107 |
| [17] | Ying J, Chen X, Ding X, et al. The XA21 binding protein XB25 is required for maintaining XA21-mediated disease resistance. Plant Journal , 2013, 73 (5) : 814–823. DOI:10.1111/tpj.2013.73.issue-5 |
| [18] | Park C, Peng Y, Chen X, et al. Rice XB15, a protein phosphatase 2C, negatively regulates cell death and XA21-mediated innate immunity. PLoS Biology , 2008, 6 (11) : 1910–1926. |
| [19] | Peng Y, Bartley L, Chen X, et al. OsWRKY62 is a negative regulator of basal and Xa21 mediated defense against Xanthomonas oryzae pv. oryzae in Rice. Molecular Plant , 2008, 1 (3) : 446–458. DOI:10.1093/mp/ssn024 |
| [20] | Wang Y, Pi L, Chen X, et al. Rice XA21 binding protein 3 is a ubiquitin ligase required for full Xa21-mediated disease resistance. Plant Cell , 2006, 18 (12) : 3635–3646. DOI:10.1105/tpc.106.046730 |
| [21] | Park C, Ronald PC. Cleavage and nuclear localization of the rice XA21 immune receptor. Nature Communications , 2012, 3 (2) : 177–180. |
| [22] | Park C, Sharma R, Lefebvre B, et al. The endoplasmic reticulum-quality control component SDF2 is essential for XA21-mediated immunity in rice. Plant Science , 2013, 210 (9) : 53–60. |
| [23] | Xin L, Kapos P, Zhang Y. NLRs in plants. Current Opinion in Immunology , 2015, 32 : 114–121. DOI:10.1016/j.coi.2015.01.014 |
| [24] | Zhou T, Wang Y, Chen J, et al. Genome-wide identification of NBS genes in japonica rice reveals significant expansion of divergent non-TIR NBS-LRR genes. Molecular Genetics & Genomics , 2004, 271 (4) : 402–415. |
| [25] | Chen L, Hamada S, Fujiwara M, et al. The Hop/Sti1-Hsp90 chaperone complex facilitates the maturation and transport of a PAMP receptor in rice innate immunity. Cell Host & Microbe , 2010, 7 (3) : 185–196. |
| [26] | Chen X, Shan J, Chen D, et al. A B-lectin receptor kinase gene conferring rice blast resistance. Plant Journal for Cell & Molecular Biology , 2006, 46 (5) : 794–804. |
| [27] | Fukuoka S, Saka N, Koga H, et al. Loss of function of a proline-containing protein confers durable disease resistance in rice. Science , 2009, 325 (5943) : 998–1001. DOI:10.1126/science.1175550 |
| [28] | Nan J, Li Z, Wu J, et al. Molecular mapping of the Pi2/9 allelic gene Pi2-2 conferring broad-spectrum resistance to Magnaporthe oryzae in the rice cultivar Jefferson. Rice , 2012, 5 (1) : 1–7. DOI:10.1186/1939-8433-5-1 |
| [29] | Zhu X, Shen C, Yang J, et al. The identification of Pi50(t), a new member of the rice blast resistance Pi2/Pi9 multigene family. Theoretical & Applied Genetics , 2012, 124 (7) : 1295–1304. |
| [30] | Su J, Wang W, Han J, et al. Functional divergence of duplicated genes results in a novel blast resistance gene Pi50 at the Pi2/9 locus. Theoretical & Applied Genetics , 2015, 128 (11) : 2213–2225. |
| [31] | Ma J, Lei C, Xu X, et al. Pi64, encoding a novel CC-NBS-LRR protein, confers resistance to leaf and neck blast in rice. Molecular Plant-Microbe Interactions , 2015, 28 (5) : 558–568. DOI:10.1094/MPMI-11-14-0367-R |
| [32] | Hayashi N, Inoue H, Kato T, et al. Durable panicle blast-resistance gene Pb1 encodes an atypical CC-NBS-LRR protein and was generated by acquiring a promoter through local genome duplication. Plant Journal for Cell & Molecular Biology , 2010, 64 (3) : 498–510. |
| [33] | Zeng X, Yang X, Zhao Z, et al. Characterization and fine mapping of the rice blast resistance gene Pia. Science China Life Sciences , 2011, 54 (4) : 372–378. DOI:10.1007/s11427-011-4154-1 |
| [34] | Wang Z, Yano M, Yamanouchi U, et al. The Pib gene for rice blast resistance belongs to the nucleotide binding and leucine-rich repeat class of plant disease resistance genes. Plant Journal , 1999, 19 (1) : 55–64. DOI:10.1046/j.1365-313X.1999.00498.x |
| [35] | Shang J, Tao Y, Chen X, et al. Identification of a new rice blast resistance gene, Pid3, by genomewide comparison of paired nucleotide-binding site-leucine-rich repeat genes and their pseudogene alleles between the two sequenced rice genomes. Genetics , 2009, 182 (4) : 1303–1311. DOI:10.1534/genetics.109.102871 |
| [36] | Zhai C, Lin F, Dong Z, et al. The isolation and characterization of Pik, a rice blast resistance gene which emerged after rice domestication. New Phytologist , 2011, 189 (1) : 321–334. DOI:10.1111/j.1469-8137.2010.03462.x |
| [37] | Rai A, Kumar S, Gupta S, et al. Functional complementation of rice blast resistance gene Pi-k h(Pi54)conferring resistance to diverse strains of Magnaporthe oryzae. Journal of Plant Biochemistry & Biotechnology , 2011, 20 (20) : 55–65. |
| [38] | Ashikawa I, Hayashi N, Yamane H, et al. Two adjacent nucleotide-binding site-leucine-rich repeat class genes are required to confer Pikm-specific rice blast resistance. Genetics , 2008, 180 (4) : 2267–2276. DOI:10.1534/genetics.108.095034 |
| [39] | Yuan B, Zhai C, Wang W, et al. The Pik-p resistance to Magnaporthe oryzae in rice is mediated by a pair of closely linked CC-NBS-LRR genes. Theoretical & Applied Genetics , 2011, 122 (122) : 1017–1028. |
| [40] | Takahashi A, Hayashi N, Miyao A, et al. Unique features of the rice blast resistance Pish locus revealed by large scale retrotransposon-tagging. Bmc Plant Biology , 2010, 10 (1) : 1–14. DOI:10.1186/1471-2229-10-1 |
| [41] | Hayashi K, Yoshida H. Refunctionalization of the ancient rice blast disease resistance gene Pit by the recruitment of a retrotransposon as a promoter. Plant Journal for Cell & Molecular Biology , 2009, 57 (3) : 413–425. |
| [42] | Bryan G, Wu K, Farrall L, et al. A single amino acid difference distinguishes resistant and susceptible alleles of the rice blast resistance gene Pi-ta. Plant Cell , 2000, 12 (11) : 2033–2046. DOI:10.1105/tpc.12.11.2033 |
| [43] | Zhou B, Qu S, Liu G, et al. The eight amino-acid differences within three leucine-rich repeats between Pi2 and Piz-t resistance proteins determine the resistance specificity to Magnaporthe grisea. Molecular Plant-Microbe Interactions , 2006, 19 (11) : 1216–1228. DOI:10.1094/MPMI-19-1216 |
| [44] | Hua L, Wu J, Chen C, et al. The isolation of Pi1, an allele at the Pik locus which confers broad spectrum resistance to rice blast. Theoretical & Applied Genetics , 2012, 125 (5) : 1047–1055. |
| [45] | Lee S, Song M, Seo Y, et al. Rice Pi5-mediated resistance to Magnaporthe oryzae requires the presence of two coiled-coil-nucleotide-binding-leucine-rich repeat genes. Genetics , 2009, 181 (4) : 1627–1638. DOI:10.1534/genetics.108.099226 |
| [46] | Qu S, Liu G, Bellizzi M, et al. The broad-spectrum blast resistance gene Pi9 encodes a nucleotide-binding site-leucine-rich repeat protein and is a member of a multigene family in rice. Genetics , 2006, 172 (3) : 1901–1914. |
| [47] | Jie C, Liu W, Zhuang J, et al. A Pid3 allele from rice cultivar Gumei2 confers resistance to Magnaporthe oryzae. Journal of Genetics & Genomics , 2011, 38 (5) : 209–216. |
| [48] | Fukuoka S, Yamamoto S, Mizobuchi R, et al. Multiple functional polymorphisms in a single disease resistance gene in rice enhance durable resistance to blast. Scientific Reports , 2014, 4 : 4450. |
| [49] | Liu X, Lin F, Wang L, et al. The in silico map-based cloning of Pi36, a rice coiled-coil nucleotide-binding site leucine-rich repeat gene that confers race-specific resistance to the blast fungus. Genetics , 2007, 176 (4) : 2541–2549. DOI:10.1534/genetics.107.075465 |
| [50] | Lin F, Chen S, Que Z, et al. The blast resistance gene Pi37 encodes a nucleotide binding site leucine-rich repeat protein and is a member of a resistance gene cluster on rice chromosome 1. Genetics , 2007, 177 (3) : 1871–1880. DOI:10.1534/genetics.107.080648 |
| [51] | Liu Y, Liu B, Zhu X, et al. Fine-mapping and molecular marker development for Pi56(t), a NBS-LRR gene conferring broad-spectrum resistance to Magnaporthe oryzae in rice. Theoretical & Applied Genetics , 2013, 126 (4) : 985–998. |
| [52] | Cesari S, Thilliez G, Ribot C, et al. The rice resistance protein pair RGA4/RGA5 recognizes the Magnaporthe oryzae effectors AVR-Pia and AVR1-CO39 by direct binding. Plant Cell , 2013, 25 (4) : 1463–1481. DOI:10.1105/tpc.112.107201 |
| [53] | Lyer-Pascuzzi A, Mccouch S. Genetic and functional characteriza-tion of the rice bacterial blight disease resistance gene xa5. Phytopathology , 2008, 98 : 289–295. DOI:10.1094/PHYTO-98-3-0289 |
| [54] | Yuan M, Chu Z, Li X, et al. Pathogen-induced expressional loss of function is the key factor in race-specific bacterial resistance conferred by a recessive R gene xa13 in rice. Plant & Cell Physiology , 2009, 50 (5) : 947–955. |
| [55] | Liu Q, Yuan M, Zhou Y, et al. A paralog of the MtN3/saliva family recessively confers race-specific resistance to Xanthomonas oryzae in rice. Plant Cell & Environment , 2011, 34 (11) : 1958–1969. |
| [56] | Sun X, Cao Y, Yang Z, et al. Xa26, a gene conferring resistance to Xanthomonas oryzae pv. oryzae in rice, encodes an LRR receptor kinase-like protein. Plant Journal , 2004, 37 : 517–527. DOI:10.1046/j.1365-313X.2003.01976.x |
| [57] | Xiang Y, Cao Y, Xu C, et al. Xa3, conferring resistance for rice bacterial blight and encoding a receptor kinase-like protein, is the same as Xa26. Tag Theoretical & Applied Genetics. theoretische Und Angewandte Genetik , 2006, 113 (7) : 1347–1355. |
| [58] | Gu K, Yang B, Tian D, et al. R gene expression induced by a type-Ⅲ effector triggers disease resistance in rice. Nature , 2005, 435 (7045) : 1122–1125. DOI:10.1038/nature03630 |
| [59] | Yoshimura S, Yamanouchi U, Katayose Y, et al. Expression of Xa1, a bacterial blight-resistance gene in rice, is induced by bacterial inoculation. Proceedings of the National Academy of Sciences of the United States of America , 1998, 95 (4) : 1663–1668. DOI:10.1073/pnas.95.4.1663 |
| [60] | Tian D, Yin Z. The rice TAL effector-dependent resistance protein XA10 triggers cell death and calcium depletion in the endoplasmic reticulum. Plant Cell , 2014, 26 (1) : 497–515. DOI:10.1105/tpc.113.119255 |
| [61] | Wang C, Zhang X, Fan Y, et al. XA23 Is an executor r protein and confers broad-spectrum disease resistance in rice. Molecular Plant , 2015, 8 (2) : 290–302. DOI:10.1016/j.molp.2014.10.010 |
| [62] | Park C, Chen S, Shirsekar G, et al. The Magnaporthe oryzae effector AvrPiz-t targets the RING E3 ubiquitin ligase APIP6 to suppress pathogen-associated molecular pattern-triggered immunity in rice. Plant Cell , 2012, 24 (11) : 4748–4762. DOI:10.1105/tpc.112.105429 |
| [63] | Hogenhout S, Hoorn R, Terauchi R, et al. Emerging concepts in effector biology of plant-associated organisms. Molecular plant-microbe interactions:MPMI , 2009, 22 (2) : 115–122. DOI:10.1094/MPMI-22-2-0115 |
| [64] | Wu J, Kou Y, Bao J, et al. Comparative genomics identifies the Magnaporthe oryzae avirulence effector AvrPi9 that triggers Pi9-mediated blast resistance in rice. New Phytologist , 2015, 206 (4) : 1463–1475. DOI:10.1111/nph.13310 |
| [65] | Chan H, Shirsekar G, Bellizzi M, et al. The E3 ligase APIP10 connects the effector AvrPiz-t to the NLR receptor Piz-t in rice. Plos Pathogens , 2016, 12 (3) : e1005529. DOI:10.1371/journal.ppat.1005529 |
| [66] | Yang DL, Yang YN, He ZH, et al. Roles of plant hormones and their interplay in rice immunity. Molecular Plant , 2013, 6 (3) : 675–685. DOI:10.1093/mp/sst056 |
| [67] | Bari R, Jones J. Role of plant hormones in plant defence responses. Plant Molecular Biology , 2009, 69 (4) : 473–488. DOI:10.1007/s11103-008-9435-0 |
| [68] | Chen Z, Iyer S, Caplan A, et al. Differential accumulation of salicylic acid and salicylic acid-sensitive catalase in different rice tissues. Plant Physiology , 1997, 114 (1) : 193–201. |
| [69] | Silverman P, Seskar M, Kanter D, et al. Salicylic acid in rice(biosynthesis, conjugation, and possible role). Plant Physiology , 1995, 108 (2) : 633–639. |
| [70] | Ganesan V, Thomas G. Salicylic acid response in rice:influence of salicylic acid on H2O2 accumulation and oxidative stress. Plant Science An International Journal of Experimental Plant Biology , 2001, 160 (6) : 1095–1106. |
| [71] | Bart R, Chern M, Vegasánchez M, et al. Rice Snl6, a cinnamoyl-CoA reductase-like gene family member, is required for NH1-mediated immunity to Xanthomonas oryzae pv. oryzae. Plos Genetics , 2010, 6 (9) : 110–117. |
| [72] | Shimono M, Sugano S, Nakayama A, et al. Rice WRKY45 plays a crucial role in benzothiadiazole-inducible blast resistance. Plant Cell , 2007, 19 (6) : 2064–2076. DOI:10.1105/tpc.106.046250 |
| [73] | Mei C, Qi M, Sheng G, et al. Inducible overexpression of a rice allene oxide synthase gene increases the endogenous jasmonic acid level, PR gene expression, and host resistance to fungal infection. Molecular plant-microbe interactions:MPMI , 2006, 19 (10) : 1127–1137. DOI:10.1094/MPMI-19-1127 |
| [74] | Patkar R, Benke P, Qu Z, et al. A fungal monooxygenase-derived jasmonate attenuates host innate immunity. Nature Chemical Biology , 2015, 11 (9) : 733–740. DOI:10.1038/nchembio.1885 |
| [75] | Liu Q, Chen Y, Pan H, et al. Emerging connections in the ethylene signaling network. Trends in Plant Science , 2009, 14 (5) : 270–279. DOI:10.1016/j.tplants.2009.02.007 |
| [76] | Broekaert W, Delauré S, Bolle M, et al. The role of ethylene in host-pathogen interactions. Annual Review of Phytopathology , 2006, 44 : 393–416. DOI:10.1146/annurev.phyto.44.070505.143440 |
| [77] | Loon L, Geraats B, Linthorst H. Ethylene as a modulator of disease resistance in plants. Trends in Plant Science , 2006, 11 (4) : 184–191. DOI:10.1016/j.tplants.2006.02.005 |
| [78] | Singh M, Lee F, Counce P, et al. Mediation of partial resistance to rice blast through anaerobic induction of ethylene. Phytopathology , 2004, 94 (8) : 819–825. DOI:10.1094/PHYTO.2004.94.8.819 |
| [79] | Iwai T, Miyasaka A, Seo S, et al. Contribution of ethylene biosynthesis for resistance to blast fungus infection in young rice plants. Plant Physiology , 2006, 142 (3) : 1202–1215. DOI:10.1104/pp.106.085258 |
| [80] | Helliwell E, Wang Q, Yang Y. Transgenic rice with inducible ethylene production exhibits broad-spectrum disease resistance to the fungal pathogens Magnaporthe oryzae and Rhizoctonia solani. Plant Biotechnology Journal , 2013, 11 (1) : 33–42. DOI:10.1111/pbi.12004 |
| [81] | Zhu S, Gao F, Cao X, et al. The rice dwarf virus P2 protein interacts with ent-kaurene oxidases in vivo, leading to reduced biosynthesis of gibberellins and rice dwarf symptoms. Plant Physiology , 2005, 139 (4) : 1935–1945. DOI:10.1104/pp.105.072306 |
| [82] | Qin X, Liu J, Zhao W, et al. Gibberellin 20-oxidase gene OsGA20ox3 regulates plant stature and disease development in rice. Molecular plant-microbe interactions:MPMI , 2013, 26 (2) : 227–239. DOI:10.1094/MPMI-05-12-0138-R |
| [83] | Kim T, Wang Z. Brassinosteroid signal transduction from receptor kinases to transcription factors. Annual Review of Plant Biology , 2010, 61 (4) : 681–704. |
| [84] | 高静, 张昊, 王凤茹, 等. 油菜素内酯提高水稻抗病性的分子机制. 植物保护学报 , 2016, 43 (2) : 347–348. |
| [85] | Li J, Chory J. A putative leucine-rich repeat receptor kinase involved in brassinosteroid signal transduction. Cell , 1997, 90 (5) : 929–938. DOI:10.1016/S0092-8674(00)80357-8 |
| [86] | Jia L, Wen J, Lease K, et al. BAK1, an Arabidopsis LRR receptor-like protein kinase, interacts with BRI1 and modulates brassinosteroid signaling. Cell , 2002, 110 (2) : 213–222. DOI:10.1016/S0092-8674(02)00812-7 |
| [87] | He Z, Wang Z, Li J, et al. Perception of brassinosteroids by the extracellular domain of the receptor kinase BRI1. Science , 2000, 288 (5475) : 2360–2363. DOI:10.1126/science.288.5475.2360 |
| [88] | Vleesschauwer D, Buyten E, Satoh K, et al. Brassinosteroids antagonize gibberellin-and salicylate-mediated root immunity in rice. Plant Physiology , 2012, 158 (4) : 1833–1846. DOI:10.1104/pp.112.193672 |
| [89] | Hamon M, Cossart P. Histone modifications and chromatin remodeling during bacterial infections. Cell Host & Microbe , 2008, 4 (2) : 100–109. |
| [90] | Ma K, Flores C, Ma W. Chromatin configuration as a battlefield in plant-bacteria interactions. Plant Physiology , 2011, 157 (2) : 535–543. DOI:10.1104/pp.111.182295 |
| [91] | Ding B, Bellizzi M, Ning Y, et al. HDT701, a histone H4 deacetylase, negatively regulates plant innate immunity by modulating histone H4 acetylation of defense-related genes in rice. Plant Cell , 2012, 24 (9) : 3783–3794. DOI:10.1105/tpc.112.101972 |
| [92] | Rao Y, Li Y, Qian Q. Recent progress on molecular breeding of rice in China. Plant Cell Reports , 2014, 33 (4) : 551–564. DOI:10.1007/s00299-013-1551-x |
| [93] | 何峰, 张浩, 刘金灵, 等. 水稻抗稻瘟病天然免疫机制及抗病育种新策略. 遗传 , 2014, 36 (8) : 756–765. |
| [94] | 马军韬, 张国民, 辛爱华, 等. 水稻品种抗稻瘟病分析及基因聚合抗性改良. 植物保护学报 , 2016, 43 (2) : 177–183. |
| [95] | Zheng Y. Genome-wide association study of flowering time and grain yield traits in a worldwide collection of rice germplasm. Nature Genetics , 2012, 44 (1) : 32–39. |
| [96] | Kump K, Bradbury P, Wisser R, et al. Genome-wide association study of quantitative resistance to southern leaf blight in the maize nested association mapping population. Nature Genetics , 2011, 43 (2) : 163–168. DOI:10.1038/ng.747 |
| [97] | Kang H, Zhang Y, Wang Y, et al. Molecular Dissection of the Complex Genetic Architecture of Rice Immunity to the Blast Fungus Magnaporthe oryzae using GWAS[C]. Jeju, Korea:International Plant and Animal Genome Conference, 2013. |
| [98] | Li T, Liu B, Spalding M, et al. High-efficiency TALEN-based gene editing produces disease-resistant rice. Nature Biotechnology , 2012, 30 (5) : 390–392. DOI:10.1038/nbt.2199 |
| [99] | Pennisi E. The CRISPR craze. Science , 2013, 341 (6148) : 833–836. DOI:10.1126/science.341.6148.833 |
| [100] | Wang Y, Cheng X, Shan Q, et al. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew. Nature Biotechnology , 2014, 32 (9) : 947–951. DOI:10.1038/nbt.2969 |
| [101] | Feng G, Xiao Z, Feng J, et al. DNA-guided genome editing using the Natronobacterium gregoryi Argonaute. Nature Biotechnology , 2016, 34 (7) : 768–773. DOI:10.1038/nbt.3547 |
| [102] | Gonsalves D. Control of papaya ringspot virus in papaya:a case study. Annual Review of Phytopathology , 1998, 36 (1) : 415–437. DOI:10.1146/annurev.phyto.36.1.415 |
| [103] | Nowara D, Gay A, Lacomme C, et al. HIGS:host-induced gene silencing in the obligate biotrophic fungal pathogen Blumeria graminis. Plant Cell , 2010, 22 (9) : 3130–3141. DOI:10.1105/tpc.110.077040 |
| [104] | Nunes C, Dean R. Host-induced gene silencing:a tool for understanding fungal host interaction and for developing novel disease control strategies. Molecular Plant Pathology , 2012, 13 (5) : 519–529. DOI:10.1111/mpp.2012.13.issue-5 |
| [105] | 邹德堂, 姜思达, 赵宏伟, 等. 广谱抗性基因Pi9在黑龙江省水稻品种中的分布. 东北农业大学学报 , 2016, 47 (7) : 1–8. |
| [106] | 马建, 马小定, 赵志超, 等. 水稻抗稻瘟病基因Pi35功能性分子标记的开发及其应用. 作物学报 , 2015, 41 (12) : 1779–1790. |





