2. 复旦大学生命科学学院 遗传工程国家重点实验室,上海 200433
2. State Key Laboratory of Genetic Engineering, Institute of Plant Biology, School of Life Sciences, Fudan University, Shanghai 200433
在真核生物中,蛋白质的合成过程极其复杂,合成的主要场所是细胞质中的核糖体。对于一些大分子蛋白,例如,约占细胞总蛋白1/3的分泌蛋白和膜蛋白等需要进入内质网进行进一步的折叠和修饰,只有正确折叠和修饰后的蛋白质才能被转运到细胞质、细胞核、线粒体等细胞器中发挥生物功能[1, 2]。蛋白质折叠过程精细复杂,容易受生长发育期或外界环境的影响,导致蛋白不能折叠或者错误折叠[3]。
内质网拥有一套完备的系统,用于清除分泌途径中的未折叠蛋白和错误折叠蛋白。当遭遇干旱、高温、盐害等胁迫后,蛋白折叠需求超过蛋白折叠和降解系统的能力时,细胞内会积累大量未折叠蛋白或者错误折叠蛋白,导致内质网胁迫(ER stress),细胞会启动未折叠蛋白应答途径(unfolded protein response,UPR),调节一系列下游基因如分子伴侣等帮助蛋白正确折叠,或加速错误折叠蛋白的降解[4, 5]。
在研究植物内质网胁迫应答中,主要利用内质网胁迫诱导剂衣霉素(tunicamycin,TM)和二硫苏糖醇(dithiothreitol,DTT)处理植物(细胞),诱导内质网胁迫。衣霉素能特异阻断内质网中蛋白折叠过程中的糖基蛋白N-糖基化,未正确糖基化的糖基蛋白不能被分子伴侣识别,无法正确折叠,造成内质网中错误折叠蛋白的积累。二硫键的形成对于许多蛋白的折叠也很重要,还原剂DTT能破坏蛋白折叠过程中二硫键形成所需要的氧化条件,导致细胞质和内质网中某些蛋白错误折叠。通过检测下游基因,例如,帮助内质网蛋白折叠的分子伴侣BiP等的上调表达来判断植物是否受到内质网胁迫。本文对前人关于内质网胁迫应答途径的研究进展进行总结,并对植物中膜结合转录因子和内质网胁迫之间的关系进行阐述,以期为进一步讨论内质网胁迫的研究者提供一些参考。
1 蛋白折叠与内质网胁迫应答内质网是真核细胞重要的细胞器,所有分泌蛋白在合成之后都需要进入内质网进一步加工折叠,才能被分泌到细胞外,发挥功能。在内质网内进行的修饰和加工主要包括糖基化、羟基化、酰基化、二硫键形成等。在内质网内,蛋白折叠主要通过一些分子伴侣和辅助因子来协同完成。分子伴侣可以与非天然状态的蛋白质结合,以帮助他们稳定原生状态,分子伴侣并不直接参与折叠过程,它们只是防止蛋白错误聚集,提高折叠的效率[6]。
1.1 蛋白折叠方式内质网中聚集大量错误折叠或未折叠的蛋白会对细胞产生一种毒害作用,是内质网胁迫的主要原因。因此,调控蛋白正确折叠是细胞维持自我平衡的关键。初生肽链通过Sec61易位子复合体进入内质网的蛋白折叠调控通路[7],根据初生蛋白是否具有糖基化位点分为两种蛋白折叠通路:(1)糖基化蛋白被糖基转移酶(oligosaccharide transferase,OST)识别后进入钙联蛋白/钙网蛋白(CNX/CRT)循环中进行折叠;(2)非糖基化蛋白主要依赖内质网腔内的分子伴侣BiP以及其它辅助因子组成复合体进行蛋白折叠[8]。
1.1.1 糖基蛋白在内质网中的折叠方式糖基蛋白在内质网中的折叠主要是一种由凝集素CNX/CRT介导的蛋白折叠方式。糖基化是一种重要的翻译后修饰,在肽链生物合成后,糖链在酶的催化下被接到肽链上的特定糖位点,称为蛋白质糖基化。大部分在内质网合成的蛋白最终都会被N-糖基化[9]。糖链的存在对肽链的折叠、糖蛋白的进一步成熟、分拣、投送以及最后的定位都有重要影响[10]。对于初生糖蛋白的折叠主要有4种方式:OST介导的N-糖基化修饰、结合蛋白折叠装置CNX/CRT、UGGT催化重新糖基化和ERAD途径执行降解。
(1)OST介导的N-糖基化修饰:Asn-X-Ser/Thr三个氨基酸残基的序列称为糖基化位点(其中X是除Pro以外的任意氨基酸)。N-糖基化是指蛋白的糖链与肽链的天冬酰胺氮(Asn-X-Ser序列)以共价键连接[11]。含有糖基位点的初生多肽链通过Sec61复合体进入内质网,OST将预先装配好的脂(长醇)连低聚糖转移到糖基蛋白的天冬酰胺残基上。通过不断加入低聚糖形成一个LLO单元Glc3Man9GlcNAc2。通过葡糖苷酶Ⅰ和Ⅱ分别除去末端α-1,2-葡萄糖和α-1,3-葡萄糖得到蛋白折叠的核心寡糖Glc1Man9GlcNAc2。衣霉素(tunicamy-cin,TM)诱导内质网胁迫的主要原理就是通过阻断天冬酰胺连接的N-糖基化过程,导致低聚糖不能连接到初生蛋白上,其它分子伴侣因无法识别初生蛋白上的糖链而不能帮助蛋白折叠,导致初生多肽链成为未折叠蛋白积累在内质网中,引起内质网胁迫。拟南芥中糖基转移酶OST由5个亚基组成:DAD1、DAD2、STT3A/STT3B、DGL1和HAP6。其中At-DAD1和At-DAD2被认为是参与到糖基转移酶OST的识别定位[12],过表达At-DAD1和At-DAD2能抑制紫外损伤引起的DNA片段化[13]。在拟南芥中,单突变体stt3a-1和stt3b-1植物仍然能生存,但是双突变体stt3a-1stt3b-1植物会胚胎致死。DGL1(defective glycosylation1-1)是糖基化转移酶中重要的亚基,如果在拟南芥中突变掉DGL1,会削弱蛋白的糖基化,影响细胞分化和生长[14]。表明OST对于植物生长非常重要并且不同亚基有功能冗余的现象[15]。
(2)结合蛋白折叠装置CNX/CRT:外源凝集素钙联蛋白(calnexin,CNX)和钙网蛋白(calreticulin,CRT)是凝集素样分子伴侣,具有相似的专一性,参与糖蛋白的折叠质量控制过程,是内质网中蛋白折叠的主要场所,进入该场所必须是先被低聚糖链修饰后的糖蛋白。钙联蛋白存在于内质网中需Ca2+的凝集素样分子伴侣蛋白。钙联蛋白是一种膜定位蛋白,钙网蛋白定位在内质网腔,可结合新合成的未折叠完全的肽链的寡糖链,防止蛋白质彼此错误聚集。在胁迫情况下,蛋白折叠的核心寡糖Glc1Man9GlcNAc2,结合到有效的质量控制系统CNX/CRT,进入CNX/CRT折叠循环途径。
(3)UGGT催化重新糖基化:当糖蛋白折叠不完全或折叠错误,由UDP葡萄糖糖蛋白糖基转移酶(UDP-glucose:glycoprotein glucosyltransferase,UGGT)催化其重新糖基化[16]。UGGT不能识别折叠正确的蛋白质,但UGGT能够识别不完全折叠的糖蛋白,通过糖苷Ⅱ和UGGT逐步除去末端的寡糖形式的α-1,3-葡萄糖,重新添加并且催化其葡萄糖基化,指导蛋白再次进入CNX/CRT循环。
(4)ERAD途径执行降解:无法恢复正确构象的糖蛋白,为避免在内质网过度堆积造成危害,则从CNX/CRT循环中释放出来,由葡萄糖苷酶I和葡萄糖苷酶Ⅱ共同作用下,α甘露糖苷酶样蛋白EDEM(ER degradation-enhancing α mannosidase like protein)结合[17],去掉部分甘露醇形成的Man5GlcNAc2形式,被外源凝集素OS9识别C链末端α-1,6-甘露糖残基后进入泛素-蛋白酶体ERAD途径中将其降解。ERAD途径使永久错误折叠的蛋白质返回胞液,被蛋白酶体降解[18]。
1.1.2 非糖基蛋白在内质网中的折叠方式初生蛋白通过Sec61复合体后,没有糖基化位点的蛋白与定位于内质网上的热激蛋白ERdj3B直接结合后,招募BiP[19]。在哺乳动物细胞中,类基质衍生因子SDF2(stromal-derived factor-2)与其它辅助因子包括BiP和ERdj3B形成复合物来帮助初生蛋白折叠[20]。PDI也能与非糖基化蛋白作用帮助蛋白形成二硫键。正确折叠的糖基蛋白和非糖基蛋白被挑选后通过分泌途径转运到其它细胞器中。糖蛋白EFR发挥功能必须依赖于SDF2和ERdj3B。EFR是拟南芥抗病途径PRRs之一,能识别细菌EF-Tu,帮助植物抵抗细菌病害。当用内质网胁迫诱导剂TM和DTT处理植物,能诱导SDF2转录,EFR的蛋白量增加;去掉SDF2后,会导致EFR蛋白不稳定[21]。说明内质网胁迫下非糖基化蛋白的正确折叠也是非常重要的。
1.2 蛋白折叠过程中的辅助因子在内质网折叠过程中,蛋白折叠需要分子伴侣或协同分子伴侣的辅助调节,分子伴侣能够帮助稳定初生蛋白的状态,防止蛋白错误折叠。分子伴侣是指一类在序列上没有相关性但有共同功能的蛋白质,它们在细胞内帮助其他含多肽的结构完成正确的组装,而且在组装完毕后与之分离,不构成这些蛋白质结构执行功能时的组分。在蛋白折叠中,分子伴侣能够与其它非自然态的蛋白短暂互作后帮助它们进入自然态,虽然不能指导蛋白折叠后输出内质网,但它们能够提高蛋白折叠效率。热休克蛋白HSP70就是已知的一大类分子伴侣。
1.2.1 BiPBiP(binding protein)属于热休克蛋白HSP70家族,是内质网腔内含量很高的分子伴侣[22]。能够与Sec61易位子复合物一起结合到进入内质网的初生多肽链,避免初生多肽链表面的疏水结构间聚合。BiP是免疫球蛋白重链结合蛋白,在N端有核酸结合位点和ATP酶活性。在与ADP结合的状态下,BiP对蛋白有高亲和的结合力;而在有ATP结合的时候,BiP能释放结合的蛋白[23]。BiP对蛋白的结合和释放过程由辅助因子DnaJ调控,主要是通过促进ATP水解或者ATP:ADP的交换来完成[24]。在拟南芥中有3个BiP基因:BiP1、BiP2和BiP3。BiP1和BiP2蛋白序列非常相似,在植物生长过程中表达量较高,受内质网胁迫诱导剂TM/DTT和高温诱导上调。BiP3正常情况下表达量很低,但是受内质网胁迫的诱导大幅度上调表达[25-27]。拟南芥BiP基因表达还受其他环境胁迫的调控,例如干旱、冷、病虫害等[28, 29]。在大豆和烟草中过量表达BiP[30],植物耐旱性提高[31]。在植物体内敲掉BiP2后,植物对病虫害侵染过程非常敏感,PR1的转录水平下降,其中PR1是联系分泌系统和系统获得性抗性系统之间的重要基因[32]。另外,在植物生长发育过程中,当特定细胞中分泌蛋白表达量较高或蛋白折叠错误时,BiP基因也上调表达,表明BiP蛋白在蛋白折叠过程中发挥重要作用[33]。水稻BiP1主要在种子成熟过程中表达,大幅度抑制(BiP1 KD)或者显著过量表达(BiP1 OE)BiP1基因不仅改变了种子的表型和胚乳细胞的胞内结构,还降低了胞内贮藏蛋白的浓度、淀粉的积累和籽粒的重量[34]。
1.2.2 CNX/CRT外源凝集素钙联蛋白(Calnexin,CNX)和钙网蛋白(Calreticulin,CRT)是凝集素样分子伴侣[35],具有相似的专一性,参与糖蛋白的折叠质量控制过程,是内质网中蛋白折叠的主要场所。在胁迫情况下,首先葡萄糖苷酶I快速酶切低聚糖A链上α-1,2键连接的第14位葡萄糖,随后葡萄糖苷酶Ⅱ酶解去除A链上α-1,3键连接的第13位葡萄糖,形成蛋白折叠的核心寡糖Glc1Man9GlcNAc2,结合凝集素进入CNX/CRT折叠循环途径[3]。在拟南芥中,CRT有3个异构体(CRT1-3),CNX有2个异构体(CNX1-2)。CNX的胞外域与CRT结构非常相似,都有球形结构域、脯氨酸结构域(富含脯氨酸的发卡结构)和C结构域。两者的主要区别在于CNX是Ⅱ型膜蛋白,在脯氨酸和C结构域之间含有跨膜域[36, 37]。CRT和CNX的N末端球形β-san-dwich结构域都能作用于初生糖蛋白上的单葡糖苷低聚肽链[3]。CRT和CNX的N结构域在植物中非常保守,有2个标志性基序:KHEQKLDCGGGYVLL和IMFGPDICG。尽管拟南芥CRTs的N结构域序列中含有一个潜在的N糖基化位点,但是至今没有报道称拟南芥CRTs被富含甘露糖的低聚糖糖基化。CRT和CNX的P结构域包含2个三联重复,通过与Ca2+结合维持发卡构型。拟南芥突变掉CRT3后,植物出现bril-9的表现,表明CRT参与调控BRl1受体的折叠。小麦(Triticum aestivum)CRT(Ta-CRT)受干旱诱导上调表达,在烟草中过量表达Ta-CRT能提高植物耐旱性[38]。
1.2.3 GRP94与BiP分子伴侣类似,GRP94也是一类内质网中含量较高的分子伴侣,受葡萄糖调控。在拟南芥中只有一个GRP94的同源基因SHD(SHEPHERD),受TM和DTT诱导上调表达。通过生化和细胞生物学手段发现,SHD在体内有分子伴侣活性,并且底物非常特异。将SHD敲掉后植株表现为顶端大小异常,类似clavata突变体的表型,遗传学分析表明在高温胁迫的条件下CLV蛋白的合成需要SHD参与[39]。
1.2.4 Ero1在内质网中,蛋白二硫键的形成需要在氧化状态下进行。在酵母中,氧化还原物主要有内质网氧化还原酶1(ER oxidoreductase 1,Ero1p)[40]。Ero1p是一个定位在内质网上的膜结合蛋白,在二硫键形成中能直接传递电子到分子氧。拟南芥中也有两个同源基因:AERO1和AERO2,也参与内质网胁迫[41]。水稻的ERO1也定位在内质网,对水稻胚乳中蛋白的二硫键形成非常重要[42]。
1.2.5 PDIs氧化还原蛋白PDI(蛋白二硫桥异构酶),参与钙联蛋白/钙网蛋白循环,帮助蛋白折叠形成构型稳定的二硫键[43]。在酵母中,ERp57类硫氧还蛋白结合到CNX1/CRT1的复合物上与糖蛋白相互作用。目前不知道拟南芥是否存在与ERp57同样功能的PDIs,但是在拟南芥中的同源物是PDI家族。拟南芥基因组中共编码12个PDIs,其中9个含有信号肽和内质网定位信号。PDIs在催化过程中需要不同的氧化还原物,用于氧化半胱氨酸形成二硫键,还原异构化过程中非天然键。错误折叠的蛋白质暴露部分疏水区,PDI直接以二硫键与目的糖蛋白相连,然后将其引入ERAD途径[18]。PDIs不仅催化形成二硫键,而且当蛋白需要恢复到天然状态时,PDIs能够催化非天然键异构化或重组[41]。
1.2.6 DnaJ哺乳动物ERdj同源物DnaJ属于Hsp40家族成员,参与调控细胞生物学过程。在内质网胁迫反应中,作为辅助因子通过促进ATP水解或者ATP:ADP的交换来调控BiP对蛋白的结合和释放过程[24]。酵母DnaJ基因在拟南芥中的同源基因是Scj1p,命名为At-J1、At-J2、At-J3,但是在拟南芥中的作用知之甚少[44]。Yang等[45]报道称拟南芥含有DnaJ和ERdj5C结构域的THERMOSENSTITIVE MALE STERILE1蛋白,在花粉管的耐热胁迫中发挥作用。目前水稻中DnaJ功能研究比较少。Yamamoto等[46]发现水稻OsDnaJ能够与增殖细胞核抗原蛋白OsPCNA相互作用,调控DNA复制和DNA修复过程。最近Zhu等[47]在水稻中鉴定出叶绿体定位的OsDjA7/8,降低OsDjA7/8的表达会引起水稻叶绿体发育受损,导致幼苗出现白化致死。
2 经典未折叠蛋白应答途径真核细胞的蛋白合成和折叠是一个非常精细控制的过程。为了维持内质网功能的精确性,细胞演化出一种蛋白监控信号通路——未折叠蛋白应答途径UPR[48-51]。当非正确折叠的蛋白质在内质网内聚积,会引发UPR信号转导途径,通过调节翻译水平或转录水平来帮助蛋白正确折叠。其中主要是靠一类定位在内质网膜上的膜结合转录因子传导信号,目前哺乳动物中已知的有3条途径:IRE1-XBPl[52-54]、ATF6-S2P[55]和PERK/ATF4[56]。
2.1 IRE l介导的XBP mRNA非常规剪接途径IRE1(inositol requiring enzyme 1)在酵母中是已经报道的唯一一个内质网胁迫感应器,在动物和植物未折叠蛋白应答中也扮演着非常重要的角色。IRE1是一个单次跨膜的内质网驻留蛋白质,具有胞质激酶活性与内切核酸酶RNase活性。内质网中未折叠蛋白质累积时,IRE1蛋白形成多聚体,激活IRE1的蛋白激酶活性并发生自身磷酸化,进一步激活其内切核酸酶活性,对动物XBP1和酵母HAC1的mRNA进行非常规剪切,剪切掉一部分核苷酸,重新编码新的蛋白形式。该形式具有转录激活活性和核定位信号,能够进入细胞核,调节帮助蛋白折叠的UPR基因,从而缓解内质网胁迫。
2.2 ATF6-S2P水解途径哺乳动物中的ATF6(activating transcription factor 6)属于一个内质网Ⅱ型bZIP家族跨膜转录因子,N端位于细胞质中,包含一个碱性亮氨酸拉链结构域,C端位于内质网腔。在正常情况下,全长的ATF6被BiP滞留在内质网;在内质网胁迫下,ATF6被COPⅡ通过膜泡运输转运到高尔基体,由定位于高尔基体的两个蛋白酶S1P与S2P分步酶切,酶切后的形式不含跨膜域,通过核定位信号进入细胞核,启动下游UPR基因表达,而这种酶切方式也称之为膜内介导的蛋白质水解方式(regulated-intramembrane proteolysis,RIP)。
2.3 PERK/ATF4-CHOP选择翻译性途径PERK(PKR-like ER kinase)是一个定位在内质网的含有跨膜域的激酶,C末端朝向胞质,N末端朝向内质网腔,具有丝/苏氨酸蛋白激酶活性,目前只在动物中发现。翻译起始因子eIF2的α亚基(eIF2α)结合GTP后能与tRNA形成翻译起始复合物(eIF2-GTP-Met-tRNA)。在内质网应激时,PERK磷酸化真核起始因子eIF2α上的第51位丝氨酸。eIF2α被磷酸化后,能抑制翻译起始复合物中GDP与GTP的交换,抑制蛋白质的翻译与合成,从而减少进入内质网的蛋白。
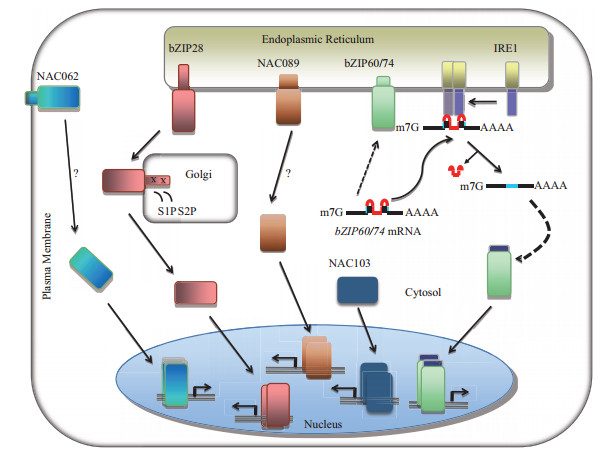
3 植物膜结合转录因子与内质网胁迫应答植物与其他生物的一个重要区别在于植物几乎是不能移动的,因此植物在生长发育过程中不得不忍受和适应环境的改变。蛋白质的正确折叠是其行使功能的前提和基础,而逆境胁迫可能引起蛋白错误折叠,从而影响逆境条件下植物的生长和发育。因此,深入研究植物蛋白折叠调控的遗传分子机制,可为通过提高细胞蛋白折叠能力来增强植物抗逆性奠定基础。目前在拟南芥中,bZIP家族和NAC家族膜结合转录因子在调控内质网胁迫信号途径方面发挥重要作用(图 1)。
3.1 AtbZIP28途径正常情况下,拟南芥膜结合转录因子bZIP28通过跨膜域定位在ER中,当感应到胁迫后(内质网胁迫、高温等),bZIP28进入高尔基体内被丝氨酸蛋白酶S1P和S2P分步酶切,去掉跨膜域,通过核定位信号进入细胞核,上调一系列与UPR相关的基因来帮助蛋白折叠[25, 58-61]。在哺乳动物中,ATF6被BiP滞留在内质网,在内质网胁迫下,COPⅡ通过膜泡运输将ATF6转运到高尔基体[55]。植物中有许多被预测是停靠在内质网出口位点的移动高尔基体,不需要中间囊泡的参与,就能将内质网蛋白转运到高尔基体;尽管如此,多数认为内质网到高尔基体的转运仍与COPⅡ装置有关。最近的研究证明植物中bZIP28的亚细胞器间的移动也受COPⅡ的调控[62]。有趣的是,虽然植物和动物中S1P和S2P的序列比较保守,但植物的bZIP28和动物的ATF6的蛋白序列同源性较低,只有S1P的酶切位点序列比较保守。
3.2 AtbZIP60/OsbZIP74途径拟南芥bZIP60(水稻bZIP74)和AtbZIP28的活化方式不一样。AtbZIP60/OsbZIP74的mRNA能够形成二级发夹结构被IRE1识别后在mRNA水平进行非常规剪切。当内质网中未折叠蛋白质积累,IRE1蛋白形成多聚体,激活IRE1的蛋白激酶活性,并促进自体磷酸化,进一步激活内切核酸酶活性,识别bZIP60 mRNA上的双茎环结构,去除掉bZIP60 mRNA上的23个核苷酸后导致阅读框移码,提前出现终止密码子,重新编码成一个不含跨膜域的蛋白形式。该形式具有转录激活活性和核定位信号,能够进入细胞核,上调UPR基因表达,促进细胞生存[63-65]。植物的IRE1蛋白与酵母和动物的IRE1蛋白序列同源性较高,但植物中的IRE1蛋白的剪接底物的核苷酸序列与酵母和动物的IRE1蛋白的剪接底物的核苷酸序列的相似性较低。在拟南芥内质网胁迫过程中,膜相关转录因子bZIP60通过IRE1调控的非常规mRNA剪切活化进核后,既可以通过之前报道的UPRE-Ⅰ或ERSE-Ⅰ调控下游基因,又可以通过UPRE-Ⅲ诱导NAC103的表达,然后通过其编码的转录因子调控下游基因的表达,更好地帮助蛋白质折叠[66]。
3.3 NAC062途径内质网、高尔基体、囊泡、质膜和疏水区域(植物中的液泡,动物的溶酶体)组成真核的分泌系统。NAC062(也称为ANAC062或NTL6)是植物特异转录因子NAC家族的成员,其C-末端具有疏水跨膜域。内质网胁迫诱导剂TM可诱导NAC062的表达上调,且这种上调在bzip60突变体中被完全抑制,而在bzip28突变体中不受影响;酵母单杂交和EMSA实验分析证明bZIP60可以直接结合到NAC062启动子上。正常情况下NAC062定位在质膜上,内质网胁迫下,被未知的方式活化后,从质膜转移到细胞核,活化后的NAC062可以结合到BiP等UPR基因的启动子,调控促进细胞生存的基因[67]。
3.4 NAC089途径在拟南芥内质网胁迫应答中,bZIP28、bZIP60和NAC062都是帮助细胞生存的正调控因子,而膜结合转录因子NAC089(也称为ANAC089)却具有促进细胞死亡的功能。通过内质网胁迫诱导剂TM和DTT处理,NAC089受内质网胁迫诱导上调,且这种上调在bzip60和bzip28单突变体中部分降低,而在双突变体zip28zip60中被完全抑制,说明NAC089的上调依赖于bZIP28和bZIP60。NAC089也是一个膜结合转录因子,正常情况下定位在内质网,当内质网胁迫严重的时候,NAC089被未知的方式活化,去掉跨膜域进入细胞核,调控包括NAC094、MC5和BAG6等与细胞死亡有关基因表达;诱导表达去除跨膜域的NAC089会导致类caspase 3/7活性增加和DNA片段化等现象。这些研究证明NAC089在内质网胁迫下从内质网膜进入细胞核介导细胞死亡[68]。
4 植物细胞死亡因子与内质网胁迫应答在动物中,线粒体诱导的细胞死亡是动物细胞死亡的主要途径,例如BCL-2家族通过调控线粒体膜完整性来调节细胞死亡。目前植物中没有发现BCL-2家族同源基因,但是在植物中仍然存在内质网胁迫诱导的程序性细胞死亡(PCD)。
4.1 异三聚体G蛋白拟南芥异源三聚体G蛋白在胁迫信号诱导的程序性细胞死亡中扮演重要的角色。异三聚体G蛋白由α、β和γ三种亚基构成,Gα本身具有内在的GTPase活性。Wang等[69]报道异三聚体G蛋白参与内质网胁相关的程序性细胞死亡。在拟南芥中,Gβ敲除后,在含有内质网胁迫诱导剂的培养基上比对照野生型和Gα生长得好,暗示Gβ(AGB1)在植物中能促进细胞死亡,但其作用机制未见报道。事实上,Chen等[70]最近的报道表明Gβ敲除后,突变体比野生型对照对内质网胁迫诱导剂更敏感,AGB1是促进细胞生存的,但其作用机制有待进一步研究。由于G蛋白复合体的复杂性,对于了解它在内质网胁迫中的功能将是一大挑战。
4.2 BAG蛋白BAG是BCL2家族基因,在哺乳动物中和酵母中非常保守,具有包括从抑制生长到促进细胞死亡多种功能[71]。在拟南芥中发现有7个BAG同源物[72]。Williams等[73]报道拟南芥AtBAG7定位在内质网,对于维持未折叠蛋白应答是必须的。首先通过双分子荧光实验和酵母双杂交实验发现AtBAG7与分子伴侣AtBiP2具有相互作用;加入内质网胁迫诱导剂TM后会加速AtBAG7突变体的细胞死亡;抑制AtBAG7的表达会导致植物对内质网胁迫、热胁迫和冷胁迫敏感;而当在AtBAG7突变体中加入一些分子伴侣后能成功恢复为野生型的表型;说明AtBAG7能与分子伴侣BiP2相互作用后,调控高温和冷害下引起的未折叠蛋白应答,延迟内质网胁迫引起的程序性细胞死亡。过表达BAG6在酵母和动物中诱导程序性细胞死亡,表明BAG6在植物中是促进细胞死亡的基因[74]。在番茄中过表达BAG4能够提高番茄对不同胁迫的抗性,包括紫外损伤、冷害、盐害和氧化损伤等。
4.3 BI-1在酵母和哺乳动物中,BI-1(Bax inhibitor-1)是被发现的第一个抑制Bax活性的细胞死亡基因,在哺乳动物细胞中,BI-1不与Bax/Bak,而能与BCL-2相互作用。Kawai等[75]在拟南芥中构建地塞米松(DEX)诱导的Bax蛋白植株。在DEX处理后,转基因植物表现出显著的细胞死亡特征:细胞皱缩、膜的破损以及其它细胞死亡表型。当在转基因Bax中转入花椰菜花叶病毒35S启动子驱动的AtBI-1载体后,能够维持细胞的完整性,抑制死亡。Sanchez等[76]通过侵染病原体丁香假单胞菌筛选得到,AtBI-1可以抑制酵母的致死表型和哺乳动物凋亡蛋白Bax的活性。伤口或病原体也会上调AtBI-1的表达,这表明其参与生物和非生物胁迫途径。Coi1是促进死亡的基因,在遭遇害虫、病原体等后会大幅度上调;在coi1突变体背景下,AtBI-1会显著降低,进一步说明拟南芥AtBI-1在功能上类似BI-1,通过抑制死亡基因来促细胞生存。
4.4 Metacaspase在哺乳动物中caspase在细胞凋亡中起着重要作用[5]。植物细胞中没有发现细胞凋亡现象。植物中存在的Metacaspase虽然没有caspase活性,但被认为与动物细胞凋亡中的caspase高度同源[77-79]。拟南芥中有9个metacaspase,其中metacaspase-8已经报道受UVC和过氧化氢大幅度上调[80],激活细胞死亡途径。此外,在植物木质部形成、重金属胁迫和抗病等都检测到与PCD相关的类caspase活性[81-83]。
5 内质网胁迫与植物生长发育和环境胁迫应答蛋白折叠过程对环境胁迫非常敏感。在细胞遭受逆境的情况下,错误折叠和未折叠的蛋白大量聚集在内质网,对细胞产生毒害作用。为了维持细胞生存,细胞会启动内质网胁迫应答,包括调节基因的表达和蛋白质的翻译在一定程度上消除和减轻内质网上蛋白质折叠的负荷,从而达到缓解胁迫、保护细胞的作用。因此,植物的内质网胁迫应答与多种环境胁迫应答密切相关。
5.1 内质网胁迫与植物生长发育拟南芥NAC103编码植物特有的转录因子,其表达受内质网胁迫的诱导,过量表达该基因后拟南芥生长发育延迟、叶片锯齿形、花器官异常。IRE1A、IRE1B和bZIP28都参与未折叠蛋白经典反应途径,然而正常情况下ire1a、ire1b、bzip28三突变体会出现花粉活力下降[84]。拟南芥膜结合转录因子ANAC089在内质网胁迫中调控细胞死亡,同时也能够负调控拟南芥的开花过程。长日照条件下,相对于野生型对照,过表达ANAC089ΔC(去掉跨膜域的形式)转基因植株出现开花延迟和株型矮小的现象[85]。Li等[86]通过QTL定位分析发现ANAC089(FSQ6)也是果糖信号的抑制子。
5.2 内质网胁迫与其他环境胁迫在拟南芥中膜结合转录因子bZIP17受高盐胁迫后活化,诱导盐胁迫应答相关基因上调表达[87-89]。同时该途径在脱落酸(ABA)调控的种子萌芽过程中也发挥着重要作用,负调控ABA信号通路。bZIP28和bZIP60在UPR调控通路中发挥作用外,也参与高温胁迫途径[63, 90]。分子伴侣BiP2受高温诱导上调表达,在bZIP28突变体中诱导上调幅度下降,表明在高温胁迫后bZIP28通过调控BiP2应对外界的高温应答[90]。
拟南芥bZIP60除了受内质网胁迫的诱导上调,也受盐胁迫诱导上调。Fujita等[91]过表达AtbZIP60全长提高了拟南芥耐盐性,说明在盐应激应答中激活了AtbZIP60蛋白。AtbZIP60在水稻中的同源基因是OsbZIP74,也受高温和水杨酸活化[65]。
拟南芥中至少存在两个IRE1蛋白,即IRE1A和IRE1B。IRE1A和IRE1B都受内质网胁迫试剂TM、病原菌侵染和水杨酸SA诱导上调表达。进一步证明IRE1A是在病原菌侵染条件下通过剪切bZIP60 mRNA发挥作用。UPR信号通路中的IRE1/bZIP60在植物病原菌应答反应中发挥重要作用,IRE1A和IRE1B在病原菌侵染调控中部分功能冗余,同时IRE1并不是完全依赖bZIP60应对植物免疫应答[92]。
环境胁迫容易导致未折叠蛋白聚集,造成细胞毒害,因此缓解这种毒害的内质网胁迫途径与很多环境胁迫有着千丝万缕的联系。利用细胞生物学、生物化学、遗传学和分子生物学等多种方法研究植物中内质网胁迫途径,有助于深入理解植物抗逆的分子和遗传基础,利于充分挖掘与干旱、高温、盐害等环境胁迫相关耐逆基因,且有助于改善作物的抗逆性,已成为作物抗性育种的重要方向。
6 总结与展望蛋白折叠是真核细胞基础生物学过程。内质网是真核细胞重要的细胞器,所有分泌蛋白和大部分膜蛋白都需要在内质网内进行加工才能被转运到其他细胞器,进而被分泌到细胞外发挥生物学功能。当植物遭受高温、高盐等逆境时,会导致未折叠和错误折叠蛋白聚集在内质网,产生内质网胁迫。然而由于酵母和哺乳动物中关键的调节在植物中都未发现同源基因,导致植物内质网胁迫途径研究进展非常缓慢。膜结合转录因子bZIP28和bZIP60(对应哺乳动物ATF6和XBP1)的发现,为研究植物内质网胁迫开辟了新的途径。此外,植物特异的NAC家族的膜结合转录因子NAC062和NAC089也参与植物内质网胁迫应答。内质网胁迫与各种环境胁迫密切相关,植物内质网胁迫应答信号途径的阐明为理解植物逆境响应过程中的作用机理提供了基础。随着研究的深入也产生了一些问题,需要进一步的研究。
6.1 质膜结合转录因子如何感应内质网胁迫目前关于未折叠蛋白应答的报道都主要集中在内质网相关的信号,其他分泌系统对内质网胁迫的作用目前还不清楚,质膜结合转录因子NAC062的发现扩展了植物中的UPR途径,然而质膜结合蛋白如何感应内质网内的错误折叠蛋白的目前并不是很清楚。在之前的报道中,内质网不仅在蛋白质生物合成,而且在合成ER膜以及质膜形成必须的脂质和甾醇方面起着重要作用。Sriburi等[93, 94]报道哺乳动物中的XBP1(在植物中对应bZIP60),在特定的分泌细胞中被激活,上调脂肪酸和磷脂质合成以驱动粗糙内质网的扩展。Bommias等[95]在2009年报道过表达哺乳动物ATF6(在植物中对应bZIP28)的活化形式,也诱导脂质生物合成及ER扩展且不依赖XBP1。Seo等[96]报道NAC062受冷诱导活化,进入细胞核调控抗病相关基因,提高植物抗病能力,冷害胁迫期间NAC062的活化可能由膜组成和流动性改变导致。我们推测在内质网胁迫下,错误折叠的蛋白质在ER中积累后影响质膜脂质成分,然后被NAC062或其相互作用组分所感知,从而导致NAC062活化进入细胞核调节UPR应答。总之,相信随着对内质网信号通路研究的不断深入,将会有更多的新组分以及新机制被发现,对现有的信号通路进行完善和补充。
6.2 内质网胁迫下如何平衡细胞生存和死亡在植物中如何调节细胞生存和死亡,目前机制并不清楚。内质网胁迫条件下未折叠蛋白反应UPR最重要的生理作用之一是确保细胞生存[97, 98]。当降低bZIP28、bZIP60或者NAC062表达水平都会降低细胞对内质网胁迫的抗性,表明这几个膜结合转录因子是帮助抵抗内质网胁迫,提高细胞生存能力。IRE1是一个定位在内质网的单次跨膜蛋白,具有胞质激酶活性和内切核酸酶RNase活性。当内质网中未折叠蛋白质积累时,IRE1蛋白形成多聚体,激活IRE1的蛋白激酶活性并发生自身磷酸化,进一步激活其内切核酸酶活性。在内质网胁迫不严重的情况下,IRE1对底物有高度特异性,主要剪切有发夹结构的mRNA,例如bZIP60;然而当这种胁迫继续延长,IRE1会失去底物特异性,利用核酸酶活性通过RIDD(IRE1-dependent decay)途径降解与内质网膜相关mRNAs,引起细胞死亡,细胞是怎样决定IRE1的作用目前还是未知的。在拟南芥中,内质网胁迫激活S2P-bZIP28和IRE1-bZIP60途径,促进下游基因表达提高细胞生存能力;然而活化后的bZIP28和bZIP60也能诱导死亡相关因子NAC089基因上调表达;在我们的研究中还发现NAC089在胁迫处理前期就被活化,但是NAC089的下游调控细胞死亡的基因只有在胁迫非常严重时才被诱导,我们推测NAC089可能受未知的方式控制,在特定的时候被释放来调控细胞死亡。
细胞在内质网胁迫下是如何决定细胞生存与死亡的命运,仍然是未来研究的一大挑战。解决这些问题,将有助于我们了解植物如何协调生长与应对外界环境胁迫,帮助我们更加精确地调控植物内质网胁迫应答,提高植物/作物的抗逆性。
| [1] | Sitia R, Braakman I. Quality control in the endoplasmic reticulum protein factory. Nature , 2003, 426 (6968) : 891–894. DOI:10.1038/nature02262 |
| [2] | Kanapin A, Batalov S, Davis MJ, et al. Mouse proteome analysis. Genome Res , 2003, 13 (6B) : 1335–1344. |
| [3] | Hammond C, Braakman I, Helenius A. Role of N-linked oligosaccharide recognition, glucose trimming, and calnexin in glycoprotein folding and quality-control. Proc Natl Acad Sci USA , 1994, 91 (3) : 913–917. DOI:10.1073/pnas.91.3.913 |
| [4] | Vitale A, Boston RS. Endoplasmic reticulum quality control and the unfolded protein response:Insights from plants. Traffic , 2008, 9 (10) : 1581–1588. DOI:10.1111/tra.2008.9.issue-10 |
| [5] | Ron D, Walter P. Signal integration in the endoplasmic reticulum unfolded protein response. Nat Rev Mol Cell Bio , 2007, 8 (7) : 519–529. DOI:10.1038/nrm2199 |
| [6] | Otero JH, Lizak B, Hendershot LM. Life and death of a BiP substrate. Semin Cell Dev Biol , 2010, 21 (5) : 472–478. DOI:10.1016/j.semcdb.2009.12.008 |
| [7] | Kornfeld R, Kornfeld S. Assembly of asparagine-linked oligosaccharides. Annu Rev Biochem , 1985, 54 : 631–664. DOI:10.1146/annurev.bi.54.070185.003215 |
| [8] | Howell SH. Endoplasmic reticulum stress responses in plants. Annu Rev Plant Biol , 2013, 64 : 477–499. DOI:10.1146/annurev-arplant-050312-120053 |
| [9] | Helenius A, Aebi M. Roles of N-linked glycans in the endoplasmic reticulum. Annu Rev Biochem , 2004, 73 : 1019–1049. DOI:10.1146/annurev.biochem.73.011303.073752 |
| [10] | Hubbard SC, Ivatt RJ. Synthesis and processing of asparagine-linked oligosaccharides. Annu Rev Biochem , 1981, 50 : 555–583. DOI:10.1146/annurev.bi.50.070181.003011 |
| [11] | Pattison RJ, Amtmann A. N-glycan production in the endoplasmic reticulum of plants. Trend Plant Sci , 2009, 14 (2) : 92–99. DOI:10.1016/j.tplants.2008.11.008 |
| [12] | Gallois P, Makishima T, Hecht V, et al. An Arabidopsis thaliana cDNA complementing a hamster apoptosis suppressor mutant. Plant J , 1997, 11 (6) : 1325–1331. DOI:10.1046/j.1365-313X.1997.11061325.x |
| [13] | Danon A, Rotari VI, Gordon A, et al. Ultraviolet-C overexposure induces programmed cell death in Arabidopsis, which is mediated by caspase-like activities and which can be suppressed by caspase inhibitors, p35 and defender against apoptotic death. J Biol Chem , 2004, 279 (1) : 779–787. DOI:10.1074/jbc.M304468200 |
| [14] | Lerouxel O, Mouille G, Andeme-Onzighi C, et al. Mutants in DEFECTIVE GLYCOSYLATION, an Arabidopsis homolog of an oligosaccharyltransferase complex subunit, show protein underglycosylation and defects in cell differentiation and growth. Plant J , 2005, 42 (4) : 455–468. DOI:10.1111/tpj.2005.42.issue-4 |
| [15] | Koiwa H, Li F, McCully MG, et al. The STT3a subunit isoform of the Arabidopsis oligosaccharyltransferase controls adaptive responses to salt/osmotic stress. Plant Cell , 2003, 15 (10) : 2273–2284. DOI:10.1105/tpc.013862 |
| [16] | Deprez P, Gautschi M, Helenius A. More than one glycan is needed for ER glucosidase Ⅱ to allow entry of glycoproteins into the calnexin/calreticulin cycle. Mol Cell , 2005, 19 (2) : 183–195. DOI:10.1016/j.molcel.2005.05.029 |
| [17] | Olivari S, Cali T, Salo KEH, et al. EDEM1 regulates ER-associated degradation by accelerating de-mannosylation of folding-defective polypeptides and by inhibiting their covalent aggregation. Biochem Biophy Res Commun , 2006, 349 (4) : 1278–1284. DOI:10.1016/j.bbrc.2006.08.186 |
| [18] | Hegde RS, Ploegh HL. Quality and quantity control at the endoplasmic reticulum. Curr Opin Cell Biol , 2010, 22 (4) : 437–446. DOI:10.1016/j.ceb.2010.05.005 |
| [19] | Jin Y, Zhuang M, Hendershot LM. ERdj3, a luminal ER DnaJ homologue, binds directly to unfolded proteins in the mammalian ER:identification of critical residues. Biochem , 2009, 48 (1) : 41–49. DOI:10.1021/bi8015923 |
| [20] | Meunier L, Usherwood YK, Chung KT, et al. A subset of chaperones and folding enzymes form multiprotein complexes in endoplasmic reticulum to bind nascent proteins. Mol Biol Cell , 2002, 13 (12) : 4456–4469. DOI:10.1091/mbc.E02-05-0311 |
| [21] | Schott A, Ravaud S, Keller S, et al. Arabidopsis stromal-derived factor2(SDF2)is a crucial target of the unfolded protein response in the endoplasmic reticulum. J Biol Chem , 2010, 285 (23) : 18113–18121. DOI:10.1074/jbc.M110.117176 |
| [22] | Kleizen B, Braakman I. Protein folding and quality control in the endoplasmic reticulum. Curr Opin Cell Biol , 2004, 16 (4) : 343–349. DOI:10.1016/j.ceb.2004.06.012 |
| [23] | Flynn GC, Chappell TG, Rothman JE. Peptide binding and release by proteins implicated as catalysts of protein assembly. Science , 1989, 245 (4916) : 385–390. DOI:10.1126/science.2756425 |
| [24] | Cheetham ME, Caplan AJ. Structure, function and evolution of DnaJ:conservation and adaptation of chaperone function. Cell Stress Chaperon , 1998, 3 (1) : 28–36. DOI:10.1379/1466-1268(1998)003<0028:SFAEOD>2.3.CO;2 |
| [25] | Liu JX, Srivastava R, Che P, et al. An endoplasmic reticulum stress response in Arabidopsis is mediated by proteolytic processing and nuclear relocation of a membrane-associated transcription factor, bZIP28. Plant Cell , 2007, 19 (12) : 4111–4119. DOI:10.1105/tpc.106.050021 |
| [26] | Iwata Y, Fedoroff NV, Koizumi N. Arabidopsis bZIP60 is a proteolysis-activated transcription factor involved in the endoplasmic reticulum stress response. Plant Cell , 2008, 20 (11) : 3107–3121. DOI:10.1105/tpc.108.061002 |
| [27] | Martinez IM, Chrispeels MJ. Genomic analysis of the unfolded protein response in Arabidopsis shows its connection to important cellular processes. Plant Cell , 2003, 15 (2) : 561–576. DOI:10.1105/tpc.007609 |
| [28] | Anderson JV, Li QB, Haskell DW, et al. Structural organization of the Spinach endoplasmic reticulum-luminal 70-kilodalton heat-shock cognate gene and expression of 70-kilodalton heat-shock genes during cold-acclimation. Plant Physiol , 1994, 104 (4) : 1359–1370. DOI:10.1104/pp.104.4.1359 |
| [29] | Jelitto-Van Dooren EPWM, Vidal S, Denecke J. Anticipating endoplasmic reticulum stress:a novel early response before pathogenesis-related gene induction. Plant Cell , 1999, 11 (10) : 1935–1943. DOI:10.1105/tpc.11.10.1935 |
| [30] | Valente MAS, Faria JAQA, Soares-Ramos JRL, et al. The ER luminal binding protein(BiP)mediates an increase in drought tolerance in soybean and delays drought-induced leaf senescence in soybean and tobacco. J Exp Bot , 2009, 60 (2) : 533–546. DOI:10.1093/jxb/ern296 |
| [31] | Alvim FC, Carolino SMB, Cascardo JCM, et al. Enhanced accumulation of BiP in transgenic plants confers tolerance to water stress. Plant Physiol , 2001, 126 (3) : 1042–1054. DOI:10.1104/pp.126.3.1042 |
| [32] | Wang D, Weaver ND, Kesarwani M, et al. Induction of protein secretory pathway is required for systemic acquired resistance. Science , 2005, 308 (5724) : 1036–1040. DOI:10.1126/science.1108791 |
| [33] | Boston RS, Fontes EBP, Shank BB, et al. Increased expression of the maize immunoglobulin binding-protein homolog b-70 in 3 zein regulatory mutants. Plant Cell , 1991, 3 (5) : 497–505. DOI:10.1105/tpc.3.5.497 |
| [34] | Yuhya W, Hiroshi Y, Youko O, et al. Expression of ER quality con-trol-related genes in response to changes in BiP1 levels in develo-ping rice endosperm. Plant J , 2011, 65 (5) : 675–689. DOI:10.1111/tpj.2011.65.issue-5 |
| [35] | Alves MS, Reis PAB, Dadalto SP, et al. A novel transcription factor, ERD15(early responsive to dehydration 15), connects endoplasmic reticulum stress with an osmotic stress-induced cell death signal. J Biol Chem , 2011, 286 (22) : 20020–20030. DOI:10.1074/jbc.M111.233494 |
| [36] | Navazio L, Miuzzo M, Royle L, et al. Monitoring endoplasmic reticulum-to-Golgi traffic of a plant calreticulin by protein glycosylation analysis. Biochem , 2002, 41 (48) : 14141–14149. DOI:10.1021/bi0204701 |
| [37] | Jia XY, He LH, Jing RL, et al. Calreticulin:conserved protein and diverse functions in plants. Physiol Plant , 2009, 136 (2) : 127–138. DOI:10.1111/ppl.2009.136.issue-2 |
| [38] | Jia XY, Xu CY, Jing RL, et al. Molecular cloning and characterization of wheat calreticulin(CRT)gene involved in drought-stressed responses. J Exp Bot , 2008, 59 (4) : 739–751. DOI:10.1093/jxb/erm369 |
| [39] | Ishiguro S, Watanabe Y, Ito N, et al. SHEPHERD is the Arabidopsis GRP94 responsible for the formation of functional CLAVATA proteins. EMBO J , 2002, 21 (5) : 898–908. DOI:10.1093/emboj/21.5.898 |
| [40] | Sevier CS, Kaiser CA. Disulfide transfer between two conserved cysteine pairs imparts selectivity to protein oxidation by Ero1. Mol Biol Cell , 2006, 17 (5) : 2256–2266. DOI:10.1091/mbc.E05-05-0417 |
| [41] | Dixon DP, Van Lith M, Edwards R, et al. Cloning and initial characterization of the Arabidopsis thaliana endoplasmic reticulum oxidoreductins. Antioxid Redox Signal , 2003, 5 (4) : 389–396. DOI:10.1089/152308603768295122 |
| [42] | Onda Y, Kumamaru T, Kawagoe Y. ER membrane-localized oxidoreductase Ero1 is required for disulfide bond formation in the rice endosperm. Proc Natl Acad Sci USA , 2009, 106 (33) : 14156–14161. DOI:10.1073/pnas.0904429106 |
| [43] | Lu DP, Christopher DA. Endoplasmic reticulum stress activates the expression of a sub-group of protein disulfide isomerase genes and AtbZIP60 modulates the response in Arabidopsis thaliana. Mol Genet Genomics , 2008, 280 (3) : 199–210. DOI:10.1007/s00438-008-0356-z |
| [44] | Schlenstedt G, Harris S, Risse B, et al. A yeast Dnaj homolog, Scj1p, can function in the endoplasmic-reticulum with Bip/Kar2p via a conserved domain that specifies interactions with hsp70s. J Cell Biol , 1995, 129 (4) : 979–988. DOI:10.1083/jcb.129.4.979 |
| [45] | Yang KZ, Xia C, Liu XL, et al. A mutation in THERMOSENSITIVE MALE STERILE 1, encoding a heat shock protein with DnaJ and PDI domains, leads to thermosensitive gametophytic male sterility in Arabidopsis. Plant J , 2009, 57 (5) : 870–882. DOI:10.1111/tpj.2009.57.issue-5 |
| [46] | Yamamoto T, Mori Y, Ishibashi T, et al. Interaction between proliferating cell nuclear antigen(PCNA)and a DnaJ induced by DNA damage. J Plant Res , 2005, 118 (2) : 91–97. DOI:10.1007/s10265-005-0197-3 |
| [47] | Zhu X, Liang S, Yin J, et al. The DnaJ OsDjA7/8 is essential for chloroplast development in rice(Oryza sativa). Gene , 2015, 574 (1) : 11–9. DOI:10.1016/j.gene.2015.07.067 |
| [48] | Woehlbier U, Hetz C. Modulating stress responses by the UPRosome:A matter of life and death. Trend Biochem Sci , 2011, 36 (6) : 329–337. DOI:10.1016/j.tibs.2011.03.001 |
| [49] | Shore GC, Papa FR, Oakes SA. Signaling cell death from the endoplasmic reticulum stress response. Curr Opin Cell Biol , 2011, 23 (2) : 143–149. DOI:10.1016/j.ceb.2010.11.003 |
| [50] | Merksamer PI, Papa FR. The UPR and cell fate at a glance. J Cell Sci , 2010, 123 (7) : 1003–1006. DOI:10.1242/jcs.035832 |
| [51] | Buchberger A, Bukau B, Sommer T. Protein quality control in the cytosol and the endoplasmic reticulum:brothers in arms. Mol Cell , 2010, 40 (2) : 238–252. DOI:10.1016/j.molcel.2010.10.001 |
| [52] | Cox JS, Shamu CE, Walter P. Transcriptional induction of genes encoding endoplasmic-reticulum resident proteins requires a transmembrane protein-kinase. Cell , 1993, 73 (6) : 1197–1206. DOI:10.1016/0092-8674(93)90648-A |
| [53] | Shen XH, Ellis RE, Lee K, et al. Complementary signaling pathways regulate the unfolded protein response and are required for C-elegans development. Cell , 2001, 107 (7) : 893–903. DOI:10.1016/S0092-8674(01)00612-2 |
| [54] | Calfon M, Zeng HQ, Urano F, et al. IRE1 couples endoplasmic reticulum load to secretory capacity by processing the XBP-1 mRNA. Nature , 2002, 420 (6912) : 202. |
| [55] | Haze K, Yoshida H, Yanagi H, et al. Mammalian transcription factor ATF6 is synthesized as a transmembrane protein and activated by proteolysis in response to endoplasmic reticulum stress. Mol Biol Cell , 1999, 10 (11) : 3787–3799. DOI:10.1091/mbc.10.11.3787 |
| [56] | Vattem KM, Wek RC. Reinitiation involving upstream ORFs regulates ATF4 mRNA translation in mammalian cells. Proc Natl Acad Sci USA , 2004, 101 (31) : 11269–11274. DOI:10.1073/pnas.0400541101 |
| [57] | Liu JX, Howell SH. Managing the protein folding demands in the endoplasmic reticulum of plants. New Phytol , 2016, 211 (2) : 418–428. DOI:10.1111/nph.13915 |
| [58] | Liu JX, Howell SH. bZIP28 and NF-Y transcription factors are activated by ER stress and assemble into a transcriptional complex to regulate stress response genes in Arabidopsis. Plant Cell , 2010, 22 (3) : 782–796. DOI:10.1105/tpc.109.072173 |
| [59] | Sun L, Lu SJ, Zhang SS, et al. The lumen-facing domain is important for the biological function and organelle-to-organelle movement of bZIP28 during ER stress in Arabidopsis. Mol Plant , 2013, 6 (5) : 1605–1615. DOI:10.1093/mp/sst059 |
| [60] | Le S, Zhang SS, Lu SJ, et al. Site-1 protease cleavage site is important for the ER stress-induced activation of membrane-associated transcription factor bZIP28 in Arabidopsis. Sci China Life Sci , 2015, 58 (3) : 270–275. DOI:10.1007/s11427-015-4807-6 |
| [61] | Song ZT, Sun L, Lu SJ, et al. Transcription factor interaction with COMPASS-like complex regulates histone H3K4 trimethylation for specific gene expression in plants. Proc Natl Acad Sci USA , 2015, 112 (9) : 2900–2905. DOI:10.1073/pnas.1419703112 |
| [62] | Srivastava R, Chen Y, Deng Y, et al. Elements proximal to and within the transmembrane domain mediate the organelle-to-organelle movement of bZIP28 under ER stress conditions. Plant J , 2012, 70 (6) : 1033–1042. DOI:10.1111/tpj.2012.70.issue-6 |
| [63] | Deng Y, Humbert S, Liu JX, et al. Heat induces the splicing by IRE1 of a mRNA encoding a transcription factor involved in the unfolded protein response in Arabidopsis. Proc Natl Acad Sci USA , 2011, 108 (17) : 7247–7252. DOI:10.1073/pnas.1102117108 |
| [64] | Nagashima Y, Mishiba K, Suzuki E, et al. Arabidopsis IRE1 catalyses unconventional splicing of bZIP60 mRNA to produce the active transcription factor. Sci Rep , 2011, 1 : 29. |
| [65] | Lu SJ, Yang ZT, Sun L, et al. Conservation of IRE1-Regulated bZIP74 mRNA unconventional splicing in Rice(Oryza sativa L.)involved in ER stress responses. Mol Plant , 2012, 5 (2) : 504–514. DOI:10.1093/mp/ssr115 |
| [66] | Sun L, Yang ZT, Song ZT, et al. The plant-specific transcription factor gene NAC103, is induced by bZIP60 through a new cis -regulatory element to modulate the unfolded protein response in Arabidopsis. Plant J , 2013, 76 (2) : 274–286. |
| [67] | Yang Z, Lu S, Wang M, et al. A plasma membrane-tethered transcription factor, NAC062/ANAC062/NTL6, mediates the unfolded protein response in Arabidopsis. Plant J , 2014, 79 (6) : 1033–1043. DOI:10.1111/tpj.12604 |
| [68] | Yang ZT, Wang MJ, Sun L, et al. The membrane-associated transcription factor NAC089 controls ER-stress-induced programmed cell death in plants. Plos Genet , 2014, 10 (3) : 119–123. |
| [69] | Wang SY, Narendra S, Fedoroff N. Heterotrimeric G protein signaling in the Arabidopsis unfolded protein response. Proc Natl Acad Sci USA , 2007, 104 (10) : 3817–3822. DOI:10.1073/pnas.0611735104 |
| [70] | Chen YN, Brandizzi F. AtIRE1A/AtIRE1B and AGB1 independently control two essential unfolded protein response pathways in Arabidopsis. Plant J , 2012, 69 (2) : 266–277. DOI:10.1111/tpj.2011.69.issue-2 |
| [71] | Takayama S, Sato T, Krajewski S, et al. Cloning and functional-analysis of Bag-1 -a novel Bcl-2-binding protein with anti-cell death activity. Cell , 1995, 80 (2) : 279–284. DOI:10.1016/0092-8674(95)90410-7 |
| [72] | Doukhanina EV, Chen S, van der Zalm E, et al. Identification and functional characterization of the BAG protein family in Arabidopsis thaliana. J Biol Chem , 2006, 281 (27) : 18793–18801. DOI:10.1074/jbc.M511794200 |
| [73] | Williams B, Kabbage M, Britt R, et al. AtBAG7, an Arabidopsis Bcl-2-associated athanogene, resides in the endoplasmic reticulum and is involved in the unfolded protein response. Proc Natl Acad Sci USA , 2010, 107 (13) : 6088–6093. DOI:10.1073/pnas.0912670107 |
| [74] | Kang CH, Jung WY, Kang YH, et al. AtBAG6, a novel calmodulin-binding protein, induces programmed cell death in yeast and plants. Cell Death Differ , 2006, 13 (1) : 84–95. DOI:10.1038/sj.cdd.4401712 |
| [75] | Kawai-Yamada M, Jin LH, Yoshinaga K, et al. Mammalian Bax-induced plant cell death can be down-regulated by overexpression of Arabidopsis Bax Inhibitor-1(AtBl-1). Proc Natl Acad Sci USA , 2001, 98 (21) : 12295–12300. DOI:10.1073/pnas.211423998 |
| [76] | Sanchez P, Zabala MD, Grant M. AtBI-1, a plant homologue of Bax Inhibitor-1, suppresses Bax-induced cell death in yeast and is rapidly upregulated during wounding and pathogen challenge. Plant J , 2000, 21 (4) : 393–399. DOI:10.1046/j.1365-313x.2000.00690.x |
| [77] | Coll NS, Vercammen D, Smidler A, et al. Arabidopsis type i metacaspases control cell death. Science , 2010, 330 (6009) : 1393–1397. DOI:10.1126/science.1194980 |
| [78] | Tsiatsiani L, Van Breusegem F, Gallois P, et al. Metacaspases. Cell Death Differ , 2011, 18 (8) : 1279–1288. DOI:10.1038/cdd.2011.66 |
| [79] | Lam E, Zhang Y. Regulating the reapers:activating metacaspases for programmed cell death. Trend Plant Sci , 2012, 17 (8) : 487–494. DOI:10.1016/j.tplants.2012.05.003 |
| [80] | Zhang LR, Xu QX, Xing D, et al. Real-Time detection of caspase-3-like protease activation in vivo using fluorescence resonance energy transfer during plant programmed cell death induced by ultraviolet c overexposure. Plant Physiol , 2009, 150 (4) : 1773–1783. DOI:10.1104/pp.108.125625 |
| [81] | Han JJ, Lin W, Oda Y, et al. The proteasome is responsible for caspase-3-like activity during xylem development. Plant J , 2012, 72 (1) : 129–141. DOI:10.1111/tpj.2012.72.issue-1 |
| [82] | Hatsugai N, Iwasaki S, Tamura K, et al. A novel membrane fusion-mediated plant immunity against bacterial pathogens. Gene Dev , 2009, 23 (21) : 2496–2506. DOI:10.1101/gad.1825209 |
| [83] | Ye Y, Li Z, Xing D. Nitric oxide promotes MPK6-mediated caspase-3-like activation in cadmium-induced Arabidopsis thaliana programmed cell death. Plant Cell Environ , 2013, 36 (1) : 1–15. DOI:10.1111/pce.2013.36.issue-1 |
| [84] | Deng Y, Srivastava R, Srivastava R, et al. Protein kinase and ribonuclease domains of IRE1 confer stress tolerance, vegetative growth, and reproductive development in Arabidopsis. Proc Natl Acad Sci USA , 2013, 110 (48) : 19633–19638. DOI:10.1073/pnas.1314749110 |
| [85] | Li JQ, Zhang JA, Wang XC, et al. A membrane-tethered transcription factor ANAC089 negatively regulates floral initiation in Arabidopsis thaliana. Sci China Life Sci , 2010, 53 (11) : 1299–1306. DOI:10.1007/s11427-010-4085-2 |
| [86] | Li P, Wind JJ, Shi XL, et al. Fructose sensitivity is suppressed in Arabidopsis by the transcription factor ANAC089 lacking the membrane-bound domain. Proc Natl Acad Sci USA , 2011, 108 (8) : 3436–3441. DOI:10.1073/pnas.1018665108 |
| [87] | Liu JX, Srivastava R, Che P, et al. Salt stress responses in Arabidopsis utilize a signal transduction pathway related to endoplasmic reticulum stress signaling. Plant J , 2007, 51 (5) : 897–909. DOI:10.1111/j.1365-313X.2007.03195.x |
| [88] | Liu JX, Srivastava R, Howell SH. Stress-induced expression of an activated form of AtbZIP17 provides protection from salt stress in Arabidopsis. Plant Cell Environ , 2008, 31 (12) : 1735–1743. DOI:10.1111/pce.2008.31.issue-12 |
| [89] | Zhou SF, Sun L, Valdés AE, et al. Membrane-associated transcription factor peptidase, site-2 protease, antagonizes ABA signaling in Arabidopsis. New Phytol , 2015, 208 (1) : 188–97. DOI:10.1111/nph.13436 |
| [90] | Gao HB, Brandizzi F, Benning C, et al. A membrane-tethered transcription factor defines a branch of the heat stress response in Arabidopsis thaliana. Proc Natl Acad Sci USA , 2008, 105 (42) : 16398–16403. DOI:10.1073/pnas.0808463105 |
| [91] | Fujita M, Mizukado S, Fujita Y, et al. Identification of stress-tolerance-related transcription-factor genes via mini-scale Full-length cDNA Over-eXpressor(FOX)gene hunting system. Biochem Biophy Res Commun , 2007, 364 (2) : 250–257. DOI:10.1016/j.bbrc.2007.09.124 |
| [92] | Humbert S, Zhong SH, Deng Y, et al. Alteration of the bZIP60/IRE1 pathway affects plant response to ER stress in Arabidopsis thaliana. PLoS One , 2012, 7 (6) : e39023. DOI:10.1371/journal.pone.0039023 |
| [93] | Sriburi R, Jackowski S, Mori K, et al. XBP1:a link between the unfolded protein response, lipid biosynthesis, and biogenesis of the endoplasmic reticulum. J Cell Biol , 2004, 167 (1) : 35–41. DOI:10.1083/jcb.200406136 |
| [94] | Sriburi R, Jackowski S, Mori K, et al. XBP1(S)and the mechanism of phospholipid biosynthesis. FASEB J , 2006, 20 (5) : A952. |
| [95] | Bommiasamy H, Back SH, Fagone P, et al. ATF6 alpha induces XBP1-independent expansion of the endoplasmic reticulum. J Cell Sci , 2009, 122 (10) : 1626–1636. DOI:10.1242/jcs.045625 |
| [96] | Seo PJ, Kim MJ, Park JY, et al. Cold activation of a plasma membrane-tethered NAC transcription factor induces a pathogen resistance response in Arabidopsis. Plant J , 2010, 61 (4) : 661–671. DOI:10.1111/tpj.2010.61.issue-4 |
| [97] | Lam E, Kato N, Lawton M. Programmed cell death, mitochondria and the plant hypersensitive response. Nature , 2001, 411 (6839) : 848–853. DOI:10.1038/35081184 |
| [98] | Lai ZB, Wang F, Zheng ZY, et al. A critical role of autophagy in plant resistance to necrotrophic fungal pathogens. Plant J , 2011, 66 (6) : 953–968. DOI:10.1111/tpj.2011.66.issue-6 |