2. 河北经贸大学生物科学与工程学院,石家庄 050061
2. College of Biology Science & Engineering, Hebei University of Economics & Business, Shijiazhuang 050061
环境低温,尤其冻害是主要的环境非生物胁迫之一,不但影响植物的地理分布,还对水稻等作物的产量有着重要的影响。我国每年因自然灾害造成的经济损失巨大[1]。随着人口的增长和耕地面积的不断减少,提高作物对恶劣环境的抵抗能力,减少低温对作物的影响,提高作物的产量,对于21世纪粮食安全有着非常重要的意义。
研究发现,经过非冰冻低温处理的植物可以获得对冰冻低温的抵抗能力,这个过程称为冷驯化(cold acclimation)。植物的冷驯化过程包括低温条件下多种细胞内生理和生化过程的改变,其中变化最显著的包括钙离子的瞬时上升[2],生长停止,组织的含水量降低,ABA含量升高,磷脂组成的变化[3],渗透调节物质如脯氨酸、甜菜碱和可溶性糖的积累等[4]。冷驯化产生的分子机制是非冰冻低温能诱导植物表达一系列冷响应蛋白(COLD REGULATED GENES,简称CORs),帮助植物抵抗冰冻低温。冷驯化现象也说明,在非冰冻低温下,植物能够感受环境温度的变化并且通过一系列信号传导机制改变自身的基因表达模式,从而适应改变的环境[5, 6]。随着分子生物学技术的发展,利用模式植物拟南芥为研究材料,人们找到了许多参与冷信号传导途径的信号组分,并且也已经将这些组分组装成一个基本清晰的冷信号调节网络。近年来,随着基因组学和蛋白组学的兴起,其研究结果也在不断地丰富和补充人们对于冷信号转导调控网络的认知。因此在本文中,我们将介绍冷信号传导机制研究的一些新进展,尤其是来自转录组学和蛋白组学的研究成果。
1 冷信号的感知植物能够感知环境温度的变化并通过改变基因表达模式来提高植物对冰冻低温的抵抗能力。但是一直以来,植物感知低温的分子机制都不是非常的清楚。TRP通道蛋白(TRANSIENT RECEPTOR PO-TENTIAL CHANNELS)是动物中的温度感受器[7]。环境温度发生改变可以激活TRP通道,改变跨膜的离子(包括钙离子)运输,起始内吞或外排等细胞过程,调节基因的表达。虽然在植物中没有TRP通道的同源基因,但是植物中是否存在类似于TRP通道的受低温激活的离子通道?最近种康课题组[8]发现水稻质膜上一个具有9次跨膜结构的蛋白COLD1(CHILLING-TOLERANCE DIVERGENCE 1)发生突变后可显著影响水稻对冷的抵抗能力。COLD1可能能够通过三亚基G蛋白来调节胞内钙离子的变化来调节植物的耐冷性。目前还不太清楚COLD1是否本身具备离子通道的活性,以及低温是如何调节COLD1活性的,因此对于COLD1是否能够作为一个低温受体来调节植物的耐冷性还有待进一步的研究。
2 冷驯化对基因表达的调控低温对转录因子CBFs(C-REPEAT BINDING FACTORS)的转录调节被认为在植物冷驯化过程中发挥着极其重要的作用。在拟南芥中,参与冷响应的CBF家族转录因子主要有CBF1、CBF2和CBF3。如果通过CRISPR/Cas9技术将拟南芥中3个CBFs都敲除,发现突变体的基础耐冻性和野生型相比没有区别或者仅有细微差异,但是突变体获得的耐冻性显著降低,说明CBFs的确在植物的冷驯化过程中发挥重要调节作用[9, 10];过表达CBF1-3可以组成型地激活COR基因,且提高植物的基础抗冻性[11, 12]。但是由组成型强启动子驱动CBF的表达会诱导COR基因在正常条件下的表达,导致植物正常条件下生长缓慢,植株变矮小[11, 12]。如果用RD29A(也称为COR78)的启动子启动CBF1的表达既可以有效地提高植物的抗冻性,又不改变植物的生长表型[12]。
CBFs通过作用于核心序列为“RCCGAC”的CRT/DRE(C-REPEAT/DEHYDRATION-RESPONS-IVE ELEMENT)顺式元件上调节下游COR基因的表达,从而增强植物对低温的耐受能力[13-15]。如受CBFs调节的COR15A[16-18]、COR78[19]、COR47[20]和COR6.6[20]所编码的蛋白高度亲水[21]。这些蛋白可以使生物膜在冻害时保持稳定,减少渗漏,并且在复苏时增加膜的可伸展性[21, 22]。此外CBFs调节的其它COR蛋白还参与低温条件下细胞的解毒作用、抗氧化反应、光合作用、木质素代谢,次级代谢、细胞多糖重塑、淀粉代谢、甾醇类激素合成和寡聚糖合成等冷响应过程[23]。因此找到受CBF直接调节的COR基因对于我们解析冷驯化的作用机理具有非常重要的意义。由于CBF1、CBF2和CBF3在拟南芥四号染色体上紧密连锁,难以获得T-DNA插入突变体的双重或者三重突变体。一直以来,科学家们基本都是利用CBF单突变体,CBF的反义抑制或者CBF的过表达转基因植物来研究受CBF调节的COR基因[24-28]。
最近杨淑华课题组[9]和朱健康课题组[10]分别利用CRISPR/Cas9系统构建了将CBF1、CBF2和CBF3同时敲除的突变体,并结合RNAseq的方法,使人们对于冷胁迫条件下受CBFs调节的基因有了一个更加清晰的认识。研究结果发现,拟南芥野生型中约有3 346[9]或4 087[10]个基因的表达水平在冷处理后有2倍以上的变化。同样的处理条件,发现在cbf123三突变体中,约有3 134[9]或者3 659[10]个基因的表达水平受冷胁迫调节。以上两个课题组都发现,只有7%[9]或10%[10]左右的COR基因受CBFs的调节。对cbf123三突变体的基础耐冻性和获得耐冻性的比较也发现,虽然冷驯化后的cbf123三突变体的耐冻能力低于野生型,但要高于没有经过冷驯化的三突变体,说明拟南芥CBF1、CBF2和CBF3缺失的情况下,植物仍然可以感受冷信号,调节COR的表达,提高植物的耐冻性,充分证明CBF之外可能还存在其它的转录因子能够调节COR基因的表达,调节植物冷驯化过程。通过对cbf123突变体与野生型植物冷响应基因的比较,朱健康课题组[10]发现有3种冷信号快速响应的转录因子HSFC1(HEAT SHOCK TRANSCRIPTION FACTOR C1)、ZAT12(ZINC FINGER OF ARABIDOPSIS THALIANA 12)和CZF1(CCCH DOMAIN CONTAINING ZINC FINGER 1)没有受到CBFs敲除的影响,可能是独立于CBFs之外的冷胁迫相关转录因子,而这3种转录因子分别过表达后都能够像CBFs的过表达植物一样,可以上调COR基因的表达[29]。
3 冷信号传导途径拟南芥CBFs的表达水平在低温下处理15 min就会迅速上升,在冷处理1-2 h后表达水平达到最高[30],说明CBFs的转录水平还受到其它转录因子的调节。研究表明,有一系列的转录因子可以直接与CBF的启动子结合,调节CBF的表达[31-34]。在这些转录因子中研究得最为清楚的是转录因子ICE1(INDUCER OF CBF EXPRESSION 1)对CBF的调节。ICE1是一种bHLH类的转录因子,与MYC-2类的顺式元件(CACATG)的结合能力要强于其他类型的MYC元件[31]。在CBF3编码框的上游1 kb以内的序列中含有5个MYC结合位点,而CBF1和CBF2的启动子区各只有一个MYC元件。ICE1功能缺失后能抑制低温促进的CBF3的表达,而对低温促进的CBF1和CBF2的表达基本没有影响[31]。因此人们认为ICE1主要参与冷胁迫对CBF3的表达水平的调节。后续的研究发现,ICE1的同源蛋白ICE2可以参与低温条件下对CBF1的表达调控[35]。
ICE1过表达后只有在低温下才能促进CBFs的表达,说明ICE1的转录活性是受低温调节的[31]。此外,研究还发现低温能够调节ICE1蛋白的稳定性。在冷处理的早期(1 h以内)ICE1蛋白比较稳定,但随着冷处理的时间延长(1 h以后),ICE1的稳定性下降,被蛋白酶体降解[36]。目前发现能够调节ICE1蛋白的冷信号组分有HOS1(HIGH EXPRESSION OF OSMOTICALLY RESPONSIVE GENE 1)[36, 37]、SIZ1(SAP AND MIZ1)[38]和OS-T1(OPEN STOMATA 1)[39]。HOS1是ICE1的E3泛素连接酶,在冷驯化的晚期(0℃处理15 h)能介导ICE1的泛素化从而使ICE1被26S蛋白酶体降解[36]。SIZ1是一种SUMO(SMALL UBIQUITIIN-LIKE MODIFIER)蛋白的E3连接酶,在冷处理时,SIZ1可以介导ICE1的SUMO化。被SUMO化后的ICE1与HOS1相互作用降低,泛素化程度减弱[38]。因此SIZ1是ICE1的正向调节因子,在冷信号的早期起着稳定ICE1蛋白的作用。但是目前还不是非常清楚冷处理的晚期,ICE1稳定性的降低是因为SIZ1的作用受到抑制还是HOS1的作用得到了增强,或者还是有其它的调控机制参与了ICE1的降解。蛋白激酶OST1是植物激素ABA信号途径中的一个重要组分。在正常情况下,OST1激酶和PP2C(PROTEIN PHOSPHATASE 2C)蛋白磷酸酶结合,活性受到抑制。ABA和受体PYR(PYRABATIN RESISTANCE)结合后[40, 41],ABA/PYR复合体和PP2C结合,解除PP2C对OST1的抑制,OST1被激活,磷酸化下游AREBs/ABFs(ABRE-binding proteins/factors)家族转录因子来调节一系列胁迫相关的基因的表达[42]。最近的研究发现:4℃处理30 min就可以显著提高OST1的激酶活性。OST1可以和HOS1竞争性地与ICE1结合,磷酸化ICE1。一方面提高ICE1蛋白的稳定性,另一方面促进ICE1的转录活性,从而激活冷信号的ICE1-CBF-COR通路[39]。Ding[39]的研究还发现冷处理30 min的情况下,植物体内ABA的含量略有减少,说明在该处理时间段内,OST1激酶活性的增加可能与ABA信号无关。PP2C磷酸酶突变体abi1-1是一个显性突变,在该突变体中突变的abi1蛋白和OST1结合非常紧密,不再受ABA/PYR受体复合物的调节[42]。在abi1-1突变体中,冷处理后OST1激酶活性不再增加,说明低温诱导OST1活性的增加同样依赖于PP2C蛋白磷酸酶对OST1抑制作用的解除[39]。目前我们对于低温胁迫如何解除PP2C对OST1的抑制作用还不太清楚,还有待于后续的研究。相信对该抑制解除机制的解析不但有助于人们理解冷信号早期的感知和启动机制,还有助于理解长时间处理后ICE1-CBF-COR冷信号途径被关闭的分子调节机制。
近年来蛋白质组学的兴起,为植物信号传导机制的研究带来了新的手段。与转录组学研究不同,蛋白组学的研究对象是生物功能的直接执行者——蛋白质,因此可以做到实时观测样品处理后蛋白丰度的变化过程。此外蛋白质组学研究还有一个优点就是能够对蛋白的翻译后修饰进行检测,从而对目的蛋白参与某个生物学过程的调控机制进行预测。利用2D-DIGE的蛋白组学方法,汤文强课题组[43]从水稻中找到26个丰度在冷处理5 min后就发生显著变化的冷快速响应蛋白。这些蛋白涉及到细胞代谢、胁迫响应、蛋白折叠、骨架重排和磷脂代谢等生物学过程。从这些蛋白中发现磷酯酶OsPLDα1的蛋白含量在冷处理5 min后能上升4倍左右。功能研究表明,OsPLDα1是水稻冷信号传导早期的重要组分之一,可能通过CBF途径调节COR基因的表达[43]。OsPLDα1能水解磷脂酰胆碱(Phosphatidylcholine)产生磷脂酸分子(Phosphatidic acid,简称PA)。PA可以与冷信号传导上游的ABI1[44]、MPK6[43, 45]、SIZ1[43]等结合,可能通过调节这些蛋白的亚细胞定位或者蛋白的活性来调节CBF的表达,从而调节植物的冷驯化过程。根据该研究成果和最近植物冷驯化机制的研究进展,我们总结整理了一个植物冷信号传导途径的模式图(图 1)。
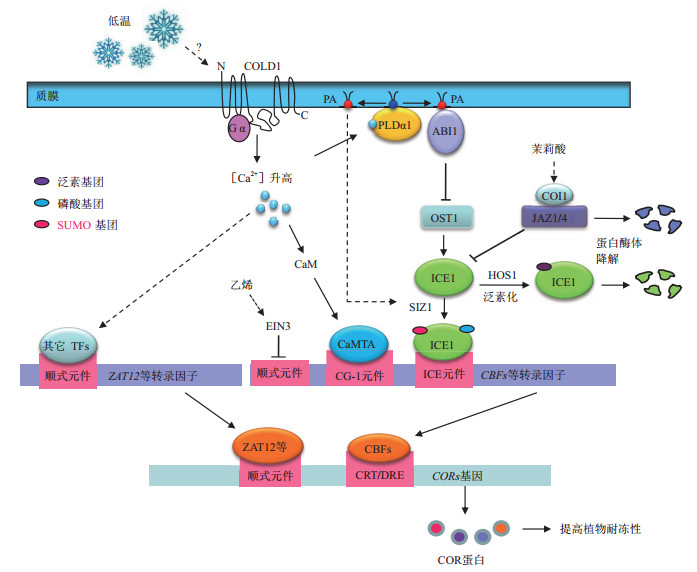
|
| 图 1 冷信号传导途径模式图 在植物受到冷刺激的情况下,跨膜蛋白COLD1通过未知机制感知冷信号后,与G蛋白(Gα)相互作用,激活质膜钙通道,促进胞内钙离子浓度上升。钙离子可以通过钙调素(CaM)激活CAMTA转录因子来调节转录因子CBFs的表达。此外钙离子还可以激活PLDα 1,从而产生各种类型的PA信号分子。PA一方面可能通过与SIZ1结合而影响SIZ1的定位或活性;另一方面可以与ABI1结合,将ABI1锚定在膜上,解除ABI1对于OST1活性的抑制。OST1激活后可以磷酸化ICE1,抑制ICE1和HOS1的相互作用,稳定ICE1蛋白的同时激活ICE1的转录活性。ICE1与CBF启动子上的ICE-box顺式元件结合促进CBF的表达。冷处理条件下,转录因子CBFs可以与CORs基因的CRT/DRE元件结合启动CORs基因的表达,增加植物对冷冻胁迫的抵抗能力。此外低温还可以通过未知的机制调节ZAT12等转录因子的表达和活性,以CBFs不依赖的方式调节CORs基因的表达。而植物激素茉莉酸和乙烯信号途径则可以分别通过调节JAZ1/4的降解来解除JAZ1/4对于ICE1的抑制作用,以及通过EIN3抑制CBF的表达 |
如上所述,虽然ICE1-CBF-COR信号通路的主要元件都已被克隆和进行功能验证,但是冷信号传导通路并不是单线传导的方式。朱健康课题组[46]此前的研究发现,与野生型相比,只有约40%的COR基因的表达水平在ice1突变体中发生了改变,说明ICE1-CBF-COR途径之外还存在其它的冷信号传导途径。此外,人们还发现许多其它的转录因子包括CAMTA3(CALMODULIN BINDING TRANSCRIPTION ACTIVATOR 3)[33]、ZAT12[47]和MYB15(MYB DOMAIN PROTEIN 15)[32]等都能够调节CBFs的表达。其中CAMTA3是一种受钙调素调节的转录因子。由于冷胁迫处理能够快速诱导植物胞内钙离子的升高[2]。升高的钙离子可以激活钙调素,通过调节CAMTA3的转录活性来调节CBF1和CBF2的表达[33]。此外,CBF的表达水平以及ICE1的转录活性还可以受EIN3(ETHYLENE INSE-NSITIVE 3)[34]和JAZ1/4(JASMONATE ZIM DOM-AIN PROTEIN 1/4)[48]等植物激素信号传导组分的调节,说明ICE1-CBF调控模块不仅是冷信号传导的重要组分,还是其他胁迫信号及植物生长发育信号调节植物冷胁迫反应的核心元件。
5 展望近年来,人们对于植物冷驯化信号传导的分子机制有了许多突破性的进展。但是从图 1中可以看出,目前我们对冷驯化信号传导机制主要还是围绕着ICE1-CBF-COR途径进行的。最近的研究结果表明,CBF和ICE1只参与一小部分COR基因的表达调控,相信今后对CBF以及ICE不依赖的冷驯化信号传导机制的研究将会逐渐占据主导地位,并丰富我们对于植物冷驯化生物学过程的理解。植物对低温的感受机制以及早期冷信号的传递机制也将是今后冷信号传导研究的热点问题之一。由于蛋白质组学研究方法的优点和此前利用蛋白组学找到植物信号传导新组分的成功案例[49],相信诸如N15和TMT(Tandem Mass Tag)标记的定量蛋白质组学方法的应用能够为解析早期植物冷信号传导的分子机制作出重要的贡献。另外,此前的研究,包括对cbf123三突变体的RNAseq研究,冷胁迫的处理时间都是几个小时甚至24 h[9, 10]。长时间的冷胁迫处理必将导致转录因子对基因表达的二级或者多级表达调控,而不能真正显示受冷信号传导组分直接调节的基因。前期的研究也表明,CBF的表达水平在冷处理15 min后就可以显著升高[30];而蛋白质组学的研究结果也显示,对植物进行5 min的冷处理就足以导致一批早期冷响应蛋白的丰度发生变化[43],说明冷信号传导事件在冷处理15 min就已经完成或者部分完成。因此,今后的冷信号传导机理尤其是早期冷信号传导途径的研究需要注意将冷处理的时间缩短到1 h以内,甚至15 min以内,这样所得到的研究结果才能够真正体现冷信号传递发生的过程,而不是体现一种长时间冷处理后,由转录因子级联调控导致的差异基因表达的结果。
| [1] | 施雅风. 全球变暖影响下中国自然灾害的发展趋势. 自然灾害学报 , 1996, 5 : 102–117. |
| [2] | Carpaneto A, Ivashikina N, Levchenko V, et al. Cold transiently activates calcium-permeable channels in Arabidopsis mesophyll cells. Plant Physiol , 2007, 143 : 487–494. |
| [3] | Webb MS, Uemura M, Steponkus PL. A comparison of freezing injury in oat and rye: two cereals at the extremes of freezing tolerance. Plant Physiol , 1994, 104 : 467–478. |
| [4] | Krasensky J, Jonak C. Drought, salt, and temperature stress-induced metabolic rearrangements and regulatory networks. J Exp Bot , 2012, 63 : 1593–1608. DOI:10.1093/jxb/err460 |
| [5] | Thomashow MF. PLANT COLD ACCLIMATION: Freezing tolerance genes and regulatory mechanisms. Annu Rev Plant Physiol Plant Mol Biol , 1999, 50 : 571–599. DOI:10.1146/annurev.arplant.50.1.571 |
| [6] | Viswanathan C, Zhu JK. Molecular genetic analysis of cold-regulated gene transcription. Philos Trans R Soc Lond B Biol Sci , 2002, 357 : 877–886. DOI:10.1098/rstb.2002.1076 |
| [7] | Zheng J. Molecular mechanism of TRP channels. Compr Physiol , 2013, 3 : 221–242. |
| [8] | Ma Y, Dai X, Xu Y, et al. COLD1 confers chilling tolerance in rice. Cell , 2015, 160 : 1209–1221. DOI:10.1016/j.cell.2015.01.046 |
| [9] | Jia Y, Ding Y, Shi Y, et al. The cbfs triple mutants reveal the essential functions of CBFs in cold acclimation and allow the definition of CBF regulons in Arabidopsis. New Phytol , 2016, 212 (2) : 345–353. DOI:10.1111/nph.14088 |
| [10] | Zhao C, Zhang Z, Xie S, et al. Mutational evidence for the critical role of CBF genes in cold acclimation in Arabidopsis. Plant Physiol , 2016, 171 : 2744–2759. |
| [11] | Jaglo-Ottosen KR, Gilmour SJ, Zarka DG, et al. Arabidopsis CBF1 overexpression induces COR genes and enhances freezing tolerance. Science , 1998, 280 : 104–106. DOI:10.1126/science.280.5360.104 |
| [12] | Gilmour SJ, Fowler SG, Thomashow MF. Arabidopsis transcriptional activators CBF1, CBF2, and CBF3 have matching functional activities. Plant Mol Biol , 2004, 54 : 767–781. DOI:10.1023/B:PLAN.0000040902.06881.d4 |
| [13] | Stockinger EJ, Gilmour SJ, Thomashow MF. Arabidopsis thaliana CBF1 encodes an AP2 domain-containing transcriptional activator that binds to the C-repeat/DRE, a cis-acting DNA regulatory element that stimulates transcription in response to low temperature and water deficit. Proc Natl Acad Sci USA , 1997, 94 : 1035–1040. DOI:10.1073/pnas.94.3.1035 |
| [14] | Liu Q, Kasuga M, Sakuma Y, et al. Two transcription factors, DREB1 and DREB2, with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in drought-and low-temperature-responsive gene expression, respectively, in Arabidopsis. Plant Cell , 1998, 10 : 1391–1406. DOI:10.1105/tpc.10.8.1391 |
| [15] | Sakuma Y, Liu Q, Dubouzet JG, et al. DNA-binding specificity of the ERF/AP2 domain of Arabidopsis DREBs, transcription factors involved in dehydration-and cold-inducible gene expression. Biochem Biophys Res Commun , 2002, 290 : 998–1009. DOI:10.1006/bbrc.2001.6299 |
| [16] | Lin C, Thomashow MF. DNA sequence analysis of a complementary dna for cold-regulated Arabidopsis gene cor15 and characterization of the COR 15 polypeptide. Plant Physiol , 1992, 99 : 519–525. DOI:10.1104/pp.99.2.519 |
| [17] | Artus NN, Uemura M, Steponkus PL, et al. Constitutive expression of the cold-regulated Arabidopsis thaliana COR15a gene affects both chloroplast and protoplast freezing tolerance. Proc Natl Acad Sci USA , 1996, 93 : 13404–13409. DOI:10.1073/pnas.93.23.13404 |
| [18] | Steponkus PL, Uemura M, Joseph RA, et al. Mode of action of the COR15a gene on the freezing tolerance of Arabidopsis thaliana. Proc Natl Acad Sci USA , 1998, 95 : 14570–14575. DOI:10.1073/pnas.95.24.14570 |
| [19] | Yamaguchi-Shinozaki K, Shinozaki K. Characterization of the expression of a desiccation-responsive rd29 gene of Arabidopsis thaliana and analysis of its promoter in transgenic plants. Mol Gen Genet , 1993, 236 : 331–340. |
| [20] | Gilmour SJ, Artus NN, Thomashow MF. cDNA sequence analysis and expression of two cold-regulated genes of Arabidopsis thaliana. Plant Mol Biol , 1992, 18 : 13–21. DOI:10.1007/BF00018452 |
| [21] | Thomashow MF. Role of cold-responsive genes in plant freezing tolerance. Plant Physiol , 1998, 118 : 1–8. DOI:10.1104/pp.118.1.1 |
| [22] | Hundertmark M, Hincha DK. LEA(late embryogenesis abundant)proteins and their encoding genes in Arabidopsis thaliana. BMC Genomics , 2008, 9 : 118. DOI:10.1186/1471-2164-9-118 |
| [23] | Janska A, Marsik P, Zelenkova S, Ovesna J. Cold stress and acclimation-what is important for metabolic adjustment?. Plant Biol(Stuttgart, Germany) , 2010, 12 : 395–405. DOI:10.1111/plb.2010.12.issue-3 |
| [24] | Seki M, Narusaka M, Abe H, et al. Monitoring the expression pattern of 1300 Arabidopsis genes under drought and cold stresses by using a full-length cDNA microarray. Plant Cell , 2001, 13 : 61–72. DOI:10.1105/tpc.13.1.61 |
| [25] | Novillo F, Alonso JM, Ecker JR, Salinas J. CBF2/DREB1C is a negative regulator of CBF1/DREB1B and CBF3/DREB1A expression and plays a central role in stress tolerance in Arabidopsis. Proc Natl Acad Sci USA , 2004, 101 : 3985–3990. DOI:10.1073/pnas.0303029101 |
| [26] | Kim YS, Lee M, Lee JH, et al. The unified ICE-CBF pathway provides a transcriptional feedback control of freezing tolerance during cold acclimation in Arabidopsis. Plant Mol Biol , 2015, 89 : 187–201. DOI:10.1007/s11103-015-0365-3 |
| [27] | Fowler S, Thomashow MF. Arabidopsis transcriptome profiling indicates that multiple regulatory pathways are activated during cold acclimation in addition to the CBF cold response pathway. Plant Cell , 2002, 14 : 1675–1690. DOI:10.1105/tpc.003483 |
| [28] | Maruyama K, Sakuma Y, Kasuga M, et al. Identification of cold-inducible downstream genes of the Arabidopsis DREB1A/CBF3 transcriptional factor using two microarray systems. Plant J , 2004, 38 : 982–993. DOI:10.1111/tpj.2004.38.issue-6 |
| [29] | Park S, Lee CM, Doherty CJ, et al. Regulation of the Arabidopsis CBF regulon by a complex low-temperature regulatory network. Plant J , 2015, 82 : 193–207. DOI:10.1111/tpj.12796 |
| [30] | Gilmour SJ, Zarka DG, Stockinger EJ, et al. Low temperature regulation of the Arabidopsis CBF family of AP2 transcriptional activators as an early step in cold-induced COR gene expression. Plant J , 1998, 16 : 433–442. DOI:10.1046/j.1365-313x.1998.00310.x |
| [31] | Chinnusamy V, Ohta M, Kanrar S, et al. ICE1: a regulator of cold-induced transcriptome and freezing tolerance in Arabidopsis. Genes Dev , 2003, 17 : 1043–1054. DOI:10.1101/gad.1077503 |
| [32] | Agarwal M, Hao Y, KapoorA, et al. A R2R3 type MYB transcription factor is involved in the cold regulation of CBF genes and in acquired freezing tolerance. J Biol Chem , 2006, 281 : 37636–37645. DOI:10.1074/jbc.M605895200 |
| [33] | Doherty CJ, Van Buskirk HA, Myers SJ, Thomashow MF. Roles for Arabidopsis CAMTA transcription factors in cold-regulated gene expression and freezing tolerance. Plant Cell , 2009, 21 : 972–984. DOI:10.1105/tpc.108.063958 |
| [34] | Shi Y, Tian S, Hou L, et al. Ethylene signaling negatively regulates freezing tolerance by repressing expression of CBF and type-A ARR genes in Arabidopsis. Plant Cell , 2012, 24 : 2578–2595. DOI:10.1105/tpc.112.098640 |
| [35] | Fursova OV, Pogorelko GV, Tarasov VA. Identification of ICE2, a gene involved in cold acclimation which determines freezing tolerance in Arabidopsis thaliana. Gene , 2009, 429 : 98–103. DOI:10.1016/j.gene.2008.10.016 |
| [36] | Dong CH, Agarwal M, Zhang Y, et al. The negative regulator of plant cold responses, HOS1, is a RING E3 ligase that mediates the ubiquitination and degradation of ICE1. Proc Natl Acad Sci USA , 2006, 103 : 8281–8286. DOI:10.1073/pnas.0602874103 |
| [37] | Lee H, Xiong L, Gong Z, et al. The Arabidopsis HOS1 gene negatively regulates cold signal transduction and encodes a RING finger protein that displays cold-regulated nucleo--cytoplasmic partitioning. Genes Dev , 2001, 15 : 912–924. DOI:10.1101/gad.866801 |
| [38] | Miura K, Jin JB, Lee J, et al. SIZ1-mediated sumoylation of ICE1 controls CBF3/DREB1A expression and freezing tolerance in Arabidopsis. Plant Cell , 2007, 19 : 1403–1414. DOI:10.1105/tpc.106.048397 |
| [39] | Ding Y, Li H, Zhang X, et al. OST1 kinase modulates freezing tolerance by enhancing ICE1 stability in Arabidopsis. Dev Cell , 2015, 32 : 278–289. DOI:10.1016/j.devcel.2014.12.023 |
| [40] | Ma Y, Szostkiewicz I, Korte A, et al. Regulators of PP2C phosphatase activity function as abscisic acid sensors. Science , 2009, 324 : 1064–1068. |
| [41] | Park SY, Fung P, Nishimura N, et al. Abscisic acid inhibits type 2C protein phosphatases via the PYR/PYL family of START proteins. Science , 2009, 324 : 1068–1071. |
| [42] | Umezawa T, Sugiyama N, Mizoguchi M, et al. Type 2C protein phosphatases directly regulate abscisic acid-activated protein kinases in Arabidopsis. Proc Natl Acad Sci USA , 2009, 106 : 17588–17593. DOI:10.1073/pnas.0907095106 |
| [43] | Huo C, Zhang B, Wang H, et al. Comparative study of early cold-regulated proteins by two dimensional difference gel electrophoresis reveals a key role for phospholipase Dα1 in mediating cold acclimation signaling pathway in rice. Molecular & Cellular Proteomics , 2016, 15 : 1397–1411. |
| [44] | Zhang W, Qin C, Zhao J, Wang X. Phospholipase D alpha 1-derived phosphatidic acid interacts with ABI1 phosphatase 2C and regulates abscisic acid signaling. Proc Natl Acad Sci USA , 2004, 101 : 9508–9513. DOI:10.1073/pnas.0402112101 |
| [45] | Yu L, Nie J, Cao C, et al. Phosphatidic acid mediates salt stress response by regulation of MPK6 in Arabidopsis thaliana. New Phytol , 2010, 188 : 762–773. DOI:10.1111/j.1469-8137.2010.03422.x |
| [46] | Lee BH, Henderson DA, Zhu JK. The Arabidopsis cold-responsive transcriptome and its regulation by ICE1. Plant Cell , 2005, 17 : 3155–3175. DOI:10.1105/tpc.105.035568 |
| [47] | Vogel JT, Zarka DG, Van Buskirk HA, et al. Roles of the CBF2 and ZAT12 transcription factors in configuring the low temperature transcriptome of Arabidopsis. Plant J , 2005, 41 : 195–211. |
| [48] | Hu Y, Jiang L, Wang F, Yu D. Jasmonate regulates the INDUCER OF CBF EXPRESSION-C-REPEAT BINDING FACTOR/DRE BINDING FACTOR1 cascade and freezing tolerance in Arabidopsis. Plant Cell , 2013, 25 : 2907–2924. DOI:10.1105/tpc.113.112631 |
| [49] | Tang W, Kim TW, Oses-Prieto JA, et al. BSKs mediate signal transduction from the receptor kinase BRI1 in Arabidopsis. Science , 2008, 321 : 557–560. DOI:10.1126/science.1156973 |




