2. 广东省林业科学研究院 广东省森林病虫害生物防治重点实验室,广州 510520
2. Guangdong Provincial Key Laboratory of Bio-control for the Forest Disease and Pest, Guangdong Academy of Forestry, Guangzhou 510520
杉木[Cunninghamia lanceolata(Lamb.)Hook]是中国特有的造林与用材树种,具有速生、材质优良、用途广等特点[1, 2]。近年来,国内多个科研院所、高校甚至是地方林业生产单位不断强化杉木优良无性系选育与快繁技术研究并取得可喜的进展。为提高繁育系数,一些研究团队还探索建立了适于杉木无性系扩繁的组织培养技术,为其规模化繁育与推广奠定了基础[3-6]。然而,为更好地应用这些组培无性系,有必要对它们的抗逆特性尤其是耐旱性进行综合测评。目前,有关杉木优良无性系耐旱性/抗旱性评价的报道尚少[7-8]。
不同杉木无性系形态相近,依靠形态特征难以对无性系种质进行准确地鉴别,而利用DNA分子标记技术构建不同无性系指纹图谱是进行种质鉴别最有效的手段。目前,适于杉木遗传多态性研究的DNA标记技术包括RAPD(random amplified polymorphic DNA)、AFLP(amplified fragment length polymorphism)、ISSR(inter simple sequence repeat)、SSR(simple sequence repeat)以及SRAP(sequence-related amplified polymorphism)等[9]。其中,针对功能序列扩增的SRAP技术具有简单、高效、稳定、部分共显性、扩增片段易测序等特点,优势明显[9-12],但利用SRAP技术构建杉木无性系指纹图谱以进行种质鉴别的研究尚未见报道。
本研究以6个杉木优良无性系组培移栽苗为试材,采用聚乙二醇(PEG-6000)溶液培养法模拟干旱胁迫,对不同无性系耐旱相关生理生化指标进行测定分析并运用隶属函数法对参试无性系耐旱性作出综合评价,以期为耐旱无性系的选用提供参考。同时,利用SRAP技术对无性系DNA分子多态性进行检测并构建不同无性系的DNA-SRAP数字指纹图谱,为无性系种质鉴别及品种保护提供依据。
1 材料与方法 1.1 材料以广东省林业科学研究院选育的6个杉木优良无性系组培移栽苗(无性系编号:T-c01、T-c04、T-c07、T-c08、T-c22、T-cF1)为试材,苗龄1年,培养基质为新鲜黄心土,苗木生长势较为一致。
1.2 方法 1.2.1 模拟干旱处理参考李树斌等[8]的方法。将参试苗木从基质中除出,用清水将根部洗净,随后移入盛有1/2 Hoagland营养液的塑料桶(外壁套上黑色塑料袋)内水培驯化2 d。随后,将试验苗木移入到-2.0 MPa聚乙二醇(PEG-6000)溶液(用1/2 Hoagland营养液配制PEG-6000溶液,PEG-6000浓度为300 g/L)进行模拟干旱胁迫处理(T),每个无性系苗木处理3个重复,每个重复3株苗,同时以1/2 Hoagland营养液培养的苗木作为对照。处理和对照试样在23℃、光周期为16/8 h光照/黑暗的培养箱内培养,24 h后,部分无性系出现脱水萎蔫现象,剪取每个重复苗木株高1/2以上部分的叶片,将每个重复3株苗木叶片进行混合作为1个待测样品,随后进行各项生理生化指标测定。
1.2.2 生理生化指标测定叶片相对含水量(RWC)测定即称取鲜叶质量后用蒸馏水浸泡24 h,随后称饱和鲜质量,最后105℃下烘干,称干质量,根据公式计算RWC。RWC=[(鲜叶质量-干质量)/(饱和鲜质量-干质量)]×100%。丙二醛(MDA)含量、脯氨酸(PRO)含量及超氧化物歧化酶(SOD)、过氧化物酶(POD)、过氧化氢酶(CAT)活性测定均采用苏州科铭生物技术有限公司提供的试剂盒进行,具体操作步骤参考说明书。
1.2.3 基因组总DNA提取采用天根生化科技(北京)有限公司提供的新型植物基因组DNA提取试剂盒(DNAsecure Plant Kit)提取杉木无性系叶片基因组总DNA,具体操作步骤参考说明书。采用1%琼脂糖凝胶电泳和紫外分光光度计测定DNA的质量和浓度,并将浓度稀释至100 ng/μL,于-20℃冰箱内保存、备用。
1.2.4 SRAP-PCR扩增采用前期建立的适于杉木SRAP-PCR扩增的反应体系及引物进行[12],反应体系25 µL,包括0.5 µL基因组总DNA(约50 ng)溶液、0.5 µL上游引物(10 µmol/L)、0.5 µL下游引物(10 µmol/L)、12.5 µL 2 × Taq Plus PCR MasterMix(购自天根生化科技有限公司)及11 µL双蒸水。扩增程序参照Li和Quiros[10]的通用程序进行,即94℃预变性5 min;94℃变性1 min,35℃退火1 min,72℃延伸1 min,5个循环;94℃变性1 min,50℃退火1 min,72℃延伸1 min,35个循环;72℃延伸10 min;4℃保温。PCR扩增完毕后,以D2000 DNA Marker(购自天根生化科技有限公司)为参照,对扩增产物作2%(g/v)琼脂糖凝胶电泳检测,用紫外凝胶成像系统显示结果,并照相记录。
1.2.5 数据处理与分析采用Excel 2010和SAS v8.1软件对生理生化指标数据进行统计分析。应用隶属函数法对各无性系耐旱性进行综合排序,计算各无性系各指标的隶属函数值,当测定指标与耐旱性为正相关时,隶属函数值计算采用公式:
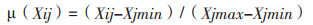 (1)
(1)
当测定指标与耐旱性为负相关时,隶属函数值计算采用公式:
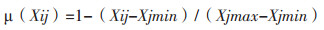 (2)
(2)
式中,μ(Xij):i无性系j性状的耐旱隶属函数值,Xij:i无性系j性状值,Xjmin:各无性系j性状中最小值,Xjmax:各无性系j性状中最大值。
随后求出各指标的隶属函数值综合平均值,平均值越大,耐旱性越强。
SRAP数据获取与分析:对SRAP-PCR电泳谱带进行记录,清晰的SRAP条带记为1,无带则记为0(紫外凝胶成像系统成像后的图片均经过反复的亮度和对比度调试,确认谱带的存在与缺失,及可辨性,对模糊不清、不可辨认的位点均按无带处理)。运用NTSYSpc v2.10e分析软件计算不同无性系间遗传相似系数。指纹图谱置信概率计算采用公式:C=1-1/2n,式中n为多态性条带数[13, 14]。
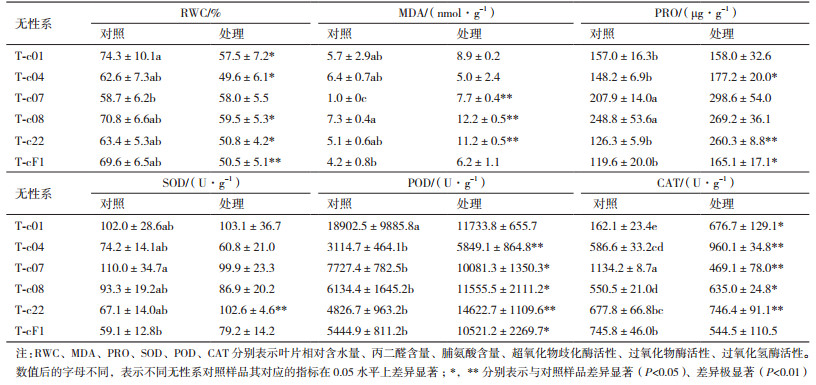
2 结果 2.1 模拟干旱条件下杉木无性系生理生化响应6个杉木无性系在干旱胁迫条件下叶片各生理生化指标均发生变化,但变化程度各异(表 1)。T-c01、T-c04、T-c08、T-c22、T-cF1等5个无性系叶片相对含水量(RWC)明显下降(P < 0.05或P < 0.01),而T-c07、T-c08、T-c22的丙二醛(MDA)含量增幅显著(P < 0.01),此时T-c04、T-c22和T-cF1脯氨酸含量(PRO)也显著提高(P < 0.05或P < 0.01)。在超氧化物歧化酶活性(SOD)方面,仅T-c22出现较明显的变化(P < 0.01);与对照相比,无性系T-c04、T-c07、T-c08、T-c22、T-cF1叶片过氧化物酶(POD)活性大幅提高(P < 0.05或P < 0.01),其中以T-c22变化最为显著,POD活性是对照的3.0倍。另外,不同无性系在干旱胁迫下调控过氧化氢酶(CAT)活性方式上有所差异,如无性系T-c01、T-c04、T-c08和T-c22 CAT活性因干旱胁迫呈上调模式(P < 0.05或P < 0.01),而无性系T-c07和T-cF1 CAT活性则表现为下调。事实上,参试的6个无性系在干旱胁迫下其生理生化指标综合调控模式也存在差异,如T-c01在RWC、MDA、PRO、SOD、POD、CAT活性分别表现为:下调-上调-上调-上调-下调-上调模式,而T-c08则表现为下调-上调-上调-下调-上调-上调模式。值得一提的是,各无性系在非干旱条件下(对照)其生理生化指标同样存在差异,表明参试的6个无性系自身的预抗性方面也有所不同。
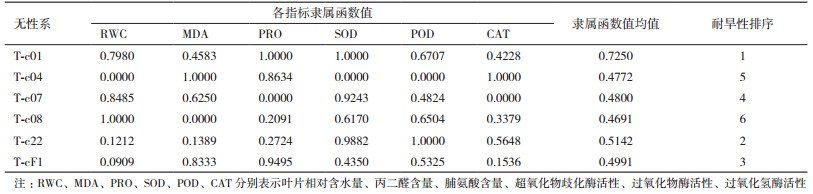
采用隶属函数法对参试无性系耐旱性进行综合评价。各无性系RWC、MDA、PRO、SOD、POD和CAT隶属函数值详见表 2,求取各无性系各指标隶属函数值均值发现,无性系T-c01耐旱性最强,而T-c08较差,6个无性系耐旱性排序为:T-c01 > T-c22 > T-cF1 > T-c07 > T-c04 > T-c08。
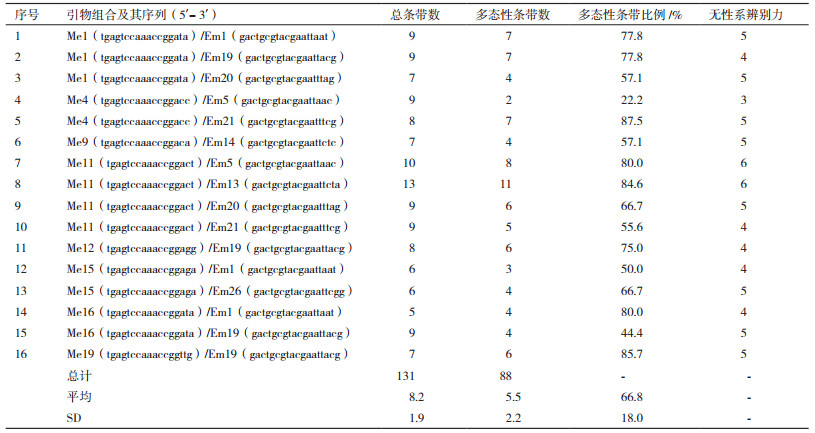
选取16对SRAP引物组合对供试的6个杉木无性系基因组DNA进行PCR扩增,共检测到131个清晰、稳定的谱带,其中多态性条带88条,多态性条带比例达66.8%(表 3)。每对引物组合可扩增出5-13条条带,平均8.2条;其中多态性条带2-11条,平均5.5条,多态性条带比例介于22.2%(引物组合Me4/Em5)-87.5%(引物组合Me4/Em21)间。不同引物组合对无性系的辨别力也有所差异,例如,利用引物组合Me11/Em5、Me11/Em13进行PCR扩增,均可将无性系一一辨清,但采用引物组合Me4/Em5则只能分辨出3类种质:(1)T-c01、T-c08、T-cF1;(2)T-c04和T-c07;(3)T-c22。
基于各无性系基因组DNA SRAP-PCR多态性信息,运用NTSYSpc v2.10e软件计算不同无性系间遗传相似系数发现(表 4),6个无性系遗传相似性(DICE系数)介于0.6909(无性系T-c07和T-c22)-0.9133(无性系T-c04和T-c07)间,但无性系相似性多数(60.0%)在0.7000-0.8000范围内。
根据多态性条带比例≥75%、无性系辨别力≥5、差异条带大小和亮度易辨且在不同无性系内分布均匀、所用引物组合类型多样等原则,从现有16对引物组合中选取3对引物组合(Me1/Em1+Me4/Em21+Me11/Em5)构建无性系指纹图谱(图 1),经数字化后,形成了无性系DNA-SRAP身份证号码,详见表 5。因3对引物组合多态性条带总数22,根据概率公式1/2n可知,在222=4 194 304种无性系中才有可能存在2个无性系电泳谱带完全相同,指纹图谱置信概率为99.99998%。因此,利用该指纹图谱能够准确地检测出其中任何一个无性系。
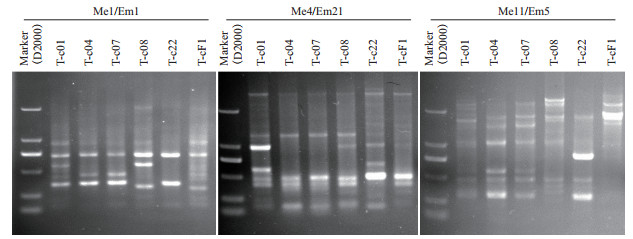
|
| 图 1 杉木无性系SRAP-PCR扩增谱带(引物组合: Me1/Em1、 Me4/Em21、 Me11/Em5) |
与其它树种相比,杉木属中等耐旱/抗旱类型[15-17]。谭建伟和陈建新[18]对杉木全分布区种源试验林气孔阻抗、蒸腾强度、根部形态及旱季生长率进行分析还发现,不同种源杉木抗旱生理、形态和生长性状差异显著,种源间抗旱力存在明显的遗传分化。而何贵平和陈益泰[19]测定了抗旱较强杉木家系和较差家系在干旱胁迫下的脯氨酸含量,结果表明不同家系抗旱性与脯氨酸累积量没有一定的相关性。在无性系水平,刘爱琴等[20]的研究显示,参试的杉木无性系对干旱胁迫反应敏感,不同无性系生理反应(无性系水势、蒸腾速率、叶绿素含量、NR活力下降、自然饱和亏、脯氨酸含量)差异明显。李树斌等[8]则采用PEG溶液培养法模拟干旱胁迫,比较了10个速生杉木无性系在干旱条件下叶片相对含水量、叶片蒸腾速率、叶片保水力、叶绿素荧光参数的变化模式,也发现不同无性系抗旱性存在较显著的差异。事实上,植物的耐旱性/抗旱性是一个综合体现。本研究同样采用PEG溶液培养法模拟干旱胁迫,当部分无性系出现脱水萎蔫现象时(培养24 h后),旋即对参试无性系RWC、MDA、PRO及保护酶系统(SOD、POD和CAT)进而测定分析,在此基础上运用隶属函数法对无性系耐旱性做出快速、综合评价(排序)。因参试无性系均为新选育并成功实现规模化组培扩繁的品系,应用潜力大,对其耐旱性做出评价显然为这些无性系下一步大规模推广应用提供重要参考。值得一提的是,不同无性系在干旱胁迫下其生理生化指标调控模式有所差异,而事实上各无性系自身的预抗性方面也有所不同,反映出不同无性系耐旱力存在一定程度的遗传分化。根据隶属函数值均值可知,无性系T-c01耐旱性较强,而T-c08耐旱性较弱。
为快速、准确鉴别各无性系,本研究利用前期建立的SRAP分子标记技术构建各无性系指纹图谱[12],其操作程序简单,稳定性强,有效降低了多种试剂操作,包括Mg2+、dNTPs、DNA聚合酶、PCR buffer、引物等对扩增结果的影响[21, 22];同时,扩增结果可采用2%(g/v)琼脂糖凝胶进行电泳检测,操作简便、效果理想且成本低。而选用其中3对引物组合(Me1/Em1+Me4/Em21+Me11/Em5)可准确鉴别出各无性系(T-c01、T-c04、T-c07、T-c08、T-c22、T-cF1),置信概率达99.99998%。为区分外观相近的4个白杨新品种及其父母本和3个近缘种,樊蓉等[23]同样利用SRAP标记技术构建不同品种的DNA-SRAP指纹图谱,效果也较为理想。另外,李婷婷等[24]联用毛细管电泳技术(CE)构建了不同来源的榉树优良单株CE-SRAP指纹图谱,为优良单株无性系分子鉴定、遗传资源管理及品种保护提供了参考依据。本研究则为SRAP技术应用于杉木指纹图谱构建及无性系种质鉴别方面提供了例证。
4 结论本研究采用PEG溶液培养法模拟干旱胁迫,对具有较大应用潜力的6个杉木优良无性系耐旱性进行综合测评,不同无性系耐旱性排序为:T-c01 > T-c22 > T-cF1 > T-c07 > T-c04 > T-c08。应用SRAP分子标记技术对参试无性系遗传多态性进行检测并构建了无性系的DNA-SRAP数字指纹图谱,为无性系种质鉴别和品种保护提供了分子依据。
| [1] | 陈岳武, 施季森. 杉木遗传改良中的若干基本问题. 南京林业大学学报:自然科学版 , 1983, 25 (4) : 5–18. |
| [2] | 郑会全, 梁瑞友, 胡德活, 等. 杉木大径材优树选择与主要经济性状变异分析. 西南林业大学学报 , 2012, 32 (4) : 26–29. |
| [3] | 欧阳磊, 郑仁华, 翁玉榛, 等. 杉木优良无性系组培快繁技术体系的建立. 南京林业大学学报:自然科学版 , 2007, 31 (3) : 47–51. |
| [4] | 陈琴, 黄开勇, 蓝肖, 等. 我国杉木组织培养技术研究进展. 世界林业研究 , 2012, 25 (6) : 58–63. |
| [5] | 张玫瑰, 马祥庆, 陈奶莲, 等. 杉木组织培养研究进展. 福建林业科技 , 2013, 40 (2) : 192–198. |
| [6] | 王港, 陈骏, 侯娜, 等. 杉木无性系规模化组培繁育技术研究. 湖北林业科技 , 2014, 43 (5) : 7–9. |
| [7] | 刘爱琴, 马祥庆, 徐凤兰, 等. 杉木无性系抗旱性层次选择. 中南林业科技大学学报 , 1998, 18 (2) : 24–26. |
| [8] | 李树斌, 丁国昌, 曹光球, 等. 不同杉木无性系模拟胁迫下抗旱性综合评价. 福建农林大学学报:自然科学版 , 2012, 41 (5) : 491–496. |
| [9] | Zheng HQ, Duan HJ, Hu DH, et al. Genotypic variation of Cunninghamia lanceolata revealed by phenotypic traits and SRAP markers. Dendrobiology , 2015, 74 : 85–94. DOI:10.12657/denbio.074.009 |
| [10] | Li G, Quiros CF. Sequence-related amplified polymorphism(SRAP), a new marker system based on a simple PCR reaction: its application to mapping and gene tagging in Brassica. Theoretical and Applied Genetics , 2001, 103 : 455–461. DOI:10.1007/s001220100570 |
| [11] | Robarts DW, Wolfe AD. Sequence-related amplified polymorphism(SRAP)markers: A potential resource for studies in plant molecular biology(1.)[J]. Applications in Plant Sciences, 2014, 2(7)pii: apps. 1400017. |
| [12] | Zheng HQ, Duan HJ, Hu DH, et al. Sequence-related amplified polymorphism primer screening on Chinese fir(Cunninghamia lanceolata(Lamb.)Hook). Journal of Forestry Research , 2015, 26 (1) : 101–106. DOI:10.1007/s11676-015-0025-0 |
| [13] | 吴渝生, 杨文鹏, 郑用琏. 3个玉米杂交种和亲本SSR指纹图谱的构建. 作物学报 , 2003, 29 (4) : 496–500. |
| [14] | 齐兰, 王文泉, 张振文, 等. 利用SRAP标记构建18个木薯品种的DNA指纹图谱. 作物学报 , 2010, 36 (10) : 1642–1648. |
| [15] | 谢寅峰, 沈惠娟, 罗爱珍, 等. 南方7个造林树种幼苗抗旱生理指标的比较. 南京林业大学学报:自然科学版 , 1999, 23 (4) : 13–16. |
| [16] | 周席华, 刘学全, 胡兴宜, 等. 鄂西北主要造林树种耐旱生理特性分析. 南京林业大学学报:自然科学版 , 2005, 29 (1) : 67–70. |
| [17] | 潘伟, 王彦辉, 史东梅, 等. 重庆市域主要树种抗旱性评价. 中国城市林业 , 2007, 5 (4) : 36–38. |
| [18] | 谭建伟, 陈建新. 杉木种源抗旱变异研究. 广东林业科技 , 1993, 4 : 21–24. |
| [19] | 何贵平, 陈益泰. 不同抗旱杉木家系在水分胁迫下脯氨酸积累的差异. 林业实用技术 , 1990, 11 : 15–17. |
| [20] | 刘爱琴, 马祥庆, 冯丽贞. 不同杉木无性系对干旱胁迫的生理响应研究. 福建林学院学报 , 1998, 18 (1) : 28–31. |
| [21] | 王新民, 郑仁华, 陈金慧, 等. 杉木SRAP-PCR体系优化. 南京林业大学学报:自然科学版 , 2014, 38 (1) : 15–20. |
| [22] | 侯娜, 毛红, 王港, 等. 杉木SRAP-PCR反应体系优化与建立. 西部林业科学 , 2014, 6 : 85–90. |
| [23] | 樊蓉, 樊军锋, 李周岐. 4个白杨新品种和父母本及近缘种的SRAP遗传分析与指纹图谱构建. 西北农林科技大学学报:自然科学版 , 2016, 44 (4) : 81–86. |
| [24] | 李婷婷, 朱锦茹, 邱帅, 等. 基于CE-SRAP标记的榉树优良单株指纹图谱构建. 植物遗传资源学报 , 2016, 17 (1) : 169–176. |