随着技术的发展,用于基因表达研究的检测方法也越来越多样化,主要包括:实时定量PCR(quantitative real-time polymerase chain reaction,qRT-PCR)、Northern印迹(northerblot)、DNA微阵列(DNA microarray)和高通量测序(High-throughput sequencing)等[1]。其中,qRT-PCR具有操作方便、灵敏性强、效率高等特点,而且对实验结果有较为准确的分析[2-4],是目前研究基因表达最常用也是最重要的一种手段,在植物基因的研究中有着较为广泛的应用[5, 6]。qRT-PCR通过检测样品cDNA中的模板量比较和分析来自不同组织或不同处理的样品中目的基因的表达量,但是其分析结果通常受样品RNA质量、cDNA合成效率以及实验条件的影响,需要内参基因来矫正cDNA样品之间的差异,从而实现对目的基因的定量和标准化,进而得到更加准确、可靠的分析数据[7]。
目前,在植物基因表达研究中常用的内参基因大多是一些管家基因,如甘油醛-3-磷酸脱氢酶(glyceraldehyde-3-phosphatedehydrogenase)基因GAPDH、肌动蛋白(actin)基因ACT、延伸因子(el-ongation factor 1-alpha)基因EF1α、18S核糖体RNA(18SrRNA)和微管蛋白(tubulin)基因TUB等[8]。这些管家基因在细胞中均有较高的表达量,且表达量相对恒定[9, 10]。一个合适的内参基因在研究目的基因的实验条件下其表达量是相对恒定的。此外,内参基因作为研究对象,其表达量也应该保持在相对稳定的水平[11, 12]。近年来有关非生物胁迫下内参基因筛选的报道很多,如Wan等[13]研究发现,EF1α是黄瓜在非生物胁迫下较稳定的基因;Chen[14]在筛选非生物胁迫下匍茎剪股颖的内参基因时,发现EF1α基因在其冷胁迫的叶中具有较好的稳定性;韩晓雪等[15]在研究非生物胁迫下的番茄中,发现Actin基因较其他几个候选内参基因稳定性更好。由此可知,EF1α和Actin基因在非生物胁迫下的不同植物中均具有较好的稳定性。
迄今为止,园林树木香樟内参基因相关研究十分有限,李勇鹏等[16]从香樟中分离和鉴定了香樟肌动蛋白基因(CcACTa、CcACTb、CcACTc和CcACTd),其中,CcACTc的核苷酸序列与樟科山鸡椒中的ACT7有较高的相似性,相似度达98%,氨基酸序列相似度达100%,而山鸡椒ACT7基因是优良候选内参基因[17]。张力维等[18]从香樟中分离得到了香樟延伸因子基因(CcEF1α)。经qRT-PCR分析表明,在低温处理下香樟CcACTc和CcEF1α基因表达量均相对恒定,是研究低温胁迫条件下较好的候选内参基因[16, 18]。但是,上述两个基因在其他非生物胁迫下的稳定性分析并未见报道,香樟非生物胁迫的应答分子机制缺乏相关研究。本实验室已成功从香樟中分离并鉴定得到4个香樟冷诱导相关转录因子基因CBF/DREB1(CRT/DRE binding factor),为了系统研究香樟CBF/DREB1基因的表达模式,本实验拟对其进行相对定量分析,而内参基因在不同逆境处理下的稳定性分析是必要的研究基础。本研究利用qRT-PCR技术分析和比较这两个基因在低温、干旱、高盐和ABA处理下表达量的稳定性,旨在为研究香樟逆境相关基因的表达模式提供内参基因。
1 材料与方法 1.1 材料 1.1.1 实验材料本实验采用生长在自然环境中6月龄,株高约30 cm的香樟(Cinnamomum camphora L.)实生幼苗作为实验材料。
1.1.2 试剂EASYspin Plus植物RNA快速提取试剂盒购自北京艾德莱公司;PrimeScript Ⅱ 1st strand cDNA Synthesis Kit试剂盒购自TaKaRa生物公司(大连);qRT-PCR试剂为FastStart Universal SYBR Green Master试剂盒(Roche,Germany);其他常规化学试剂均为国产或进口分析纯。
1.2 方法 1.2.1 香樟在不同非生物胁迫下的处理选取12株无病害、生长健壮且长势一致的香樟幼苗,置于人工气候箱中进行一周的培养,其培养条件为:光照强度3 000 lx,光照时间16/8 h(光照/黑暗),温度(25±1)℃,相对湿度75%。1周后,每3株香樟幼苗分为一组,分别做低温、干旱、高盐和ABA处理。将其中一组放入另一同型号人工气候培养箱中进行低温处理,将温度设为4℃,其他条件保持不变,分别于0、0.5、1、2、4、6、12和24 h时取其叶片,将收获的叶片放入液氮中速冻后于-80℃冰箱中保存。其余3组幼苗转入蒸馏水中,在原人工气候培养箱中进行液体培养1周,然后将3组香樟幼苗分别转入对应的处理溶液中:20% polyethylene glycol(PEG)6000用来模拟干旱胁迫、250 mmol/L NaCl溶液用来模拟高盐胁迫、ABA的处理浓度为100 μmol/L。分别于各处理0、0.5、1、2、4、6、12和24 h时取其叶片,放入液氮中速冻后于-80oC冰箱中保存。
1.2.2 总RNA的提取和cDNA的合成将各处理的材料用液氮研磨后,使用EASYspin Plus植物RNA快速提取试剂盒(北京艾德莱公司)提取总RNA,具体操作按照试剂盒说明书步骤进行。提取的总RNA用Thermo Fisher Scientific公司的微量紫外检测仪NanoDrop 2000和琼脂糖凝胶电泳进行RNA样品的浓度和质量测定。然后根据PrimeScript II 1st strand cDNA Synthesis Kit(大连TaKaRa公司)试剂盒说明书将提取的总RNA反转录成cDNA,将反转录得到的各个cDNA样品分别用去离子水稀释10倍作为模板。
1.2.3 内参基因扩增片段特异性检测以香樟叶片cDNA为模板,利用李勇鹏等[16]和张力维等[18]设计的qRT-PCR引物CcACTc-RTF、CcACTc-RTR、CcEF1α-RTF和CcEF1α-RTR进行PCR扩增(表 1),扩增产物利用1.0%琼脂糖凝胶电泳检测是否有非特异性扩增。
qRT-PCR反应按照FastStart Universal SYBR Green Master试剂盒(Roche,Germany)的说明书进行。使用CFX96实时定量PCR仪(Bio-Rad)和8连管。反应体系为10 μL:2×Power Taq PCR Master Mix 5 μL,3.4 μL的ddH2O,上下游引物各0.3 μL,cDNA模板1 μL。反应条件:94℃预变性5 min;94℃变性15 s,60℃退火30 s,72℃延伸1 min,共40次循环;熔解曲线在65-95℃之间,每0.05 s升高0.5℃。基因表达分析软件为BioRad CFX manager2.0,每个样品重复3次。每个实验设3个生物学重复。
1.2.5 引物扩增效率的计算将各反转录的cDNA样品等量混合后,等比逐级稀释6个梯度,每个梯度稀释5倍,则初始cDNA浓度分别为1、1/5、1/25、1/125、1/625、1/3 125、每个反应设3个重复。反应在qPCR分析,由PCR仪自带的计算软件CFX ManagerTMSoftware绘制出标准曲线。由公式E=10(-1/k)-1计算出扩增效率E。
1.2.6 BestKeeper软件分析BestKeeper软件根据每个样品的平均Ct值进行分析[19]。根据不同处理时间下各个样品的CT值,计算标准偏差SD,SD值越小,基因稳定性越好,若SD > 1,则认为该基因不稳定[20, 21]。
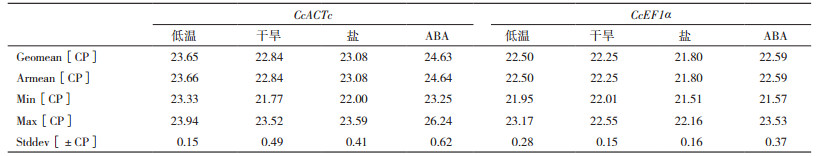
2 结果 2.1 CcACTc和CcEF1α基因片段的PCR扩增分析1%琼脂糖凝胶电泳结果(图 1)显示,在200-500 bp之间和200 bp处,CcACTc和CcEF1α基因扩增条带单一,无杂带和引物二聚体,表明CcACTc和CcEF1α基因引物特异性较高,可以用于qRT-PCR分析。
|
| 图 1 CcACTc和CcEF1α的PCR扩增产物 M :DNA Marker;1 :香樟肌动蛋白基因(CcACTc);2 :香樟延伸因子基因(CcEF1α) |
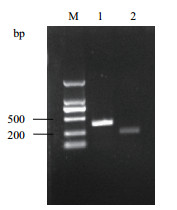
通过qRT-PCR分析,成功获得了CcACTc和CcEF1α基因引物的标准曲线。由标准曲线可知CcACTc基因的斜率k(slope)为-3.079,回归方程R2=0.994,扩增效率E=111%;CcEF1α基因的斜率k(slope)为-3.144,回归方程R2=0.997,扩增效率E=108%。以上数据中,回归方程R2均大于0.99,扩增效率在0.9-1.2之间,说明CcACTc和CcEF1α基因引物qRT-PCR体系较好,可以用于后续分析。溶解曲线结果(图 2)表明,CcACTc和CcEF1α基因都只有显著单一的熔解峰,各重复间的曲线有较好的重复性,表明引物均有良好特异性。
|
| 图 2 CcACTc基因和CcEF1α基因的Real-time PCR溶解曲线 |
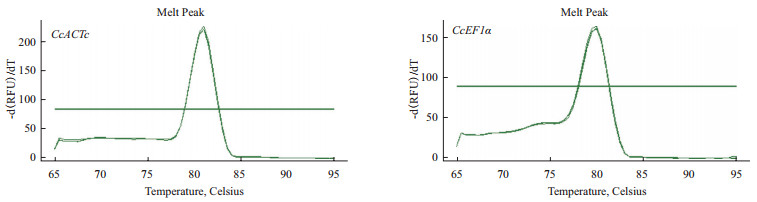
为了比较CcACTc和CcEF1α基因在各个非生物胁迫下的转录水平,通过qRT-PCR获得的Ct值来对其进行测定。qRT-PCR中基因的表达量与Ct值成反比,即Ct值越大,基因表达量越小,反之亦然。qRT-PCR结果(图 3)显示,在不同时间的低温处理下CcACTc基因的平均Ct值在23.33-23.94个循环之间,在干旱处理下的平均Ct值在21.78-23.30个循环之间,在高盐处理下的平均Ct值在22.00-23.59个循环之间,在ABA处理下的平均Ct值在24.17-26.24个循环之间;在不同时间低温处理下CcEF1α基因(图 4)的平均Ct值在21.95-23.17个循环之间,在干旱处理下的平均Ct值在22.01-22.55个循环之间,在高盐处理下的平均Ct值在21.51-22.16个循环之间;在ABA处理下的平均Ct值在21.57-23.53个循环之间。由Ct值的循环数差值,初步了解到,在低温胁迫下CcACTc基因较CcEF1α基因稳定;在干旱、高盐和ABA处理下,则CcEF1α基因更稳定。
|
| 图 3 CcACTc基因在不同非生物胁迫下的平均Ct值s |
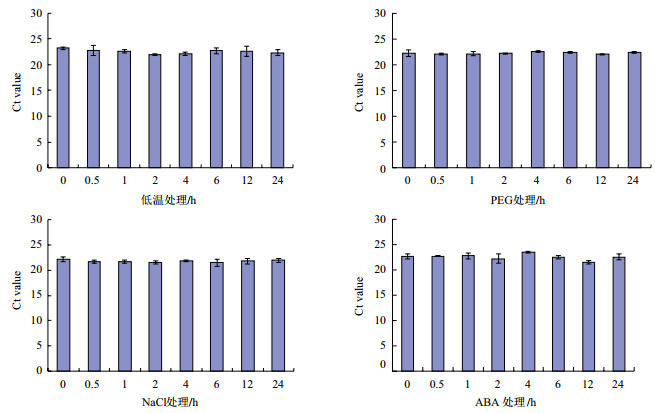
|
| 图 4 CcEF1α基因在不同非生物胁迫下的平均Ct值 |
BestKeeper软件根据候选内参基因在不同处理时间样品中的Ct值计算标准偏差SD,对基因的稳定性作分析,SD值越小,稳定性越好,若SD > 1,则说明该基因不稳定。分析结果(表 2)表明,CcACTc和CcEF1α基因在各非生物胁迫下的SD值均小于1。CcACTc基因的SD值(0.15)在低温条件下比CcEF1α基因(0.28)小,而在干旱、高盐和ABA条件下CcEF1α基因的SD值都比CcACTc基因的小。因此,在低温条件中,CcACTc基因较稳定;而在干旱、高盐和ABA胁迫中,CcEF1α基因更稳定。
qRT-PCR具有较高的灵敏性、准确性和重复性,近年来被广泛应用于基因表达的研究[22]。而在运用qRT-PCR技术时,选择合适的内参基因是基因表达研究中的关键,选择不恰当的内参基因可能会得出错误的实验结论。Actin存在于所有的真核细胞中,所表达的肌动蛋白是真核细胞中含量最丰富的蛋白,是qRT-PCR过程中的首选内参基因[23];EF1α是真核生物中一种多功能蛋白,在细胞的翻译、信号转导、凋亡、细胞骨架的形成以及癌基因的转化中都发挥着重要的作用[24],这两个基因都广泛作为内参基因应用于功能基因的表达分析研究中。此外,研究表明,Actin基因和EF1α基因经常作为内参基因应用于植物中逆境相关基因DREB的表达分析研究中。如烟草[25]、胡杨[26]、枸杞[27]和麻风树[28]等的Actin基因及马铃薯[29]、梅花[30]和莴苣[31]等的EF1α基因。然而,同一基因在不同物种或同一物中的不同处理中其稳定性有一定的差异,如在低温胁迫下,柠条锦鸡儿[32]Actin基因表达比较稳定,而在马铃薯的6个候选内参基因筛选中,Actin基因稳定性却最差[11];在干旱胁迫下,柳枝稷[33]Actin基因表达较稳定,而在油菜中[34]却表达不稳定;在脱落酸(ABA)处理下的水芹[35]和柠条锦鸡儿[32]中EF1α基因表达较稳定,而在短柄草[36]中却是较不稳定的。因此,在研究目标基因的表达模式前,对候选内参基因进行稳定性分析是非常必要的。
本研究通过qRT-PCR获得了CcACTc和CcEF1α基因在不同非生物胁迫下的平均Ct值。同时又利用BestKeeper软件对其Ct值进行了分析,依据所得SD值来判定内参基因的稳定性,分析结果发现在低温条件下,CcACTc基因的表达相对稳定,而在干旱、高盐、ABA处理的条件下,则CcEF1α基因的表达较稳定。
4 结论本研究对受不同胁迫的香樟实生苗进行了候选内参基因的分析与选择,在低温胁迫下,CcACTc基因的稳定性较好,较适合作内参基因;而在干旱、高盐和ABA胁迫中,CcEF1α基因的稳定性都较CcACTc基因高。CcEF1α基因虽然在低温条件下稳定性低于CcACTc基因,但其表达量也相对稳定,因此,CcEF1α基因则更适合作为非生物胁迫相关基因研究中的内参基因。
| [1] | Le DT, Aldrich DL, Valliyodan B, et al. Evaluation of candidate reference genes for normalization of quantitative RT-PCR in soybean tissues under various abiotic stress conditions. PLoS One , 2012, 7 (9) : 1602–1603. |
| [2] | Gachon C, Mingam A, Charrier B. Real-time PCR: what relevance to plant studies. Journal of Experimental Botany , 2004, 55 (402) : 1445–1454. DOI:10.1093/jxb/erh181 |
| [3] | Nolan T, Hands RE, Bustin SA. Quantification of mRNA using real-time RT-PCR. Nature Protocols , 2006, 1 (3) : 1559–1582. DOI:10.1038/nprot.2006.236 |
| [4] | Hu RB, Fan CM, Li HY, et al. Evaluation of putative reference genes for gene expression normalization in soybean by quantitative real-time RT-PCR. BMC Molecular Biology , 2009, 10 (1) : 93. DOI:10.1186/1471-2199-10-93 |
| [5] | Caldana C, Scheible WR, Mueller-Roeber B, et al. A quantitative RT-PCR platform for high-throughput expression profiling of 2500 rice transcription factors. Plant Methods , 2007, 3 (1) : 7. DOI:10.1186/1746-4811-3-7 |
| [6] | Gutierrez L, Mauriat M, Pelloux J, et al. Towards a systematic validation of references in real-time RT-PCR. Plant Cell , 2008, 20 (7) : 1734–1735. DOI:10.1105/tpc.108.059774 |
| [7] | Bjarnadottir H, Jonsson JJ. A rapid real-time qRT-PCR assay for ovine β-actin mRNA. Journal of Biotechnology , 2005 (2) : 173–182. |
| [8] | 牙库甫江·阿西木, 关波, 张富春. 植物基因表达转录分析中内参基因的选择与应用. 生物技术通报 , 2011 (7) : 7–11. |
| [9] | 张玉芳, 赵丽娟, 曾幼玲. 基因表达研究中内参基因的选择与应用. 植物生理学报 , 2014, 50 (8) : 1119–1125. |
| [10] | Nicot N, Hausman JF, Hoffmann L, et al. Housekeeping gene selection for real-time RT-PCR normalization in potato during biotic and abiotic stress. Journal of Experimental Botany , 2005, 56 (421) : 2907–2914. DOI:10.1093/jxb/eri285 |
| [11] | Lopez-Pardo R, Ruiz de Galarreta JI, Ritter E. Selection of housekeeping genes for qRT-PCR analysis in potato tubers under cold stress. Molecular Breeding , 2013, 31 (1) : 39–45. DOI:10.1007/s11032-012-9766-z |
| [12] | Bustin SA, Gyselman VG, Siddiqi S, et al. Cytokeratin 20 is not atissue specific marker for the detection of malignant epithelial cells in the blood of colorectal cancer patients. International Journal of Surgical Investigation , 2000, 2 (1) : 49–57. |
| [13] | Wan HJ, Zhao ZJ, Qian CT, et al. Selection of appropriate reference genes for gene expression studies by quantitative real-time polymerase chain reaction in cucumber. Analytical Biochemistry , 2010, 399 (2) : 257–261. DOI:10.1016/j.ab.2009.12.008 |
| [14] | Chen Y, Hu B, Tan Z, et al. Selection of reference genes for quantitative real-time PCR normalization in creeping bentgrass involved in four abiotic stresses. Plant Cell Reports , 2015, 34 (10) : 1825–1834. DOI:10.1007/s00299-015-1830-9 |
| [15] | 韩晓雪, 韩佳轩, 姜晶. 番茄在非生物胁迫下实时定量RT-PCR中内参基因的筛选. 分子植物育种 , 2015, 13 (4) : 822–831. |
| [16] | 李勇鹏, 张力维, 姚瑶, 等. 香樟Actin基因的克隆及表达分析. 生物技术通报 , 2015, 31 (5) : 120–127. |
| [17] | Lin LY, Han XJ, Chen YC, et al. Identification of appropriate reference genes for normalizing transcript expression by quantitative real-time PCR in Litsea cubeba. Mgg Molecular & General Genetics , 2013, 288 (12) : 727–737. |
| [18] | 张力维, 李勇鹏, 姚瑶, 等. 香樟延伸因子EF1α基因片段的克隆和表达分析. 中南林业科技大学学报 , 2015, 35 (5) : 122–128. |
| [19] | Pfaffl MW, Tichopad A, Prgomet C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: bestkeeper-excel-based tool using pair-wise correlations. Biotechnology Letters , 2004, 26 (6) : 509–515. DOI:10.1023/B:BILE.0000019559.84305.47 |
| [20] | Mehta R, Birerdinc A, Hossain N, et al. Validation of endogenous reference genes for qRT-PCR analysis of human visceral adipose samples. BMC Molecular Biology , 2010, 11 (39) : 1471–2199. |
| [21] | 苏晓娟, 樊保国, 袁丽钗, 等. 实时荧光定量PCR分析中毛果杨内参基因的筛选和验证. 植物学报 , 2013, 48 (5) : 507–518. |
| [22] | Bustin SA, Benes V, Nolan T, et al. Quantitative real-time RT-PCR-a perspective. Journal of Molecular Endocrinology , 2005, 34 (3) : 597–601. DOI:10.1677/jme.1.01755 |
| [23] | Santella L, Chun JT. Actin, more than just a house-keeping protein at the scene of fertilization. Science China Life Sciences , 2011, 54 (8) : 733–743. DOI:10.1007/s11427-011-4202-x |
| [24] | 周冰, 曹诚, 刘传暄. 翻译延伸因子1A的研究进展. 生物技术通讯 , 2007, 18 (2) : 281–284. |
| [25] | Wang XM, Dong J, Liu Y, et al. A novel dehydration-responsive element-binding protein from Caragana korshinskii is involved in the response to multiple abiotic stresses and enhances stress tolerance in transgenic tobacco. Plant Molecular Biology Reporter , 2010, 28 (4) : 664–675. DOI:10.1007/s11105-010-0196-y |
| [26] | Zhou ML, Ma JT, Zhao YM, et al. Improvement of drought and salt tolerance in Arabidopsis and Lotus corniculatus by overexpression of a novel DREB transcription factor from Populus euphratica. Gene , 2012, 506 (1) : 10–7. DOI:10.1016/j.gene.2012.06.089 |
| [27] | Wu DY, Ji J, Wang G, et al. LchERF, a novel ethylene-responsive transcription factor from Lycium chinense, confers salt tolerance in transgenic tobacco. Plant Cell Reports , 2014, 33 (12) : 2033–2045. DOI:10.1007/s00299-014-1678-4 |
| [28] | Wang LH, Gao JH, Qin XB, et al. JcCBF2 gene from Jatropha curcas improves freezing tolerance of Arabidopsis thaliana during the early stage of stress. Molecular Biology Reports , 2015, 42 (5) : 937–945. DOI:10.1007/s11033-014-3831-0 |
| [29] | Bouaziz D, Jbir R, Charfeddine S, et al. The StDREB1 transcription factor is involved in oxidative stressresponse and enhances tolerance to salt stress. Plant Cell Tiss Organ Cult , 2014, 121 (1) : 237–248. |
| [30] | Guo C, Zhang JQ, Peng T, et al. Structural and expression analyses of three PmCBFs from Prunus mume. Biologia Plantarum , 2014, 58 (2) : 247–255. DOI:10.1007/s10535-014-0393-x |
| [31] | Kudo K, Oi T, Uno Y. Functional characterization and expression profiling of a DREB2-type gene from lettuce(Lactuca sativa L.). Plant Cell Tiss Organ Cult , 2014, 116 (1) : 97–109. DOI:10.1007/s11240-013-0386-z |
| [32] | Yang Q, Yin JJ, Li G, et al. Reference gene selection for qRT-PCR in Caragana korshinskii Kom. under different stress conditions. Molecular Biology Reports , 2014, 41 (4) : 2325–2334. DOI:10.1007/s11033-014-3086-9 |
| [33] | Huang L, Yan H, Jiang X, et al. Evaluation of candidate reference genes for normalization of quantitative RT-PCR in switchgrass under various abiotic stress conditions. Bioenergy Research , 2014, 7 (4) : 1201–1211. DOI:10.1007/s12155-014-9457-1 |
| [34] | Wang Z, Chen Y, Fang H, et al. Selection of reference genes for quantitative reverse-transcription polymerase chain reaction normalization in Brassica napus under various stress conditions. Molecular Genetics and Genomics , 2014, 289 (5) : 1023–1035. DOI:10.1007/s00438-014-0853-1 |
| [35] | Jiang Q, Wang F, Li MY, et al. Selection of suitable reference genes for qPCR normalization under abiotic stresses in Oenanthe javanica(BI.)DC. PLoS One , 2014, 9 (3) : e92262. DOI:10.1371/journal.pone.0092262 |
| [36] | Hong SY, Seo PJ, Yang MS, et al. Exploring valid reference genes for gene expression studies in Brachypodium distachyon by real-time PCR. BMC Plant Biology , 2008, 8 (1) : 112. DOI:10.1186/1471-2229-8-112 |