2.中国人民解放军总后勤部卫生部药品仪器检验所,北京 100166;
3.中国科学院北京基因组研究所,北京 100101
2. Institute for Drug and Instrument Control,Health Department,the General Logistics Department of People’s Liberation Army,Beijing 100166;
3. Beijing Institute of Genomics,Chinese Academy of Sciences,Beijing 100101
现阶段最常用的三大基因编辑技术主要有锌指核酸酶技术(Zinc finger nucleases,ZFNs)、转录激活样效应子核酸酶技术(Transcription activator-like effector nucleases,TALENs)、CRISPR/Cas9技术(Clustered regularly interspaced short palindromic Repeats/Cas9 nuclease)[1]。其中TALENs技术具有无上下文效应[2]、脱靶效率低于ZFNs技术和CRISPR/Cas9技术[3]、特异性高等优点,现已成为一种广泛应用的基因定点修饰技术。
TALENs由转录激活样效应子蛋白(Transcription activator-like effector,TALE)、识别域和FokIⅡ核酸酶及切割域组成[4]。其中,TALE蛋白源自黄单胞杆菌,一般由N端、C端和中央串联重复模块组成,N端包含Ⅲ型转运信号(Translocation signal);C端包含核定位信号(Nuclear localization signal,NLS)和酸性转录激活域(Activation domain,AD);中央重复模块具有“一模块识别一碱基”的特点,且没有上下文效应,其结构高度保守,一般由34个氨基酸组成,仅在12和13位高度可变,又称这两位的氨基酸为RVD(Repeat-variable diresidue)[2, 5, 6]。本研究采用并改造TALENs的精简处理的骨架,即N端包含136个氨基酸,C端包含63个氨基酸,且C端融合有FokIⅡ型核酸内切酶[7, 8, 9, 10]。利用TALENs这些结构特点可以构建识别任何DNA序列的TALENs,包括核基因组和线粒体基因组[11, 12]。
在TALENs应用中,TALENs的中央重复模块的组装是限速步骤,目前为止,最常用的TALENs构建方法大体可分为Golden Gate法[13, 14, 15, 16]、FLASH(Fast Ligation-based automatable solid-phase high-throughput)法[17, 18]、模块组装法(unit assembly,UA)法[7]及其他方法,如LIC法(Ligation-independent cloning)[19]、ICA法(Iterative capped assembly)[20]、idTALE法[21]、REAL法(Restriction enzyme and ligation)[22]等。其中Golden Gate法、FLASH法最为省时,然而这两种方法存在的酶切连接效率问题或因PCR技术的限制性而出现非特异的问题难以避免,相比之下,UA法虽构建时间较长,但TALENs编码序列可控且没有上述问题,同时可以根据密码子简并原则改变TALEs编码序列,降低TALENs重复模块编码序列的重复性,降低mRNA的重复性,有助于提高TALENs在真核细胞内表达效率。本研究提供了TALENs的一单元模块编码序列库,并改造表达载体pCS2-peas/perr,利用UA法定向组装TALENs。鉴于目前没有报告清晰的介绍线粒体DNA(Mitochondrial DNA,mtDNA)双链断裂(Double-strand breaks,DSB)的修复机制,本研究将mtDNA靶序列随机整合于核染色体中,利用核基因组的同源重组(Homologous recombination,HR)和非同源末端连接(Non-homologous DNA end joining,NHEJ)修复方式[23],以检测新型模块组装法构建的TALENs活性,旨在为编辑mtDNA提供新方法。
1 材料与方法 1.1 材料 1.1.1 细胞HEK293F细胞为本实验室保存。
1.1.2 试剂和引物Xba I、Nhe I、AsiS I、Sac I、BamH I等限制性核酸内切酶购自NEB公司;质粒小提试剂盒购自北京庄盟国际生物公司;高纯度小提中量试剂盒购自天根生物科技公司;HA、Flag、β-actin抗体购自Sigma公司;Anti-Rabbit、Anti-mouse抗体购自CST公司;转染试剂X-tremeGENE HP DNA Transfection Reagent购自Roche公司;DMEM高糖培养基购自Invitrogen公司;胎牛血清购自Biowest公司;pUC19、pEGFP-N1质粒由本实验室保存;pCS2-peas/perr质粒购自康为世纪公司;所有引物序列(表 1)由生工生物工程(上海)股份有限公司合成;测序由华大基因公司完成。
HEK293F细胞以及HEK293F-T1细胞用含10%胎牛血清的DMEM培养基培养于37℃、5%CO2温箱中。
1.2.2 pCS2-peas/perr载体骨架的改造本研究于载体0.5单元的RVD编码序列两侧分别构建AsiS I和Xba I酶切位点,如图 1所示。实验方法如下:以pCS2-peas/perr载体质粒为模板,TALEN0.5_N-F/R为引物对,扩增得到全长质粒对,PCR产物经DpnI酶切,转化后挑单克隆菌落,pCS2-NF/CR引物对测序鉴定出阳性质粒,最终得到载体质粒pCS2-0.5U-peas/perr。

|
| A:pCS2-peas/perr载体骨架改造示意图;B:改造后0.5U序列及酶切位点详图 图 1 pCS2-0.5U-peas/perr构建示意图 |
基于TALEs自然单元模块序列[6]和UA法的人工单元模块序列[7],本研究设计的人工单元模块序列命名为偏单元(图 2),起始于TALEs自然模块的+19位氨基酸残基Leu,终止于下一个自然模块的+18位氨基酸残基Ala。于偏单元序列的两端分别构建Xba I、Nhe I同尾酶识别位点,于RVD区附近构建AsiS I酶切位点。根据AsiS I酶切位点的有无分为活动一单元模块(含AsiS I酶切位点)和固定一单元模块(不含AsiS I酶切位点)。活动一单元模块位于TALEs重复模块的首位,并命名为0序列,依次标记为A0、C0、G0、T0。固定一单元模块位于第2位及后续位置。由于氨基酸密码简并性和第4位、32位氨基酸残基的高度可变,TALEN模板序列数目模大,经排列组合计算后结果如表 2所示。选取了氨基酸序列一致的4种基本骨架a1、c1、g1、t1,碱基序列不同但差异率并非最大(折中差异性),N-LETVQRLLPVLCQAHGLTPEQVVAIASNIGGKQA-C(灰色背景部分为RVD区)。依次构建含有4种RVD(NI、HD、NN、NG)的一单元模块,依次标记为A1、A2、A3、A4,C1、C2、C3、C4,G1、G2、G3、G4,T1、T2、T3、T4,如表 3所示。

|
| A:横线部分为TALEs自然单元重复模块序列,横线外部分为末位0.5单元模块;B:UA法人工单元重复序列;C:本课题组人工单元重复序列 图 2 TALEs偏单元的氨基酸序列示意图 |
活动单元的构建以C0序列为例,构建方法如下:以C0的DNA编码序列(表 3)为模板分别合成Oligo片段C0-L1、C0-L2和C0-S1、C0-S2(表 4),C0-L1/C0-L2、C0-S1/C0-S2分别退火,得到片段C0-L、C0-S,对pUC19质粒进行BamH I/Sac I双酶切,将片段C0-S连入,再次进行Xba I/AsiS I双酶切,将片段C0-L连入,得到包含C0序列的pUC19-C0质粒(图 3)。合成A0-S1/A0-S2、G0-S1/G0-S2(NN)、T0-S1/T0-S2(序列见表 4),两两退火后,分别连入AsiS I/Nhe I双酶切后的pUC19-C0偏单元载体,得到分别包含A0、G0和T0序列的质粒pUC19-A0、pUC19-G0、pUC19-T0。从合成方法来看,A0、C0、T0和G0序列除了RVD区编码序列不同外,其他位置编码序列均相同。
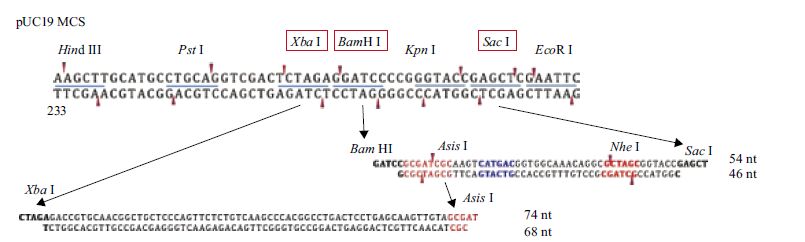
|
| 图 3 TALEs偏单元重复模块的碱基序列设计示意图 |
固定单元的构建以A1为例,构建方法如下:根据A1的DNA编码序列(表 3),合成Oligo片段A1-L1、A1-L2和A1-S1、A1-S2(表 5),分别退火,得到片段A1-L、A1-S。以A1-L1和A1-S2为引物,A1-L、A1-S为模板,扩增得到A1。以A1为骨架,RVD区分别换为其他3种RVD(HD、NN和NG)的编码序列,可得到C2、G2和T2。其他模块构建方法为,将A1-S2的RVD去分别改为其他3种RVD(HD、NN和NG)的编码序列,命名为C2-ar、G2-ar和T2-ar(表 5),分别和A1-L1组成引物对,以A1为模板,扩增得到C2、G2和T2。最终得到A1、C1、G1、T1,A2、C2、G2、T2,A3、C3、G3、T3,A4、C4、G4、T4。
在NCBI查询得到人线粒体DNA序列(NC_012920.1),利用TALE-NT 2.0(TAL Effector Nucleotide Targeter 2.0)设计一系列TALENs靶序列。经过靶位点比对和筛选,本研究选取选择线粒体DNA(NC_012920.1)的16347-16387位为靶序列,5'-TCAAATCCCTTCTCGTCCCCATGGATGACCCCCCTCAGA TA-3',并依据该靶序列设计TALEs的串联重复模块。本研究设计选取12.5个TALEs模块,Left-arm:5' -CAAATCCCTTCTC-3';Right-Arm:5'-ATCTGAGGGGGGT-3'。
用Nhe I/Sac I和Xba I/sac I分别对模块载体和目的模块序列进行酶切、连接,依次由一单元得到二单元,得到四单元,再根据设计序列进行灵活连接[7]。本研究组构建的三单元模块库,分别由4个三单元模块两两相连,得到2个六单元模块,再次相连后与活动一单元连接,最后得到13单元TALE-13U,Left-arm的构建如图 4所示,然后将其进行AsiS I和Nhe I(Xba I的同尾酶)双酶切,连接入pCS2-0.5U-peas/perr载体,得到pCS2-12.5U-L/R质粒对。
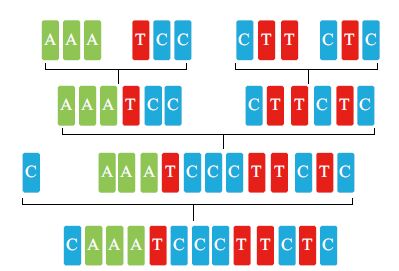
|
| 图 4 TALE-13U构建示意图 |
合成5'端和3'端分别含有Nhe I、Age I酶切位点的靶序列片段,分别对靶序列片段和pEGFP-N1质粒进行Nhe I/Age I双酶切后,将二者进行连接、转化,挑取单克隆菌落,经PCR检测,测序验证含有靶序列的目标质粒,转染培养于35 mm dish的HEK293F细胞,24 h后消化转移至10 cm dish培养,同时加G418筛选,在推荐药物浓度附近设置浓度梯度,经摸索得到G418的最适浓度为1 mg/mL。挑取单克隆细胞系,继续培养,经PCR扩增并测序,得到整合有靶序列的目标细胞系,标记为HEK293F-T1。
1.2.6 TALENs在细胞内表达活性的检测将HEK293F-T1细胞接种于两个10 cm dish中,待两盘细胞长至生长对数期且细胞密度一致(约24 h),选取其中一盘细胞瞬时转染1.2.4中构建的质粒对pCS2-12.5U-L/R,另一盘作为control,再继续培养48 h,收细胞,提取基因组,PCR扩增后测序鉴定打靶效果。PCR反应为活性检测的关键步骤,反应体系为:基因组(模板)200 ng,上/下游引物各0.5 μL,LA Taq 0.5 μL,10×LA PCR BufferⅡ 2.5 μL,dNTP Mixture(各2.5 mmol/L)4 μL,ddH2O补足至25 μL。反应程序:95℃ 2 min;95℃ 15 s、60℃ 30 s,72℃ 1 min(35个循环);72℃ 5 min。
2 结果 2.1 靶序列的确定 2.1.1 HEK293F细胞线粒体中靶序列的确定依据线粒体DNA(NC_012920.1),在靶序列附近设计引物,以提取的HEK293F基因组为模板,扩增含有靶序列的片段并测序,测序结果峰图如图 5所示。

|
| 图 5 mtDNA中靶序列测序峰图 |
提取HEK293F-T1细胞基因组并作为模板,在pEGFP-N1-T1质粒靶序列两侧设计引物,扩增靶序列,测序结果峰图 6如图所示。
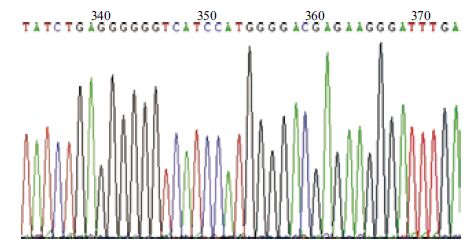
|
| 图 6 HEK293F-T1基因组中靶序列测序峰图 |
模块组装法组装TALENs,构建得到pCS2-12.5U-L/R质粒,经AsiS I/BamH I酶切鉴定,片段为1 448 bp,大小如图 7-A所示,测序结果经Blast比对,左右臂的氨基酸比对结果如图 7-B所示。

|
| 图 7 pCS2-12.5U-L/R质粒酶切(A)及pCS2-12.5U-L质粒测序比对(B)结果 |
通过提取瞬时转染pCS2-12.5U-L/R质粒的HEK293F-T1基因组,扩增、测序分析后,观察到靶序列强峰下出现套峰,结果(图 8)表明,TALENs成功的作用于HEK293F-T1基因组上的靶序列,造成双链断裂后,在细胞核内进行了修复。

|
| 图 8 pCS2-12.5U-L/R打靶结果图 |
TALENs识别域是由n个串联的高度重复的模块组成,在细胞转录与翻译过程中,能否对高度重复的序列十分准确的转录翻译成重复的蛋白模块,至今还未有研究组论证,本研究组尝试着尽量降低TALENs编码序列的重复性,而模块组装法恰好能够保证构建的TALENs编码序列的高保真性[24],使得TALENs编码序列重复性可控,提高了TALENs的表达效率。
3.2 针对mtDNA的相关编辑研究因mtDNA的碱基序列长度远远小于nDNA(Nuclear DNA),且线粒体中针对双链断裂的修复机制尚未准确解析。本研究组中将mtDNA序列整合于核基因组中,利用核基因组中的HR和NHEJ机制对其进行修复,同时考虑到TALENs中央重复模块的数目是否会影响打靶效率,本研究中设计的TALENs均选取12.5个重复模块,对nDNA打靶效果比较理想,进而为mtDNA的编辑研究奠定了基础。Bacman[12]实验组首次利用TALENs编辑mtDNA的相关研究,为本实验室提供了可参考的思路与方法,本实验组下一步将进行mtTALENs(Mitochondria-targeted TALENs)的研究。
4 结论本研究中提供的新型模块组装法可定向的组装TALENs,提高克隆效率,并增加了靶位点设计的灵活性,在表达强度和打靶效果上均可达到理想效果。
| [1] | Gaj T, Gersbach CA, Barbas CF. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering[J]. Trends in Biotechnology, 2013, 31(7):397-405. |
| [2] | Moscou MJ, Bogdanove AJ. A Simple Cipher Governs DNA Recognition by TAL Effectors[J]. Science, 2009, 326(5959):1501. |
| [3] | Chen L, Tang L, Xiang H, et al. Advances in genome editing technology and its promising application in evolutionary and ecological studies[J]. GigaScience, 2014, 3:24. |
| [4] | Christian M, Cermak T, Doyle EL, et al. Targeting DNA double-strand breaks with TAL effector nucleases[J]. Genetics, 2010, 186(2):757-761. |
| [5] | Boch J, Scholze H, Schornack S, et al. Breaking the code of DNA binding specificity of TAL-type III effectors[J]. Science, 2009, 326(5959):1509-1512. |
| [6] | Boch J, Bonas U. Xanthomonas AvrBs3 family-type III effectors:discovery and function[J]. Annual Review of Phytopathology, 2010, 48:419-436. |
| [7] | Huang P, Xiao A, Zhou MG, et al. Heritable gene targeting in zebrafish using customized TALENs[J]. Nature Biotechnology, 2011, 29(8):699-700. |
| [8] | Miller JC, Tan S, Qiao G, et al. A TALE nuclease architecture for efficient genome editing[J]. Nature Biotechnology, 2011, 29(2):143-148. |
| [9] | Szurek B, Rossier O, Hause G, et al. Type III-dependent translocation of the Xanthomonas AvrBs3 protein into the plant cell[J]. Mol Microbiol, 2002, 46(1):13-23. |
| [10] | Mussolino C, Morbitzer R, Lutge F, et al. A novel TALE nuclease scaffold enables high genome editing activity in combination with low toxicity[J]. Nucleic Acids Research, 2011, 39(21):9283-9293. |
| [11] | Li T, Liu B, Spalding MH, et al. High-efficiency TALEN-based gene editing produces disease-resistant rice[J]. Nature Biotechnology, 2012, 30(5):390-392. |
| [12] | Bacman SR, Williams SL, Pinto M, et al. Specific elimination of mutant mitochondrial genomes in patient-derived cells by mitoTALENs[J]. Nature Medicine, 2013, 19(9):1111-1113. |
| [13] | Cermak T, Doyle EL, Christian M, et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting[J]. Nucleic Acids Research, 2011, 39(12):e82. |
| [14] | Weber E, Gruetzner R, Werner S, et al. Assembly of designer TAL effectors by Golden Gate cloning[J]. PloS One, 2011, 6(5):e19722. |
| [15] | Zhang F, Cong L, Lodato S, et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription[J]. Nature Biotechnology, 2011, 29(2):149-153. |
| [16] | Sanjana NE, Cong L, Zhou Y, et al. A transcription activator-like effector toolbox for genome engineering[J]. Nature Protocols, 2012, 7(1):171-192. |
| [17] | Reyon D, Tsai SQ, Khayter C, et al. FLASH assembly of TALENs for high-throughput genome editing[J]. Nature Biotechnology, 2012, 30(5):460-465. |
| [18] | Reyon D, Maeder ML, Khayter C, et al. Engineering customized TALE nucleases (TALENs) and TALE transcription factors by fast ligation-based automatable solid-phase high-throughput (FLASH) assembly[J]. Current Protocols in Molecular Biology, 2013, Chapter 12:Unit 12.6. |
| [19] | Schmid-Burgk JL, Schmidt T, Kaiser V, et al. A ligation-independent cloning technique for high-throughput assembly of transcription activator-like effector genes[J]. Nature Biotechnology, 2013, 31(1):76-81. |
| [20] | Briggs AW, Rios X, Chari R, et al. Iterative capped assembly:rapid and scalable synthesis of repeat-module DNA such as TAL effectors from individual monomers[J]. Nucleic Acids Research, 2012, 40(15):e117. |
| [21] | Li L, Piatek MJ, Atef A, et al. Rapid and highly efficient construction of TALE-based transcriptional regulators and nucleases for genome modification[J]. Plant Mol Biol, 2012, 78(4-5):407-416. |
| [22] | Sander JD, Cade L, Khayter C, et al. Targeted gene disruption in somatic zebrafish cells using engineered TALENs[J]. Nature Biotechnology, 2011, 29(8):697-698. |
| [23] | Larsen NB, Rasmussen M, Rasmussen L J. Nuclear and mitochond-rial DNA repair:similar pathways?[J]. Mitochondrion, 2005, 5(2):89-108. |
| [24] | 沈延,肖安,黄鹏,等. 类转录激活因子效应物核酸酶(TALEN)介导的基因组定点修饰技术[J]. 遗传, 2013, 35(4):395-409. |









