纳豆激酶(Nattokinase,NK)是由枯草芽孢杆菌(Bacillus subtilis)代谢产生的一种丝氨酸蛋白酶,是目前国内外研究和开发溶栓药物的热点[1]。产生纳豆激酶的菌种大多来源于日本纳豆或中国豆豉等传统的发酵食品经筛选获得,这些筛选方法是建立在纤溶活性基础上的,如纤维平板法和纤维降解法[2],然而有很多类似的酶具有相同的纤溶活性,如subtilisin DJ-4、subtilisin DFE和纤溶酶BSF1[3, 4, 5],对纳豆激酶产生菌的筛选带来了干扰。纳豆激酶的基因aprN,在这些纤溶活性的酶中具有专一性[6],因此,采用PCR方法检测纳豆激酶基因和测定纤溶活性两者相结合的方法为一种高效筛选获得纳豆激酶产生菌的途径,几乎未检索到这方面的报道。
目前纳豆激酶发酵活性或产量不高,阻碍了产业化开发进程。近年来通过基因工程手段,研究人员获得了一些纳豆激酶的基因工程菌种,但酶活力或产量也并不理想。在基因水平上,纳豆激酶产生菌(纳豆菌)的基因组信息获得了解析并已经公开发表[7],因此,从分子水平上对纳豆激酶进行研究奠定了基础。各国学者对纳豆激酶发酵工艺[8, 9, 10]、分离纯化技术[11]、高产菌株诱变与选育[12]以及纳豆激酶代谢动力学和作用机理等[13, 14]诸多方面进行了大量研究,获得许多较为重要的进展。同时继续寻找不同来源的纳豆激酶高产菌,以得到具有工业化潜在应用价值的新菌株,也越来越受到人们的关注。本课题组经多年的研究,从发酵豆制品中选育出1株纳豆激酶高产菌株,前期对纳豆激酶酶学性质、发酵培养、分离纯化及体外溶栓特性等方面进行过研究[15, 16, 17],现在正进行纳豆激酶保健产品的开发和应用研究。然而,前期的工作未对纳豆激酶和菌株名称进行确切的鉴定,一直是以测定纤溶活性这种传统方法为基础进行研究的。本研究拟利用检测纳豆激酶分子(蛋白和基因)与测定纤溶活性相结合的方法对纳豆激酶进行鉴定,同时结合分子生物学方法将该高产菌株的名称定名到亚种的水平,旨在为产纳豆激酶菌株的鉴定提供借鉴和参考。
1 材料与方法 1.1 材料 1.1.1 供试样品分离菌株的样品分别来源于中国传统食品自然发酵的豆豉;枯草芽孢杆菌枯草亚种(B. subtilis subsp. subtilis)168菌株来自于中国普通微生物菌种保藏管理中心。
1.1.2 主要试剂凝血酶、人纤维蛋白原均购自中国药品生物制品检定所;酵母粉、胰蛋白胨为Oxoid公司产品;其他试剂均为国产分析纯试剂。
1.1.3 培养基LB培养基:酵母粉5 g/L,胰蛋白胨10 g/L,NaCl 10 g/L,pH7.2(固体加1.5%琼脂粉);发酵培养基:葡萄糖5 g/L,蛋白胨10 g/L,酵母粉5 g/L,NaCI 5 g/L,pH值7.2-7.4。
1.2 方法 1.2.1 菌种的分离纯化用无菌牙签挑取少量豆豉表面的黏性物质,接种到10 mL无菌水的试管中充分搅拌,制成悬浊液。取少量悬浊液,涂布在LB培养基平板上,待风干后倒置于37℃恒温培养箱中培养12-24 h,挑取平板上长出的有皱褶的白色菌落,镜检观察。继续划线纯化1次,挑取单菌落,保存于LB培养基斜面上。
1.2.2 纤溶活性测定采用纤维蛋白平板法测定纳豆激酶活性[18]。将分离获得的不同菌株接种到液体发酵培养基中于37℃下振荡培养过夜,发酵液8 000 r/min离心5 min得到上清粗酶液。取粗酶液10 μL点于纤维蛋白平板上,倒置于37℃恒温培养箱中培养18 h后测量透明圈直径,计算纳豆激酶的纤溶活性,筛选获得活性最高的菌株,编号为Td。
1.3 菌株鉴定 1.3.1 菌株形态及生理生化特性测定观察高产菌株Td的菌落和菌体形态,参照《常见细菌鉴定手册》并与枯草芽孢杆菌枯草亚种168对比进行生理生化鉴定试验[19]。
1.3.2 分子鉴定及系统发育树构建参照《分子克隆实验指南》描述的方法提取菌株Td的基因组DNA[20]。分别应用16S引物(27F:5'-AGAGTTTG-ATCCTGGCTCAG-3';1492R:5'-GGTTACCTTGTTA-CGACTT-3')和解旋酶gyrA引物(gyrA-42F:5'-CA-GTCAGGAA ATGCGTACGTCCTT-3';gyrA-1066R:5'-CAAGGTAATGCTCCAGGCATTGCT-3')进行PCR扩增[21],目的产物纯化后由北京华大基因有限公司进行测序。将获得的16S rDNA和gyrA序列在GenBank数据库中进行BLAST比对,确定与其亲缘关系最近的属种,并从数据库下载获得相关种属的gyrA基因序列,构建系统发育树。序列比对用ClustalX 2.1进行,系统进化树的构建采用MEGA 5.0软件进行,应用Neighbour-Joining方法进行bootstrap分析,重复次数为1 000。
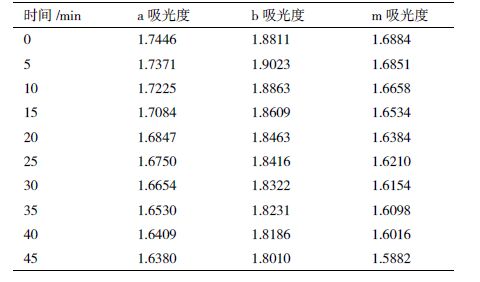
1.3.3 基因组DNA-DNA杂交分析根据上述方法提取菌株Td和菌株168的基因组DNA,用仪器DU800进行DNA-DNA杂交分析。通过超声断裂法(400 W,2 s,6次)剪切基因组DNA,使DNA片段在1 000-2 000 bp之间。将剪切过的DNA用0.1×SSC精确配置成OD260为1.5-2.0。取Td、168(分别标记为a,b)各300 µL及两者各150 µL的混合物(标记为m),分别加到1.5 mL离心管中,于沸水浴中变性10 min,并用预热的枪头吸取75 µL预热的10×SSC溶液加入变性样品中,吹打混匀,使终浓度为2×SSC,固定温度为最适复性温度(Tor),以2×SSC为空白对照,复性45 min,在260 nm波长下,每隔25 s测一次吸光值,作出复性曲线,计算复性速率Va、Vb、Vm,并用下式计算DNA-DNA杂交度D%=[4Vm-(Va+Vb)]/2(VaVb)1/2。
1.4 纳豆激酶的鉴定 1.4.1 SDS-PAGE凝胶电泳检测纳豆激酶产量配制浓度为5%浓缩胶、12%分离胶的聚丙烯酰胺凝胶用于纳豆激酶产量的SDS-PAGE分析,每个点样孔加入1.2.2中描述的发酵液16 µL进行电泳。电泳完毕后对凝胶用考马斯亮蓝R-250染色蛋白质条带,使用quantity one软件对蛋白进行定量分析。
1.4.2 MALDI-TOF质谱分析鉴定纳豆激酶将菌株Td培养得到的发酵液进行SDS-PAGE电泳分离,切胶回收目的大小的条带,用胰蛋白酶(Trypsin)进行胶内酶切处理来进行MALDI-TOF质谱分析。获得的肽质量指纹图谱应用MASCOT 2.0软件进行NCBI蛋白质数据库的检索分析。
1.4.3 纳豆激酶基因的克隆根据1.4.2的结果,并参照GenBank公布的纳豆激酶基因来设计一对引物,序列分别为F:5'-TCAGCATAATGAACATTTACTCA-3'和R:5'-GGTTGATCCTCCGGTGCTTGTGA-3'。参照1.3.2中方法提取菌株Td基因组DNA为模板进行PCR扩增纳豆激酶基因。PCR反应体系(50 μL):10×rTaq Reaction Buffer 5 μL,rTaq DNA聚合酶2.5 U,上下游引物(10 mmol/L)各2 μL,10 mmol/L dNTPs 3 μL,补足ddH2O至50 μL。反应条件:94℃预变性5 min后进入循环,94℃ 50 s,56℃ 40 s,72℃ 2 min,循环30次,最后72℃延伸10 min。PCR产物经纯化后连接到质粒pMD18-T载体上,连接产物转化大肠杆菌DH5α感受态细胞,碱裂解法提取大肠杆菌转化子质粒。经酶切鉴定后,将验证为阳性的重组质粒送至北京华大基因有限公司进行序列测定。
2 结果 2.1 纳豆激酶高产菌株的筛选从供试样品中共分离得到18株菌株,将筛选得到的菌株经过再次纯化、发酵培养,用纤维蛋白法测其纳豆激酶的活性大小。根据在纤维蛋白原平板上产生透明圈的大小(图 1),最终确定1号菌株为高产菌株,编号为Td。

|
| 1:菌株Td;2-9:分离获得的其它编号菌株的纤溶活性大小 图 1 不同菌株发酵液的纤溶活性 |
在LB培养基平板上于37℃恒温培养24 h后,菌株Td的菌落呈圆形,表面粗糙有皱褶、微突起、边缘不整齐、无光泽、呈乳白或灰白色,菌落直径3-4 mm。显微镜下观察菌体呈杆状,单个或链状排列,大小(2.0-3.0)µm×(0.6-0.8)µm,有鞭毛,能运动,芽孢呈椭圆或柱状。
以标准菌株168作为对照菌株,与菌株Td共同进行生理生化特性的试验。结果(表 1)表明,菌株Td与菌株168生理生化特性相同,因此可以确定菌株Td符合芽孢杆菌属(Bacillus)的特征。
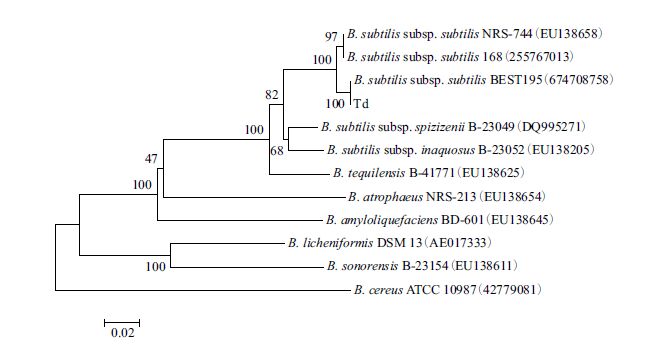
根据16S rDNA序列的测定结果,与GenBank 中已经登录的序列进行BLAST检索比对,结果显示菌株Td的16S rDNA与芽孢杆菌属(Bacillus)不同种具有高度相似,同源性高达99%以上;而基因gyrA进行BLAST检索比对结果表明,与枯草芽孢杆菌枯草亚种(B. subtilis subsp. subtilis)的相似性最高,达100%。根据BLAST检索结果,筛选芽孢杆菌属相关物种的gyrA基因序列与供试菌株Td的序列进行系统发育树的构建。结果(图 2)表明,菌株Td与枯草芽孢杆菌枯草亚种聚为一个分支上,而与其他物种相距较远。
|
| 图 2 芽孢杆菌属部分菌种的gyrA基因系统发育树(括号内数字表示GenBank的登录号) |
经DU-800测定与分析,根据表 2的结果,计算菌株Td与标准菌株168的复性速率为Va=0.002 7和Vb=0.002 3,混合样品的复性速率Vm=0.002 4,代入杂交率计算公式得出菌株Td与标准菌株168的DNA杂交率D%=92.3%,显著高于80%的阈值,因此可以确定两者为同一个亚种。
综合形态特征和分子鉴定的结果,鉴定菌株Td为枯草芽孢杆菌枯草亚种。
2.3 纳豆激酶的活性与鉴定 2.3.1 纳豆激酶的溶解纤维蛋白活性纳豆激酶活性越高,在纤维蛋白平板上的透明圈直径则越大。根据活性测定结果(图 1),Td产生的透明圈明显比其它菌株的大,因此菌株Td产生的纳豆激酶活性最高,纤溶活性达237 U/mL。
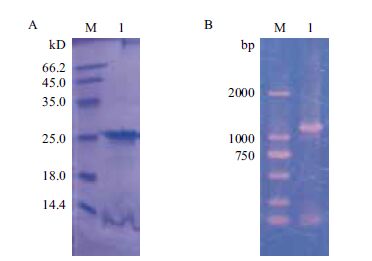
2.3.2 SDS-PAGE电泳分析菌株Td发酵液进行SDS-PAGE凝胶电泳的结果(图 3-A)显示,泳道中的蛋白质杂质谱带较少,其中有1条明显较亮的蛋白质谱带。与标准分子量蛋白比较和计算,得出此条带的蛋白分子量大小约为27 kD;应用quantity one软件计算蛋白产量可达300 mg/L,占总蛋白的40%以上。
|
| (A)菌株Td的发酵液SDS-PAGE电泳图,M:protein marker,1:菌株Td的发酵液样品;(B)菌株Td的PCR扩增纳豆激酶基因的电泳图,M:DNA marker DL2000,1:菌株Td的PCR产物 图 3 纳豆激酶的SDS-PAGE和PCR扩增基因的电泳图 |
从图 3-A凝胶中挖取最亮的蛋白质条带采用胰蛋白酶进行胶内酶切后用于MALDI-TOF分析。根据肽质量指纹谱和数据库检索结果,MASCOT 值最高达150,与枯草芽孢杆菌的蛋白酶Subtilisin E相似性最高;验证了7条序列,分别为:GAYSLSLR、GFFLFVEGGR、GSSIFGLAPGK、SSGTSYPDVLK、VCNYVSWIK、GSSIFGLAPGK、LNTLETEEWFFK,这与纳豆激酶的序列完全相同。
2.3.4 纳豆激酶的基因序列根据PCR扩增条带(图 3-B)和基因序列测定(图 4)结果,获得了菌株Td的纳豆激酶全基因序列。菌株Td的纳豆激酶基因全长为1 143 bp,以GTG 为起始密码子,起始密码子上游 -17- -11 bp位点为核糖体结合位点SD 序列,SD序列上游是A/T含量高达72%的转录调控区域。随后是由1 143 bp组成的开放阅读框架,编码379个氨基酸,其中氨基酸序列的前27个氨基酸为信号肽序列,随后的77个氨基酸为前导肽序列,最后的275个氨基酸为成熟肽。基因的3'末端是3个连续的终止密码子TAA、TAG 和TAA,转录终止序列位于成熟蛋白区域C端下游7 bp处,该段终止序列可形成茎环结构,即ρ因子非依赖性终止序列。

|
| 图 4 菌株Td的纳豆激酶基因及编码蛋白质序列 |
纳豆激酶(nattokinase)是枯草芽孢杆菌产生的一种胞外丝氨酸蛋白激酶,在血栓、血脂、血压、抗癌、糖尿病等方面均有预防和治疗的功能,已为国际社会所接受,但是对其产生菌的定名却存在争议。目前已经发表的文章采用的纳豆枯草芽孢杆菌(Bacillus subtilis subsp. natto)或枯草芽孢杆菌纳豆亚种(Bacillus subtilis subsp. natto),严格意义上来说并非一个合法有效的名称,只是人们的传统习惯命名。目前有效发表的枯草芽孢杆菌有3个亚种,分别为B. subtilis subsp. subtilis、B. subtilis subsp. spizizenii和B. subtilis subsp. inaquosorum,已为研究人员所接受[22]。本研究也遵从此命名法,认为定名为枯草芽孢杆菌枯草亚种(Bacillus subtilis subsp. subtilis)较为适宜。
枯草芽孢杆菌及其近缘种群(简称枯草群)是一群表型相似的革兰氏阳性、产芽孢的杆菌,但是单一的鉴定技术不能完全胜任枯草群菌株的鉴定,特别是通过生理生化等表型特征分析的方法。虽然16S rDNA基因序列广泛应用于细菌鉴定或构建细菌的系统进化关系,但对于枯草群的菌种由于序列间的相似度太高而不能有效区分开来,因此通过表型特性和16S rDNA序列分析将菌株鉴定到种缺乏有力的证据。近年来研究者发现以编码蛋白的基因作为系统发育鉴定标记可以弥补16S rDNA基因的不足,如gyrA基因、gyrB基因、ropD基因等[23, 24]。Rooney 等[21]利用gyrA 基因研究了枯草芽孢杆菌种间的复杂性发现,枯草芽孢杆菌的不同菌株间存在很大的差异;可以基于gyrA 基因的差异分为不同的群。本研究结果显示,单独利用gyrA 基因进行BLAST 搜索时,所获信息主要是Rooney 等登录的信息,利用这些信息构建系统发育树时,可以发现菌株Td与已知菌株间的亲缘关系,因此根据NCBI上登录的gyrA 基因信息来鉴定枯草芽孢杆菌是可行的。
应用全基因组DNA杂交方法作为公认的标准来划分细菌的种已为广大分类学家所接受,并且普遍认为,细菌种内DNA-DNA杂交同源性为60%-100%;80%-90%以上的杂交同源性属于种内同一亚种水平;60%-70%以上的杂交同源性属于种内不同亚种水平;而20%-60%的同源性则代表着待定菌株是与参比菌株靠近的种。本研究中菌株Td与枯草芽孢杆菌枯草亚种标准菌株168的DNA-DNA杂交同源性达到了92.3%,显著高于同一亚种的DNA杂交同源性标准;同时参照16S rDNA和gyrA的基因序列信息,因此将菌株Td定名为枯草芽孢杆菌枯草亚种。
纳豆激酶的名称最初命名为BSP[25],随后改为subtilisin NAT[6]。比较纳豆激酶的氨基酸序列与其他几种枯草杆菌蛋白酶发现它们有较高的同源性,与枯草杆菌蛋白酶E(Subtilisin E)的同源性达到99.5%,与枯草杆菌蛋白酶amylosacchariticus、subtilisin BPN和subtilisin Carlsberg的同源性也分别达到99.3%、86%和72%[6]。本研究应用MALDI-TOF质谱分析获得的结果与subtilisin E相似性最高,这可能与两者序列高度相似有关。利用PCR克隆获得的纳豆激酶全基因序列推导出的氨基酸序列也与已报道的纳豆激酶的氨基酸序列完全一致。通过对菌株Td发酵液进行SDS-PAGE测定纳豆激酶的分子量大小、MALDI-TOF分析氨基酸序列特征和PCR克隆全基因序列,三者取得了一致的结果,相互印证了纳豆激酶的氨基酸和核酸序列,为鉴定纳豆激酶提供了依据。另外,本研究成功地克隆了纳豆激酶全长基因,这为下一步的基因工程菌株的构建和纳豆激酶的活性改造奠定了基础。
4 结论根据菌体形态、生理生化特征和16S rDNA、gyrA基因序列、DNA-DNA杂交率等实验结果,鉴定菌株Td为枯草芽孢杆菌枯草亚种(Bacillus subtilis subsp. subtilis);通过MALDI-TOF质谱分析和PCR克隆基因,鉴定了菌株Td产生的纳豆激酶的分子特征;SDS-PAGE和纤溶活性测定表明菌株Td是一株高产纳豆激酶菌株。
| [1] | Sumi H, Hamada H, Tsushima H, et al. A novel fibrinolytic enzyme(nattokinase)in the vegetable cheese Natto:a typical and popular soybean food in the Japanese diet[J]. Experientia, 1987, 43(10):1110-1111. |
| [2] | Ku TW, Tsai RL, Pan TM. A simple and cost-saving approach to optimize the production of subtilisin NAT by submerged cultivation of Bacillus subtilis Natto[J]. Journal of Agricultural and Food Chemistry, 2009, 57(1), 292-296. |
| [3] | Choi NS, Chang KT, Jae MP, et al. Cloning, expression, and fibrin(ogen)olytic properties of a subtilisin DJ-4 gene from Bacillus sp. DJ-4[J]. FEMS Microbiology Letters, 2004, 236(2):325-331. |
| [4] | Peng Y, Yang XJ, Xiao L, et al. Cloning and expression of a fibrinolytic enzyme(subtilisin DFE)gene from Bacillus amyloliquefaciens DC-4 in Bacillus subtilis[J]. Research in Microbiology, 2004, 155(3):167-173. |
| [5] | Agrebi R, Haddar A, Hmidet N, et al. BSF1 fibrinolytic enzyme from a marine bacterium Bacillus subtilis A26:purification, biochemical and molecular characterization[J]. Process Biochemistry, 2009, 44(11):1252-1259. |
| [6] | Nakamura T, Yamagata Y, Ichishima E. Nucleotide sequence of the subtilisin NAT gene, aprN, of Bacillus subtilis(natto)[J]. Bioscience, Biotechnology, and Biochemistry, 1992, 56(11):1869-1871. |
| [7] | Nishito Y, Osana Y, Hachiya T, et al. Whole genome assembly of a natto production strain Bacillus subtilis natto from very short read data[J]. BMC Genomics, 2010, 11:243-254. |
| [8] | 周伏忠, 贾蕴莉, 陈国参, 等. 纤溶酶高产菌株筛选及其液体发酵条件研究[J]. 河南科学, 2008, 26(1):125-130. |
| [9] | 熊迎新, 尹宗宁, 李婉宜. 纳豆激酶固体浅盘发酵工艺的优化[J]. 中国生化药物杂志, 2005(5):15-17. |
| [10] | Cho YH, Song JY, Kim KM, et al. Production of nattokinase by batch and fed-batch culture of Bacillus subtilis[J]. New Biotechnology, 2010, 27(4):341-346. |
| [11] | Wang C, Du M, Zheng D, et al. Purification and characterization of nattokinase from Bacillus subtilis natto B-12[J]. Journal of Agricultural and Food Chemistry, 2009, 57(20):9722-9729. |
| [12] | Chen PT, Chao YP. Enhanced production of recombinant nattokinase in Bacillus subtilis by the elimination of limiting factors[J]. Biotechnology Letters, 2006, 28(19):1595-1600. |
| [13] | Kim JY, Gum SN, Paik JK, et al. Effects of nattokinase on blood pressure:a randomized, controlled trial[J]. Hypertension Research, 2008, 31(8):1583-1588. |
| [14] | Yang NC, Chou CW, Chen CY, et al. Combined nattokinase with red yeast rice but not nattokinase alone has potent effects on blood lipids in human subjects with hyperlipidemia[J]. Asia Pacific Journal of Clinical Nutrition, 2009, 18(3):310-317. |
| [15] | 陈晓飞, 杜迅, 刘安邦, 等. 纳豆激酶酶学性质的初步研究[J]. 河南科学, 2008(10):1212-1214. |
| [16] | 陈晓飞, 周伏忠, 陈国参, 等. 纳豆激酶的分离纯化及体外溶栓特性研究[J]. 大豆科学, 2010, 29(2):295-298. |
| [17] | 陈晓飞, 周伏忠, 陈国参, 等. 纳豆激酶酶学性质研究[J]. 河南科学, 2010, 28(1):41-43. |
| [18] | Astrup T, Mullertz S. The fibrin plate method for estimating fibrinolytic activity[J]. Archive of Biochemistry and Biophysics. 1952, 40(2):346-351. |
| [19] | 东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京:科学出版社, 2001. |
| [20] | 萨姆布鲁克, 拉塞尔著;黄培堂, 等译. 分子克隆实验指南[M].第3版. 北京:科学出版社, 2008. |
| [21] | Rooney AP, Price NPJ, Ehrhardt C, et al. Phylogeny and molecular taxonomy of the Bacillus subtilis species complex and description of Bacillus subtilis subsp. inaquosorum subsp. nov. [J]. International Journal of Systematic and Evolutionary Microbiology, 2009, 59:2429-2436. |
| [22] | 曹凤明, 杨小红, 马鸣超, 等. 枯草芽孢杆菌近缘种群鉴定方法研究进展[J]. 微生物学通报, 2014, 41(5):968-974. |
| [23] | Chun J, Bae KS. Phylogenetic analysis of Bacillus subtilis and related taxa based on partial gyrA gene sequence[J]. Antonie Van Leeuwenhoek International Journal of General and Molecular Microbiology, 2000, 78(2):123-127. |
| [24] | Yamamoto S, Harayama S. Phylogenetic relationships of Pseudomonas putida strains deduced from the nucleotide sequences of gyrB, rpoD and 16S rRNA genes[J]. International Journal of Systematic Bacteriology, 1998, 48:813-819. |
| [25] | Kamata H, Yamagata Y, Nakamura, et al. Characterization of the complex between a macroglobulin and a serine proteinase from Bacillus natto[J]. Agricultural and Biological Chemistry, 1989, 53:2695-2702. |