结缕草(Zoysia Willd)隶属于禾本科(Grami-neae)画眉草亚科(Eragrostoideae),是一种多年生C4型低矮草本植物,主要分布于亚洲和大洋洲。该属植物的5个种,即日本结缕草(Zoysia japonica Steud.)、中华结缕草(Zoysia sinica Hance)、大穗结缕草(Zoysia macrostachya Franch. et Sav)、沟叶结缕草(Zoysia matrella)和细叶结缕草(Zoysia tenuifolia Willd. ex Trin)[1],具有耐高温和干旱、耐修剪、匍匐生长、耐践踏的优点,是开发作为草坪草的理想草种。目前在全球种植的结缕草品种有17个左右,广泛用于运动场草场、公用绿地、家庭草坪、高尔夫球场果岭和固沙保土草坪的建植。由于多变的自然环境,草坪的完整性和观赏性常常受到低温、干旱和盐碱等非生物因素的威胁。抗逆机制和抗逆育种研究仍然是结缕草育种工作的重要内容之一。
近年来人们对模式植物拟南芥、水稻等植物材料的抗逆分子机制研究结果为结缕草的相关研究提供了参考。对拟南芥的研究发现,转录因子DREB(Dehydration responsive element binding protein)在逆境应答中起重要作用。DREB属于AP2超家族,包括6个亚组,即DREB1-DREB6,其中DREB1和DREB2与植物逆境响应关系最密切,能特异结合启动子中含有的DRE/CRT顺式元件,激活许多逆境诱导基因的表达,增强植物对逆境的忍耐能力。其中,DREB1B/CBF1、DREB1C/CBF2和DREB1A/CBF3主要与低温应答相关[2, 3, 4, 5],DREB1D/CBF4主要参与干旱胁迫相应[6],而DREB1E/DDF2和DREB1F/DDF1主要参与盐胁迫响应[7]。DREB2组有8个基因,主要对干旱、高盐和高温胁迫响应[8, 9, 10, 11]。在其他多种植物中,包括水稻[12]、玉米[13]、大麦[14, 15]、小麦[16]、油菜[17]等农作物,黑麦草[18, 19]、狗牙根[20]等草坪草,均克隆获得了多个DREB1和DREB2基因,它们在基因序列和功能上都有不同程度的相似性。对同一物种不同种质的比较研究结果表明,不同种质之间抗逆能力差异与DREB1/CBFs和DREB2的基因结构或表达差异相关。例如,玉米的ZmDREB2.7的表达与幼苗的抗旱性相关[13];不同生态型拟南芥中,CBF2基因结构和表达、CBFs基因调节子的表达差异与种质抗寒能力差异相关[21, 22, 23];大麦的一个CBF基因簇与耐冻和越冬性的遗传位点FR-2的遗传定位一致[15];一粒小麦(Triticum monococcum)中含有11个CBF基因的基因簇被定位到 5号染色体的Fr-Am2抗冻位点上[16];冬小麦中CBF14的拷贝数比春小麦的高[24]。结缕草作为一种多耐逆性的草种,其DREB基因与抗逆性的关系值得研究。
本研究以我国山东胶州湾的结缕草‘胶东青’变种为材料,克隆获得到一个新DREB A-2亚组基因,分析该基因在低温、干旱和高盐胁迫下的表达调节,以期为结缕草DREB基因研究积累资料。
1 材料与方法 1.1 材料试验用植物材料为日本结缕草‘胶东青’变种(Zoysia japonica var. pallida cv Jiaodong),由青岛海源草坪有限公司赠送。RNA 提取用 RNAPrep pure 植物总RNA提取试剂盒(北京天根生物工程公司),mRNA反转录用M-MLV(Progema),质粒提取试剂盒、琼脂糖DNA快速胶回收试剂盒和T-载体(pUM-T)连接试剂盒均购自北京百泰克生物工程有限公司,PCR扩增用的普通PCR mix 和高保真的Pfu mix 购自TaKaRa公司。试验用引物均由上海生物工程有限公司合成,引物名称及序列见表 1。
从‘胶东青’草坪中采集匍匐茎段回温室内培养,基质为营养土、珍珠岩和蛭石以2∶1∶1比例的混合物,培养温度25℃-28℃,光周期14 h/d。室内培养6月后,选取生长状态良好、长势基本一致的材料进行逆境处理,方法如下:
(1)低温胁迫:材料转到植物生长箱中培养,4℃恒温,光周期14 h/d;(2)PEG模拟干旱胁迫:用PEG6000浓度为20%的营养液浇灌根部至流出溶液为培养基质体积的两倍以上,然后将培养钵放到托盘中,在托盘加浅层浇灌液,温室内继续培养;(3)高盐胁迫:用NaCl浓度为200 mmol/L的营养液浇灌根部,方法同PEG处理。
3种逆境胁迫时间分为2、24和72 h。以室温培养的材料为低温处理的对照,以浇灌营养液的材料为高盐胁迫和PEG模拟干旱胁迫处理的对照。取对照和逆境处理材料上部第1-2展开叶,液氮速冻后贮存于-80℃冰箱中。
1.2.2 RNA的提取与cDNA的制备将叶片在液氮中研磨,用植物总RNA 提取试剂盒提取RNA,电泳检测RNA的完整性,用NanoDrop2000超微量分光光度计(美国Thermo公司)检测RNA浓度。再以Oligo dT为引物,用M-MLV将mRNA反转录为cDNA,具体操作按照说明书进行。以Actin1为内标基因,用其cDNA特异性引物Pactin-F和Pactin-R对cDNA 进行PCR扩增,检测反转录效果。
1.2.3 DREB2.2基因的克隆以低温胁迫2 h的cDNA 为模板,用引物P1、P2和 Pfu mix进行PCR扩增,产物在1%的琼脂糖凝胶上电泳。回收DNA片段,连接到载体pUM-T上,并测序。对测序获得的基因序列,用Conserved Domain Search Service(CD Search)工具(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)预测其保守结构域。蛋白质的等电点、分子量和二级结构用ExPASy网站相关软件完成;亚细胞定位信息用PSORT Prediction(http://psort.hgc.jp/)预测;蛋白模型用Built Model(http://swissmodel.expasy.org/interactive)以及RaptorX(http://raptorx.uchicago.edu/)工具预测。用DNA man 6.0制作基因的系统进化树。
1.2.4 逆境胁迫下基因表达模式以cDNA 为模板,先用Pactin-F和Pactin-R对内标基因进行23个循环的PCR扩增,产物在1.5%的琼脂糖凝胶上电泳,确定条带亮度基本一致时各处理的cDNA用量。然后以该用量的cDNA为模板,分别用引物PL-F+PLS-R和PS-F+PLS-R组合,对各处理cDNA 进行31个循环的PCR扩增,产物在1.5%的琼脂糖凝胶上电泳,比较各处理条带亮度。
2 结果 2.1 RNA提取和反转录效果提取的RNA电泳结果显示,各样品中28S和18S条带清晰完整,两条带亮度比例为2∶1左右。用Nano Drop 2000超微量分光光度计检测结果表明,各样品RNA的A260/280比值均大于2,说明RNA无明显的蛋白质污染。对RNA进行反转录制备获得的cDNA样品,用Actin1的cDNA特异性引物进行PCR扩增,结果都产生了约450 bp左右的特异带,说明所有样品RNA反转录效果良好。
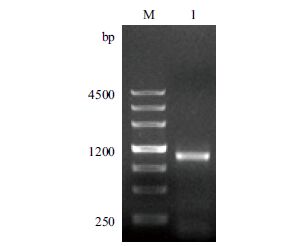
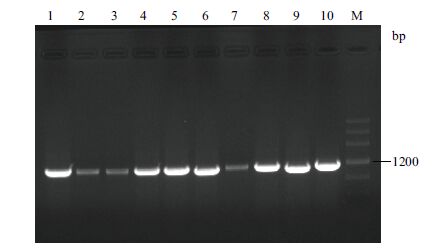
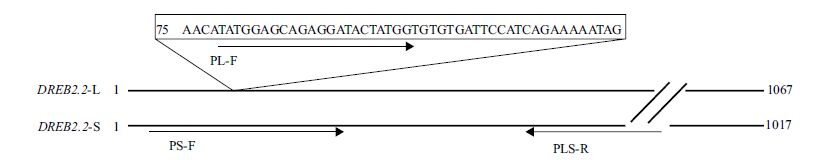
2.2 DREB2.2基因的克隆以低温处理2 h 样品的cDNA 为模板,用DREB2.2基因特异性引物进行PCR扩增,获得了1 000 bp左右的特异带(图 1)。回收该条带并克隆到pUM-T载体中。对10个可能重组子进行PCR鉴定,结果(图 2)显示,有7个在1 000 bp附近均产生高亮度的特异带,其中1号、6号和9号的条带位置略低,4号、5号、8号和10号的条带略高,推测PCR产物中可能存在两种长短不同的转录本。选择6号和9号、8号和10号重组子进行测序,结果(图 3)证实了我们的推测,前两者克隆的核酸序列长1 017 bp,后两者克隆的核酸序列从第75位碱基开始有50 bp的插入序列,总长1 067 bp。
|
| M:DNA 分子量标准;1:DREB2.2 扩增产物 图 1 DREB2.2基因的RT-PCR扩增 |
|
| M:DNA 分子量标准;1-10:可能重组子 图 2 DREB2.2核酸片段与克隆载体重组子的PCR鉴定 |
|
| 方框内为DREB2.2-L含有的50 bp插入序列;PL-F和PS-F分别为DREB2.2-L和DREB2.2-S特异的上游引物位置,PLS-R为两序列的共同下游引物位置 图 3 DREB2.2基因两个转录本的序列比较 |
本研究将DREB2.2的长、短转录本分别命名为DREB2.2-L和DREB2.2-S。由于含有50 bp的插入序列,DREB2.2-L的阅读框提前终止,仅推测编码65个氨基酸。DREB2.2-S有完整的阅读框,推测编码蛋白含338个氨基酸(图 3)。这两个序列均已登录到NCBI网站上,登录号分别为KP676131 和KP676132。
DREB2.2-S推测编码的蛋白质分子量37.3 kD,等电点 pI为4.86。氨基酸序列的疏水性/亲水性分析结果表明,第24-33位氨基酸区域的亲水性最强,疏水区域中,第156-164位氨基酸疏水性最强。氨基酸序列中18%为α-螺旋,5%为β-折叠,76%为无规则卷曲。第89-145位氨基酸构成一个AP2结构域(图 4-A),其三维模型与拟南芥ERF1A(At4g17500)中的AP2/ERF结构域PDB2gcc:A的模型高度相似(P-value:2.94e-04)。亚细胞定位分析结果显示,第23-32位氨基酸(PGRKKRPRRS)为细胞核定位序列(NLSs)。DREB2.2-S整体蛋白的结构模型预测,见图 4-B。

|
| 图 4 DREB2.2-S蛋白AP2/EREBP结构域(A)和3D模型(B) |
在NCBI网站上进行蛋白BLAST分析结果(图 5)表明,与DREB2.2-S相似度最高的前5个基因分别来自高粱(Sorghum bicolor),谷子(Setaria italic)、玉米(Zea mays)、羊草(Leymus chinensis)和四倍体小麦硬粒亚种(Triticum turgidum subsp. Durum),它们与DREB2.2-S在核定位序列和AP2结构域序列上高度相似,但在基因的C端差异较大。基因系统进化分析结果(图 6)显示,DREB2.2-S被归到A2亚组,与高粱、玉米、谷子中的同源基因遗传距离较近。

|
| SbDREB2A:高粱(AFI71292);SiDREB2B:谷子(XP_004962409.1);ZmDREB2A:玉米(AFI71292);LcDREB2A:羊草(AEC53579.1);TtDRF1.3:四倍体小麦硬粒亚种(AFO10999.1)。划线下方为核定位序列,方框内为AP2/EREBP DNA 结合结构域 图 5 DREB2.2-S与5个相似度最高的直系同源基因氨基酸序列的比较 |

|
| A-1-A-6:分别代表DREB转录因子亚家族的6个亚组,方框内为结缕草‘胶东青’的DREB2.2-S基因。Zj:结缕草(Zoysia japonica);Sb:高粱(Sorghum bicolor);Zm:玉米(Zea mays);Si:谷子(Setaria italic);Lc:羊草(Leymus chinensis);Tt:四倍体小麦硬粒亚种(Triticum turgidum subsp. Durum);Ta:小麦(Triticum aestivum);Tm:一粒小麦(Triticum monococcum);Bd:二穗短柄草(Brachypodium distachyon);Os:水稻(Oryza.sativa);At:拟南芥(Arabidopsis thaliana) 图 6 DREB2.2-S基因的系统发育树 |
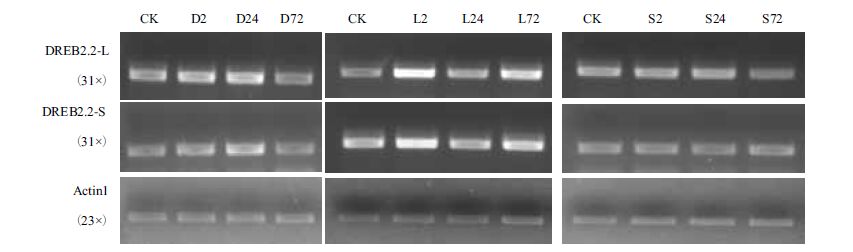
以Actin基因为内标,采用半定量PCR技术研究DREB2.2基因在低温、干旱和高盐胁迫下的表达变化。在相同cDNA用量条件下,Actin1基因进行了23个循环的扩增,DREB2.2-L和DREB2.2-S进行了31个循环的扩增。产物经1.5%的琼脂糖凝胶电泳检测,结果(图 7)显示,在正常生长条件下,DREB2.2-S和DREB2.2-L的扩增均获得了比较亮的特异带,说明两者均为组成型较高水平表达。干旱胁迫2 h和24 h时,两种序列均略受上调表达;低温胁迫时,两序列均受上调表达,胁迫2 h时表达量最高;在高盐胁迫下,两序列表达均无明显响应。总体上,相同条件下DREB2.2-L的条带亮度均比DREB2.2-S的稍亮一些,说明前者的表达量略高
|
| 字母D、L和S分别代表干旱、低温和高盐胁迫处理,字母后的数字代表胁迫处理时间 图 7 逆境胁迫下DREB2.2的表达 |
结缕草作为一种多耐逆植物,其DREB基因结构、功能和表达模式值得研究。Wang等[25]克隆获得了首个结缕草DREB A-2亚组的基因(ZjDREB-2.1),过量表达该基因的拟南芥植株的抗寒性明显高于野生型。冯勋伟和才宏伟[26]以日本北海道地区的耐寒生态型室兰(Murorann)和日本九州地区的不耐寒生态型俵山北(Tawarayamakita)为材料,克隆获得了结缕草的首个DREB A-1亚组基因ZjCBF,在拟南芥中超表达该基因可增强植株抗冻能力。本研究以结缕草‘胶东青’变种为材料,克隆获得该属植物的第2个DREB A-2亚组的基因,命名为DREB2.2。
结缕草DREB2.2基因有两个转录本,长转录本DREB2.2-L在第75位碱基处插入50 bp的序列,导致阅读框提前终止,仅编码65个氨基酸,短转录本DREB2.2-S 包含完整的阅读框,推测编码蛋白含338个氨基酸,与ZjDREB2.1氨基酸序列相似度为33.14%。在DREB亚家族基因的进化系统中,DREB2.2-S 属于A-2组,与高粱、玉米、谷子的同源基因遗传距离较近,而与ZjDREB2.1处于A-2亚组中的不同分枝上,推测结缕草的这两个基因有不同的禾本科DREB2基因祖先。DREB2基因的多种转录剪接方式在其他禾本科植物中也有报道。玉米的ZmDREB2A有两个转录本,长转录本ZmDREB2A-L多含一个53 bp的序列,编码区提前终止,仅推测编码89个氨基酸,而短转录本ZmDREB2A-S编码蛋白属于DREB2亚组[27]。大麦的HvDRF1和小麦的TaDRF1基因有3个转录本,其中两个转录本可以翻译产生DREB2类转录因子[28]。Qin等[27]根据他们对玉米ZmDREB2A基因的研究结果认为,单子叶和双子叶植物在一些DREB2基因活性调控方式上有差异,前者通过转录剪接调节,后者通过磷酸化调节,本研究结果支持了这个观点。由于有生理活性的DREB2基因的超表达影响植株的生长发育[9, 27],采用可变剪接产生无活性转录本的调节方式,可能有利于减轻基因的负面效应,使植株正常生长。
多数DREB2基因具有逆境应答特性,不同物种中DREB2基因的逆境响应模式有差异。拟南芥中的DREB2A、DREB2B基因的表达受干旱和高盐诱导,而对低温诱导无明显响应[29]。菊花(Dendranthema vestitum)DvDREB2A基因表达受低温诱导的强度是干旱和高盐诱导强度的8-15倍左右[30]。珍珠狼尾草(Pennisetum glaucum)PgDREB2A基因表达对干旱胁迫响应较早,但在低温胁迫下上调幅度比在干旱胁迫和高盐胁迫下的高[31]。胡杨(Populus euphratica)的PeDREB2基因对高盐胁迫响应较低温和干旱胁迫的滞后[32]。玉米ZmDREB2A基因的两个长、短转录本在常温下表达量非常低,在低温和盐胁迫下两者均上调表达,其中有生理功能的ZmDREB2A-S上调幅度更大[27]。本研究表明,结缕草的DREB2.2的两个转录本在正常生长条件下有表达,在干旱胁迫下,两种剪接本在24 h内均略上调表达;在低温胁下,两序列均在2 h时表达量最高;高盐胁迫下,两序列表达均无明显响应。在相同条件下,DREB2.2-L的表达量均略高于DREB2.2-S的量或与其相当。转基因研究表明,DREB2基因超表达后可增强植株抵抗低温、干旱和高盐等非生物逆境的能力。拟南芥组成型活性DREB2A基因的超表达明显增强拟南芥植株的抗旱能力,但未增强抗冻能力[9]。超表达PgDREB2A基因的烟草植物抗高盐和干旱胁迫能力增强[33]。Chen 等[34]用大豆转录因子 GmDREB2 转化拟南芥,干旱处理(19 d 不浇水)后野生型全部死亡,而转基因植株的成活率可高达 45.9%,证明转基因植株抗旱能力有明显提高。组成型表达玉米ZmDREB2A-S的拟南芥植株抗旱能力增强,植株矮壮,发育迟缓。结缕草DREB2.2-S编码的蛋白质属于DREB2亚组转录因子,在氨基酸序列上与高粱、谷子、玉米的同源基因高度相似,推测它具有DREB2亚组基因类似的生理功能,参与结缕草抗逆生理过程。
4 结论结缕草‘胶东青’DREB2.2基因含DREB2.2-L和DREB2.2-S两个转录本,前者阅读框不完整,推测后者编码的蛋白质属于DREB转录因子亚家族A-2亚组,与高粱、谷子、玉米的同源基因亲缘关系较近。DREB2.2-L和DREB2.2-S为组成型表达,表达量受低温和干旱诱导上调,可能参与结缕草抗逆生理过程。
| [1] | 董厚德, 宫丽君, 等. 中国结缕草生态学及其资源开发与应用[M]. 北京: 中国林业出版社, 2001: 105-113. |
| [2] | Medina J, Bargues M, Terol J, et al. The Arabidopsis CBF gene family is composed of three genes encoding AP2 domain-containing proteins whose expression is regulated by low temperature but not by abscisic acid or dehydration[J]. Plant Physiology, 1999, 19: 463-470. |
| [3] | Stocking EJ, Gilmour SJ, Thomashow MF. Arabidopsis thaliana CBF1 encodes an AP2 domain-containing transcriptional activator that binds to C-repeat/DRE, a cis-acting DNA regulatory element that stimulates transcription in response to low temperature and water deficit[J]. The Proceedings of the National Academy of Sciences USA, 1997, 94: 1035-1040. |
| [4] | Kasuga M, Liu Q, Miura S, et al. Improving plant drought, salt, and freezing tolerance by gene transfer of a single stress inducible transcription factor[J]. Nature Biotechnology, 1999, 17: 287-291. |
| [5] | Gilmour SJ, Sebolt AM, Salazar MP, et al. Overexpression of the Arabidopsis CBF3 transcriptional activator mimics multiple biochemical changes associated with cold acclimation[J]. Plant Physiology, 2000, 124: 1854-1865. |
| [6] | Haake V, Cook D, Riechrmnn JL, et al. Transcription factor CBF4 is a regulator of drought adaptation in Arabidopsis[J]. Plant Physiology, 2002, 130: 639-648. |
| [7] | Magome H, Yamaguchi S, Hanada A, et al. Dwarf and delayed-flowering 1, a novel Arabidopsis mutant deficient in gibberellin biosynthesis because of overexpression of a putative AP2 transcription factor[J]. Plant Journal, 2004, 37: 720-729. |
| [8] | Nakashima K, Shinwari ZK, Sakuma Y. Organization and expression of two Arabidopsis DREB2 genes encoding DRE-binding proteins involved in dehydration and high salinity-responsive expression[J]. Plant Molecular Biology, 2000, 42: 657-665. |
| [9] | Sakuma Y, Maruyama K, Osakabe Y, et al. Functional analysis of an Arabidopsis transcription factor, DREB2A, involved in drought-responsive gene expression[J]. The Plant Cell, 2006, 18: 1292-1309. |
| [10] | Lim CJ, Hwang JE, Chen H, et al. Over-expression of the Arabidopsis DRE/CRT-binding transcription factor DREB2C enhances thermo tolerance[J]. Biochemical and Biophysical Research Communications, 2007, 362: 431-436. |
| [11] | Chen H, Eun JH, Lim CJ, et al. Arabidopsis DREB2C functions as a transcriptional activator of HsfA3 during the heat stress response[J]. Biochemical and Biophysical Research Communications, 2010, 401: 238-244. |
| [12] | Mao D, Chen C. Colinearity and similar expression pattern of rice DREB1s reveal their functional conservation in the cold-responsive pathway[J]. PLoS One, 2012, 7: e47275. |
| [13] | Liu S, Wang X, Wang H, et al. Genome-wide analysis of ZmDREB genes and their association with natural variation in drought tolerance at seedling stage of Zea mays L.[J]. PLoS Genetics, 2013, 9: e1003790. |
| [14] | Choi DW, Rodriguez EM, Close TJ. Barley cbf3 gene identification, expression pattern, and map location[J]. Plant Physiology, 2002, 129: 1781-1787. |
| [15] | Stockinger EJ, Skinner JS, Gardner KG, et al. Expression levels of barley Cbf genes at the frost resistance-H2 locus are dependent upon alleles at Fr-H1 and Fr-H2[J]. Plant Journal, 2007, 51: 308-321. |
| [16] | Knox AK, Li C, Vágújfalvi A, et al. Identification of candidate CBF genes for the frost tolerance locus Fr-Am2 in Triticum monococcum[J]. Plant Molecular Biology, 2008, 67: 257-270. |
| [17] | Jaglo KR, Kleff S, Amundsen KL, et al. Components of the Arabidopsis C-repeat / dehydration-responsive element binding factor cold response pathway are conserved in Brassica napus and other plant species[J]. Plant Physiology, 2001, 127: 910-917. |
| [18] | Xiong Y, Fei S. Functional and phylogenetic analysis of a DREB/CBF-like gene in perennial ryegrass(Lolium perenne L.)[J]. Planta, 2006, 224: 878-888. |
| [19] | Zhao H, Bughrara SS. Isolation and characterization of cold-regulated transcriptional activator LpCBF3 gene from perennial ryegrass(Lolium perenne L.)[J]. Molecular Genetics and Genomics, 2008, 279: 585-594. |
| [20] | 谢永丽. 草坪草狗牙根中抗逆基因BeDREB的克隆及功能鉴定[J]. 中国生物化学与分子生物学报, 2005, 21: 521. |
| [21] | Zhen Y, Ungerer MC. Clinal variation in freezing tolerance among natural accessions of Arabidopsis thaliana[J]. New Phytologist, 2008, 177: 419-427. |
| [22] | Zhen Y, Ungerer MC. Relaxed selection on the CBF/DREB1 regulatory genes and reduced freezing tolerance in the Southern Range of Arabidopsis thaliana[J]. Molecular Biology and Evolution, 2008, 25: 2547-2555. |
| [23] | McKhann HI, Gery C, Bérard A, et al. Natural variation in CBF gene sequence, gene expression and freezing tolerance in the Versailles core collection of Arabidopsis thaliana[J]. BMC Plant Biology, 2008, 8: 105-122. |
| [24] | Badawi M, Danyluk J, Boucho B, et al. The CBF gene family in hexaploid wheat and its relationship to the phylogenetic complexity of cereal CBFs[J]. Molecular Genetics and Genomics, 2007, 277: 533-554. |
| [25] | Wang Z, Zhang F, Xuan J, et al. Isolation and expression profiles of the ZjDREB1 gene encoding a DRE-binding transcription factor from zoysiagrass Zoysia japonica[J]. Journal of Horticultural Science and Biotechnology, 2012, 87: 77-83. |
| [26] | 冯勋伟, 才宏伟. 结缕草CBF 基因的同源克隆及其转基因拟南芥的抗寒性验证[J]. 作物学报, 2014, 40: 1572-1578. |
| [27] | Qin F, Kakimoto M. Regulation and functional analysis of ZmDREB2A in response to drought and heat stresses in Zea mays L.[J]. The Plant Journal, 2007, 50: 54-69. |
| [28] | Xue GP, Loveridge CW. HvDRF1 is involved in abscisic acid-mediated gene regulation in barley and produces two forms of AP2 transcriptional activators, interacting preferably with a CT-rich element[J]. The Plant Journal, 2004, 37: 326-339. |
| [29] | Liu Q, Kasuga M, Sakuma Y. Two transcription factors, DREB1 and DREB2, with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in drought- and low-temperature-responsive gene expression, respectively, in Arabidopsis[J]. The Plant Cell, 1998, 10: 1391-1406. |
| [30] | Liu L, Zhu K, Yang Y, et al. Molecular cloning, expression profiling and trans-activation property studies of a DREB2-like gene from chrysanthemum(Dendranthema vestitum)[J]. Journal of Plant Research, 2008, 121: 215-226. |
| [31] | Agarwal P, Agarwal PK, Nair S, et al. Stress-inducible DREB2A transcription factor from Pennisetum glaucum is a phosphoprotein and its phosphorylation negatively regulates its DNA-binding activity[J]. Molecular Genetics and Genomics, 2007, 277: 189-198. |
| [32] | Chen J, Xia X, Yin W. Expression profiling and functional characte-rization of a DREB2-type gene from Populus euphratica[J]. Biochemical and Biophysical Research Communications, 2009, 378: 483-487. |
| [33] | Agarwal P, Agarwal PK, Joshi AJ, et al. Overexpression of PgDREB2A transcription factor enhances abiotic stress tolerance and activates downstream stress-responsive genes[J]. Molecular Biology Reports, 2010, 37: 1125-1135. |
| [34] | Chen M, Wang QY. A soybean DRE-binding transcription factor, conferred drought and high-salt tolerance in transgenic plants[J]. Biochemical and Biophysical Research Communications, 2007, 353: 299-305. |





