2. 吉林农业大学生命科学学院,长春 130118
2. College of Life Sciences,Jilin Agricultural University,Changchun 130118
Boyer等[1]指出环境问题将成为影响作物产量的一个重要因素。虽然无法精确计算出环境因素对农作物产量影响的具体数值,但可耕地面积的逐渐减少势必导致粮食作物产量的持续下滑[2, 3]。2007年,世界粮农组织统计出目前全球只有3.5%的土地未受到环境因素影响[4]。这为人们敲响了警钟,在不久的将来粮食问题可能又会成为社会亟待解决的主要问题。为了降低环境因素对粮食产量的影响,选育抗逆性强的优质作物新品种已经成为农业科研工作者的主要目标。农作物新品种选育的方法主要为传统遗传育种与基因工程育种。由于基因工程育种具有目的性强,育种周期短,可克服远缘杂交不亲和障碍等优点,该方法已经成为育种的主要手段。而基因工程育种所需要的表达调控元件以及抗逆基因的挖掘成为重中之重。
CBF(C-repeat binding factor,CBF)转录因子是AP2/EREBP转录因子家族的一个亚家族,它是植物调节逆境胁迫的重要转录因子之一[5]。CBF能够与启动子中一个5 bp大小的核心片段(CCGAC)相结合,从而调控该基因的转录水平[6, 7]。CBF基因的表达能够被多种非生物胁迫以及上游转录因子所调控[8, 9, 10]。目前,多种植物CBF转录因子参与逆境胁迫的分子机制已经被揭示,包括拟南芥[11]、水稻[12]、玉米[13, 14]、小麦[15, 16]、大豆[17, 18]和葡萄[19]等。拟南芥4个CBF基因过表达后均能够激活下游靶基因的表达,并能够提高植物的抗寒能力[20]。AtCBF2基因的负调控作用能够对AtCBF1及AtCBF3起到适当调节下游基因表达强度,从而适度的调节植物对低温以及相关胁迫的应答[21]。目前,拟南芥CBF基因的信号转导网路已经十分清楚,大麦与番茄等植物CBF基因在耐盐等信号转导中的作用机制也得到了初步证实[22, 23]。这些研究成果均说明CBF转录因子在调节植物耐寒、耐盐等非生物胁迫过程中起着重要的调控作用。
本研究以野生山葡萄为材料,在已经成功分离了山葡萄VaCBF1、VaCBF2和VaCBF3的基础上[24],分别将3个基因构建到植物过表达载体中,利用浸花法将目的基因转入拟南芥基因组中,并对野生型拟南芥与转基因拟南芥进行盐胁迫处理,以探究其在植物盐胁迫中的作用。
1 材料与方法 1.1 材料实验所用转基因植物材料为哥伦比亚野生型拟南芥(Arabidopsis thaliana col-0)。山葡萄VaCBF1、VaCBF2和VaCBF3基因(GenBank登录号:DQ51-7296、DQ517297和DQ517298)由本实验室克隆。农杆菌EHA105和植物表达载体pBASTA由本实验室保存。克隆载体pMD18-T,限制性内切酶、T4连接酶、Ex Taq聚合酶均购自大连宝生物工程有限公司。凝胶回收试剂盒和质粒小提试剂盒购自爱思进生物技术(杭州)有限公司。PCR引物(表 1)及基因测序均由北京金唯智生物科技有限公司完成。
分别以VaCBF1、VaCBF2和VaCBF3的克隆载体质粒为模板,利用基因特异性引物进行PCR扩增。将3个PCR产物分别与表达载体pBASTA同时使用EcoR I与Xba I进行双酶切,回收目的基因与载体大片段后,利用T4 DNA连接酶进行连接,并将连接产物转化大肠杆菌DH5α感受态细胞。提取阳性菌液质粒DNA并进行酶切鉴定,将阳性重组质粒分别命名为pB-CBF1、pB-CBF2和 pB-CBF3。将3个重组质粒送交北京金唯智生物科技有限公司进行测序。
1.2.2 表达载体向农杆菌中的转化将农杆菌EHA105感受态细胞置于冰上缓慢融化,将重组质粒pB-CBF1、pB-CBF2和 pB-CBF3分别加入感受态细胞中,冰浴30 min。将离心管放置在液氮中冷冻1 min,然后在37℃水浴中温育5 min。温育过后,每个离心管中分别加入1 mL无抗生素的YEP培养基,在28℃摇床中振荡培养2-4 h。将菌液离心浓缩后涂于含有50 mg/L卡那霉素和100 mg/L利福平的固体YEP培养基上,28℃培养2-3 d,挑取单菌落后进行PCR鉴定。
1.2.3 农杆菌介导的浸花法转化拟南芥将野生型拟南芥播种于培养钵中,待拟南芥长至抽薹初期时剪掉所有拟南芥的主茎。当周围的茎抽薹后除去已经开的花准备浸染。同时准备农杆菌菌液,将农杆菌离心沉淀后使用渗透缓冲液(5%蔗糖,0.02% SilwettL-77)将菌液OD600调至0.9-1.0。将拟南芥花序部分倒置于渗透缓冲液中浸泡1-3 min,之后使用保鲜膜密封后黑暗下平放24 h,最后置于培养室中正常培养。待种子成熟时收取种子,以备后续鉴定。
1.2.4 转基因拟南芥的筛选与单拷贝株系鉴定将4℃春化2 d后的T0代转基因拟南芥种子播种于培养钵中,待长至四叶期时喷施1% Basta,每隔1 d喷施1次,共计喷施3次。观察拟南芥幼苗生长情况,始终保持绿色的为阳性转基因植株,慢慢枯萎死亡的为非转基因植株。将阳性转基因植株移栽至无Basta的培养钵中继续培养,并收获T1代转基因拟南芥种子。将T1代转基因拟南芥种子播种于含有1% Basta的MS固体培养基中,每个株系播种约100棵。统计各株系死亡率后计算各个转基因株系是否符合孟德尔遗传规律的分离比,从而鉴定单拷贝株系。将单拷贝株系移栽后收取T2代拟南芥种子,准备用于根系的生长发育实验。
1.2.5 转基因拟南芥的根系发育分析将WT与VaCBF1、VaCBF2和VaCBF3的T2代转基因拟南芥种子春化处理2 d,经70%酒精消毒30 min后,点播于MS固体培养基中。培养7 d后,分别将各个株系植株移栽至含有150 mmol/L NaCl的MS固体培养基中进行盐胁迫处理。处理7 d后观察各株系表型变化,并分别统计主根伸长长度、侧根数以及侧根长度。每个实验3次重复,并利用SPSS软件分析各组数据差异的显著性(P<0.05)。
2 结果 2.1 山葡萄CBF基因植物表达载体的构建提取表达载体pB-CBF1、pB-CBF2和 pB-CBF3的质粒DNA,利用EcoR I与Xba I进行双酶切鉴定,结果(图 1)显示3个质粒各切出一条约756 bp、762 bp与720 bp的特异条带。这证实山葡萄VaCBF1、VaCBF2和VaCBF1基因已经成功插入到表达载体中。将pB-CBF1、pB-CBF2和 pB-CBF3的质粒DNA测序后证实,载体插入片段与目的基因序列完全一致,未发现有碱基突变。

|
| M:分子量标准DL2000;1,2:pB-CBF1酶切产物;3,4:pB-CBF2酶切产物;5,6:pB-CBF3酶切产物 图 1 表达载体酶切验证 |
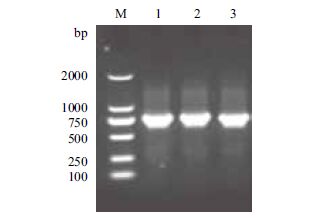
分别挑取转化后YEP平板上的单菌落,接种于5 mL液体YEP培养基中摇菌。待菌液生长至OD600为0.8左右时各取1 mL菌液作为模板,利用基因特异性引物(表 1)进行PCR验证阳性菌落。PCR结果(图 2)显示,3种农杆菌中均含有转化的目的基因,证实VaCBF1、VaCBF2和VaCBF1基因分别成功转入农杆菌中。
|
| M:分子量标准DL2000;1-3:转VaCBF1、VaCBF2及VaCBF3农杆菌PCR产物 图 2 农杆菌转化子的PCR鉴定 |
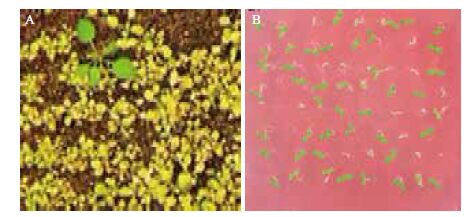
待T0代转基因拟南芥生长至四叶期时利用Basta筛选阳性转基因植株,喷施3次Basta后非转基因植株死亡,而转基因植株仍可以继续生长(图 3-A)。将阳性转基因植株移栽至正常土壤的营养钵中继续生长,并收取该株系T1代种子。将T1代转基因拟南芥进行春化后播种于含有1% Basta的MS固体培养基中进行再次筛选,并计算转基因植株死亡率(图 3-B)。OE-CBF1、OE-CBF2和OE-CBF3三个转基因株系的分离比分别为3.26∶1、3.51∶1与2.88∶1,基本符合孟德尔遗传定律,证实3个转基因株系为单拷贝株系。
|
| A:Basta筛选转基因植株;B:Basta筛选单拷贝植株 图 3 转基因拟南芥的筛选(A)及单拷贝株系鉴定(B) |
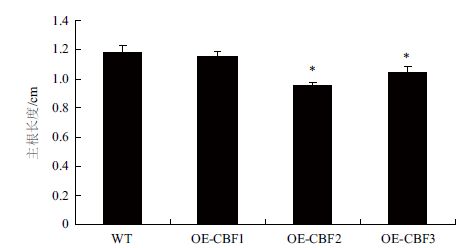
将WT与3个转基因株系T2代种子播种于MS固体培养基中生长,待生长至7 d时测量各个株系的主根长度。结果(图 4)显示,WT与OE-CBF1的主根长度相一致,而OE-CBF2与OE-CBF3的主根长度要显著短于WT及OE-CBF1。这说明在正常生长情况下,VaCBF2及VaCBF3的过表达在一定程度上对拟南芥主根生长造成了抑制。
|
| *表示P<0.05;**表示P<0.01,下同 图 4 转基因拟南芥主根生长状态分析 |
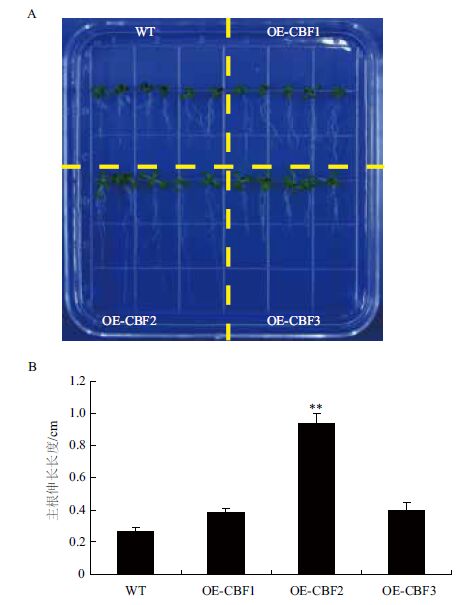
将WT与3个转基因株系T2代种子播种于MS固体培养基中,待生长至7 d后移栽至含有150 mmol/L NaCl的MS固体培养基中进行盐胁迫处理。胁迫处理8 d后观察各个株系根系的生长状态,发现OE-CBF2株系的主根长度显著长于其他3个株系(图 5)。而OE-CBF2在正常生长情况下的主根长度却显著短于WT植株(图 4),产生这种现象的原因可能是在盐胁迫下VaCBF2转录因子基因能够激活一系列盐应答基因的表达,增强了植物对盐胁迫的抵抗能力,从而降低了盐胁迫对主根生长的影响。
|
| A:WT与转基因植株在盐胁迫下的表型分析;B:盐胁迫下WT与转基因植株主根伸长长度分析 图 5 盐胁迫下拟南芥表型(A)及主根伸长分析(B) |
另外,在盐胁迫后,OE-CBF1的侧根数量显著多于其他3个株系(图 6-A)。这说明在盐胁迫条件下VaCBF1可能对植物侧根数起着一定的调控作用。同时,OE-CBF1、OE-CBF2和OE-CBF3三个株系的侧根长度均显著长于WT(图 6-B)。这说明VaCBF1、VaCBF2和VaCBF3在盐胁迫中对植物侧根的伸长均起着重要调控作用。
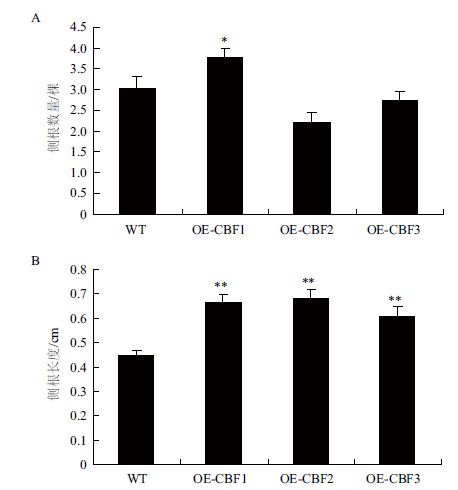
|
| A:盐胁迫下WT与转基因植株侧根数量分析;B:盐胁迫下WT与转基因植株侧根长度分析 图 6 盐胁迫下WT与转基因拟南芥侧根生长状态分析 |
植物在长期的进化过程中,逐渐形成了自身对环境的适应机制,当植物受到外界刺激后能够通过植物细胞内基因表达变化来实现和调节胁迫应答反应。1997年,Stockinger等[6]在拟南芥中首先发现一个能够与C重复序列相结合的因子,即CBF转录因子,并且命名为AtCBF1。在正常生长条件下,拟南芥3个CBF基因的过表达植株均表现出生长延迟的现象[11]。将玉米ZmCBF3基因转入水稻中进行过表达,在正常生长条件下也发现转基因植株的生长情况受到了一定程度的抑制[25]。而本研究结果显示OE-CBF2与OE-CBF3在正常生长条件下的主根长度均小于野生型植株。产生此现象的原因可能是由于转录因子可直接调控多个下游靶基因,从而导致多个基因的过表达。这种多基因过表达可能会增加植物内能源消耗,最终导致植物根系生长发育的延迟。
当植物处于非正常生长条件时,植物受到胁迫刺激后CBF基因的过表达就会调节下游相关基因的表达从而抵御外界环境的刺激[23, 26]。CBF调控的下游靶基因主要有COR15A、RD29A与KIN2等与逆境胁迫密切相关基因,这些靶基因直接调控植物对不同逆境胁迫的应答。在干旱及低温处理条件下,拟南芥CBF基因过表达植株生存率明显高于野生型对照[11]。在干旱胁迫下,水稻ZmCBF3的过表达株系也表现出较强的耐旱能力[25]。本研究中将转基因拟南芥进行盐胁迫处理,结果发现OE-CBF2的主根伸长长度显著长于其他植株。而番茄属的两个CBF基因(SlCBF1与ShCBF1)在拟南芥中过表达后,在盐胁迫条件下转基因植株的根系生长发育明显优于WT[23, 26]。Duan等[27]报道,在盐胁迫下植物侧根的生长处于停滞状态,直到生长条件恢复正常后才能开始继续生长发育。而本研究中在盐处理后OE-CBF1的侧根数量明显多于WT,而且OE-CBF1、OE-CBF2和 OE-CBF3的侧根长度均显著长于WT,表明VaCBF1、VaCBF2和VaCBF3的过表达在一定程度上缓解了盐胁迫对植物根系生长发育的抑制作用。说明山葡萄CBF基因可能通过调节植物根系发育来调节植物对盐胁迫的应答。但本研究只局限于植物根部发育的变化,该基因调控植物根系发育的分子机制仍不清楚,还有待于进一步的研究来揭示。
4 结论本研究通过构建山葡萄VaCBF1、VaCBF2及VaCBF3基因的植物过表达载体,并分别将3个表达载体转入拟南芥中,获得3个转基因株系OE-CBF1、OE-CBF2与OE-CBF3。对野生型拟南芥与转基因拟南芥进行盐胁迫处理,发现OE-CBF2转基因植株的主根伸长长度显著长于其它植株,3个转基因株系的侧根长度也明显长于野生型植株。
| [1] | Boyer JS. Plant productivity and environment[J]. Science, 1982, 218(4571): 443-448. |
| [2] | Lobell DB, Schlenker W, Costa-Roberts J. Climate trends and global crop production since 1980[J]. Science, 2011, 333(6042): 616-620. |
| [3] | 张俊环, 王玉柱, 孙浩元, 等. 外源水杨酸对低温下杏花抗氧化酶和CBF转录因子表达的影响[J]. 植物生理学报, 2014, 50(2): 171-177. |
| [4] | Cramer GR, Urano K, Delrot S, et al. Effects of abiotic stress on plants: a systems biology perspective[J]. BMC Plant Biol, 2011, 11: 163. |
| [5] | Yamaguchi-Shinozaki K, Shinozaki K. Transcriptional regulatory networks in cellular response and the tolerance to dehydration and cold stresses[J]. Annu Rev Plant Biol, 2006, 57: 781-803. |
| [6] | Stockinger EJ, Gilmour SJ, Thomashow MF. Arabidopsis thaliana CBF1 encodes an AP2 domain-containing transcriptional activator that binds to the C-repeat/DRE, a cis-acting DNA regulatory element that stimulates transcription in response to low temperature and water deficit[J]. Proc Natl Acad Sci USA, 1997, 94: 1035-1040. |
| [7] | Thomashow MF. Plant cold acclimation: Freezing tolerance genes and regulatory mechanisms[J]. Annual Review of Plant Physiology and Plant Molecular Biology, 1999, 50: 571-599. |
| [8] | 林元震, 郭海, 刘纯鑫, 等. 赤桉抗寒转录因子ICE1基因的分子克隆与表达分析[J]. 植物生理学报, 2011, 47: 488-494. |
| [9] | Chinnusamy V, Ohta M, Kanrar S, et al. ICE1, a regulator of cold-induced transcriptome and freezing tolerance in Arabidopsis[J]. Gene Dev, 2003, 17(8): 1043-1054. |
| [10] | Wang Y, Jiang CJ, Li YY, et al. CsICE1 and CsCBF1: two transcription factors involved in cold responses in Camellia sinensis[J]. Plant Cell Rep, 2012, 31(1): 27-34. |
| [11] | Liu Q, Kasuga M, Sakuma Y, et al. Two transcription factors, DREB1 and DREB2 with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in drought- and low-temperature-responsive gene expression, respectively, in Arabidopsis[J]. Plant Cell, 1998, 10(8): 1391-1406. |
| [12] | Satoko M, Mizoi J, Yoshida T, et al. Comprehensive analysis of rice DREB2-type genes that encode transcription factors involved in the expression of abiotic stress-responsive genes[J]. Mol Genet Genomics, 2010, 283(2): 185-196. |
| [13] | Qin F, Kakimoto M, Sakuma Y, et al. Regulation and functional analysis of ZmDREB2A in response to drought and heat stresses in Zea mays L.[J] Plant J, 2007, 50(1): 54-69. |
| [14] | Zhou ML, Tang YX, Wu YM. Genome-wide analysis of AP2/ERF transcription factor family in Zea mays[J]. Curr Bioinform, 2012, 7(3): 324-332. |
| [15] | Sazegari S, Niazi A. Isolation and molecular characterization of wheat(Triticum aestivum)dehydration responsive element binding factor(DREB)isoforms[J]. Aust J Crop Sci, 2012, 6(6): 1037-1044. |
| [16] | Shen YG, He SJ, Zhang WK, et al. An EREBP/AP2-type protein in Triticum aestivum was a DRE-binding transcription factor induced by cold, dehydration and ABA stress[J]. Theor Appl Genet, 2003, 106(5): 923-930. |
| [17] | Jing T, Chang Q, Li W, et al. Stress-inducible expression of GmDREB1 conferred salt tolerance in transgenic alfalfa[J]. Plant Cell Tiss Org, 2010, 100(2): 219-227. |
| [18] | Mizoi J, Ohori T, Moriwaki T, et al. GmDREB2A; 2, a canonical dehydration-responsive element-binding protein2-type transcription factor in soybean, is posttranslationally regulated and mediates dehydration-responsive element-dependent gene expression[J]. Plant Physiol, 2013, 161(1): 346-361. |
| [19] | Xiao H, Siddigua M, Braybrook S, et al. Three grape CBF/DREB1 genes respond to low temperature, drought and abscisic acid[J]. Plant Cell Environ, 2006, 29(7): 1410-1421. |
| [20] | Gilmour SJ, Fowler SG, Thomashow MF. Arabidopsis transcriptional activators CBF1, CBF2, and CBF3 have matching functional activities[J]. Plant Mol Biol, 2004, 54(5): 767-781. |
| [21] | Novillo F, Alonso JM, Ecker JR, et al. CBF2/DREB1C is a negative regulator of CBF1/DREB1B and CBF3/DREB1A expression and plays a central role in stress tolerance in Arabidopsis[J]. Proc Natl Acad Sci USA, 2004, 101(11): 3985-3990. |
| [22] | Skinner JS, Zitzewitz J, Szucs P, et al. Structural, functional, and physogenetic characterization of a large CBF gene family in barley[J]. Plant Mol Biol, 2005, 59(4): 533-551. |
| [23] | Zhang L, Li Z, Li J, et al. Ectopic overexpression of SsCBF1, a CRT/DRE-binding factor from the nightshade plant Solanum lycopersicoides, confers freezing and salt tolerance in transgenic Arabidopsis[J]. PLoS One, 2013, 8(6): e61810. |
| [24] | 王法微, 刘洋, 吴学彦, 等. 山葡萄VaCBF1转录因子基因的克隆与表达分析[J]. 西北农林科技大学学报: 自然科学版, 2013, 12(41): 86-92. |
| [25] | Xu MY, Li LH, Fan YL, et al. ZmCBF3 overexpression improves tolerance to abiotic stress in transgenic rice(Oryza sativa)without yield penalty[J]. Plant Cell Rep, 2013, 30(10): 1949-1957. |
| [26] | Li ZJ, Zhang LL, Li JF, et al. Isolation and functional characterization of the ShCBF1 gene encoding a CTR/DRE-binding factor from the wild tomato species Solanum habrochaites[J]. Plant Physiol Bioch, 2014, 74: 294-303. |
| [27] | Duan LN, Dietrich D, Ng CH, et al. Endodermal ABA signaling promotes lateral root quiescence during salt stress in Arabidopsis seedlings[J]. Plant Cell, 2013, 25(1): 324-341. |





