2. 山西师范大学生命科学学院,临汾 041004 ;
3. 北京师范大学生命科学学院, 北京 100875
2. College of Life Science,Shanxi Normal University,Linfen 041004 ;
3. College of Life Science,Beijing Normal University,Beijing 100875
PCR 技术,自1983 年美国人Kary Mullis 发明来,已经成为分子生物学及其相关领域最基本、重要的技术方法。如今,从基因扩增与基因检测基因克隆、基因改造、遗传分析等都已离不开CR 技术[1, 2]。另外,在病原微生物检测、法医鉴定、古研究食品安全及品种鉴定等多领域PCR 技术也挥着重要作用[3, 4, 5, 6, 7]。
PCR 方法的前提条件是获得DNA 或RNA 反转的cDNA 模板。DNA 的提取方法主要有十二烷基酸钠(Sodium dodecyl sulfonate,SDS) 提取法[8]十六烷基三乙基溴化铵(Hexadecyltrimethyl ammoniumromide,CTAB)提取法[9],或者直接从试剂公购买提取试剂盒。这些方法虽能得到较高质量的NA,但需样品量较大,且操作步骤较繁琐,效率低,难以满足对大量样品检测的需求。另外,对于品检测实验需要的模板量少,常规DNA 提取方法取的大量DNA 未被充分利用,造成了浪费。为解以上问题,研究人员开发多种简单、快速的获得NA 模板的方法,如微量玻璃粉吸附、Chelex-100、微波处理、TE 缓冲液沸水煮、NaOH 溶液处理,通过处理植物叶片、真菌菌丝等样品获得模板NA[10, 11, 12, 13, 14, 15, 16]。GE 公司的Whatman FTA 卡仅用一张卡就可以完成室温下DNA 和RNA 的采集、运输、化和储存,为血样、口腔细胞、植物DNA 等样本保存和分析提供了简单、可靠的方法[17, 18]。但是,些方法多为传统方法的改良,程序较复杂或需要殊的仪器、试剂或相关配套产品,因此成本较高。
对于丝状真菌而言,遗传转化子筛选和野外样菌株鉴定往往需要对大量样本进行PCR 扩增,且需的DNA 模板量又很少,而丝状真菌细胞壁结构杂又无法使用普通的菌落 PCR[19, 20]。因此,本究以尖孢镰刀菌(Fusarium oxysporum)为材料,合前人研究成果,建立一种用于丝状真菌PCR 模DNA 的快速制备方法,即利用NaOH 溶液对丝真菌的菌丝进行简单的沸水浴处理,再加入Tris-Cl 中和即可得到适用于PCR 的 DNA 模板,该方经济、简单、快速、高效,旨在为丝状真菌转化的高通量筛选和分离菌株的快速鉴定提供技术持。
1 材料与方法 1.1 材料本实验室保存的尖孢镰刀菌甘蓝专化型A8 和他丝状真菌菌株18 个。尖孢镰刀菌和其他真菌菌接种于含100 mg/L 氨苄青霉素钠和头孢噻肟钠的DA 平板上,28℃培养4 d,备用。
1.2 方法 1.2.1 菌丝处理取 4 个1.5 mL 离心管,分别标记Ⅰ、Ⅱ、Ⅲ、Ⅳ,Ⅰ号离心管加入无菌水50 μL,号离心管加入1×TE 缓冲液50 μL,Ⅲ号离心管入100 mmol/L Tris-HCl 缓冲液(pH8.0)50 μL,号离心管加入50 mmol/L NaOH 溶液50 μL。用无牙签挑取少量菌丝到各离心管中,涡旋打散菌丝,水浴10 min。Ⅳ号离心管中再加入5 μL 1 mol/LTris-HCl(pH8.0)缓冲液。12 000 r/min 离心10 min。上清至新离心管中,即为制备的模板DNA,-20℃存备用。
1.2.2 PCR 扩增以1.2.1 得到的DNA 为模板,SDS取法得到的100 ng/μL DNA(CK+)和无菌水(CK-)别为阳性和阴性对照,引物选用2 对真菌检测通用引物和10 对尖孢镰刀菌特异引物(表 1)。aq DNA 聚合酶选自于5 家公司:天根Taq DNAolymerase( 货号:ET101-01-03)、TaKaRa Ex Taq货号:RR001A)、全式金 EasyTaq DNA polymerase货号AP111-01)、博迈德 Taq DNA polymerase(货:PC0101)和康为世纪Taq DNA polymerase(货:CW0680),编号为T1、T2、T3、T4 和T5( 编顺序与上述提到的公司顺序无对应关系)。除酶适性实验中用到5 种酶外,其余均使用酶T1。PCR应体系为20 μL :模板2.0 μL(模板用量实验中分用0.2、0.5、1.0、2.0、4.0 和8.0 μL),10×Taquffer 2 μL,2.5 mmol/L dNTP 1.6 μL,10 μmol/L 正向引物各0.5 μL,5 U/μL Taq 酶0.2 μL,加ddH2O足到20 μL。PCR 反应程序为:95℃ 3 min ;95℃0 s,56℃ 30 s,72℃ 2 min,30 个循环;72℃ 8in。PCR 反应完成后,向PCR 产物中加入4 μL×loading buffer,混匀,取10 μL 进行琼脂糖电泳测,Marker 为全式金trans2K Plus DNA Marker。
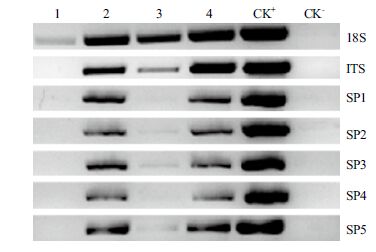
用4 种方法制备的模板DNA 进行PCR 扩增,果如图 1 所示。用1×TE 缓冲液沸水浴(方法Ⅱ)备的模板DNA 和50 mmol/L NaOH 溶液沸水浴后加1/10 体积1 mol/L Tris-HCl(pH8.0)缓冲液(方Ⅳ)制备的模板DNA 与阳性对照(CK+)扩增结一致,2 对通用引物及5 对特异引物都能得到清的电泳条带,而100 mmol/L Tris-HCl缓冲液(pH8.0)水浴(方法Ⅲ)制备的模板DNA 只有通用引物扩得到了清晰的条带,特异引物扩增只得到微弱的带,无菌水沸水浴(方法Ⅰ)制备的模板DNA 只18S rDNA 通用引物得到微弱条带,其余引物均没条带。所以,方法Ⅱ和Ⅳ所制备的模板DNA,用1 酶进行扩增,能得到理想的结果。
|
| 图 1 不同方法制备的尖孢镰刀菌DNA 模板PCR 扩增结果 1-4 :分别为I、II、III、IV 方法;CK+ :阳性对照;CK- :阴性对照 |
用实验室常用的5 个不同公司的Taq DNA 聚合,以方法Ⅱ和Ⅳ所制备的DNA 为模板进行PCR,果如图 2 所示。方法Ⅳ得到的模板DNA 用5 个公的酶扩增都能得到清晰条带,但条带亮度有所不,而以方法Ⅱ得到的模板DNA,只有用T1 酶才扩增出条带。所以,方法Ⅱ所得到的模板DNA 可用来进行PCR 检测,但需要选择活性较高的酶,检测前需要进行预备实验,检验所用酶是否能用该方法制备的模板DNA ;而方法Ⅳ得到的模板NA 对酶的适应性较好,5 种酶都可用,比方法Ⅱ具优势。

|
| 图 2 不同方法制备的尖孢镰刀菌模板DNA 用不同Taq DNA 聚合酶的PCR 扩增结果 A :方法II ;B :方法IV |
由于使用沸水浴制备的模板DNA 都是微量的,法准确测得其浓度,故而使用不同模板DNA 溶液体积进行实验。用方法Ⅳ制备的模板DNA 0.2-8.0L 进行PCR 反应,结果(图 3)显示,模板用量为 μL 时,条带最清晰,模板量高于或低于2 μL 条带度减弱。这是由于该方法制备的模板DNA 中含有他杂质(如NaOH、Tris-HCl 等),当加入的模板高时会抑制Taq 酶的活性,模板量低,DNA 量少,条带也较弱。因此,使用方法Ⅳ制备的模板DNA 时,量控制在1-4 μL 为佳。

|
| 图 3 方法IV 制备的尖孢镰刀菌不同模板DNA 用量PCR 扩增结果 1-6 :DNA 模板用量分别为0.2、0.5、1.0、2.0、4.0 和8.0 μL |
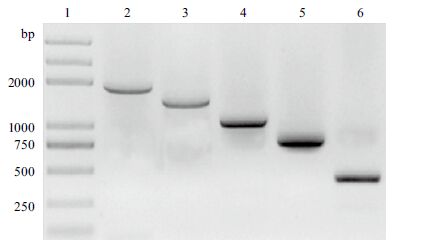
以方法Ⅳ制备的DNA 为模板,重新选用5 对扩片段大小不同的引物,进行PCR 扩增,结果(图 4)示,扩增大小从396-1978 bp 均得到单一清晰条带,段范围已能满足真菌检测的需要。
|
| 图 4 方法IV 制备的尖孢镰刀菌模板DNA 不同大小DNA 片段的PCR 扩增结果 |
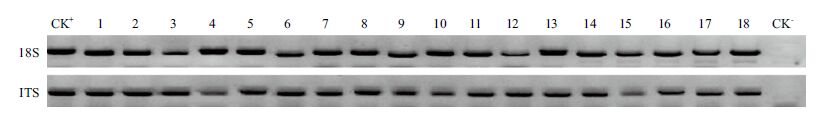
选用本实验室保存的丝状真菌菌株18 个,挑取丝用方法Ⅳ制备模板DNA,扩增其18S rDNA 和ITS 片段,结果如图 5 所示。18 个菌株与阳性对照样都能得到清晰单一的条带,说明方法Ⅳ除可以备尖孢镰刀菌模板DNA 外,对其他丝状真菌也可制备适合PCR 的模板。
|
| 图 5 其他丝状真菌菌株18S rDNA 及ITS 扩增结果 1. Epicoccum sorghinum ;2.Paraphaeosphaeria angularis ;3.Sclerotinia sclerotiorum ;4.Fusarium equiseti 5.Glomerella acutata ;6.Colletotrichum simmondsii ; 7.Nigrospora sphaerica ;8.Botryotinia fuckeliana ;9.Gibberella zeae ;10.Engyodontium album ;11.Colletotrichum capsici ;12.Sarocladium attenuatum ; 13.Curvularia lunata ;14.Nectria cinnabarina ;15.Fusarium sacchari ;16.Setosphaeria rostrata ;17.Gibberella avenacea ;18.Bipolaris panicimiliacei |
本研究结果表明,方法Ⅳ可以制备优质的PCR板DNA。实际应用中可以根据需要减少或加大aOH 溶液体积,再用1/10 体积的1 mol/L Tris-HClpH8.0)缓冲液中和即可,根据体积变化适当增加减少菌丝量。
用NaOH 溶液直接处理粗样品的方法在水稻、麦和海洋浮游生物等研究中都有应用,如汪秀峰[16]用250 mmol/L 的NaOH 溶液处理水稻叶片,理过后用HCl 和Tris-HCl 中和,而后取出处理过水稻叶片作为PCR 模板,但该法每次PCR 得用1块叶片,加大了样品的使用量,同时也给PCR 增了额外的步骤;刘鹏等[13]用100 mmol/L 的NaOH液处理小麦幼叶,但还需经酚氯仿抽提,异丙醇淀,比传统方法只是省去了液氮速冻和样品研磨,不够简化;刘泳等[14]用125 mmol/L 的NaOH 处海洋浮游生物,25℃处理8 h 以上,然后99℃加10 min,再加入HCl、Tris-HCl 和TritonX-100 混,再次99℃加热10 min,此法虽简化了步骤,但耗时过长。以上的方法与本研究建立的方法相似,都不够简化,这可能与样品的复杂性相关,有些骤不可或缺,否则无法达到PCR 模板的要求。对丝状真菌,陈吉良等[12]建立了一种用10%(W/V)无菌 Chelex-100 树脂溶液经沸水浴制备PCR 模板NA 的方法,但样品在处理前需要液氮适当研磨。法步骤简单,但需要特殊试剂,成本相对较高;力等[15]也建立了一种微波处理置于 10 × TE 缓液中的菌丝获得DNA 的方法,但未对该方法获得的模板DNA 进行Taq DNA 聚合酶适应性的研究,本研究发现TE 处理(方法Ⅱ)的样品具有Taq选择性,只适用于少数Taq DNA 聚合酶。
相对于上述方法,本研究建立的方法具有以下点:(1)直接从菌落挑取菌丝,不需要液体培养,需要磨样,减化了实验步骤,大大减少了实验时,同时也减少了被污染的几率;(2)不需使用液,只需普通的NaOH 和Tris-HCl 试剂,且用量少,器需要普通离心机、水浴锅或者电磁炉即可,成低廉;(3)实验人员可避免接触酚、氯仿等毒性机试剂,安全可靠。
4 结论本研究以尖孢镰刀菌为材料,通过对4 种方法比较筛选,建立了一种快速制备丝状真菌PCR 模DNA 的方法。该方法制备模板DNA,无需液氮研磨,只需少量50 mmol/L NaOH 溶液及1 mol/Lris-HCl,沸水浴10 min 及离心10 min 即可,可采实验室常用的各种Taq DNA 聚合酶扩增出18SDNA 和ITS 片段及400 bp-2 000 bp 大小范围的特片段。另外,本法也适用于其他丝状真菌,可为验室大量丝状真菌的转化子及菌株的鉴定提供经、 简单、 快速、 安全和高效的方法。
| [1] | Dieffenbach CW, Dveksler GS. PCR primer : a laboratoryanual[M]. Cold Spring Harbor Laboratory Press, 2003. |
| [2] | 钟肖芬, 邓日强. Bt 毒素蛋白基因的 PCR 鉴定及定位[J]. 微物学报, 2001, 41(3): 293-297. |
| [3] | Brown KA, Brown TA. Biomolecular archaeology[J]. Annu Revnthropol, 2013, 42 : 159-174. |
| [4] | Gausterer C, Stein C, Stimpfl T. Application of direct PCR in aorensic case of yew poisoning[J]. Int J Legal Med, 2012, 126(2): 15-319. |
| [5] | 曹凤明, 沈德龙, 李俊, 等. 应用多重 PCR 鉴定微生物肥料常芽孢杆菌[J]. 微生物学报, 2008, 48(5): 651-656. |
| [6] | 戚华雄, 杨蜀兰. 用聚合酶链式反应技术鉴定杂交稻种子纯度初步研究[J]. 湖北农业科学, 1999(1): 12-14. |
| [7] | 杨冬燕, 杨小柯, 杨永存, 等. 潲水油聚合酶链式反应鉴定技研究[J]. 中国食品卫生杂志, 2011, 23(3): 255-260. |
| [8] | Edwards K, Johnstone C, Thompson C. A simple and rapid methodor the preparation of plant genomic DNA for PCR analysis[J].ucleic Acids Res, 1991, 19(6): 1349. |
| [9] | Stewart Jr CN, Via LE. A rapid CTAB DNA isolation techniqueseful for RAPD fingerprinting and other PCR applications[J].iotechniques, 1993, 14(5): 748-750. |
| [10] | Griffin D, Kellogg C, Peak K, et al. A rapid and efficient assay forxtracting DNA from fungi[J]. Lett Appl Microbiol, 2002, 34(3): 10-214. |
| [11] | Tendulkar SR, Gupta A, Chattoo BB. A simple protocol for isolationf fungal DNA[J]. Biotechnol Lett, 2003, 25(22): 1941-1944. |
| [12] | 陈吉良, 黄小龙, 吴安迪, 等. 一种快速高效提取病原真菌NA 作为PCR 模板的方法[J]. 菌物学报, 2011, 30(1): 47-149. |
| [13] | 刘鹏, 邱晓东, 郭智慧, 等. 小麦幼叶 DNA 快速提取新方法及SR-PCR 扩增[J]. 江苏农业科学, 2011(4): 36-37. |
| [14] | 刘泳, 杨官品. 碱煮法: 一种制备海洋浮游生物 PCR 模板NA 的实用方法[J]. 实验与技术, 2004, 28(7): 1-4. |
| [15] | 潘力, 崔翠, 王斌. 一种用于 PCR 扩增的丝状真菌 DNA 快速取方法[J]. 微生物学通报, 2010, 37(3): 450-453. |
| [16] | 汪秀峰, 杨剑波, 向太和, 等. 一种叶片直接用作 PCR 扩增的方法及其应用[J]. 中国水稻科学, 2002, 16(1): 67-70. |
| [17] | Borman AM, Fraser M, Linton CJ, et al. An improved protocol forhe preparation of total genomic DNA from isolates of yeast andould using Whatman FTA filter papers[J]. Mycopathologia,010, 169(6): 445-449. |
| [18] | Johanson HC, Hyland V, Wicking C, et al. DNA elution from buccalells stored on Whatman FTA Classic Cards using a modifiedethanol fixation method[J]. Biotechniques, 2009, 46(4): 09-311. |
| [19] | Baldini A, Ross M, Nizetic D, et al. Chromosomal assignment ofuman YAC clones by fluorescence in situ hybridization : Use ofingle-yeast-colony PCR and multiple labeling[J]. Genomics,992, 14(1): 181-184. |
| [20] | Ward AC. Rapid analysis of yeast transformants using colony-CR[J]. Biotechniques, 1992, 13(3): 350. |





